‘湘辣14号’叶绿体基因组密码子偏好性分析


摘 要:为阐明‘湘辣14号’叶绿体基因组密码子的使用模式,本研究以‘湘辣14号’(Capsicum annuum cv ‘Xiangla NO.14’)叶绿体基因组52条蛋白编码基因序列(CDS)为材料,采用Codon W1.4.2等软件分析‘湘辣14号’密码子碱基组成及使用偏好性,并利用中性绘图、奇偶偏好(PR2-plot)、有效密码子数(ENC-plot)分析了影响密码子偏好性的主要因素,并进一步确定其最优密码子。结果表明,‘湘辣14号’密码子GCall平均含量为38.70%,且GC1(47.29%)>GC2(39.74%)>GC3(29.05%),ENC介于34.10~54.31之间,平均值为46.69。有29个密码子的同义密码子相对使用度(RSCU)>1,且以A或U结尾,密码子第3位碱基偏好使用A/U结尾。ENC与GC3呈极显著相关,而与GCall、GC1、GC2不相关。绘图分析表明,自然选择和突变均对‘湘辣14号’密码子偏好性产生重要影响。共筛选出GCA、GCU、AGA等19个最优密码子,其中10个以A结尾,9个以U结尾,说明‘湘辣14号’密码子第3位碱基偏爱使用A/U结尾。本研究为茄科辣椒属叶绿体基因工程的开展提供了理论依据。
关键词:‘湘辣14号’;叶绿体基因组;密码子偏好性;最优密码子
中图分类号:S602 文献标志码:A 文章编号:1008-1038(2025)01-0047-09
DOI:10.19590/j.cnki.1008-1038.2025.01.008
Analysis of Codon Usage Bias in Chloroplast Genome
of Capsicum annuum cv‘Xiangla NO.14’
HE Daoshan1, YANG Xiaoqiang2, QIN Yalin2, HE Haiyan1, XIE Wei1, LUO Zhiguo1, LI Peng1*
(1. Xiangtan Institute of Agricultural Sciences, Xiangtan 411134, China; 2. Hunan University of Humanities, Science and Technology, College of Agriculture and Biotechnology, Loudi 417000, China)
Abstract: In order to confirm the codon usage pattern of chloroplast genome of Capsicum annuum cv ‘Xiangla NO.14’, the 52 protein coding genes of the chloroplast genome of ‘Xiangla NO.14’ were selected as materials in this work, and base composition and codon usage bias of ‘Xiangla NO.14’ were detected using Codon W1.4.2. The main factors affecting codon preference were analyzed by neutral plot, PR2-plot and ENC-plot, and the optimal codon was further determined. The results indicated that the average ratio of GCall in the ‘Xiangla NO.14’ chloroplast genome was 38.70%, with GC1(47.29%)>GC2(39.74%)>GC3(29.05%). The ENC value varied from 34.10 to 54.31, with an average of 46.69. There were 29 codons with RSCU>1 and ended with A or U, which revealed that the third base of the codon preferred to end with A/U. ENC was significantly correlated with GC3, but not with GCall, GC1 and GC2. The graphical analysis showed that codon usage bias was affected by natural selection and mutation in the ‘Xiangla NO.14’ chloroplast genome. A total of 19 optimal codons such as GCA, GCU, AGA were screened out, and ten codons ended with A, and nine codons ended with U. The third base of codon preferred to use A/U ending. The findings obtained in this work provided a theoretical basis for the development of chloroplast genetic engineering of Capsicum in Solanaceae.
Keywords: Capsicum annuum cv ‘Xiangla NO.14’; chloroplast genome; codon usage bias; optimal codons
密码子是生物活动中最基本的信息单元,在传递遗传信息过程中扮演着不可替代的角色[1]。64种密码子因密码子简并性的存在只编码20种氨基酸。在核基因组中,除色氨酸(Trp)和甲硫氨酸(Met)外,其他氨基酸均由2~6个同义密码子编码[2-3]。密码子偏好性是指同一密码子在不同的基因组或基因中非随机的使用现象。密码子偏好性是特定生物体所特有的,受GC含量、基因表达水平和基因长度等因素的影响[4-5],且会影响外源基因的表达效率、信使RNA的合成、蛋白质的翻译速度及折叠等生物学功能[6-8]。考虑到不同的密码子模式具有重要的生物学效应,在特定的基因组或基因中识别密码子的使用偏好性对于阐明生物长期进化过程中的表达规律以及利用叶绿体基因组技术改良物种性状具有重要的意义[9-10]。
目前,解释密码子使用偏好性的最流行且被广大学者普遍认可的假说是突变-选择平衡,该理论认为密码子的使用反映了三种进化力量的综合效应:突变压力、自然选择和群体内的遗传漂变[11-13]。研究表明,紫菜薹(Brassica rapa var. purpuraria)叶绿体密码子偏好使用A/U碱基,主要受突变压力的影响[14],自然选择、碱基突变均对豇豆(Vigna unguiculata)密码子的偏好性产生了重要影响[15];这些研究表明,密码子偏好性使用模式和其他相关指标为研究不同物种复杂的进化规律提供了简单且直观的策略。
‘湘辣14号’(Capsicum annuum cv ‘Xiangla NO.14’)属茄科(Solanaceae)辣椒属(Capsicum)一年生植物,是湖南地区重要的蔬菜和香料作物。‘湘辣14号’具有皮薄肉脆、果实细长、辣味上佳、味道鲜美等特点,在辣椒市场中具有较强的竞争力,其营养价值和商业价值都比较高,深受国内外广大消费者的喜爱。近年来,‘湘辣14号’在湖南省栽培面积和产值迅猛发展,已成为湖南地区农民增收、乡村振兴的重要农作物之一。目前,研究者们从种质资源[16]、高产高效栽培技术集成[17]、遗传育种[18]等方面开展了大量的研究,随着‘湘辣14号’叶绿体全基因组序列的拼接和组装的完成,目前尚未对‘湘辣14号’叶绿体基因组密码子使用偏好性进行相关研究。本研究以‘湘辣14号’叶绿体基因组52条蛋白编码基因序列(coding DNA sequence,CDS)为材料,对‘湘辣14号’密码子碱基组成及使用偏好性进行分析,并明确影响‘湘辣14号’密码子偏好性的主要因素,从而在‘湘辣14号’叶绿体基因组筛选出最优密码子,以期为开展‘湘辣14号’品种鉴定、性状改良及外源基因的优化表达等方面提供理论支撑。
1 材料与方法
1.1 序列的获得
根据‘湘辣14号’的GenBank登录号(OR551752.1)在NCBI数据库下载整个序列,共87条蛋白编码基因序列(coding DNA sequence,CDS),利用Excel 2016挑选出起始密码子为ATG,终止密码子为TAG、TGA 或TAA的CDS序列,但删除长度小于300 bp且重复的CDS序列,最后共得到52条CDS序列作为本研究密码子偏好性分析的样本。
1.2 密码子使用偏好性及相关指标分析
通过在线程序CodonW 1.3对‘湘辣14号’的同义密码子相对使用度(relative synonymous codon usage,RSCU)、有效密码子数(ENC)、总GC含量(GCall)、第3位碱基GC使用频率(GC3s),以及密码子第3位各碱基使用频率(A3s、T3s、C3s和G3s)分别进行计算[19]。RSCU值和ENC值一起被用来描述‘湘辣14号’的密码子使用偏好性。当RSCU大于1时,为‘湘辣14号’的高频密码子且偏好性较强;RSCU等于1,则表明‘湘辣14号’密码子无使用偏向;RSCU小于1,表明密码子偏好性较弱。采用EMBOSS软件对‘湘辣14号’密码子的GC1、GC2、GC3和GC 12含量进行计算。
1.3 中性绘图分析
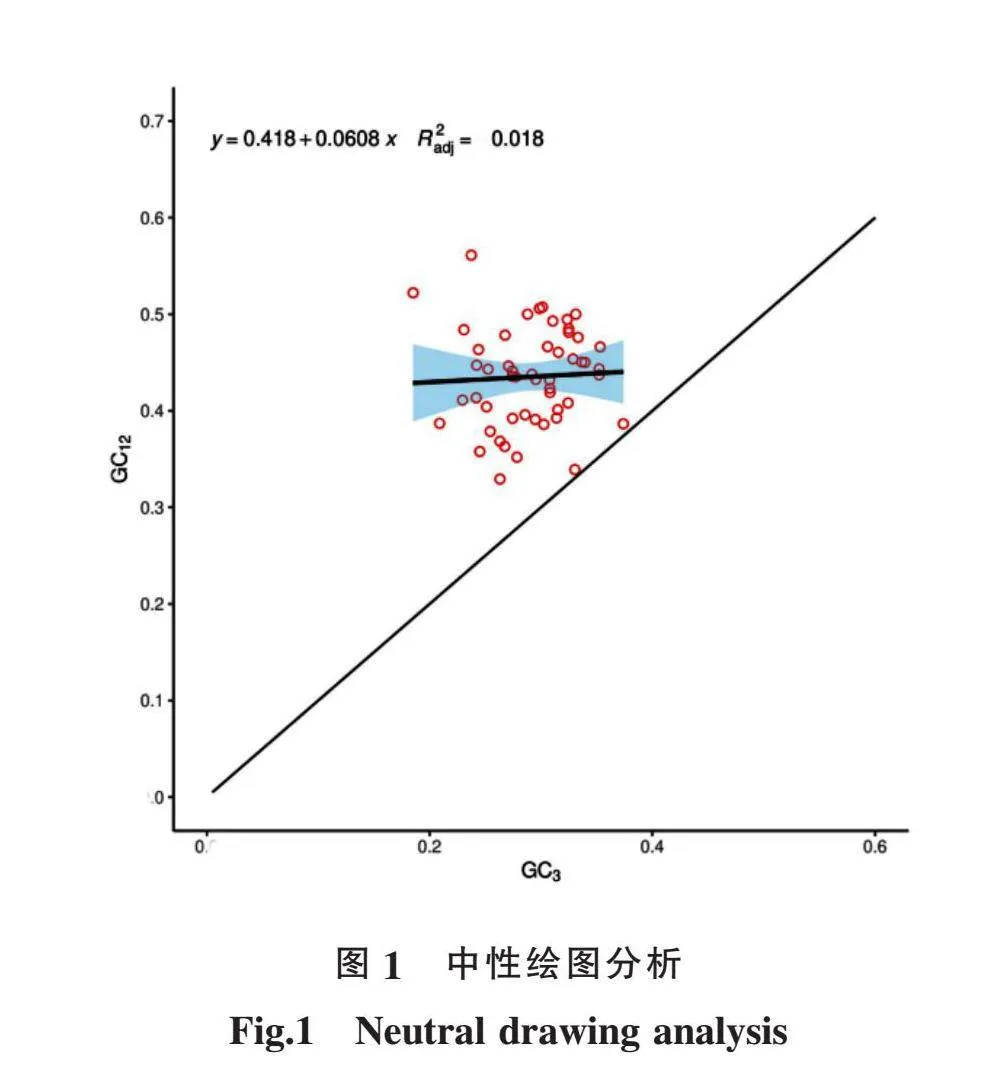
根据Wei等[20]的方法,对‘湘辣14号’密码子第3位的GC含量(GC3)进行计算,分别取GC3和GC12为横坐标、纵坐标,制作散点图进行相关性分析。
1.4 ENc-plot分析
采用ENC-plot绘图法分析可以确定影响‘湘辣14号’密码子使用偏好性的关键因素。ENC值取值为20~61之间,与密码子使用偏好性存在负相关关系。分别采用‘湘辣14号’的GC3s和有效密码子ENC作为横纵坐标,制作‘湘辣14号’的ENc-plot 散点图,其中有效密码子ENC的计算公式为ENC=2+GC3s +29/[GC3s2 +(1-GC3s)2],并制作其标准曲线,当散点越接近ENC标准曲线,则表明碱基突变对‘湘辣14号’密码子偏好性的形成起主导作用,当散点处于ENC标准曲线下方,则表示自然选择对‘湘辣14号’密码子偏好性的形成起主导作用[21]。
1.5 PR2-plot绘图分析
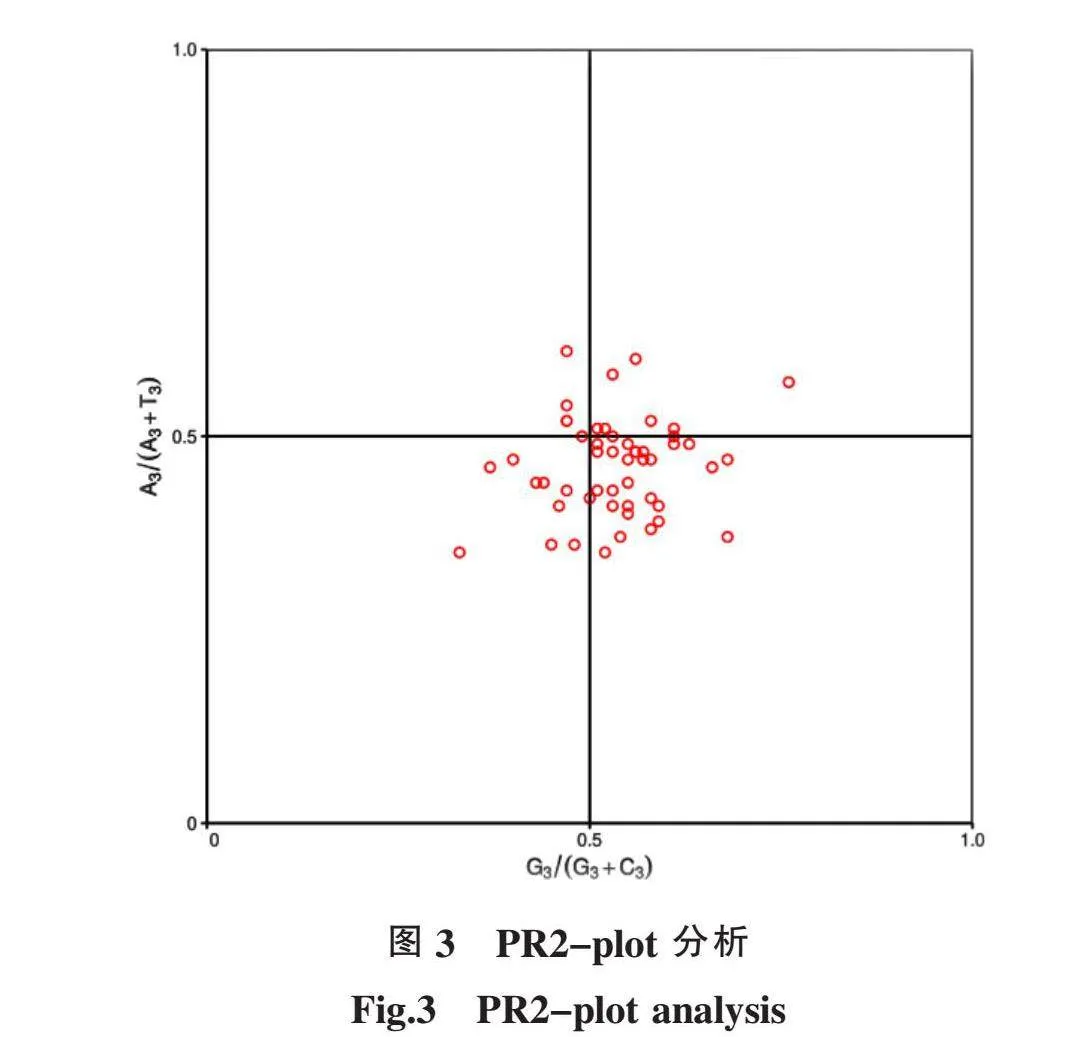
利用PR2-plot对‘湘辣14号’密码子的第3位碱基(A、T、C、G)的组成情况进行分析。计算‘湘辣14号’密码子第 3位各碱基的比例,以A3/(A3+T3)、G3/(G3+C3) 分别为Y轴、X轴,通过Matlab R2016a制作PR2-plot散点图,散点与中心点的距离则表示‘湘辣14号’密码子的偏倚程度及方向[22]。
1.6 最优密码子的鉴定
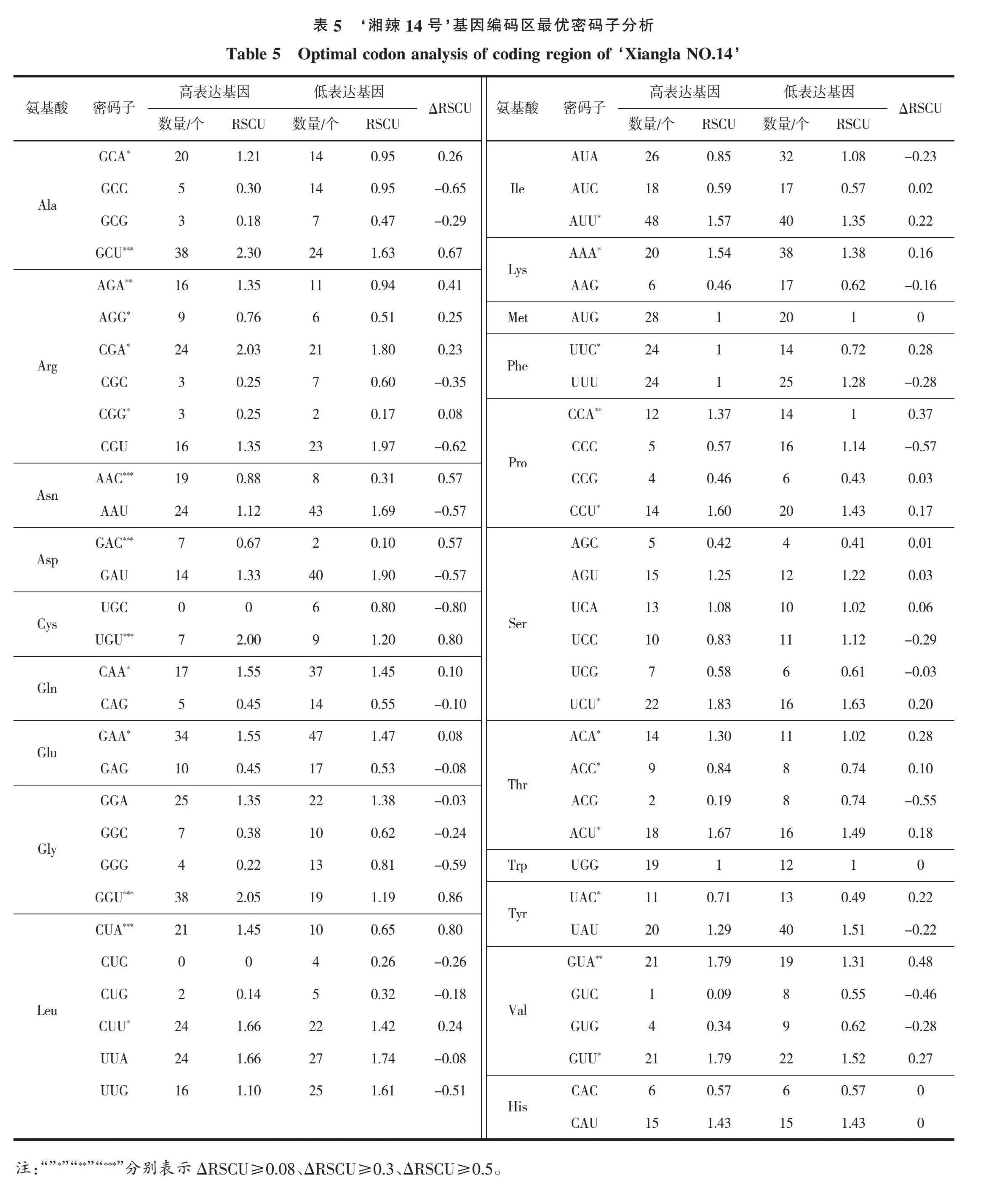
根据密码子的RSCU值大小来确定高频使用密码子[23]。以ENC分析作为偏好性标准,对52条‘湘辣14号’CDS序列进行排序,选择5%的高偏向性(ENC值小于30)和低偏向性(ENC值大于55)数据集[24]。计算ΔRSCU=高表达基因的RSCU-低表达基因的RSCU,并同时满足RSCU>1且ΔRSCU≥0.08这两个条件的密码子为最优密码子。
2 结果与分析
2.1 密码子的碱基组成分析
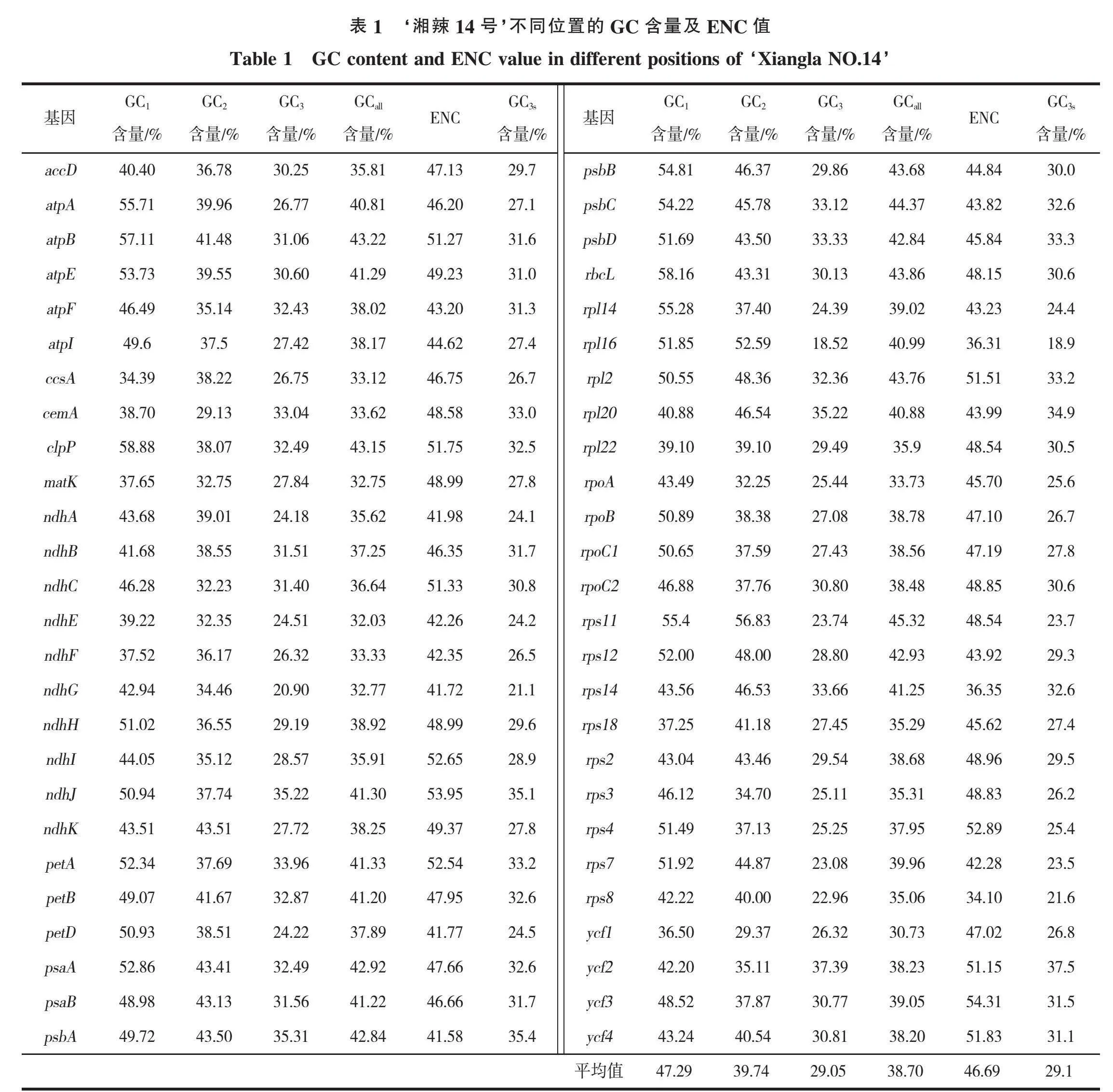
由表1可知,‘湘辣14号’密码子的总GC含量(GCall)为38.70%,GC1、GC2、GC3分别为47.29%、39.74%、29.05%,其中GC1含量最高,而GC3含量最低,GC含量在‘湘辣14号’叶绿体基因组中分布不平衡,表明‘湘辣14号’在其密码子第3位碱基优先使用A或T。ENC值介于34.1~54.31之间,平均值为46.69,第3位同义密码子的GC3s为29.10%,说明‘湘辣14号’密码子偏好性较弱且不明显。
由表2可知,GCall与GC1、GC2、GC3三个指标间存在极显著相关;GC1分别与GC2和GCall两个指标间存在极显著相关;ENC和GC3存在极显著相关,且与GC1和GC2不相关;揭示GC3在‘湘辣14号’密码子偏好性形成过程中的影响作用大于GC2与GC1。
2.2 相对同义密码子使用度分析
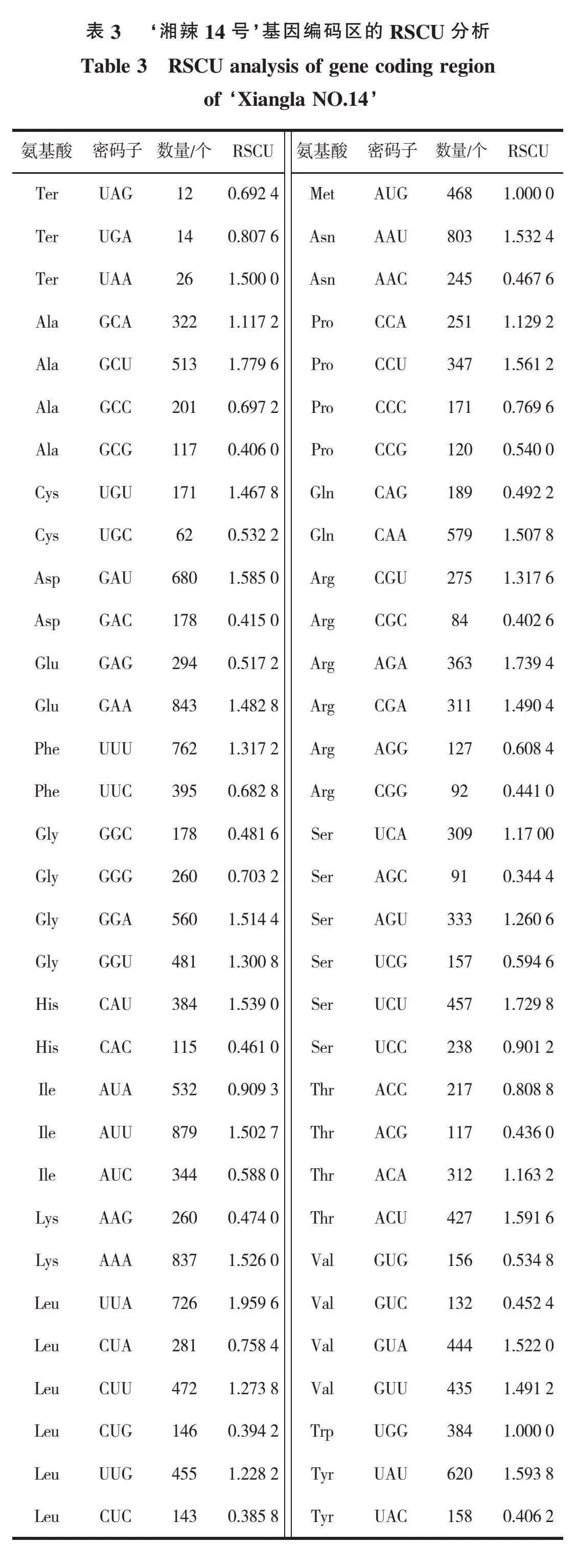
由表3可知,在‘湘辣14号’叶绿体基因组中共发现21 055个密码子,总编码20种氨基酸,密码子数介于12~879个,其中密码子UAG为12个,编码异亮氨酸(Ile)的AUU最多,为879个。RSCU值介于 0.344 4~1.959 6之间,使用频率最高氨基酸为亮氨酸(Leu),其RSCU值为1.959 6,丝氨酸(Ser)的使用频率最低,其RSCU值为0.344 4,在‘湘辣14号’叶绿体基因组中RSCU>1的高频密码子共有30个,其中以U、A、G结尾的密码子分别为16、13、1个,表明‘湘辣14号’偏好使用NNA或NNU的密码子。此外,蛋氨酸(Met)和色氨酸(Trp)的使用频率为1,说明‘湘辣14号’的AUG和UGG密码子不表现出偏好性。
2.3 中性绘图分析
中性绘图(图1)分析说明,‘湘辣14号’GC12和GC3之间的相关关系,量化自然选择、突变与‘湘辣14号’密码子偏好性形成的关系。GC3取值介于18.52%~37.39%之间,GC12取值为31.76%~57.86%,‘湘辣14号’基因全部落在对角线之上,回归系数为0.060 8,r2为0.018,GC3与GC12并不存在显著相关性,说明影响‘湘辣14号’偏好性形成的主导因素为自然选择,但突变在一定程度上影响‘湘辣14号’密码子的使用偏好性。
2.4 ENC-plot绘图分析
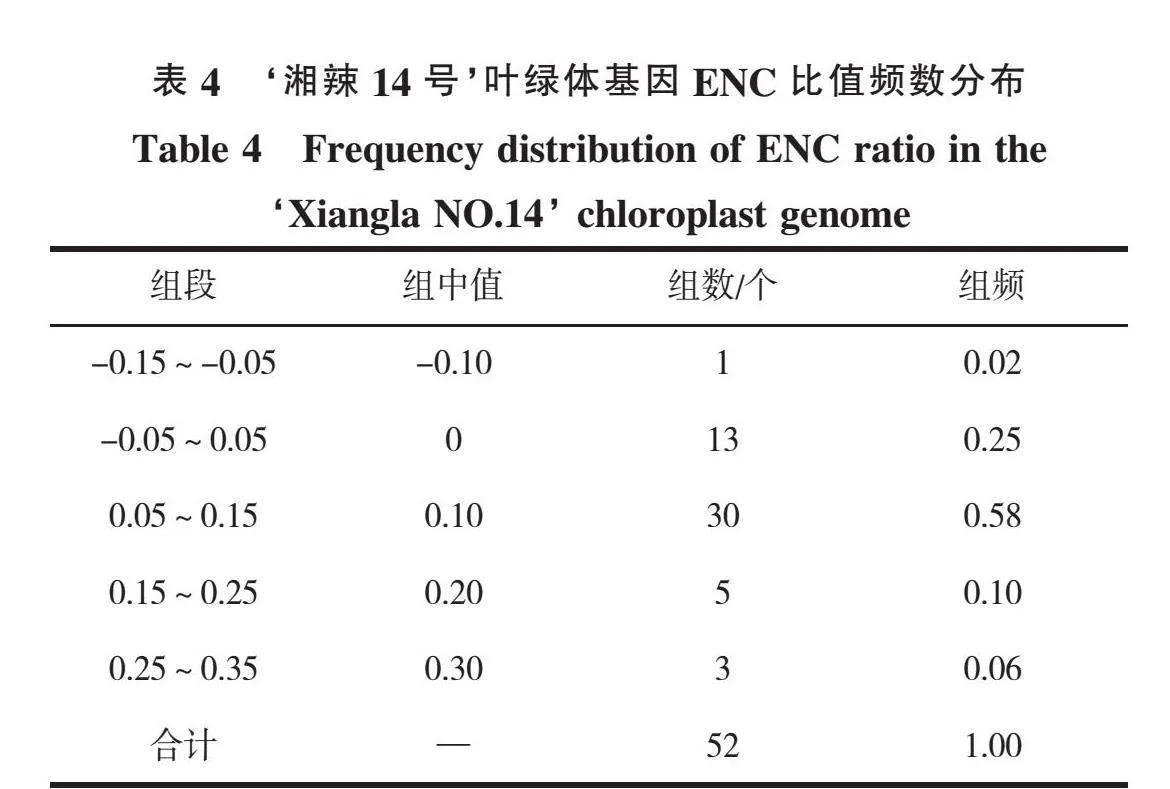
ENC-plot绘图分析结果显示(见图2),少数几个‘湘辣14号’叶绿体基因位于标准曲线上方,只有少数基因处于ENC标准曲线附近,说明自然选择和突变都对‘湘辣14号’密码子偏好性的形成产生影响。位于标准曲线下方的基因数量较多且相距较远,说明自然选择主导了‘湘辣14号’密码子偏好性的形成。ENC比值频数分析说明,共有13个基因与ENC预期值(-0.05~0.05)相符,有39个基因与ENC预期值差距较大(表4),说明在‘湘辣14号’叶绿体基因组中只有少数基因的ENC期望值与预期值相符。
2.5 PR2-plot绘图分析
分析A、T、C、G碱基的使用频率有助于阐明‘湘辣14号’密码子偏好性(见图3)。其中大多数基因分布于平面图的下半方,其中右下方最多,其次是左下方,再其次是右上方,最后是左上方,说明4种碱基的使用频率为T>A、G>C,这说明‘湘辣14号’密码子的偏好性受多种因素影响,包括碱基突变、自然选择等。
2.6 最优密码子的确定
在‘湘辣14号’叶绿体基因组中有24个高频密码子(ΔRSCU≥0.08),9个高频密码子以U结尾,2个高频密码子以G结尾,按照RSCU值>1 且ΔRSCU≥0.08条件,共在‘湘辣14号’中确定19个最优密码子,分别为GCA、GCU、AGA、CGA、UGU、CAA、GAA、GGU、AUU、CUA、CUU、AAA、CCA、CCU、UCU、ACA、ACU、GUA、GUU(表5),这些最优密码子全部以碱基A或U结尾。
3 结论与讨论
密码子偏好性分析是研究原核生物和真核生物进化特征的有效手段[25]。大量研究表明,多种因素都对密码子偏好性有影响,如基因片段大小、基因表达丰度、tRNA丰度等[26]。研究显示,影响密码子偏好性最主要的原因是碱基突变和自然选择,被广泛应用于阐述物种密码子使用的偏好性[27]。碱基组成和自然选择对植物进化过程中的密码子偏好性形成有重要的影响。本研究在‘湘辣14号’叶绿体基因组中,GC3与GCall和ENC呈极显著相关,与GC1和GC2不相关,说明自然选择对‘湘辣14号’密码子使用偏好性的形成起主导作用。
叶绿体密码子的碱基组成对生物的进化历程有着重要的影响。本研究发现‘湘辣14号’总GC含量(GCall)为38.70%,GC1为47.29%,GC2为39.74%,GC3为29.05%,GC3与GCall和ENC三者之间具有极显著相关,而与GC1和GC2三者之间不存在相关性,表明‘湘辣14号’密码子的使用偏好性为NNU/NNA,这与同科植物番茄(Solanum lycopersicum L.)[28]、辣椒属等密码子的使用模式相似[29]。‘湘辣14号’共得到30个高频密码子(RSCU值gt;1),其中有29个以碱基A或U结尾,再次验证‘湘辣14号’偏好使用NNU/NNA类型的密码子。
本研究发现‘湘辣14号’密码子的ENC平均值为46.69,且高于45,35个CDS的ENC>45,揭示这些CDS密码子的使用偏好性较弱。对‘湘辣14号’密码子的中性绘图分析表明,叶绿体基因全部位于标准曲线之上,且GC3与GC1和GC2中的A、T、C、G组成差异较大,GC12含量明显高于GC3,进一步表明‘湘辣14号’密码子偏好性的主要影响因素为自然选择,这与黄荆坪竹根椒(C. annuu L. cv HuangJingping)[30]、梁山慈竹(Dendrocalamus farinosus)[31]的结果相符。ENC-plot分析表明,自然选择比突变更加影响‘湘辣14号’密码子偏好性,该结果与叶琦等[32]对迎春樱桃(Prunus discoidea)、与胡晓艳等[33]对酸枣(Ziziphus jujuba var.spinosa)的研究结果相一致。影响紫花苜蓿(Medicago sativa)[34]密码子偏好性形成的主要原因则是突变。在芒果(Mangifera indicate L.)[35]密码子偏好性形成中突变和选择起同等重要作用。这些结果揭示了影响不同植物密码子的使用偏好性因素可能有所不同。
在‘湘辣14号’叶绿体基因组中共确定GCA、GCU、AGA等19个最优密码子,这些最优密码子全部以碱基A或U结尾,进一步证实了‘湘辣14号’密码子偏向使用NNA或 NNU模式,其使用模式与黄荆坪竹根椒[30]、牛茄子(Solanum capsicoides)[36]、黄果枸杞(Lycium ruthenicum)[37]等植物密码子使用模式一致。同时,‘湘辣14号’密码子第3位的高AU含量可能是造成这种使用模式的原因之一。在设计‘湘辣14号’外源基因时,优先考虑A或U结尾的最优密码子,可在一定程度上提高‘湘辣14号’外源基因表达效率,从而为‘湘辣14号’的性状改良及进化规律的研究提供理论支撑。
参考文献:
[1] CHEN Y, SHI Y, DENG H, et al. Characterization of the porcine epidemic diarrhea virus codon usage bias[J]. Infection, Genetics and Evolution, 2014, 28: 95-100.
[2] KANYEMA M M, CHENG M, LUO J, et al. Comprehensive codon usage analysis of the African Swine Fever Virus[J]. Acta Virologica, 2023, 67: 11562.
[3] XU Q, CAO J, RAI K R, et al. Codon usage bias of goose circovirus and its adaptation to host[J]. Poultry Science, 2024, 103(7): 103775.
[4] KOMAR A A. The Yin and Yang of codon usage[J]. Human Molecular Genetics, 2016, 25(R2): R77-R85.
[5] YU C H, DANG Y, ZHOU Z, et al. Codon usage influences the local rate of translation elongation to regulate co-translational protein folding[J]. Molecular Cell, 2015, 59(5): 744-754.
[6] YANG Q, LYU X, ZHAO F, et al. Effects of codon usage on gene expression are promoter context dependent[J]. Nucleic Acids Research, 2021, 49(2): 818-831.
[7] LIU Y, YANG Q, ZHAO F. Synonymous but not silent: The codon usage code for gene expression and protein folding[J]. Annual Review of Biochemistry, 2021, 90(1): 375-401.
[8] PANJA A S. The systematic codon usage bias has an important effect on genetic adaption in native species[J]. Gene, 2024: 148627.
[9] WU P, XIAO W, LUO Y, et al. Comprehensive analysis of codon bias in 13 Ganoderma mitochondrial genomes[J]. Frontiers in Microbiology, 2023, 14: 1170790.
[10]" GENG X, HUANG N, Zhu Y, et al. Codon usage bias analysis of the chloroplast genome of cassava[J]. South African Journal of Botany, 2022, 151: 970-975.
[11]" ZALUCKI Y M, POWER P M, JENNINGS M P. Selection for efficient translation initiation biases codon usage at second amino acid position in secretory proteins[J]. Nucleic Acids Research, 2007, 35(17): 5748-5754.
[12]" HU H, DONG B, FAN X, et al. Mutational bias and natural selection driving the synonymous codon usage of single-exon genes in rice (Oryza sativa L.)[J]. Rice, 2023, 16(1): 11.
[13]" 胡国平, 唐可兰, 刘阳华, 等. 三樟黄贡椒叶绿体基因组特征研究[J]. 长江蔬菜, 2023(14): 31-35.
[14]" 王传之, 周贤玉, 李扬眉, 等. 紫菜薹叶绿体全基因组序列及其系统发育分析[J]. 西北农业学报, 2024, 33(8): 1483-1494.
[15]" 仇学文, 李丹, 甘玉迪, 等. 豇豆叶绿体基因组密码子使用偏好性分析[J]. 核农学报, 2023, 37(6): 1118-1131.
[16]" 方萍萍, 张婷, 韦静, 等. 64份白辣椒种质资源的遗传多样性分析[J]. 浙江农业科学, 2024, 65(5): 1056-1063.
[17]" 王立浩, 张宝玺, 张正海, 等. 辣椒遗传育种研究进展[J]. 园艺学报, 2020, 47(9): 1727-1740.
[18]" 张瑞芳. 设施辣椒优质高产高效栽培新技术[J]. 长江蔬菜, 2024(5): 25-28.
[19]" ZHANG R, ZHANG L, Wang W, et al. Differences in codon usage bias between photosynthesis-related genes and genetic system-related genes of chloroplast genomes in cultivated and wild solanum species[J]. International Journal of Molecular Sciences, 2018, 19(10): 3142.
[20]" WEI L, HE J, JIA X, et al. Analysis of codon usage bias of mitochondrial genome in Bombyx mori and its relation to evolution[J]. BMC Evolutionary Biology, 2014, 14: 1-12.
[21]" NOVEMBRE J A. Accounting for background nucleotide composition when measuring codon usage bias[J]. Molecular Biology and Evolution, 2002, 19(8): 1390-1394.
[22]" SUEOKA N. Near homogeneity of PR2-bias fingerprints in the human genome and their implications in phylogenetic analyses[J]. Journal of Molecular Evolution, 2001, 53: 469-476.
[23]" YU T, LI J, YANG Y, et al. Codon usage patterns and adaptive evolution of marine unicellular cyanobacteria Synechococcus and Prochlorococcus[J]. Molecular Phylogenetics and Evolution, 2012, 62(1): 206-213.
[24]" CUI G, WANG C, WEI X, et al. Analysis of synonymous codon usage of the complete chloroplast genome in Phleum pratense cv. Minshan[J]. International Journal of Agriculture amp; Biology, 2020, 24(2): 352-358.
[25]" BIN W, JING Y, JING L, et al. Codon usage bias and determining forces in green plant mitochondrial genomes[J]. Journal of Integrative Plant Biology, 2011, 53(4): 324-334.
[26]" MORTON B R, WRIGHT S I. Selective constraints on codon usage of nuclear genes from Arabidopsis thaliana[J]. Molecular Biology and Evolution, 2007, 24(1): 122-129.
[27]" LIU Q, XUE Q. Comparative studies on codon usage pattern of chloroplasts and their host nuclear genes in four plant species[J]. Journal of Genetics, 2005, 84: 55-62.
[28]" 陆奇丰, 黄至欢, 骆文华. 番茄WRKY转录因子密码子偏性分析[J]. 分子植物育种, 2020, 18(18): 5908-5916.
[29]" 刘潮, 韩利红, 代小波, 等. 辣椒属叶绿体基因组特征及进化[J]. 热带作物学报, 2022, 43(3): 447-454.
[30]" 龚意辉, 唐诗眙, 周桂花, 等. 黄荆坪竹根椒叶绿体基因组密码子使用偏好性及影响因素分析[J]. 江苏农业科学, 2023, 51(20): 28-34.
[31]" 魏亚楠, 龚明贵, 白娜, 等. 梁山慈竹叶绿体基因组密码子偏好性分析[J]. 浙江农林大学学报, 2024, 41(4): 1-10.
[32]" 叶琦, 宋炎峰, 李蒙, 等. 迎春樱桃叶绿体基因组特征及其密码子使用偏好性分析[J]. 分子植物育种, 2022, 20(14): 4576-4585.
[33]" 胡晓艳, 许艳秋, 韩有志, 等. 酸枣叶绿体基因组密码子使用偏性分析[J]. 森林与环境学报, 2019, 39(6): 621-628.
[34]" 喻凤, 韩明. 紫花苜蓿叶绿体基因组密码子偏好性分析[J]. 广西植物, 2021, 41(12): 2069-2076.
[35]" 唐玉娟, 赵英, 黄国弟, 等. 芒果叶绿体基因组密码子使用偏好性分析[J]. 热带作物学报, 2021, 42(8): 2143-2150.
[36]" 龚秋怡, 董姝洁, 许琴, 等. 牛茄子叶绿体全基因组特征及系统发育分析[J]. 中草药, 2024, 55(12): 4150-4158.
[37]" 杜雨, 李效雄, 贾西贝, 等. 枸杞叶绿体基因组密码子偏好性分析[J]. 中草药, 2024, 55(4): 1316-1325.

