西瓜NPR基因家族的鉴定及其在盐胁迫下的表达分析

摘" " 要:以拟南芥NPR基因家族成员蛋白序列为查询序列,在西瓜基因组数据库中鉴定西瓜NPR基因家族成员,并对其进行生物信息学分析及盐胁迫下的表达分析。共鉴定到4个成员,将其命名为ClNPR1~ClNPR4,其编码蛋白的理化性质、保守功能结构域、重要氨基酸残基及motif分析结果与其他物种有较高的一致性,顺式作用元件分析显示,ClNPRs与非生物胁迫相关。300 mmol·L-1 NaCl处理耐盐材料和盐敏感材料,分别取盐胁迫0、8、24 h的叶片进行4个NPR基因的表达模式分析,结果表明,ClNPR1在盐敏感和耐盐材料中都显著上调表达,但盐胁迫24 h,ClNPR1在盐敏感材料中显著下调表达,在耐盐材料中虽然下调但还保持较高的表达水平,且在耐盐材料中的表达量显著高于盐敏感材料;盐胁迫24 h,ClNPR2和ClNPR3在盐敏感和耐盐材料中都显著上调表达,在耐盐材料中的表达量均显著高于盐敏感材料。推测ClNPRs基因在西瓜响应盐胁迫的过程中扮演重要角色。
关键词:西瓜;NPR家族蛋白;盐胁迫;表达
中图分类号:S651 文献标志码:A 文章编号:1673-2871(2025)01-016-09
Identification of NPR gene family and its expression under salt stress in watermelon
JIA Yunhe
(Horticulture Branch, Heilongjiang Academy of Agricultural Sciences, Harbin 150069, Heilongjiang, China)
Abstract: Using Arabidopsis NPR gene family members as query sequences, watermelon NPR gene family members were identified in watermelon genome database, and their bioinformatics and expression under salt stress were analyzed. A total of four members were identified and named ClNPR1-ClNPR4. Their physicochemical properties, conserved functional domains, important amino acid residues and motif analysis had high consistency with those reported in other species. Cis-acting element analysis showed that NPR gene was associated with abiotic stress in watermelon. Salt-tolerant and salt-sensitive material were treated with 300 mmol·L-1 NaCl, and the expression patterns of 4 NPR genes were analyzed in leaves under salt stress for 0, 8 and 24 h. The results showed that the expression of ClNPR1 was significantly up-regulated in both salt-sensitive and salt-tolerant materials, but the expression of ClNPR1 was significantly down-regulated in salt-sensitive materials for 24 h under salt stress. The expression level of ClNPR1 was significantly higher in salt-tolerant materials than in salt-sensitive materials. After 24 h of salt stress, ClNPR2 and ClNPR3 were significantly up-regulated in both salt-sensitive and salt-tolerant materials, and their expression level in salt-tolerant materials were significantly higher than those in salt-sensitive materials. It is speculated that ClNPRs play an important role in watermelon response to salt stress.
Key words: Watermelon; NPR family protein; Salt stress; Expression
我国是全球西瓜生产与消费第一大国。据FAO统计,2020年我国西瓜种植面积140.59万hm2,占全球的46.05%;产量6 024.69万t,占全球的59.29%,面积和产量均居世界第1位[1]。2021年产量达6 101.4万t[2]。西瓜在土壤含盐量0.2%以下时可以正常生长[3],可以利用盐碱地土壤富含钾的特点种植出高品质西瓜[4]。然而,土壤含盐量高于0.3%的盐胁迫会导致西瓜减产25%~35%[5],西瓜幼苗的耐盐性更弱[6]。我国天然盐碱地的面积约为14.8亿hm2,由于肥水的不合理应用造成的次生盐渍化土壤面积不断增加[7],盐胁迫严重制约着我国西瓜优质高效可持续性发展。
盐胁迫会对植物产生渗透胁迫和离子毒害[8];过氧化氢和丙二醛的含量显著增加[9],形成氧化胁迫,破坏细胞膜[10];造成气孔关闭,光合作用效率下降[11]。植物会对盐胁迫产生响应,如调节 Ca2+水平、维持较高的K+/Na+离子比率进行离子平衡,降低Na+的积累[12];积累脯氨酸进行渗透调节[13];提高超氧化物歧化酶(SOD)、过氧化物酶(POD)和过氧化氢酶(CAT)的活性来清除活性氧[14];多种激素通过协同或拮抗作用参与植物盐胁迫的响应[15]。
随着分子生物学技术的发展,已经从水稻[16]、玉米[17]、小麦[18]、拟南芥[19]等多种作物中鉴定到盐胁迫响应基因。NaCl胁迫处理后,与二倍体相比,四倍体西瓜幼苗受DNA超甲基化调控发生了一些基因的上调表达[20]。通过RNA-seq分析,共鉴定出21 069个基因,KEGG表明,一个TCP 转录因子在根和叶中同时存在,推测其可能受miR319调控,从而提高西瓜的耐盐性[21]。转录组学和代谢组学联合分析揭示了盐胁迫后使用外源海藻糖处理,西瓜幼苗根系bHLH家族转录因子在缓解盐胁迫过程中发挥了重要作用[22]。西瓜7个ClTPS基因广泛表达于根、茎、叶、花,盐胁迫对ClTPS3的诱导作用显著,ClTPS3过表达拟南芥的耐盐性显著提高[23]。
NPR1(non-expressor of PR genes)是植物水杨酸(SA,salicylic acid)信号通路的重要组成部分;也参与了水杨酸与茉莉酸/乙烯信号途径的相互作用[24]。NPR基因为组成型表达,NPR蛋白以寡聚体形式存在于细胞质中,当植物细胞内SA浓度上升时,寡聚体NPR发生解聚并进入到细胞核中,继而在植物系统获得抗性、PR基因表达等过程中发挥作用[25]。在拟南芥中,NPR基因家族共有6个成员:NPR1、NPR2、NPR3、NPR4、BOP2/NPR5和BOP1/NPR6 [26-28]。NPR蛋白含有3个典型的结构域,分别是位于N端的BTB/POZ结构域、位于中心区域的多个锚蛋白重复序列(ANK repeat)和位于C端NPR1_like_C结构域[29]。拟南芥的6个NPR 蛋白家族成员根据进化关系及功能分成三类,AtNPR1和AtNPR2为第一类,是水杨酸的受体蛋白,对水杨酸介导的植物免疫反应有正调控作用;AtNPR3和AtNPR4为第二类,同样作为水杨酸的受体蛋白,但对拟南芥抗病过程起负调控作用[19,30];AtNPR5和AtNPR6为第三类,蛋白C端不含 NPR1_like_C 结构域,参与叶、花发育。NPR1基因在植物抗病过程中发挥重要作用。在苹果中发现8个NPR1同源基因,经过分析鉴定,苹果NPR蛋白同样可以被分成三类,分类结果与拟南芥类似,在接种后的抗感品种中表达量差异显著[31]。在水稻中鉴定到3个NPR1同源基因,参与不同的抗病反应[32]。在香蕉中鉴定出15个NPR基因,其中MaNPR4和MaNPR11基因参与香蕉抗枯萎病过程[33]。在低温下,细胞质NPR1寡聚物释放单体,并转运到细胞核,与热休克转录因子1(HSFA1)相互作用,促进耐冷基因的表达,提高拟南芥的耐冷性[34]。
关于植物NPR蛋白对非生物胁迫响应的研究较少,笔者利用生物信息学方法鉴定西瓜NPR基因,分析其结构,探究其在盐胁迫下的表达模式,以期为挖掘耐盐相关基因、探究西瓜耐盐分子机制提供参考依据。
1 材料与方法
1.1 西瓜NPR基因家族成员鉴定
在西瓜基因组数据库CuGenDBv2(cucurbitgenomics.org)中,以6个已报道的拟南芥NPR蛋白为查询序列进行BLASTP搜索(E 值≤1×10-5)。通过SMART网站分析所得西瓜NPR成员蛋白序列结构域,去除冗余序列。利用在线网页ExPASy(http://web. expasy.org/ protparam/)对西瓜NPR蛋白序列的分子质量、理论等电点、稳定性、亚细胞定位等进行预测分析。
1.2 西瓜NPR家族成员的系统发育及结构分析
从TAIR (https://www. arabidopsis.org/)下载6个拟南芥NPR蛋白的氨基酸序列。使用MEGA X 软件,输入西瓜和拟南芥的NPR蛋白序列,使用MUSCLE进行多序列比对,输出数据以MEGA格式保存,采用邻接法构建系统发育树,重复值为1000,用iTOL (https://iTOL .embl.de/)对系统发育树进行可视化。
从西瓜基因组中获得NPR家族成员的外显子、内含子位置,使用MEME在线网站(https://meme-suite. org/)预测西瓜NPR蛋白中的保守基序,参数设置如下:最大基序数为10,最小图案宽度为6,最大图案宽度为100。使用NCBI保守结构域数据库CDD (https://www.ncbi.nlm.nih. gov/Structure/bwrpsb/bwrpsb.cgi)进行结构域分析,确定所有西瓜NPR蛋白序列的结构域类型和位置。通过PlantCARE(http://bioin‐ formatics.psb.ugent.be/webtools/ plantcare/html/)在线网站对家族成员上游2000 bp启动子序列的顺式作用元件进行预测。利用TBtools软件对西瓜NPR成员的保守基序、外显子-内含子结构及顺式作用元件进行可视化分析。
1.3 西瓜NPR家族成员的多序列比对及共线性分析
经过CLUSTALW分析和Espript可视化,多序列比对西瓜、拟南芥及水稻的NPR家族成员。运用TBtools软件的McScanX 功能对西瓜、甜瓜及拟南芥基因组进行共线性分析。
1.4 西瓜NPR家族成员在盐胁迫下的转录组分析
试验材料为前期筛选到的西瓜耐盐材料和盐敏感材料。于2024年2月在黑龙江省农业科学院园艺分院温室中使用无菌基质播种育苗。耐盐材料为野生西瓜PI296341,盐敏感材料为栽培品种分离出的纯合自交系。在幼苗2叶1心时,将育苗花盆置于托盘中,对照组浇清水,处理组浇300 mmol·L-1 NaCl溶液。分别采集盐胁迫0、8、24 h的耐盐材料和盐敏感材料幼苗叶片样品,-80 ℃冷冻保存。盐胁迫0、8 h的耐盐材料和盐敏感材料样品送华大基因公司测序。从转录组数据库中提取西瓜NPR基因的FPKM(每百万fragments中来自某一基因每千碱基长度的fragments数),通过计算3个生物学重复值平均数获得每个处理表达量值。
1.5 西瓜NPR基因家族成员在盐胁迫下的表达验证
使用Primer Premier 5.0软件设计4对NPR基因荧光标记定量引物,1对内参基因特异性引物(表1)。使用北京百泰克RNA提取试剂盒从耐盐材料和盐敏感材料的叶片中提取总RNA,在Veriti96 孔热循环仪(ABI,美国)中进行逆转录。qRT-PCR试验在QuantStudio 7 Flex实时荧光定量 PCR 仪上进行(ABI,美国)。反应体系10 μL:2×SYBR Premix EX TaqTM 5μL,10 μmol·L-1上下游引物各0.4 μL,cDNA 1 μL、ddH2O 3.2 μL。PCR扩增反应程序:95 ℃预变性30 s;95 ℃变性5 s,60 ℃退火34 s,40个循环。对熔解曲线进行分析,以验证数据的准确性,荧光定量试验进行3次重复。采用SPSS 25.0统计软件进行差异显著性分析,采用Excel软件绘图。
2 结果与分析
2.1 西瓜ClNPR基因家族鉴定
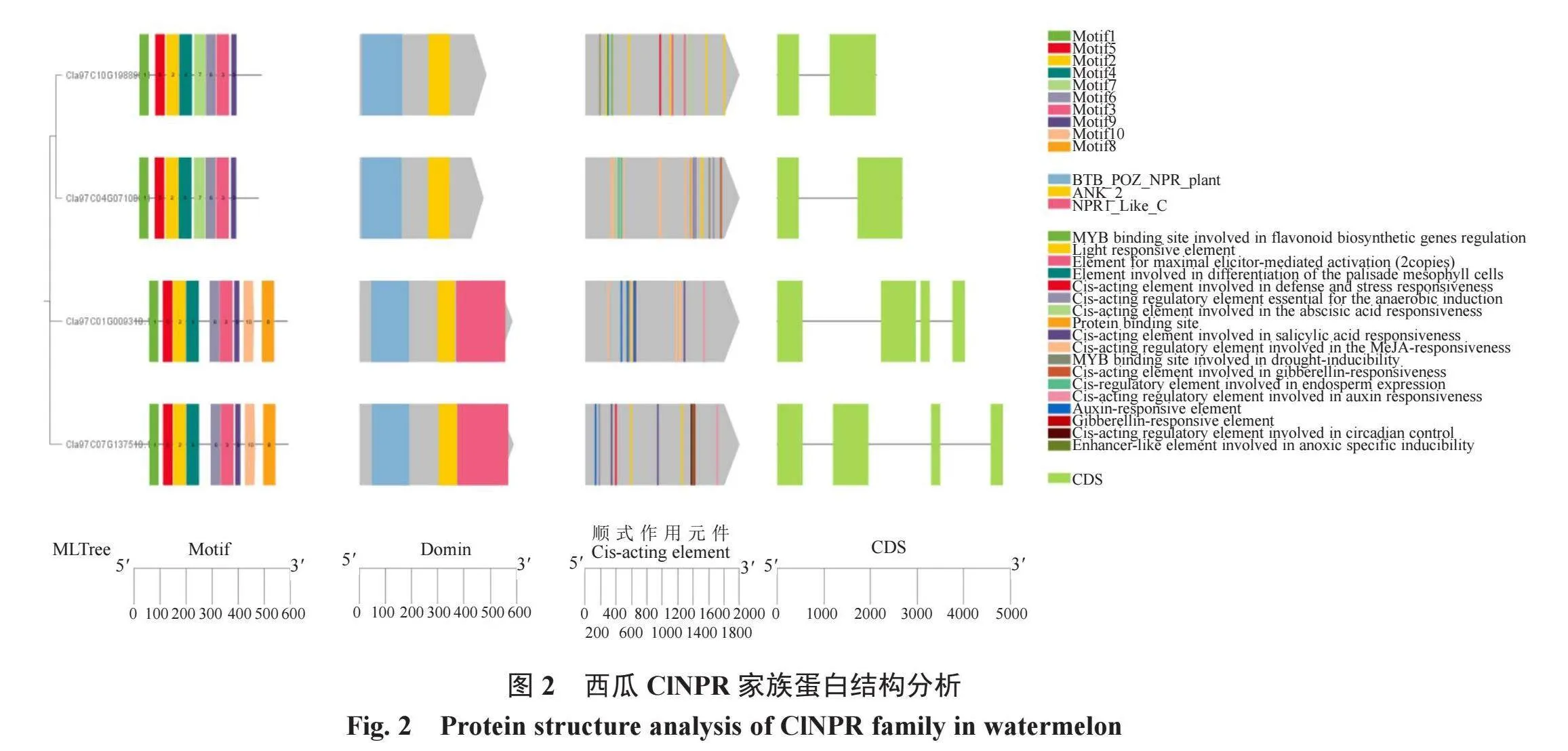
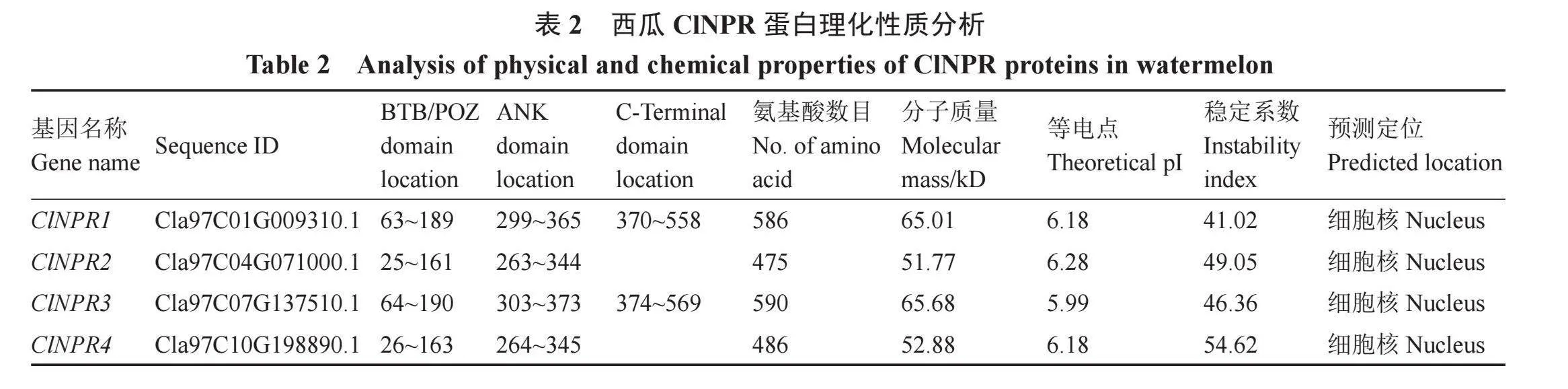
在西瓜基因组数据库中搜索NPR蛋白,经过SMART网站鉴定,西瓜基因组中共有4个NPR基因,分别位于01、04、07、10号染色体,分别命名为ClNPR1、ClNPR2、ClNPR3、ClNPR4。每个基因编码蛋白的具体理化性质见表2。ClNPR1和ClNPR3基因编码的蛋白含有保守的BTB/POZ、ANK repeat、NPR1_Like_C结构域,而ClNPR2和ClNPR4基因编码的蛋白无NPR1_Like_C结构域。编码蛋白质含有475~590个氨基酸,分子质量51.77~65.68 kD、等电点5.99~6.28、稳定系数41.02~54.62,亚细胞定位于细胞核内。
2.2 西瓜NPR家族成员的系统发育及序列分析
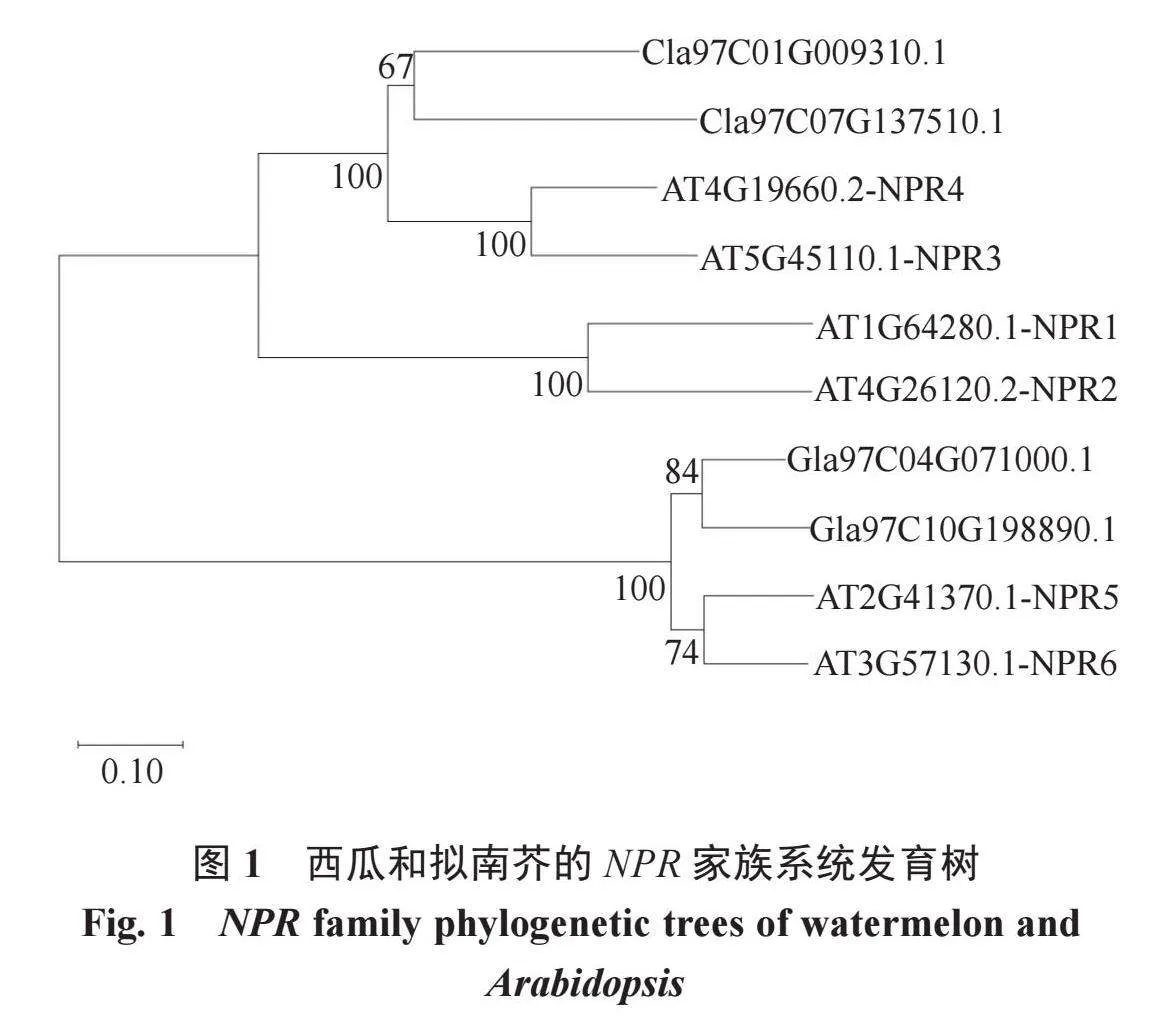
利用MEGA X软件对西瓜和拟南芥共10个NPR蛋白的全长序列进行比对,同时构建系统发育树。从种间进化树分支情况看,西瓜中4个NPR蛋白在2个分支中各有2个成员,其中ClNPR1和ClNPR3聚在一起,ClNPR2和ClNPR4聚在一起(图1)。
西瓜ClNPR基因家族序列分析表明,ClNPR1和ClNPR3基因DNA序列含有4个外显子和3个内含子,编码区包括3个domain,domain由10个motif组成。ClNPR2和ClNPR4基因DNA序列含有2个外显子和1个内含子,编码区包括2个domain,domain由8个motif组成(图2)。
对ClNPRs基因5’端上游2000 bp的序列分析发现,与其发生作用的顺式元件主要与激素(茉莉酸13个、水杨酸4个、生长素4个、赤霉素2个、ABA2个)、MYB(2个)、防御与胁迫(2个)、光响应(11个)等相关(图2、图3)。
2.3 西瓜NPR家族成员的多序列比对及共线性分析
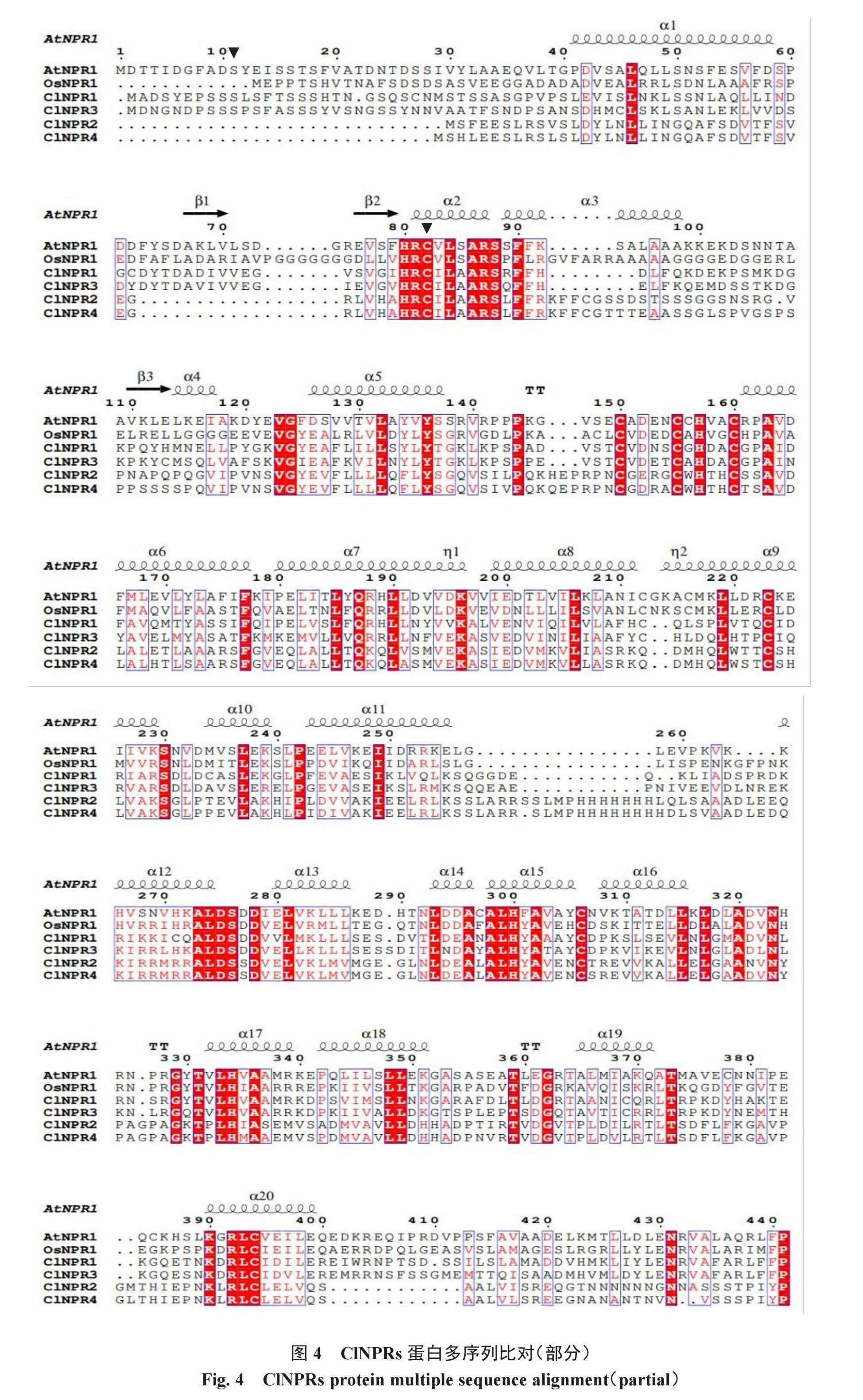
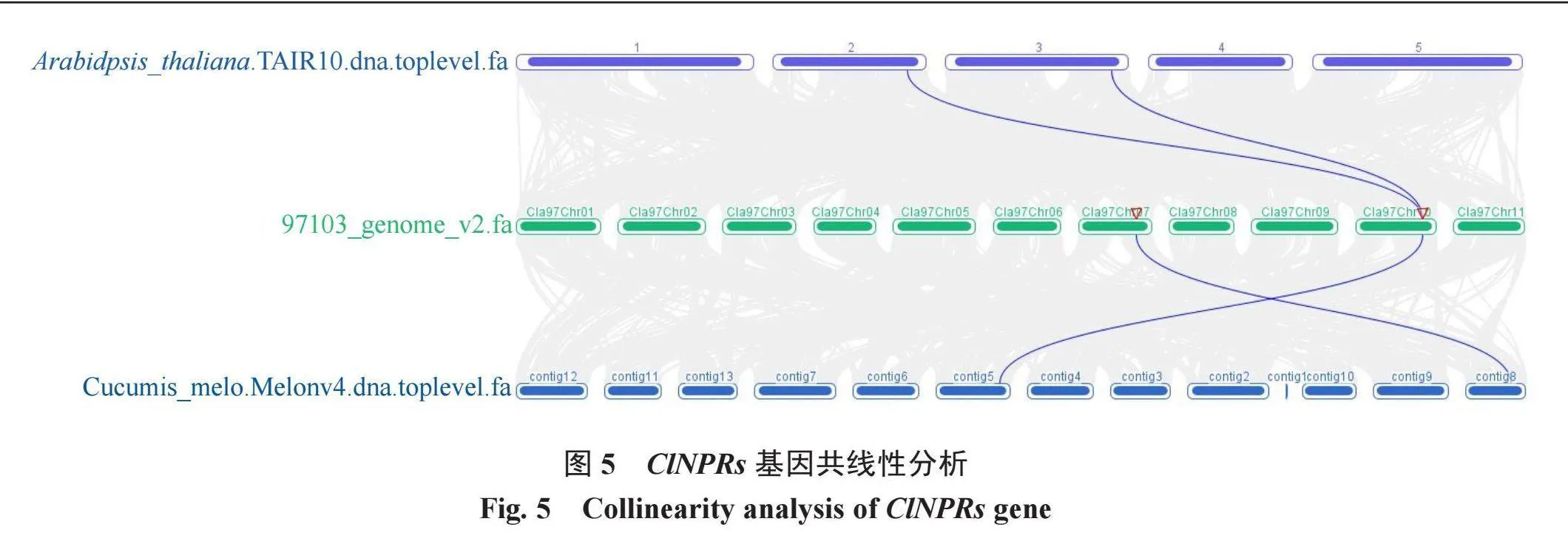
经过CLUSTALW分析和Espript可视化,多序列比对显示,NPR蛋白重要的功能结构域、氨基酸残基在多物种间高度保守,表明NPR基因在植物中行使相似功能(图4)。西瓜、甜瓜、拟南芥基因组共线性分析表明,ClNPR3和ClNPR4与甜瓜、拟南芥存在共线性(图5)。
2.4 西瓜ClNPR基因家族转录组分析
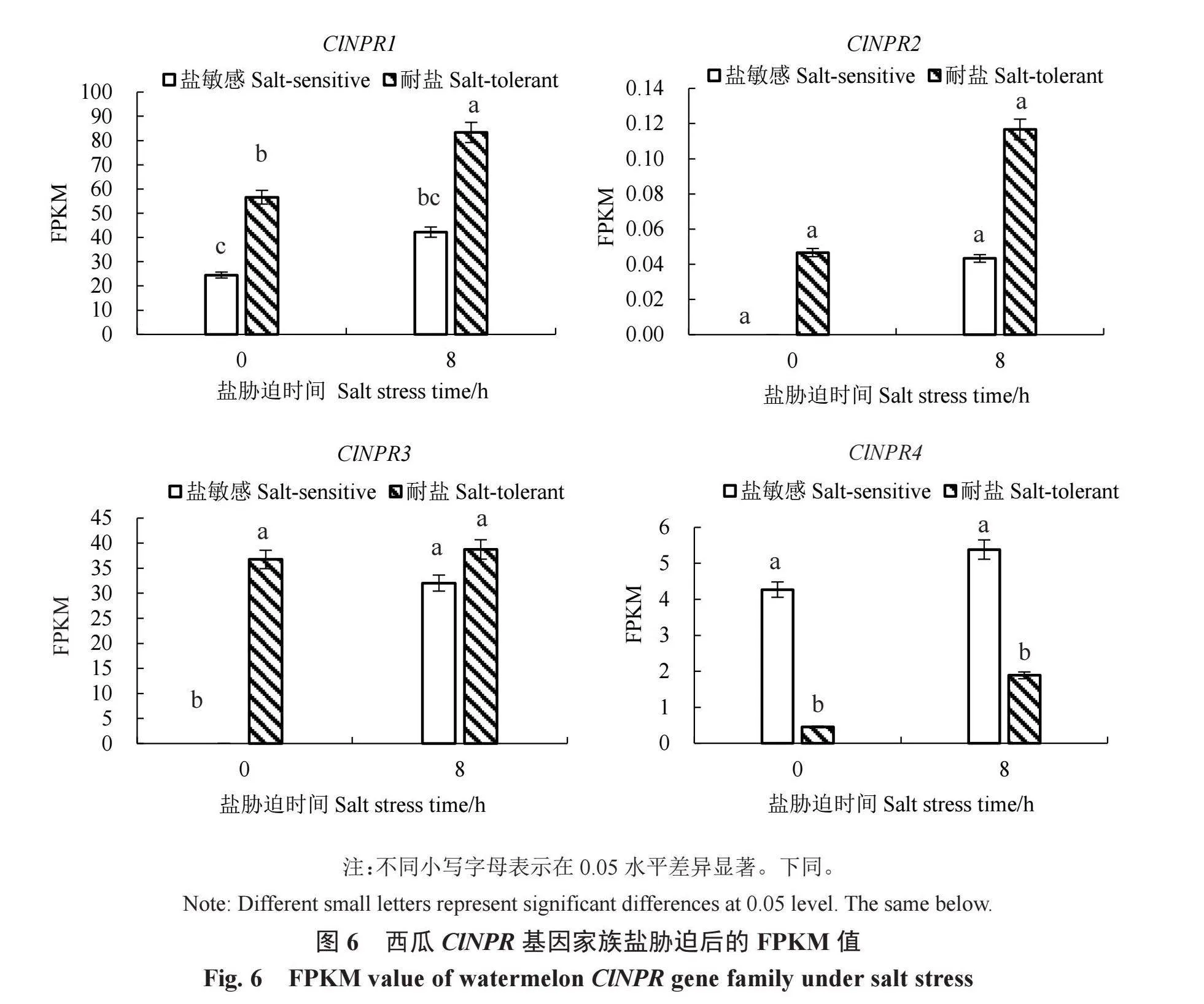
为了解西瓜NPR基因家族成员对盐胁迫的响应模式,从转录组数据中查找4个NPR基因的FPKM值,分析4个基因在NaCl处理下的表达情况(图6)。结果表明,与清水对照相比,NaCl胁迫处理8 h后,ClNPR1基因在盐敏感和耐盐材料中均上调表达,且耐盐材料的表达量显著高于盐敏感材料,清水对照的耐盐材料的ClNPR1基因表达量显著高于盐敏感材料;ClNPR2和ClNPR3基因的表达趋势与ClNPR1基因相似,但盐敏感材料和耐盐材料之间无显著差异;与清水对照相比,NaCl胁迫处理8 h后,ClNPR4基因在盐敏感和耐盐材料中都上调表达,但耐盐材料的表达量显著低于盐敏感材料,且清水对照的耐盐材料的ClNPR4基因表达量显著低于盐敏感材料。
2.5 西瓜ClNPR基因家族表达分析
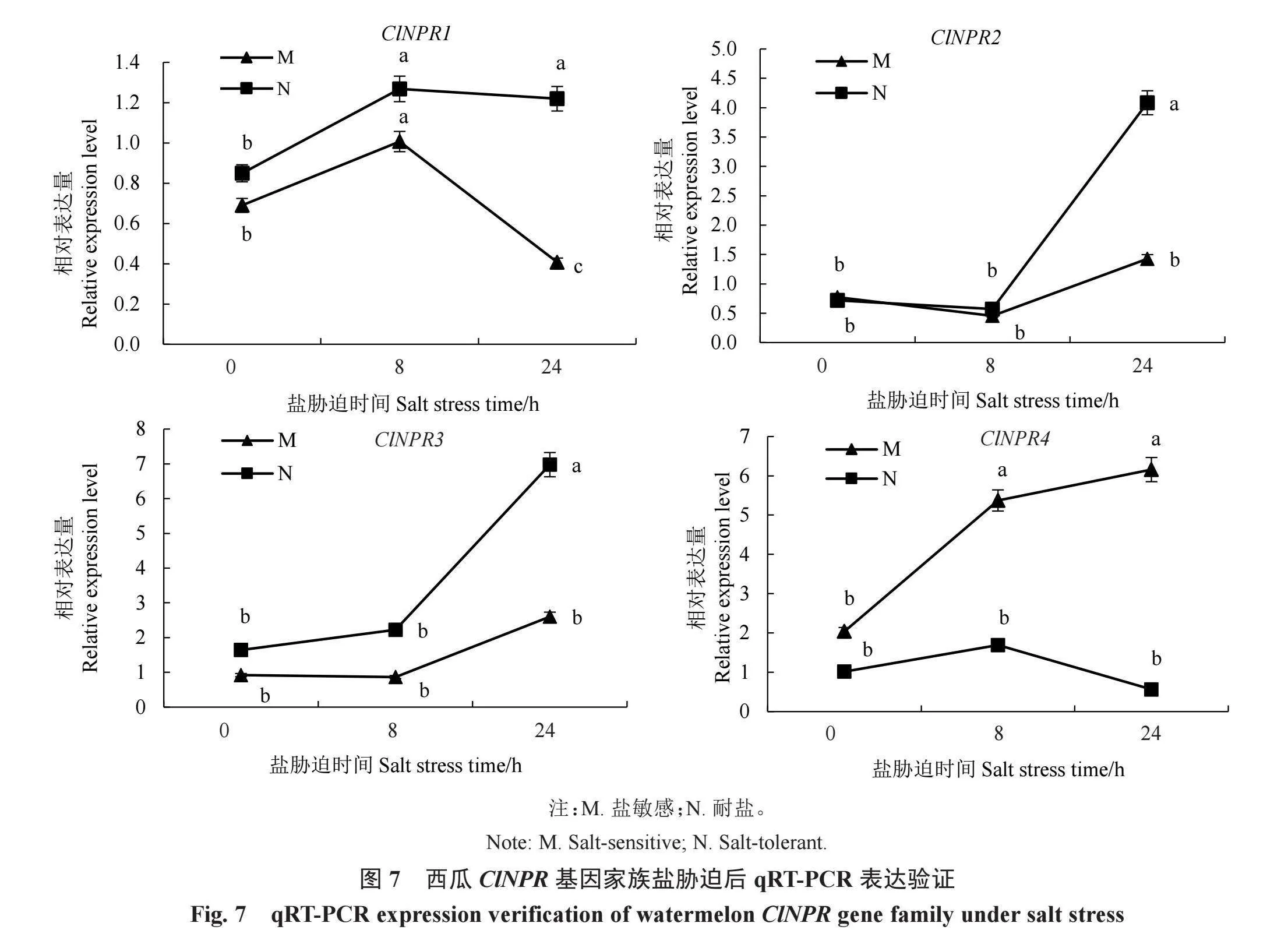
qRT-PCR结果表明,与对照相比,NaCl胁迫8 h,ClNPR1基因在盐敏感和耐盐材料中都显著上调表达;但盐胁迫24 h,ClNPR1基因在盐敏感材料中显著下调表达,在耐盐材料中虽然下调但还保持较高的表达水平,在耐盐材料中的表达量显著高于盐敏感材料。NaCl胁迫24 h,ClNPR2和ClNPR3基因在盐敏感和耐盐材料中都上调表达,在耐盐材料中的表达量显著高于盐敏感材料。NaCl胁迫后,ClNPR4基因在盐敏感材料中显著上调表达,在耐盐材料中先上调再下调表达,且在耐盐材料中的表达量显著低于盐敏感材料(图7)。
3 讨论与结论
笔者应用生物信息学分析的方法在西瓜基因组中鉴定出4个NPR基因,分别位于西瓜01、04、07、10号染色体。串联重复在植物基因家族成员增加中起关键作用[35],拟南芥基因组中包含6个NPR基因,分别位于1、2、3、4、5号染色体,其中AtNPR2(AT4G26120)和AtNPR4(AT4G19660)都位于4号染色体,表明拟南芥4号染色2个NPR基因为串联重复产生;在生菜基因组中鉴定到4个NPR基因,3个位于5号染色体,1个位于8号染色体,表明生菜5号染色体的3个NPR基因为串联重复产生[36];西瓜NPR基因家族未发现串联重复和片段重复事件,说明4个ClNPR基因具有不同的起源。
植物中NPR蛋白家族具有3个保守结构域:BTB/POZ结构域、ANK repeat结构域和NPR1_like_C结构域。西瓜ClNPR基因家族序列分析表明,ClNPR1和ClNPR3编码的蛋白包含BTB/POZ、ANK repeat和NPR1_like_C结构域,与AtNPR4(AT4G19660)、AtNPR3(AT5G45110)为同源基因;ClNPR2和ClNPR4编码的蛋白缺少NPR1_like_C结构域,与AtNPR5(AT2G41370)、AtNPR6(AT3G57130)为同源基因;这可能由于NPR基因在进化过程中,不同成员间功能差异性造成的,推测ClNPR2和ClNPR4可能参与不同的生物进程[30-31]。motif 1、5、2、4组成BTB/POZ结构域,motif 6、3、9组成 ANK repeat domains结构域,motif 9、10组成C-Terminal结构域,motif 7属于DUF(未知)结构域,这与前人的研究相符[37]。西瓜中4个NPR基因具有不同的外显子和内含子结构,说明他们表达时要进行选择性剪切。研究表明,选择性剪接在植物的生长发育过程以及响应非生物胁迫中发挥重要的调控作用[38],在苹果中,NPR正是通过选择性剪切发挥抗病作用的[31]。Ser 11高度保守,因此西瓜4个NPR蛋白能够在SAR诱导剂存在的情况下发生磷酸化,并参与调节植物的胁迫响应;Cys 82高度保守,在不受诱导的状态下,NPR低聚体不会解聚成为单体进入核内,不会诱导SAR的建立[37]。
拟南芥NPR1基因启动子中存在3个W-box,能与SA诱导表达的 WRKY蛋白特异性结合,在转录水平对NPR1基因的表达进行调控[39]。对水稻中OsNPR1基因起始位点上游1005 bp启动子区的顺式作用元件分析表明,启动子区包含W-box和ASF1,分别是转录因子WRKY和亮氨酸拉链(bZIP)的结合域,这两个区域是SA诱导SAR所必需的[40]。CDK8和WRKY18蛋白与OsNPR1基因启动子结合,不但促进了NPR1 表达,还促进了RNA聚合酶Ⅱ与NPR1靶基因PR1启动子及其编码区的结合,正调控其表达。NPR1在SA存在下,与RNA聚合酶Ⅱ一起富集CDK8、WRKY18和TGA 转录因子,从而促进自身表达以建立植物免疫的机制[41]。以上研究都表明植物NPR基因启动子对于响应胁迫的作用至关重要。西瓜NPR基因家族的启动子区存在许多和激素及防御相关的顺式作用元件,所有基因均含有水杨酸响应元件,同时还鉴定出茉莉酸响应元件,这预示着西瓜NPR基因家族成员可能在植物逆境胁迫反应中发挥重要作用。
拟南芥npr1-5突变体,缺乏npr1依赖的SA信号通路,而nudt7(nudix hydrolase7),既表达npr1依赖的SA信号通路,也表达npr1独立的SA信号通路。与野生型(Col-0)相比,npr1-5突变体对盐胁迫敏感,而nudt7突变体则具有耐受性。与野生型和nudt7突变体相比,急性盐胁迫导致npr1-5根的膜电位去极化最强,钠和质子流入最多,钾流失最多,且长期盐胁迫导致npr1-5突变体茎部钠含量最高,钾含量最低。以上结果表明,依赖npr1的SA信号对控制Na+进入根组织及其随后的长距离运输到茎部,以及防止钾的流失至关重要,可见npr1依赖的SA信号通路是拟南芥耐盐的核心[42]。在高粱中鉴定出5个NPR1基因,qRT-PCR表明,SbNPR1基因可能是高粱响应盐胁迫的一个功能基因[43]。qRT-PCR表明,在大白菜中鉴定到11个NPR基因,均在盐胁迫后6~12 h显著表达,推断大白菜BrNPR家族基因极可能参与了植物对盐胁迫的响应,并起到正向调控的作用[44]。马铃薯StNPR4具有典型的NPR1家族的功能结构域,启动子上具有响应生物胁迫和非生物胁迫的顺式作用元件,受SA诱导表达,转基因马铃薯在高盐胁迫下生根率更高,说明StNPR4在马铃薯非生物胁迫中发挥重要作用[45]。在本研究中,西瓜ClNPR1、ClNPR2、ClNPR3都在盐胁迫后上调表达,且在耐盐材料中的表达量高于盐敏感材料,推测西瓜NPR基因为西瓜盐胁迫响应途径中的功能基因,此结果与前人的研究相互印证。
综上所述,笔者在西瓜全基因组中鉴定出4个NPR基因,其编码蛋白的理化性质、保守功能结构域、重要氨基酸残基及motif分析结果与其他物种有较高的一致性,顺式作用元件分析表明,西瓜NPR与非生物胁迫相关。ClNPRs基因在盐胁迫24 h的盐敏感材料和耐盐材料中显著差异表达,推测ClNPRs基因在西瓜响应盐胁迫的过程中扮演重要角色。研究结果为挖掘耐盐相关基因及探究西瓜耐盐分子机制提供了参考依据。
参考文献
[1] 柯小娟,张蕾琛,张华峰,等.短蔓西瓜遗传育种研究进展[J].蔬菜,2023(2):33-38.
[2] 张磊,武竞春,张海英,等.西瓜重要性状功能基因研究进展[J].园艺学报,2023,50(12):2748-2764.
[3] 刘鑫,史庆华,王硕硕,等.盐胁迫对不同西瓜品种幼苗生长和生理指标的影响[J].山东农业科学,2018,50(1):55-59.
[4] 范学山,庄定云,张慧,等.浙江沿海地区耐盐西瓜品种对比试验[J].浙江农业科学,2021,62(5):881-883.
[5] ROMIC D,ONDRASEK G,ROMIC M,et al.Salinity and irrigation method affect crop yield and soil quality in watermelon (Citrullus lanatus L.) growing[J].Irrigation and Drainage,2008,57(4):463-469.
[6] 佘德琴,顾绘.不同配比育苗基质提高西瓜幼苗耐盐性研究[J].上海农业学报,2013,29(6):111-113.
[7] 于贝贝,何莉莉,杨生茂,等.木醋液改良盐碱土的研究进展[J].国土与自然资源研究,2024(1):62-67.
[8] HANSEN T B,JENSEN T I,CLAUSEN B H,et al.Natural RNA circles function as efficient micro RNA sponges[J].Nature,2013,495(7441):384-388.
[9] EKINCI M,YILDIRIM E,TURAN M.Ameliorating effects of hydrogen sulfide on growth,physiological and biochemical characteristics of eggplant seedlings under salt stress[J].South African Journal of Botany,2021,143:79-89.
[10] AHMAD P,ABD_ALLAH E F,ALYEMENI M N,et al.Exogenous application of calcium to 24-epibrassinosteroid pre-treated tomato seedlings mitigates NaCl toxicity by modifying ascorbate-glutathione cycle and secondary metabolites[J].Scientific Reports,2018,4:e05005.
[11] DUBY G,BOUTRY M.The plant plasma membrane proton pump ATPase:A highly regulated P-type ATPase with multiple physiological roles[J].Pflügers Archiv-European Journal of Physiology,2009,457(3):645-655.
[12] CU M,CHEN P,MENG S,et al.The Arabidopsis phosphatase PP2C49 negatively regulates salt tolerance through inhibition of At HKT1;1[J].Journal of Integrative Plant Biology,2021,63(3):528-542.
[13] 许祥明,叶和春,李国凤.植物抗盐机理的研究进展[J].应用与环境生物学报,2000,6(4):379-387.
[14] LU Z Q,LIU D L,LIU S K.Two rice cytosolic ascorbate peroxidases differentially improve salt tolerance in transgenic Arabidopsis[J].Plant Cell Reports,2007,26(10):1909-1917.
[15] 姚曼红,刘琳,曾幼玲.五大类传统植物激素对植物响应盐胁迫的调控[J].生物技术通报,2011(11):1-5.
[16] WEI H,WANG X M,ZHANG Z P,et al.Uncovering key salt-tolerant regulators through a combined eQTL and GWAS analysis using the super pan-genome in rice[J].National Science Review,2024,11(4):nwae043.
[17] MA L L,ZHANG M Y,CHEN J,et al.GWAS and WGCNA uncover hub genes controlling salt tolerance in maize (Zea mays L.) seedlings[J].Theoretical and Applied Genetics,2021,134(10):3305-3318.
[18] QUAMRUZZAMAN M,MANIK S M N,SHABALA S,et al.Genome-wide association study reveals a genomic region on 5AL for salinity tolerance in wheat[J].Theoretical and Applied Genetics,2022,135(2):709-721.
[19] ZHANG Y L,CHENG Y T,QU N,et al.Negative regulation of defense responses in Arabidopsis by two NPR1 paralogs[J].Plant Journal,2006,48(5):647-656.
[20] 朱红菊.四倍体西瓜幼苗耐盐机制研究[D].武汉:华中农业大学,2019.
[21] ZHU Y C,YUAN G P,GAO B W,et al.Comparative transcriptome profiling provides insights into plant salt tolerance in watermelon (Citrullus lanatus)[J].Life,2022,12(7):1033.
[22] YUAN G P,SUN D X,AN G L,et al.Transcriptomic and metabolomic analysis of the effects of exogenous trehalose on salt tolerance in watermelon (Citrullus lanatus)[J].Cells,2022,11(15):2338.
[23] YUAN G P,LIU J P,AN G L,et al.Genome-wide identification and characterization of the trehalose-6-phosphate synthetase (TPS) gene family in watermelon (Citrullus lanatus) and their transcriptional responses to salt stress[J].International Journal of Molecular Science,2022,23(1):276.
[24] EL OIRDI M,ABD EL RAHMAN T,RIGANO L,et al.Botrytis cinerea manipulates the antagonistic effects between immune pathways to promote disease development in tomato[J].Plant Cell,2011,23(6):2405-2421.
[25] 杨文博,严汉池.植物水杨酸受体NPR结构与功能研究进展[J].中国野生植物资源,2023,42(11):70-76.
[26] LIU G S,HOLUB E B,ALONSO J M,et al.An Arabidopsis NPR1-like gene,NPR4,is required for disease resistance[J].Plant Journal,2005,41(2):304-318.
[27] NORBERG M,HOLMLUND M,NILSSON O.The BLADE ON PETIOLE genes act redundantly to control the growth and development of lateral organs[J].Development,2005,132(9):2203-2213.
[28] HEPWORTH S R,ZHANG Y L,MCKIM S,et al.Blade-on-petiole-dependent signaling controls leaf and floral patterning in Arabidopsis[J].Plant Cell,2005,17(5):1434-1448.
[29] BOYLE P,LE SU E,ROCHON A,et al.The BTB/POZ domain of the Arabidopsis disease resistance protein NPR1 interacts with the repression domain of TGA2 to negate its function[J].Plant Cell,2009,21(11):3700-3713.
[30] DING Y L,SUN T J,AO K,et al.Opposite roles of salicylic acid receptors NPR1 and NPR3/NPR4 in transcriptional regulation of plant immunity[J].Cell,2018,173(6):1454-1467.
[31] ZHANG J K,JIAO P,ZHANG C,et al.Apple NPR1 homologs and their alternative splicing forms may contribute to SA and disease responses[J].Tree Genet and Genomes,2016,12(5):92.
[32] YUAN Y X,ZHONG S H,LI Q,et al.Functional analysis of rice NPR1-like genes reveals that OsNPR1/NH1 is the rice orthologue conferring disease resistance with enhanced herbivore susceptibility[J].Plant Biotechnology Journal,2007,5(2):313-324.
[33] 任陪娣,谢碧玉,陈翠,等.香蕉NPR1基因家族的鉴定及在枯萎病菌胁迫下表达分析[J].植物遗传资源学报,2019,20(6):1621-1629.
[34] OLATE E,JIMENEZ-GOMEZ J M,HOLUIGUE L,et al.NPR1 mediates a novel regulatory pathway in cold acclimation by interacting with HSFA1 factors[J].Nature Plants,2018,4(10):811-823.
[35] MUSAVIZADEH Z,NAJAFI-ZARRINI H,KAZEMITABAR S K,et al.Genome-wide analysis of potassium channel genes in rice: Expression of the OsAKT and OsKAT genes under salt stress[J].Genes,2021,12(5):784.
[36] 王祖弦,王晨晨,刘雪,等.生菜NPR家族基因鉴定与分析[J/OL].分子植物育种,[2023-7-19].https://kns.cnki.net/kcms2/detail/46.1068.S.20230719.0908.002.html.
[37] SPOEL S H,MOU Z L,TADA Y,et al.Proteasome-mediated turnover of the transcription coactivator NPR1 plays dual roles in regulating plant immunity[J].Cell,2009,137(5):860-872.
[38] LALOUM T,MARTIN G,DUQUE P.Alternative splicing control of abiotic stress responses[J].Trends in Plant Science,2018,23(2):140-150.
[39] YU D Q,CHEN C H,CHEN Z X.Evidence for an important role of WRKY DNA binding proteins in the regulation of NPR1 gene expression[J].Plant Cell,2001,13(7):1527-1539.
[40] HWANG S H,HWANG D J.Isolation and characterization of the rice NPR1 promoter[J].Plant Biotechnoloy Reports,2010,4(1):29-35.
[41] CHEN J,MOHAN R,ZHANG Y Q,et al.NPR1 promotes its own and target gene expression in plant defense by recruiting CDK81[J].Plant Physiology.2019,181(1):289-304.
[42] JAYAKANNAN M,BOSE J,BABOURINA O,et al.The NPR1-dependent salicylic acid signalling pathway is pivotal for enhanced salt and oxidative stress tolerance in Arabidopsis[J].Journal of Experimental Botany,2015,66(7):1865-1875.
[43] 杜巧丽,方远鹏,蒋君梅,等.高粱病程相关基因非表达子1(NPR1)基因家族鉴定及胁迫应答分析[J].核农学报,2021,35(5):1074-1083.
[44] 王攀,蔡兆明,廖静静,等.大白菜NPR家族基因鉴定及表达模式分析[J].分子植物育种,2021,19(3):746-758.
[45] 李登高,林睿,穆青慧,等.马铃薯StNPR4基因的克隆与功能分析[J].植物研究,2022,42(5):821-829.

