青海干旱盐碱地区不同生境土壤微生物多样性分析


摘 要 为研究不同生境土壤微生物群落多样性及其与土壤理化性质的关系,采集青海海西格尔木周边耕作土壤、非耕作土壤、荒漠土壤及盐碱土壤,测定其理化性质,利用Illumina-MiSeq平台进行16S扩增子测序,分析不同类型土壤微生物群落组成和多样性,研究土壤菌群与土壤理化性质的对应关系,为干旱盐碱条件下植物抗逆生长提供理论数据。结果表明:4种土壤的微生物群落多样性为耕作土gt;盐碱土gt;非耕作土gt;荒漠土。细菌优势类群(门、属)的组成及丰度在不同地区间存在差异。其中,耕作土壤的优势微生物为变形菌门、拟杆菌门、放线菌门、芽孢单菌门;非耕作土壤的芽孢单菌门丰度相对于耕作土壤降低,而拟杆菌门和放线菌门的丰度则较高。荒漠土壤和盐碱土壤内的优势微生物为变形菌门、拟杆菌门、放线菌门和厚壁菌门,厚壁菌门在盐碱土壤中的丰度高于荒漠土壤,放线菌门则相反。鞘脂单胞菌属是耕作土壤的最优势属,盐单胞菌属为非耕作土壤、荒漠土壤和盐碱土的最优势属。理化因子与土壤微生物群落相关性分析显示,电导率、水溶性盐含量与土壤微生物群落呈极显著正相关,有机碳含量和微生物群落多样性不具有显著相关性。表明电导率和水溶性盐含量是影响柴达木干旱盐碱土壤微生物群落多样性的主要因子。
关键词 土壤微生物;群落;多样性;理化性质
土壤是一种复杂生态系统[1],影响土壤生态系统多样性的因素有很多,如土壤营养[2]、水分含量、土壤pH[3-4]、温度[5]等非生物因素和植被覆盖度、动物、土壤微生物等生物因素,其中土壤微生物是影响土壤生态系统的关键因素之一。土壤微生物的生长、代谢直接影响土壤理化性质和植物生长状况[6]。土壤微生物群落多样性存在地区性差异,在不同生境下,土壤微生物群落多样性也不一致。
微生物在土壤有机质分解和土壤环境改善方面具有重要的作用。邵微等[7]研究发现,随着微生物群落多样性增加,土壤生态系统稳定性及其应对环境恶化的缓冲能力有明显提高。土壤中存在着数量庞大且种群复杂的微生物[8-9],在调节土壤理化性质中发挥了重要作用。研究表明,植物生长与其根际微生物多样性密切相关[10]。微生物能够分解土壤有机质以方便植物吸收利用,微生物自身分泌物还能影响植物根系发育,间接影响植物吸收土壤内营养物质[11],提高植物对所处环境的适应性,降低环境中胁迫因子对植物的危害[12-13]。植物根系分泌物也会进入土壤,成为土壤微生物的碳源和能源。因此,土壤微生物与植物间存在协同进化关系[14]。
柴达木盆地位于中国西北干旱地区,土壤荒漠化、盐碱化现象常见。干旱、盐碱等因素对本地区生物多样性具有明显的影响。现阶段对柴达木盆地不同生境的土壤理化性质及土壤微生物群落组成和多样性研究较少,对土壤微生物群落结构和植物多样性的影响仍不清楚。本文拟对柴达木地区不同生境土壤中微生物群落多样性展开分析,通过16S测序,分析不同生境土壤的微生物多样性和群落结构,了解土壤微生物群落多样性特征[15],结合土壤理化性质,研究土壤微生物群落多样性与土壤类型的关系,重点关注干旱盐碱地区耕作对土壤微生物的影响,以了解该地区农业生产对土壤微生物多样性的影响,为本地区作物增产增收提供基础数据。
1"材料与方法
1.1 材 料
土壤样品于2021年2月采自青海省格尔木市(36°41'70″N,95°08'07″E,海拔2 780 m)。采集耕地内距枸杞()植株的Lycium barbarum1m土壤作为耕作土(CS);耕地周边距离500 m的土壤作为非耕作土(NCS);耕地周边荒漠作为荒漠土(DS);耕地周边盐碱滩地土壤作为盐碱土(SS)。取样前将土壤表面的石头、固体盐壳等移除。利用多点采样法进行土壤收集,取距离地面10~15 cm处土壤。对于每种类型的土样,于 1m边长正方形的顶点处及中心采集5 份质量相当的土样混合成一个有代表性的土样。每种类型土壤采取5份样品,共20份土壤样品,分别为CS1~5,NCS1~5,DS1~5和SS1~5。土壤采集后立即密封处理,并且在冷藏(冰袋)条件下送回实验室。置于液氮内保存,用于土壤微生物的DNA提取。
1.2 方 法
1.2.1 土壤理化性质分析 土壤电导率(EC)及水溶性盐含量(WSS)采用电导率仪(DDSJ-319L)测定;土壤pH采用笔式pH计(PHB-10)测定 。1.2.2 土壤有机碳(TOC)测定 称取干燥土样0.5 g于聚四氟乙烯坩埚中,加入5mL重铬酸钾标准溶液,后加入5 mL浓硫酸混匀,在200℃的加热板上加热10min,待冷却后将样品转入锥形瓶中。以邻菲罗啉作为指示剂,用溶液进FeSO4行滴定。
1.2.3 DNA测序及数据处理 准确称取0.1g土样,按MoBio强力土壤微生物DNA提取试剂盒(MagPure Soil DNA LQ Kit)说明书分别提取各样地土样总DNA。利用Illumina-MiSeq平台进行16S扩增子测序。使用Trimmomatic(ver-sion 0.35)[16],软件扫描rawdata剔除质量较低序列;使用Flash(version1.2.11)[17]软件开展序列拼接,得到完整的pairedend序列。使用QI-IME中的split-libraries(version1.8.0)[18]软件筛选,得到clean tags。用UCHIME(versio n 2.4.2)[19]软件去除cleantags的嵌合体,得到validtags。对16S序列划分操作分类单元(oper-ational taxonomic unit,OTU),划定阈值为97%。使用Vsearch(version 2.4.2)[20]软件和RDP classifier Naive Bayesian分类算法[21]对validtags进行OTU分类,在不同分类界元上统计不同土壤的细菌群落组成。基于物种分类分析,绘制物种分类条形图及物种丰度热图。对微生物多样性进行Alpha多样性分析和Beta多样性分析。序列经重取样统一测序深度(16S16S为60000条)后,对Shannon指数、Simpson指数进行计算。以OTU丰富度对序列数作图,进行稀释分析。采用主坐标分析(PcoA)法分析微生物群落间的相似性。
1.3 数据分析
用Excel 2016整理数据,用SPSS21.0计算数据,均以“平均值±标准误”的形式表示。采用Duncan’s法进行多重比较,以Plt;0.05为差异显著,Plt;0.01为差异极显著。用配对t检验比较土壤间细菌群落多样性的差异性;用典范对应分析(CCA)检验微生物多样性与土壤理化性质间的关系。
2 结果与分析
2.1 土壤理化性质
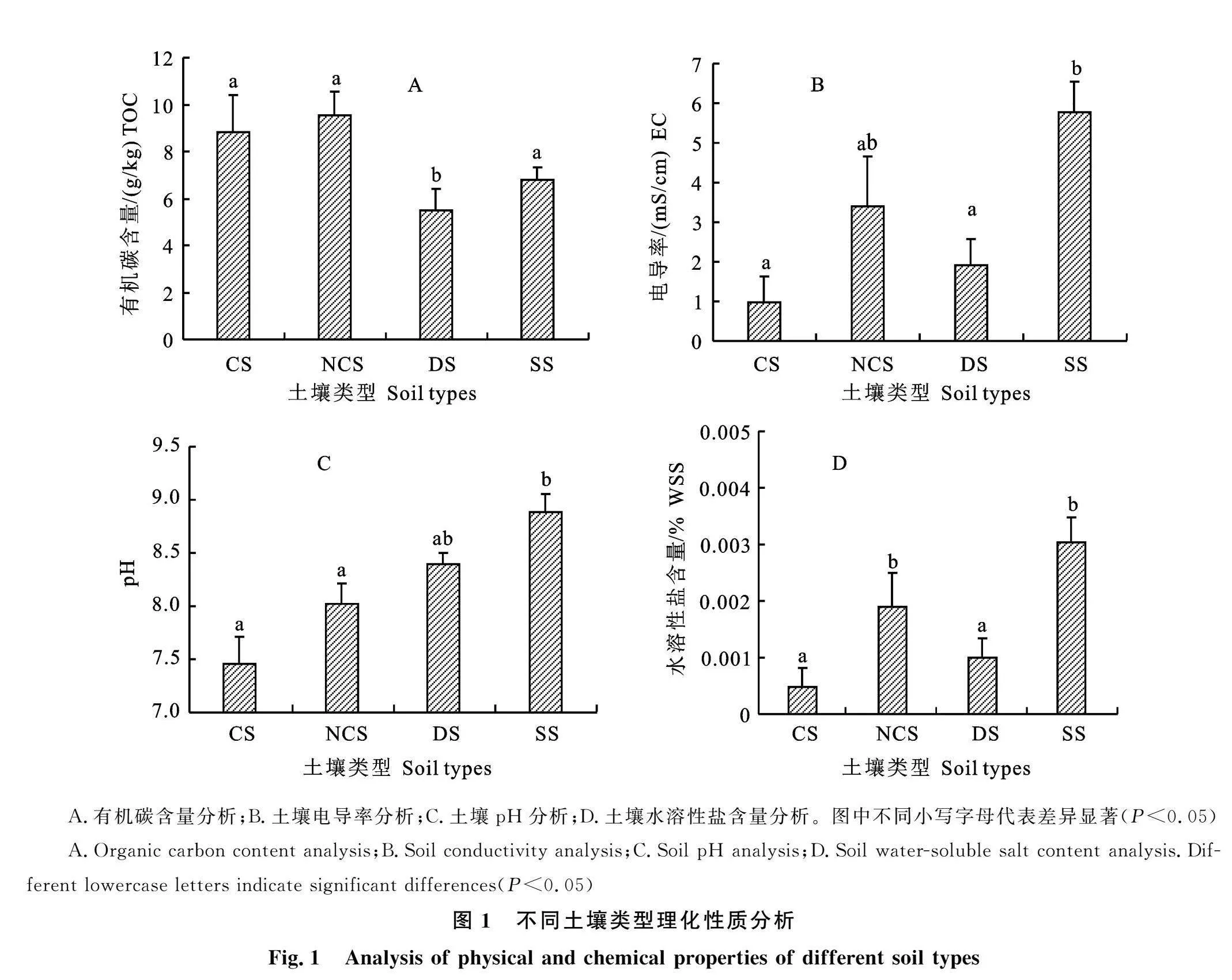
理化性质测定结果表明,DS组TOC含量最低,与CS组、NCS组、SS组差异显著(Plt;0.05);对EC分析发现,CS组电导率最低,SS组最高,差异极显著(Plt;0.01)(图1-B);SS组平均pH大于其余组,且SS与CS、NCS之间存在显著差异(Plt;0.05)(图1-C);CS组水溶性盐含量最低,且与、组差异显著(图),推测是由NCSSS1-D于植物生长及耕作过程影响土壤内离子含量,从而影响土壤电导率,水溶性盐含量与土壤电导率密切相关。
2.2 土壤微生物多样性及群落结构
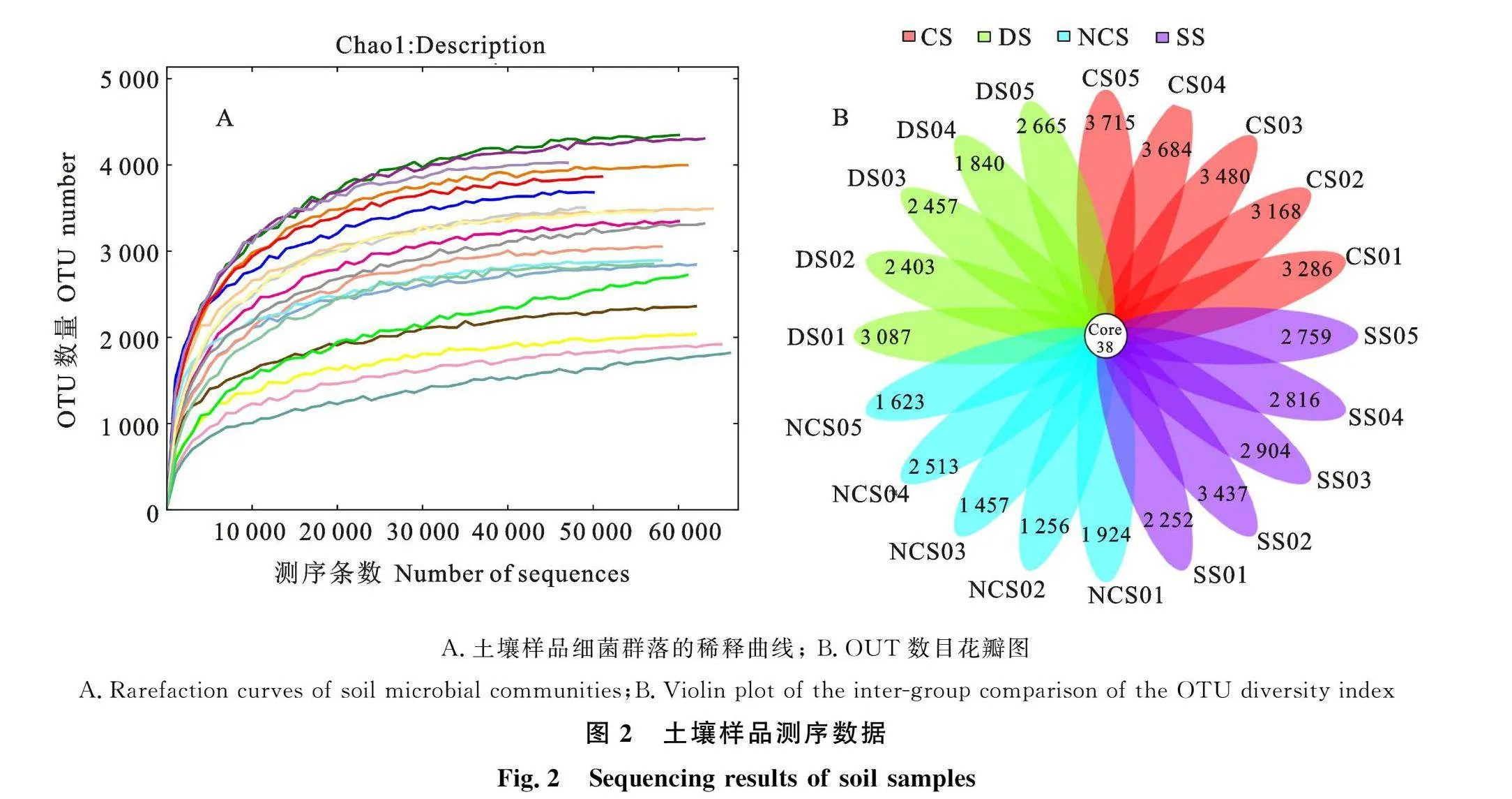
2.2.1 16S测序 测序结果显示,样品测序覆盖度在0.99,表明测序结果能较真实反映土壤内微生物群落结构组成(图2).
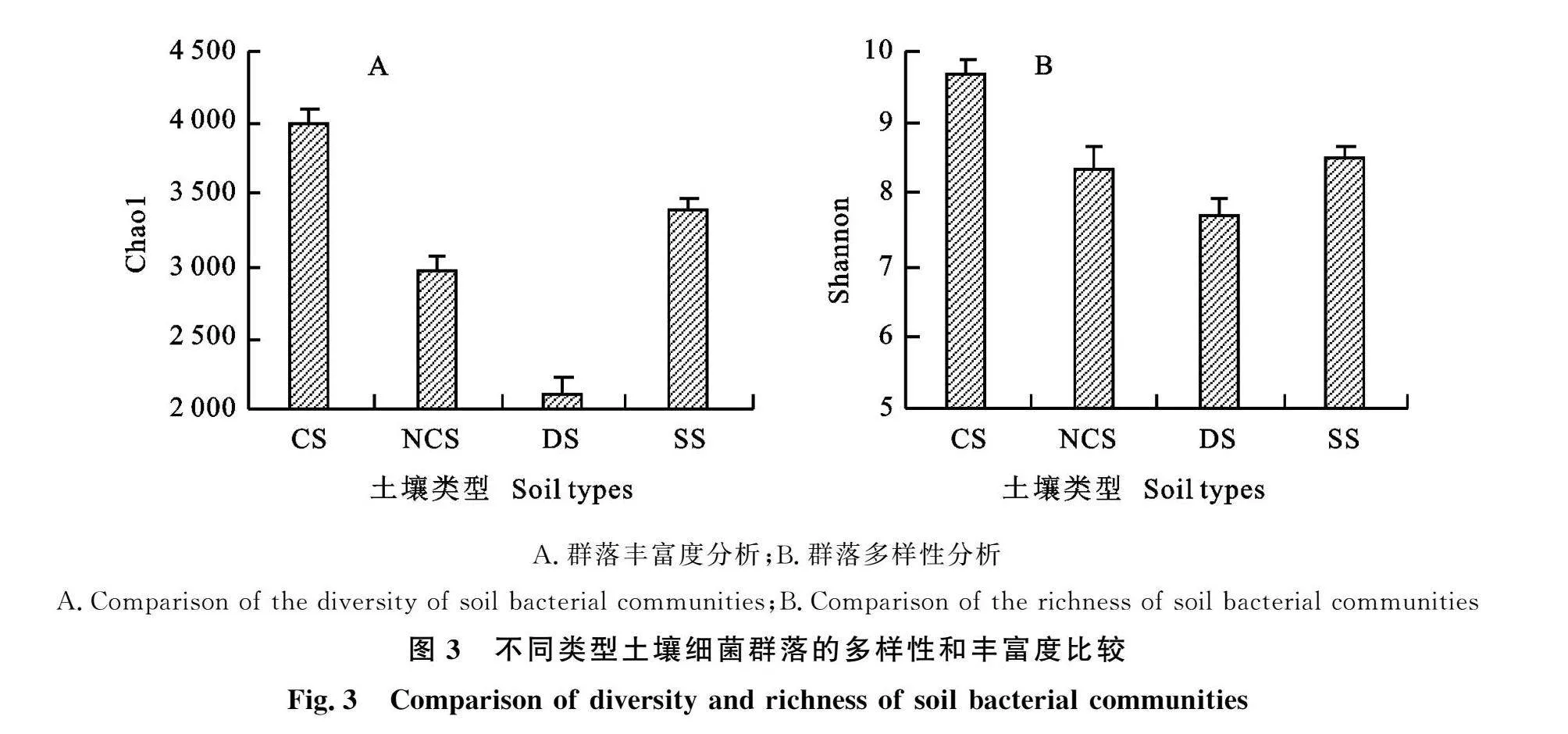
2.2.2 微生物群落多样性 不同土壤的细菌群落多样性和丰度差异显著(图3),细菌群落多样性为CSgt;SSgt;NCSgt;DS。其中,CS组细菌群落多样性和丰度均显著高于NCS组;SS组细菌群落的多样性和丰度均显著高于DS组。
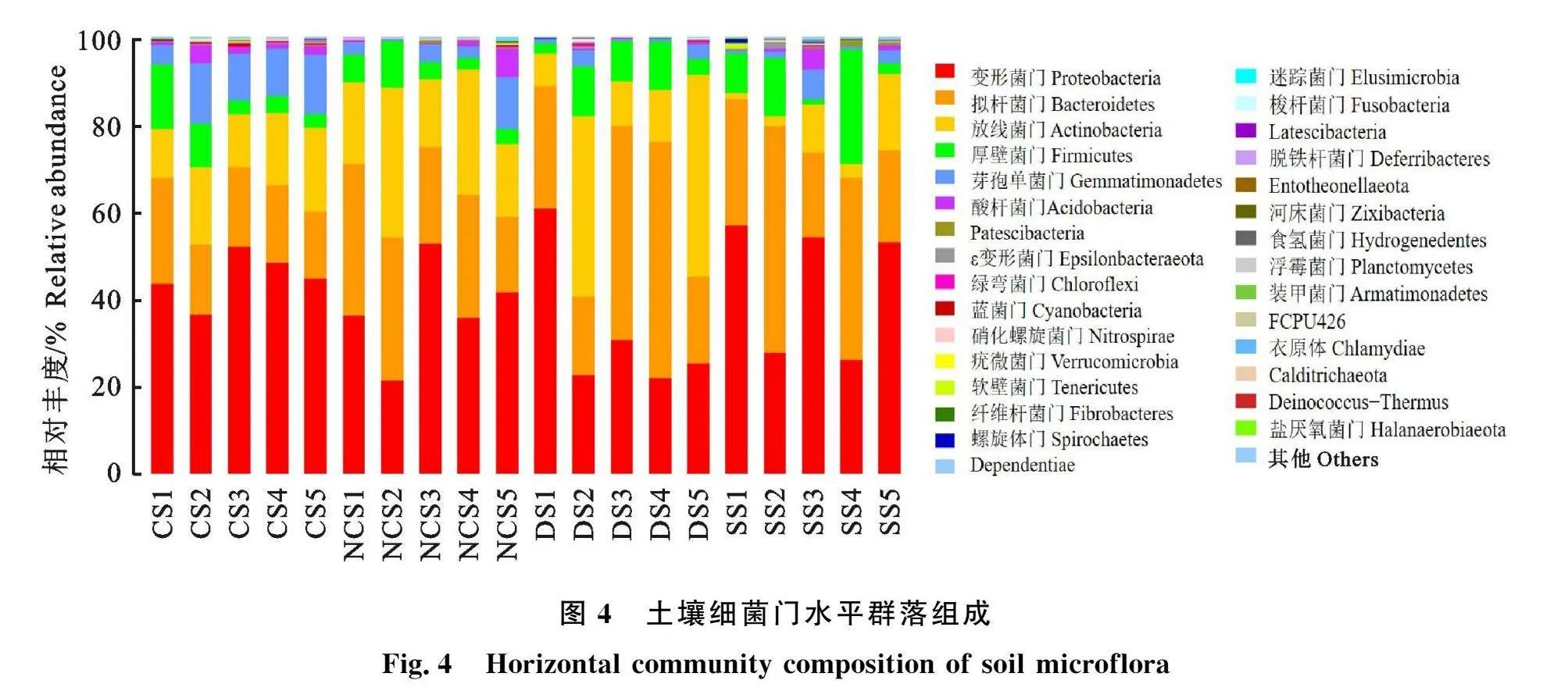
2.2.3 土壤微生物群落组成 由图4可知,CS组细菌群落中,优势类群依次是变形菌门(Pro-teobacteria)(36.55% ~ 52.10%)、拟杆菌门(Bacteroidetes)(15.25% ~24.39%)、放线菌门(Actinobacteria)(11.07%~19.37%)、芽孢单菌门(Gemmatimonadetes)(4.28% ~13.62%);NCS组细菌群落中,优势类群分别是变形菌门(36.31% ~52.73%)、拟杆菌门(17.26% ~34.74%)、放线菌门(18.66%~34.35%);DS组细菌群落中优势类群分别为变形菌门(21.80%~30.65%)、拟杆菌门(18.11%~60.84%)、放线菌门(7.39% ~46.37%)、厚壁菌门(Firmicutes)(2.42%~11.31%);SS组细菌群落中,优势类群分别为变形菌门(25.90%~56.95%)、拟杆菌门(19.61% ~52.00%)、厚壁菌门(13.31% ~ 26.33%)、放线菌门(2.36%~ 17.63%)。
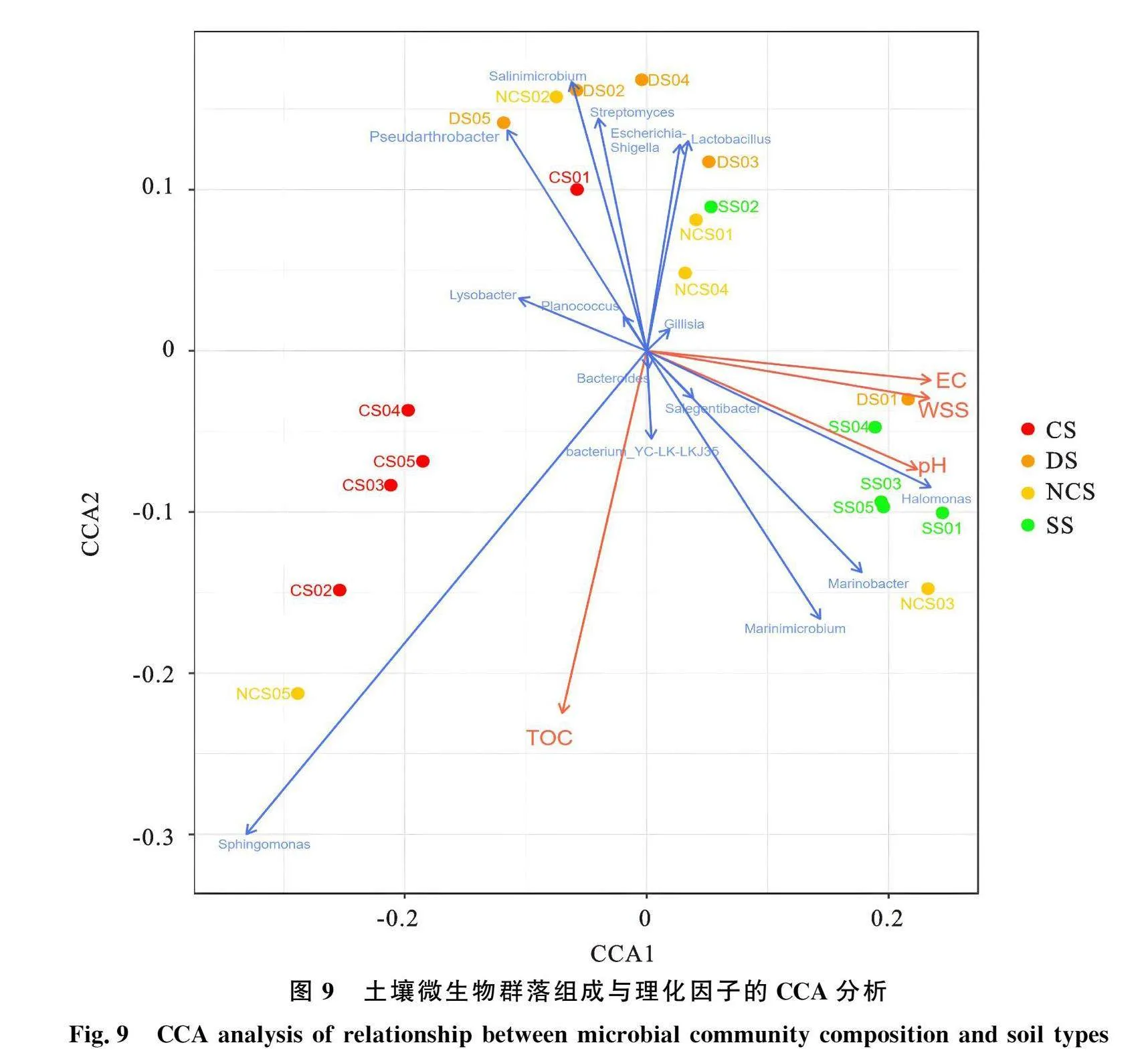
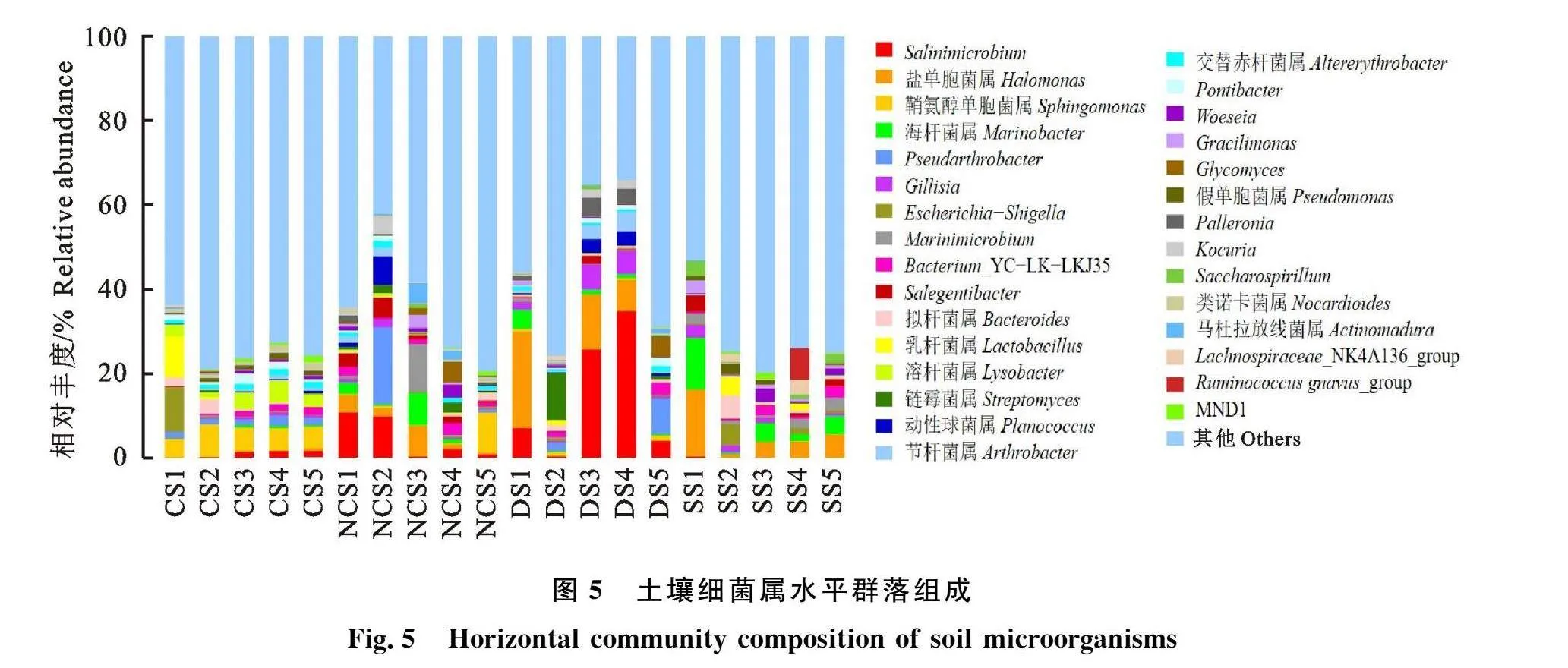
从属水平上看,鞘脂单胞菌属(Sphin-gomonas)、Pseudarthrobacter、Salinimicrobium是CS组土壤细菌群落的优势属(丰度gt;1%),其中鞘脂单胞菌属的丰度最高(4.55%~7.81%);盐单胞菌属(Halomonas)、海杆菌属(Mari-nobacter)、bacterium_YC_LK_LKJ35是NCS组土壤细菌群落的优势属(丰度gt;1%);盐单胞菌属、Salinimicrobium、Gillisia为DS组土壤细菌群落的优势属(丰度gt;1%)(图5),盐单胞菌属、海杆菌属、Gillisia以及Marinimicrobium为SS组土壤细菌群落的优势属(丰度gt;1%)(图6)。
2.3 土壤细菌群落多样性分析
土壤理化性质与土壤微生物群落多样性密切相关。相似性分析(ANOSIM)结果表明,CS组、NCS组、DS组以及SS组间微生物群落多样性具有极显著差异(Plt;0.01)。NCS组的拟杆菌门和放线菌门的丰度明显高于CS组,但两者变形菌门的丰度无显著差异,相比较而言,NCS组芽孢单菌门的丰度降低。在属水平上,CS组的优势属种类和NCS组优势属具有较大差异。NCS组的优势属,例如盐单胞菌属、海杆菌属、bactrium-YC-LK-LKJ35等在CS组中丰度较低,差异显著(Plt;0.05),而NCS组非优势属的鞘脂单胞菌属、Pseudarthrobacter、Salinimicrobium在CS组中富集成为优势属。盐单胞菌属在DS组和SS组中丰度均较高。SS组的优势属,例如Marin-imicrobium、海杆菌属、拟杆菌属等在DS组中丰度显著较低,而SS组中的非优势菌属Salinimi-crobium、Gillisia、Paiieronia等在DS组中富集成为优势属(图6)。
2.4 Alpha多样性分析
利用Chao 1指数、Shannon指数及Simpson指数对4种土样进行Alpha多样性分析。结果表明,CS组的Chao 1指数均值为3983.6 NCS组的Chao 1指数均值为2 966.7;DS组的Chao1指数最小,其均值为2 133.0;SS组的Chao1指数均值为3393.9。通过Wilcox检验计算发现,CS组与NCS组和DS组存在极显著差异 (P lt;0.01),但与SS组差异不显著(Pgt;0.05);DS组与NCS组和SS组存在显著差异(Plt;0.05);NCS组与SS组差异不显著(Pgt;0.05)。CS组、NCS组、DS组、SS组的Shannon指数依次为 9.82、8.477、7.34、8.51,Simpson指数依次为1.00、0.98、0.98、0.99,表明CS组土样中的微生物丰富度较高,且均匀性较好。对Shannon指数的Wilcox检验计算发现,CS组与其余三组存在显著性差异(Plt;0.05),其余组间差异不显著;对Simpson指数的Wilcox检验计算发现,仅CS组与DS组、SS组与DS组存在显著差异(Plt;0.05),其余组间无显著差异(图7)。
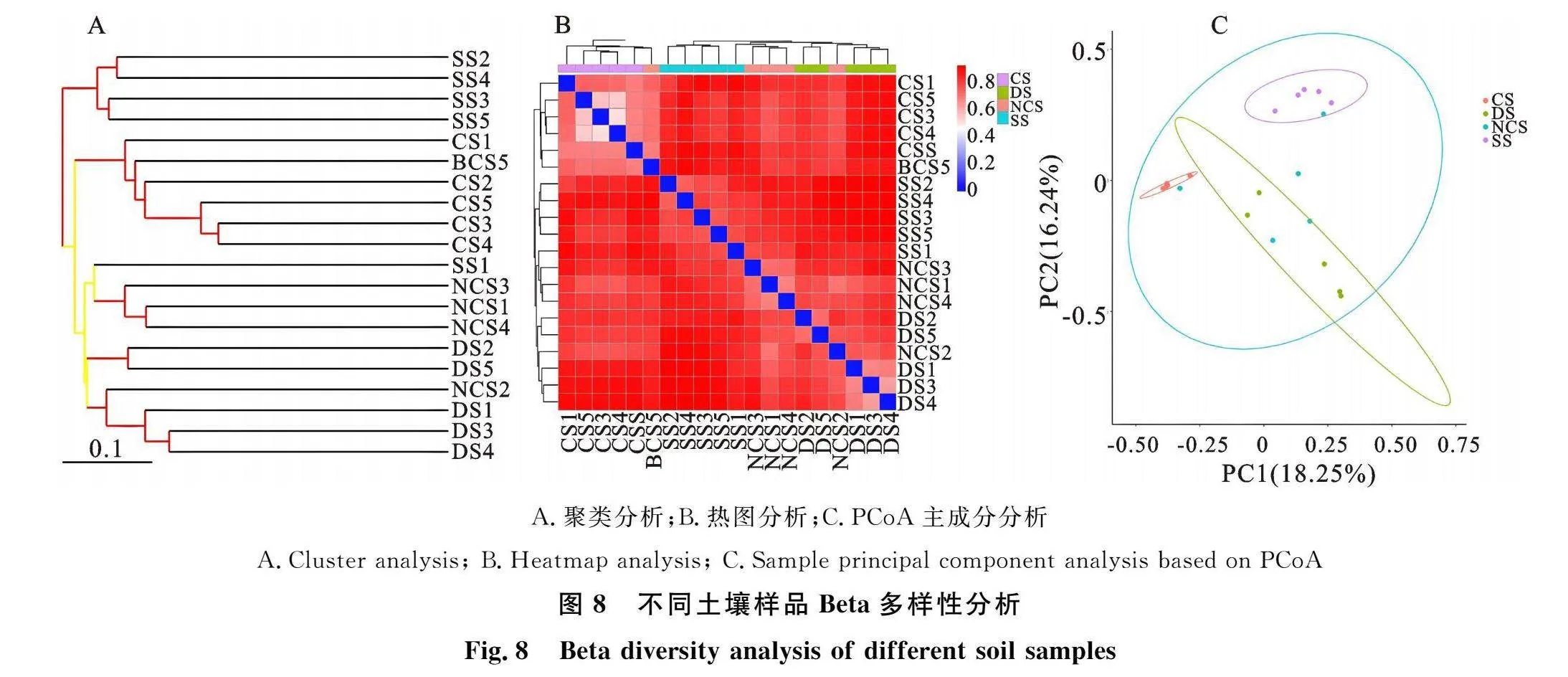
2.5 Beta多样性分析
对土壤样品组间细菌群落微生物Beta多样性进行聚类分析发现,4种土壤样品间,同组样本细菌群落聚类;利用4种土壤样本的OTU丰度信息计算BrayCruits,对不同土壤样本的细菌群落结构差异进行主坐标分析(PCoA),发现不同土壤样本间细菌群落结构具较好的分离特征(图8)。
2.6 微生物群落多样性与土壤理化性质的关系
相关性分析结果表明,TOC含量与EC和pH均呈现极显著负相关(Plt;0.01),TOC含量与细菌群落多样性呈负相关,但并不显著。EC与pH呈显著正相关(Plt;0.05),EC与细菌群落多样性极显著正相关()。土壤与土Plt;0.01pH壤内细菌群落多样性呈正相关,但不显著。水溶盐含量与土壤细菌群落多样性呈极显著正相关(Plt; 0.01)。多样性分析结果表明,EC和水溶性盐是影响土壤内微生物群落多样性的主要因子。
CCA分析结果表明,TOC与EC、pH和WWS含量呈负相关,后三者呈正相关。与土壤微生物相关性分析表明,有机碳含量与Sphin-gomonas菌属呈正相关,电导率、pH和水溶性盐含量与盐单胞菌属(Halomonas)、海杆菌属(Marinobacter)、Marinimicrobium菌属呈正相关。4种土壤与Lactobacillus菌属、Escherichia-Shigella菌属、Streptomyces菌属、Salinimi-crobium菌属、Pseudarthrobacter菌属呈负相关(图9)。
3 讨 论
近年来,随着土壤微生物研究的增多,土壤微生物功能被不断发掘。李艳玲[22]研究了根际微生物对作物生长及其调节因子的影响,发现有多种化合物与植物及微生物的生长代谢密切关联;卢玉秋[23]分析了根际微生物对作物生长及其调节因子的影响,发现微生物群落结构的改变会影响玉米和水稻的生长,还会调节玉米和水稻根内生长调节因子的种类和浓度。还有研究发现,土壤微生物能够促进植物激素合成,增强植物对矿质元素的吸收[18,24],提高植物抵抗生物和非生物胁迫的能力[19,25]。
土壤微生物总量、活性及有益微生物数量是判断土壤活跃度的重要数据。本研究发现,柴达木盆地干旱盐碱环境下,不同类型土壤中细菌丰富度均高于其他微生物,与崔纪超等[26]、陈菲菲等[27]的研究结果相一致,与土壤中微生物以细菌为主的结论一致[28]。本研究结果表明,耕作土壤内的微生物群落组成与包括荒漠土及盐碱土在内的非耕作土壤微生物群落之间存在明显差异。耕作土壤的优势属,如鞘脂单胞菌属和Escherichia_Shigella、溶杆菌属(Lysobacter)、乳酸菌属(Lactobacilus)、MND1等菌属在非耕作土壤、荒漠土及盐碱土中丰度较低;非耕作土壤内含量较高的菌属如bacterium_ YC _ LK _ LKJ35、Pseudarthrobacter、需盐杆菌属(Salegentibact-er)、马杜拉放线菌属(Actinomadura)、Prevotel-la-9等在耕作土壤、荒漠土壤以及盐碱土内较低;荒漠土的优势属如Salinimicrobium在耕作土壤内丰度较高,在非耕作土壤以及盐碱土壤内丰度则较低;盐碱土壤中的优势属,例如Marinimi-crobium、海杆菌属、拟杆菌属等在荒漠土壤中的丰度较低,而盐碱土壤非优势菌属的Salinimi-crobium、Gillisia、Paiieronia等在荒漠土壤中却富集成为优势属。对4种土壤内物种属水平比较发现,荒漠土和盐碱土内的嗜盐碱微生物如盐单胞菌属、Salinimicrobium、Thiohalobacter等含量远高于耕作土和非耕作土,非耕作土中的盐单胞菌属、Salinimicrobium等菌属含量也略高于耕作土,表明非耕作土、荒漠土、盐碱土的微生物群落结构与含盐量较低的耕作土有明显差异,可能会影响植物生长。4种不同类型土壤中的最优菌门均为变形菌门,这与李丹等[29]和刘欣等[30]的研究结果一致。变形菌门是目前已知的在自然界中最为普遍的菌门,在干旱盐碱生境土壤中丰度最高,可能是因为变形菌门适应能力更强[31]。土壤中的变形菌门和放线菌门主要与碳利用有关,参与土壤内有机质的转化以及土壤结构形成,本研究结果能进一步佐证该结果[32]。另外,耕作土壤中含有具生物固氮作用的芽孢杆菌属(Bacil-lus)、红杆菌属(Rhodospirillum)等,这些微生物与植物释放植物激素相关[33],在土壤源植物病原菌的生物防治方面发挥重要作用[34]。
微生物群落多样性在不同土壤类型间的差异与土壤理化性质密切相关[35]。本研究结果表明,4种不同类型土壤中,变形菌门和拟杆菌门是丰度最高的两个门,而厚壁菌门在荒漠土壤及盐碱土壤中丰度较高,在耕作土壤和非耕作土壤内丰度较低。造成不同类型土壤内细菌群落差异的主要原因有两点:一方面是因为4种类型土壤TOC含量、和电导率具有显著差异,利于某些特定pH的细菌类群增殖从而导致丰度的提高;另一方面,从土壤类型来看,由于枸杞生长代谢会改变耕作土的理化性质,使其微生物群落组成发生改变[36]。而盐碱土由于盐碱程度高,富集了较多的嗜盐微生物,导致土壤微生物多样性与其他类型土壤差异较大[37]。本试验结果也表明,含盐量较低的耕作土壤和非耕作土壤内微生物群落的相似度高于荒漠土和盐碱土;相关性分析结果表明水溶盐含量对土壤内细菌群落多样性具有重要的影响。有研究表明盐胁迫能够显著降低土壤内细菌群落的多样性[38],说明在柴达木盆地,土壤盐碱程度是影响微生物群落多样性的主要因子,但其作用效果还会受到植物根际效应的影响[39]。也有研究认为土壤内氮含量(TON)是影响土壤微生物群落结构的重要因素之一[40],但本研究并未对TON含量进行测量,其对于土壤微生物群落的影响有待进一步研究。
4 结 论
柴达木盆地干旱盐碱地区土壤中微生物群落多样性最高的是耕作土。耕作土壤的优势微生物为变形菌门、拟杆菌门、放线菌门、芽孢单菌门;非耕作土壤中拟杆菌门和放线菌门的丰度相对较高;荒漠土壤和盐碱土壤中优势微生物为变形菌门、拟杆菌门、放线菌门和厚壁菌门,厚壁菌门在盐碱土壤中的丰度相对高于荒漠土壤,而放线菌门则相反。鞘脂单胞菌属是耕作土壤的最优势属,盐单胞菌属为非耕作土壤、荒漠土壤和盐碱土壤的最优势属。理化因子与土壤微生物群落相关性分析显示,电导率、水溶性盐含量与土壤微生物群落呈显著正相关(Plt;0.01),有机碳含量和微生物群落多样性呈负相关,但不显著。表明电导率和水溶性盐含量是影响土壤内微生物群落多样性的主要因子。
参考文献 Reference:
[1] RAMIREZ K S,CRAINEJ M,FIERER N.Consistenteffects of nitrogen amendments on soil microbialcommu-nities and processes across biomes[J].Global ChangeBi-ology,2012,18:1918-1927.
[2]陈 源.中国小陇山国家级自然保护区珍稀濒危植物红豆杉(Taxus.chinesis(Pilg.)Rehd)林土壤微生物生态学研究[D].兰州:西北师范大学,2008.
[3] BROCKETTB FT,PRESCOTTC E,GRAYSTONS J.Soil moisture is the major factor influencing microbialcommunity structure and enzyme activities acrossseven-biogeoclimatic zones in western Canada[J].Soil BiologyandBiochemistry,2012,44:9-20.
[4]刘方春,邢尚军,马海林,等.持续干旱对樱桃根际土壤细菌数量及结构多样性影响[J].生态学报,2014,34(3):642-649.
[5] MATEOS-RIVERA A,YDEJ C,WILSON B,et al.Theeffect of temperature change on the microbial diversity andcommunity structure along the chronosequence of thesub-arctic glacier forefield of Styggedalsbreen (Norway)[J].FEMS Microbiolo Ecolo,2016,92:fnw038.gygy
[6]张燕燕,曲来叶,陈利顶,等.黄土丘陵沟壑区不同植被类型土壤微生物特性[J].应用生态学报,2010,21(1):165-173.
[7]邵 微,于会丽,张培基,等.不同落叶果树根际微生物群落代谢与组成的差异性研究[J].果树学报,2020,37(9):1371-1383.
[8] VENTOSA A,MELLADO E,SANCHEZ-PORRO C ,et al.Halophilic and halotolerant micro-organisms fro msoils microbiology of extreme soils[J].Springer Berli nHeidelberg,2008:87-115.
[9] HOLLISTERE B,ENGLEDOW A S,HAMMETT A J,et al.Shifts in microbial community structure along anec-ological gradient of hypersaline soils and sediments[J].The International Society for Microbial EcologyJournal,2010,4:829-838.
[10] ZHANG H,TANG M,CHEN H,et al.Effects ofa dark-septate endophytic isolate LBF-2 on the medicinal plan tLycium barbarum L[J].The Journal of Microbiology,2012,50:91-96.
[11] ASCHEHOUGE T,METLEN K L,CALLAWAYR M,et al.Fungal endophytes directly increase the competitiveeffects of an invasiveforb[J].Ecology,2012,93(1):3-8.
[12] BHATTACHARYYAP N,JHA D K.Plantgrowth-pro-motingrhizobacteria (PGPR):emergence in agriculture[J].World Journal of Microbiology and Biotechnology,2012,28(4):1327-1350.
[13] BHARTI N,BARNAWAL D,MAJI D,et al.Halotoleran tPGPRs prevent major shifts in indigenous microbial com-munity structure under salinity stress[J].Microbialog yEcology,2015,70(1):196-208.
[14] SHI SH J,NUCCIO E,HERMAND J,et al.Successionaltrajectories of rhizosphere bacterial communities over con-secutive seasons[J].mBio,2015,6(4):e00746-15.
[15]王 雪,接伟光,蔡柏岩.不同生境黄檗AM真菌菌群结构分析[J].林业科学,2012,48(9):99-107.
[16] BOLGER A M,LOHSE M,USADEL B.Trimmomatic:aflexible trimmer for Illumina sequence data[J].Bioinfor-matics, 2014,30(15):2114-2120.
[17] MAGOC T,SALZBERGS L.FLASH:fast length adjust-ment of short reads to improve genomeassemblies[J].Bioinformatics,2011,27(21):2957-2963.
[18] CAPORASO J G,KUCZYNSKI J,STOMBAUTH J,et al.QIIME allows analysis of high-throughputcommu-nity sequencing data[J].Nature Methods,2010,7(5):335-336.
[19] EDGARR C,HAASB J,CLEMENTEJ C,et al.UCH-IME improves sensitivity and speed of chimera detection[J].Bioinformatics,2011,27(16):2194-2200.
[20] ROGNES T,FLOURI T,NICHOLS B,etal.VSEARCH:a versatile open source tool formetagenomics[J].PeerJ,2016,4:e2584.
[21] WANG Q,GARRITY G M,TIEDJE J M,et al.Naiv eBayesian classifier for rapid assignment of rRNA se-quences into the new bacterial taxonomy[J].Applied andEnvironmental Microbiology, 2007,73(16):5261-5267.
[22]李艳玲.根际微生物群落对挥发性有机物和作物生长的影响[D].北京:中国农业科学院,2019.
[23]卢玉秋.微生物群落对作物生长及植物激素的影响[D].北京:中国农业科学院,2019.
[24] KUCZYNSKI J,LAUBERC L,WALTERS W A,etal.Experimental and analytical tools for studying the humanmicrobiome[J].Nature Reviews Genetics,2012,13(1):47-58.
[25] MCDONALD D,PRICE M N,GOODRICH J,et al.Animproved greengenes taxonomy with explicit ranks forec-ological and evolutionary analyses of bacteria andarchaea[J].The International Society for Microbial EcologyJournalJournal,2012,6(3):610-618.
[26]崔纪超,武小霞,林 怡,等.甘薯根际土壤微生物群落结构及多样性分析[J].西南农业学报,2022,35(9):2086-2095.
[27]陈菲菲,丛 欣,向极钎,等.应用IlluminaMiSeq高通量测序技术分析堇叶碎米荠根际土壤微生物多样性[J].湖北农业科学,2020,59(17):58-62,96.
[28] YANG S,XING S,LIU C,et al.Effects of root pruning onthe vegetative growth and fruit quality of Zhanhuadongzaotrees[J].HorticulturalScience,2010,37(1):14-21.
[29]李 丹,靳鲲鹏,李小霞,等.基于高通量测序技术的玉米不同生育时期土壤细菌多样性变化[J].山西农业科学,2019,47(9):1569-1572.
[30]刘 欣,李志英,刘瑞瑞,等.大豆不同生育期根际土壤细菌群落结构的变化[J].广西植物,2018,38(10):1363-1370.
[31] SPAIN A M,KRUMHOLZ L R,ELSHAHED M S.Abundance,composition,diversity and novelty ofsoilPro-teobacteria[J].The International Society for MicrobialE-cology JournalJournal,2009,3(8):992-1000.
[32] FIERER N,BRADFORD M A,JACKSON R B.Towardan ecological classification of soilbacteria[J].Ecology,2007,88(6):1354-1364.
[33] PALANIYANDIS A,DAMODHARAN K,YANGS H,et al.Streptomyces sp.strain PGPA39 alleviates saltstress and promotes growth of ‘Micro Tom’tomatoplants[J].Journal of Applied Microbiology,2014,117(3):766-773.
[34] BJELIC D,IGNJATOV M,MARINKOVIC J,etal.Anti-fungal activity of indigenous Bacillus spp.isolated fromsoil[J].Zbornik Matice Srpske Za PrirodneNauke,2017(133):261-269.
[35]张玉芳,李希来,高志香,等.不同施肥组合对木里矿区人工植被和土壤微生物特征的影响[J].西北农业学报,2022,31(6):741-754.
[36] KOWALCHUK G A,BUMA D S,BOER W D,etal.Effects of above-ground plant species composition anddi-versity on the diversity of soil-bornemicroorganisms[J].Antonie VanLeeuwenhoek,2002,81(1/4):509.
[37]李 岩,杨晓东,秦 璐,等.两种盐生植物根际土壤细菌多样性和群落结构[J].生态学报,2018,38(9):3118-3131.
[38] DEEPIKA S,KOTHAMASI D.Soil moisture—a regulatorofarbuscular mycorrhizal fungal community assembly andsymbiotic phosphorusuptake[J].Mycorrhiza,2015,25:67-75.
[39] BORRUSO L,BACCI G,MENGONI A,etal.Rhizo-sphere effect and salinity competing to shape microbialcommunities in Phragmites australis (Cav.)Trin.ex-Steud[J].FEMS MicrobiologyLetters,2014,359(2):193-200.
[40]李 岩,何学敏,杨晓东,等.不同生境黑果枸杞根际与非根际土壤微生物群落多样性[J].生态学报,2018,38(17):5983-5995.
Analysis of Soil Microbial Diversity in Different Habitatsina nArid,Saline-alkali Areaof QinghaiProvince
ZHANG Xuze1,2,3WANG Xueren1,4DU Meng1HAJinqiang1WANG Qi1,SHEN Yingfang1,2, 3and YANGXinguang1,2,3
(1.College of Eco-Environment and Resources,Qinghai Minzu University,Xining 810007,China; 2.KeyLaboratoryof Resource Chemistry and Eco-environmental Protection in Tibetan Plateau of State Ethnic Affairs Commission,QinghaiNationalities University,Xining 810007,China; 3.Qinghai Provincial Key Laboratory of High-value Utilization of
Characteristic Econom ic Plants,Xining 810007,China ;4.Inner Mongola Agricultural University Collegeof
Resources and Environmental Sciences, Hohhot 010018,China)
Abstract T oexplore the relationship between soil microbial community diversity and soil physicaland chemical properties across different habitats,samples were collected from cultivatedsoil,non-cul -tivated soil,desertsoil,and saline-alkali soil.The physical and chemical properties of these soilsam -ples weremeasured.For the analysis of microbial community composition and diversity,the Illumin aMisEQ platform was used for 16S amplicon sequencing.The relationship between soil flora and soilphysical and chemical properties was also analyzed.The results showed that the order of microbialcommunity diversity was cultivated soil gt; saline-alkali soil gt; non-cultivated soil gt; desert soil .Thecomposition and abundance of the dominant bacterial groups phyla and genera varied among thedif-ferent regions.The dominant bacteria in the cultivated soils wereProteobacteria,Bacteroidetes,Acti -nobacteria,and Blastomonas.Theabundance of Bacillus monophyta in non-cultivated soil was lowe rthan thatin cultivated soil,whereas the abundances of Bacteroidetes and Actinobacteria werehigher .The dominantbacterial phyla in desert and saline soils wereProteobacteria,Bacteroidetes,Actinobac -teria,and Firmicutes.Theabundance of Firmicutes was higher in saline soil than in desertsoil,where -as theopposite was true for the abundance of Actinobacteria.Sphingomonas was the most dominan tgenus incultivated soils,whereas Halomonas was the most dominant genus innon-cultivated,desert ,and salinesoils.The correlation analysis between physical and chemical factors and soil microbialcom-munity showed that electrical conductivity and water-soluble salt content were significantly positivelycorrelated with microbial community diversity (Plt;0.01),whereas organic carbon content was no tsignificantly correlatedwith bacterial community diversity.These results indicate that electricalconductivi-ty and water-soluble salt content are the main factors affecting soil microbial communitydiversity.
Key words Soil microbe;Community;Diversity;Physical and chemicalproperties
Received 2023-06-12 Returned 2023-09-27
Foundation item Scientific Research and Innovation Platform Construction Project,Central Funds in2023-Scientific Research and Innovation Teams of Forestry and Grassland Ecosystem FunctionMain-tenance and Sustainable Development and Utilization in Qinghai Province(No.30160101141);Innova-tion and Entrepreneurship Project for College Students of Qinghai Province (No.S202210748028); theProject of Qinghai MinzuUniversity(No.2021XJXS22).
First author ZHANG Xuze,male,master,associate professor.Research area:resourcemicroorgan-ism.E-mail:zxz1904@126.com
Corresponding author SHEN Yingfang,female,Ph.D,associate professor.Research area:plantos-motic stressbiology.E-mail:syfnc@126.com
YANG Xinguang,male,Ph.D,associate professor.Research area:ecological remediation. E-mail:yangxg618@163.com
(责任编辑:顾玉兰 Responsible editor:GUYulan)

