陆地棉种质资源抗黄萎病性状的SSR标记关联分析

摘 要 对棉花抗黄萎病性状进行全基因组关联分析(genome-wide association study,GWAS),发掘与其关联的标记位点、优异等位变异及典型材料,可为棉花抗黄萎病的分子育种提供理论依据。以403份陆地棉品种(系)资源为材料,利用覆盖全基因组的、有多态性的201对SSR标记,对3个环境的抗黄萎病性状进行基于混合线性模型(mixed linear model,MLM)的全基因组关联分析,检测与抗病性状显著关联的位点、优异等位变异及优异典型材料。结果表明,3个环境下各材料的相对病指平均值为53.45,平均变异系数为" 36.85%,平均偏度系数为-0.46,平均峰度系数为-0.31;201对引物共产生394个等位变异位点,GWAS结果表明有11个位点能同时在2个环境中检测到,其中有2个位点NAU2437b和NAU3493b能同时在3个环境中检测到;结合育种实际,发掘出含有优异等位变异的典型材料7份,其中鲁棉研28同时含有9个优异等位变异;从各材料不同生态区来源分析,来源于黄河流域棉区的材料具有较低的平均表型效应。
关键词 陆地棉;黄萎病;关联分析;优异等位基因
棉花黄萎病是棉花生产上主要的病害,被称为棉花的“癌症”[1]。目前,棉花黄萎病是中国棉花生产上危害最为严重的病害。尤其近年来在西北内陆棉区由于常年连作,导致棉花黄萎病害有逐渐加重的趋势,严重影响了棉花生产的发展[2]。防治黄萎病最经济有效、环保安全的方法就是培育具有抗黄萎病性的优良品种[3]。研究发现,黄萎病抗性属于数量性状遗传[4],其遗传特征还未明确,其中海岛棉对黄萎病的抗性受单基因或少数主效基因控制[5]。在棉花抗病育种中,开展抗黄萎病性状的QTL定位研究,挖掘出稳定表达的QTL和优异等位基因,对提高棉花黄萎病抗性具有重要的理论与应用价值。
国内外研究学者通过构建不同的连锁分离群体,应用各类型分子标记定位了多个与抗黄萎病性状相关的QTLs[6-17],为进一步挖掘抗病基因提供了很好的研究基础。随着分子生物学和生物统计学的进一步发展,基于连锁不平衡的关联分析方法在挖掘基因方面得到广泛应用,相对于传统的连锁分析更易找到微效基因,且群体样本收集相对容易,一定数量的自然群体或种质资源群体即可进行分析,不需要建立专门的作图群体,且定位精确度高于传统的连锁分析方法。在棉花抗病基因挖掘方面已有应用[18-21],为抗黄萎病性状的深入研究奠定了基础,但目前仍缺少主效QTL应用于棉花抗病育种中。本研究以中国不同棉区的403份陆地棉种质资源为研究材料,2016-2017年分别在新疆农垦科学院石河子黄萎病圃和博乐黄萎病圃两块发病均匀的自然黄萎病圃进行抗黄萎病性状鉴定,并结合均匀覆盖染色体的SSR标记进行全基因组关联分析研究。研究结果可为棉花抗黄萎病分子育种提供理论基础和标记来源。
1 材料与方法
1.1 试验材料
本研究选取403份陆地棉种质资源为试验材料,来自新疆农垦科学院棉花研究所多年来收集保存的国内外种质资源,其中199份来自于中国西北内陆棉区(NIR:the Northwestern Inland Region),87份来自于黄河流域棉区(YRR:the Yellow River Region),63份来自于长江流域棉区(YtRR:the Yangtze River Region),29份来自于北部特早熟棉区(NSEMR:the Northern Specific Ear ly Maturation Region),25份来自于国外(Abroad)。试验材料来源及分类与前期报道的一致[22]。
1.2 田间试验
为获取403份材料的抗黄萎病性状,试验于2016年、2017年连续两年在新疆农垦科学院棉花研究所博乐89团黄萎病圃,2017年在新疆农垦科学院棉花研究所石河子143团黄萎病圃种植各材料,两块病地均属自然黄萎病圃且发病均匀、致病性较强。各环境分别命名为:BL16、BL17和SHZ17。各试验点田间试验采用完全随机区组设计,2次重复,双行区,平均行距38 cm,平均株距9 cm,各材料人工点播种植,膜下滴灌栽培,以当地常规方法进行田间管理。
1.3 黄萎病性状调查
2016年-2017年在新疆博乐89团环境下(BL16、BL17),2017年在新疆石河子143团环境下(SHZ17),分别在黄萎病发病高峰期(博乐89团2016年-2017为每年的9月1日,石河子143团2017年为8月26日)调查对照材料‘新陆早9号’的病情指数,当对照病指达到或接近50时,开始调查全部材料的病指,每个材料调查20株,并依据国家标准(GBT 22101.5-2009)中所述的五级分类法判断发病级别,根据调查结果,参考朱荷琴等[23]的方法计算出病情指数和相对病情指数,其中:病情指数= [∑(各级病株数×相应病" 级) /调查总株数×最高病级 (4) ]×100;相对病情指数( IR) = K×鉴定材料的实际病指,其中" K=50÷对照实际病指(50为规定感病对照品种的标准病指)。最终以相对病情指数(IR)来进行后续的数据分析。
1.4 SSR标记及基因型鉴定
各试验材料的基因组DNA提取参照Paterson等[24]发表的CTAB方法进行,SSR引物选择主要结合前人已发表的棉花四倍体分子遗传图谱及在陆地棉中多态性较好的SSR标记结果,选取均匀分布在26条染色体的560对引物对群体进行PCR扩增分析。具体详见前期文献报道[22]。引物合成由生工生物工程(上海)股份有限公司完成。分子标记名称中的大写字母,如BNL、JESPR、HAU、NAU等表示引物的来源,引物名称后的小写字母“a”“b” “c”“d”等代表标记的多态性位点,且位点顺序依次按分子量由大到小排列。SSR分子标记实验操作规程及PCR扩增分析参照张军等[25]的研究,扩增后的PCR产物电泳分析采用毛细管电泳分析仪FragmentAnalyzer-XL960 SSR/Tilling系统进行,电泳结果使用系统自带软件ProSize软件查看,根据胶视图中DNA Ladder判断样品不同位点片段大小,相同位点有条带记为“1”,无条带记为“0”[26]。
1.5 数据分析
黄萎病表型性状一般性描述统计分析采用SPSS 17.0软件进行;多态性标记等位基因频率、基因多样性指数等采用PowerMarker V3.25软件进行;在群体结构分析、连锁不平衡分析的基础上,利用MLM (mixed linear model)进行性状和标记之间的关联分析,并计算标记位点在 Plt;" 0.05 和 Plt;0.01 时对表型变异的贡献率(R2)。在已获得关联位点的基础上,再进行优异等位变异的发掘,具体分析详见前期文献报道[26]。
2 结果与分析
2.1 表型性状分析
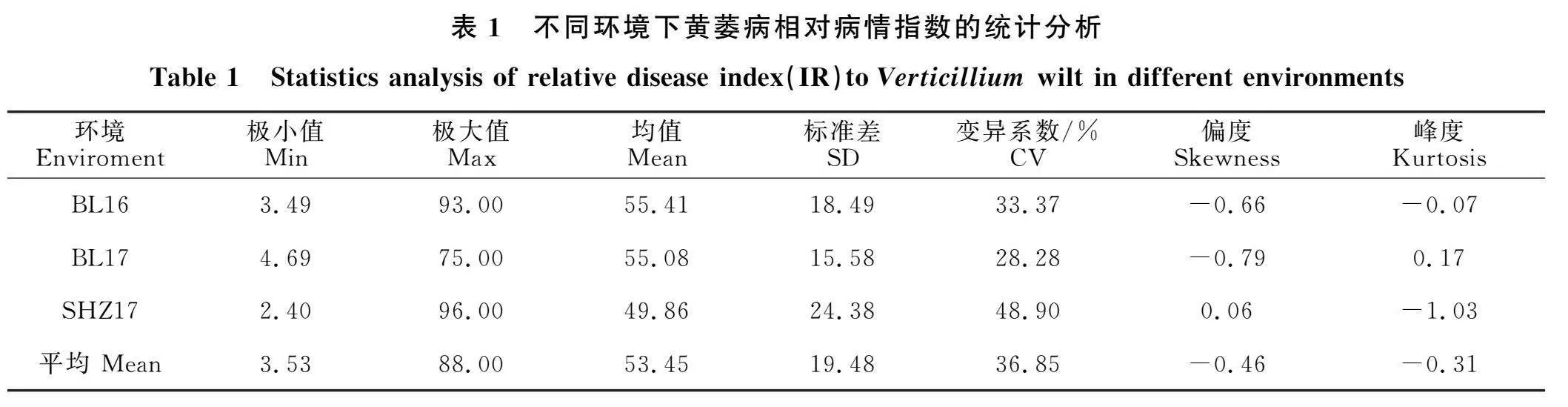
403份材料抗黄萎病性状的两年两点三环境的抗性结果见图1和表1。结果显示:相对病情指数在BL16、BL17及SHZ17环境下均值分别为55.41、55.08和49.86,3个环境下平均值为" 53.45;变异系数分别为33.37%、28.28%和" 48.90%,3个环境下平均变异系数为36.85%,表现出较高的变异系数;3个环境的相对病情指数的平均偏度系数为-0.46,平均峰度系数为" -0.3 说明不同病圃的相对病指结果均接近正态分布规律;从3个环境的相对病情指数和变异系数结果可以看出(表1),同一病圃的不同年份性状结果相对稳定,而同一年份的不同病圃性状结果表现一致性差,受环境影响较大。
2.2 SSR标记的遗传多样性
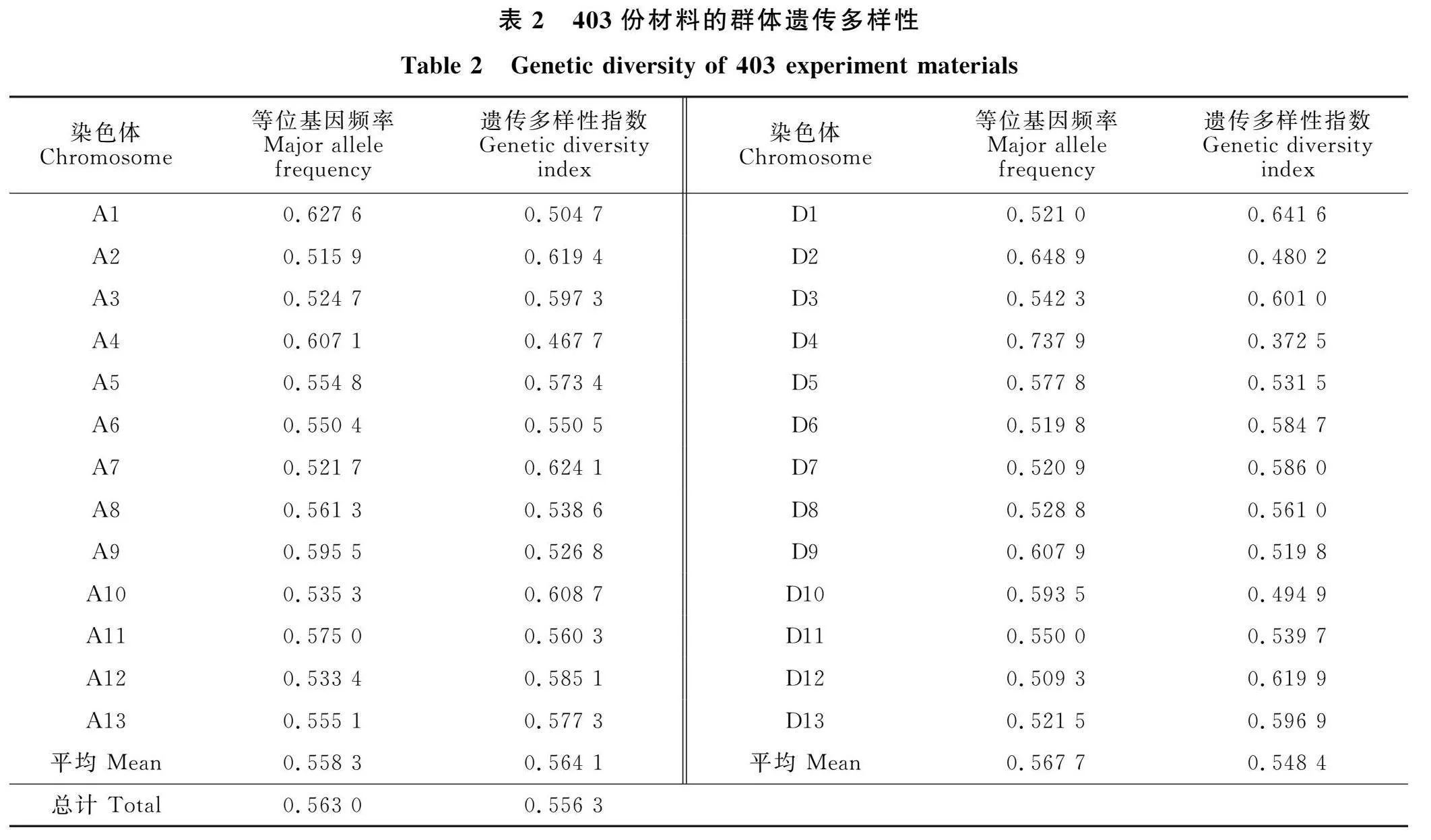
选取的560对SSR 标记中,最终获得201对标记在群体中产生多态性,基于201个SSR分子标记的遗传多样性指数结果,对403份材料的遗传多样性进行了评估。结果表明,A亚组平均等位基因频率为0.558 3,平均遗传多样性指数为0.564 "D亚组平均等位基因频率为" 0.567 7,平均遗传多样性指数为0.548 4,A亚组遗传多样性略高于D亚组,AD 全基因组的遗传多样性指数平均值为0.555 7(表2)。以上结果表明,本研究所选取的SSR引物在所选种质材料中检测到的等位基因频率和基因多样性指数的平均值较高,说明本研究所选取的陆地棉种质资源群体在基因组水平上变异比较丰富,表现出较高的遗传多" 样性。
2.3 关联分析及优异等位变异挖掘
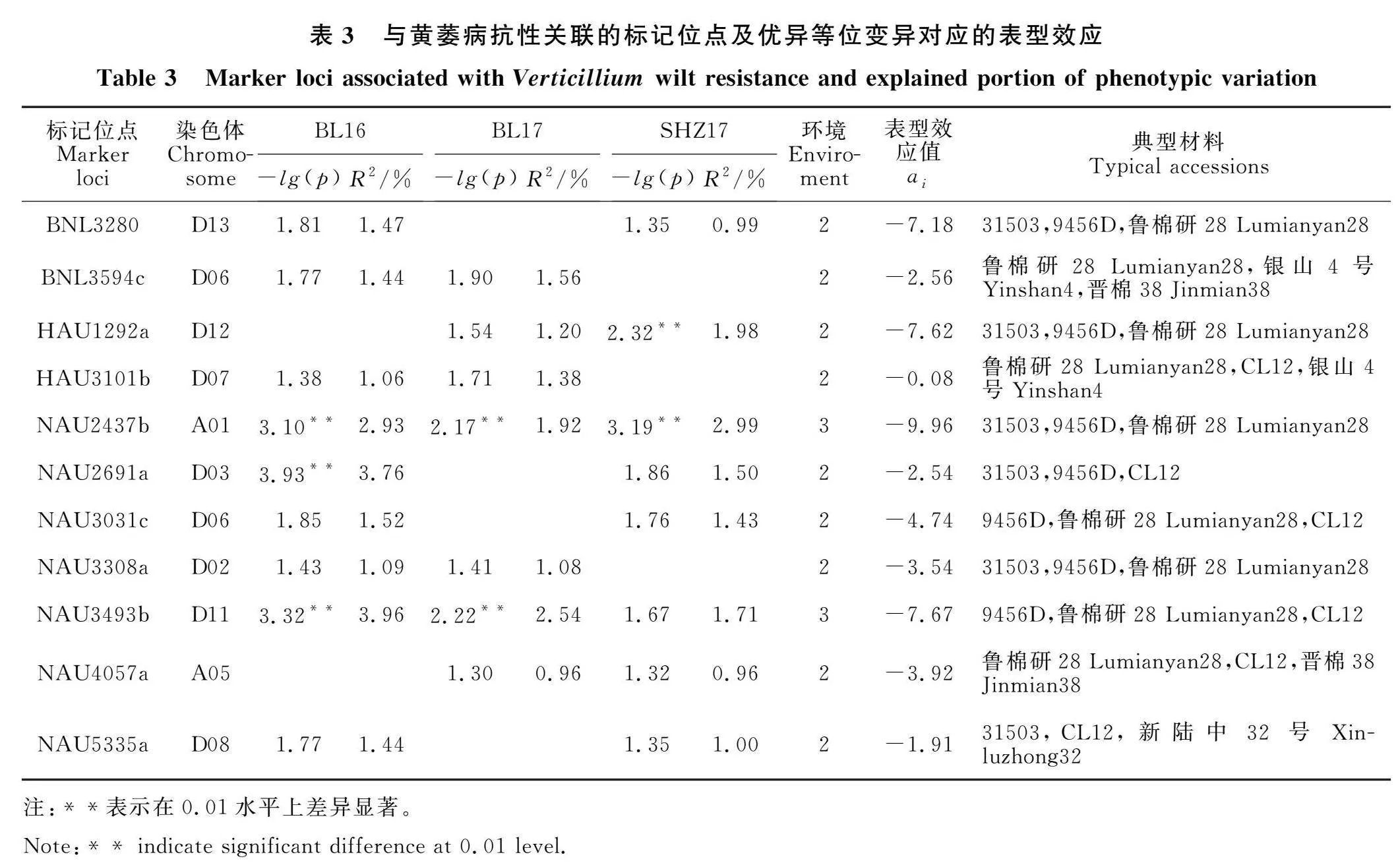
利用201对引物产生的394个等位变异位点,对403份材料进行群体结构分析、主成分分析及聚类分析的基础上[22],利用TASSEL 5.0软件的混合线性模型MLM(Q+K)程序,将上述394个位点的等位变异分别与不同环境下的抗黄萎病性状进行了关联分析。为最大程度地避免虚假关联的出现,研究结果只考虑能同时在两个以上环境中检测到的显著(Plt;0.05)关联的位点。结果表明,共11个位点能同时在两个环境中检测到,其中有2个位点NAU2437b和NAU3493b能同时在3个环境中检测到(表3);11个关联位点的等位变异表型效应值变化范围为-9.96~" -0.08;结合育种实际,以黄萎病病指最低为优异等位变异,发掘出含有优异等位变异的典型材料7份(表3),其中鲁棉研28同时含有9个优异等位变异,9456D同时含有7个优异等位变异,31503和CL12两份材料同时含有6个优异等位变异,银山4号和晋棉38两份材料同时含有2个优异等位变异,新陆中32号含有1个优异等位变异。
2.4 不同生态区种质材料抗病优异等位变异" 分析
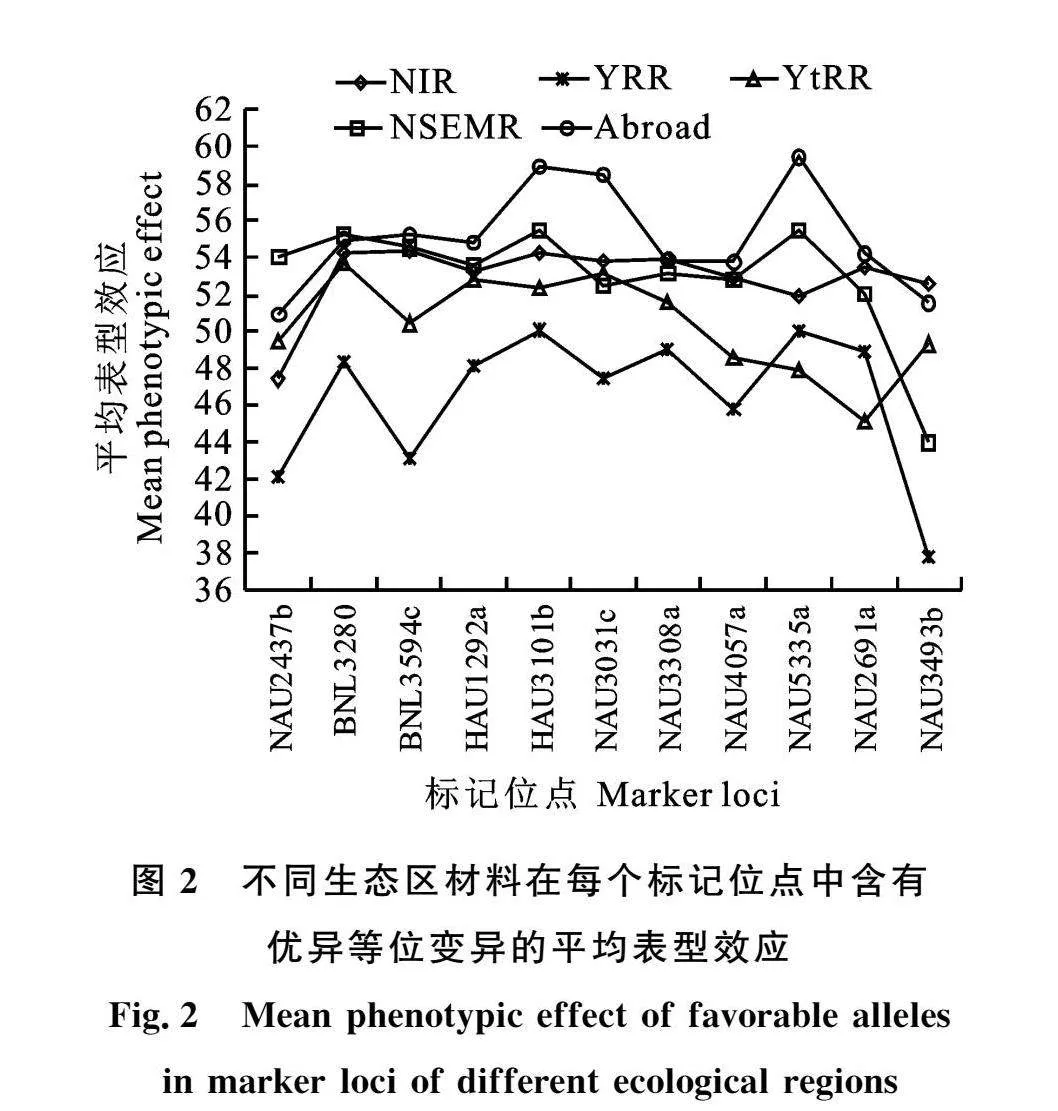
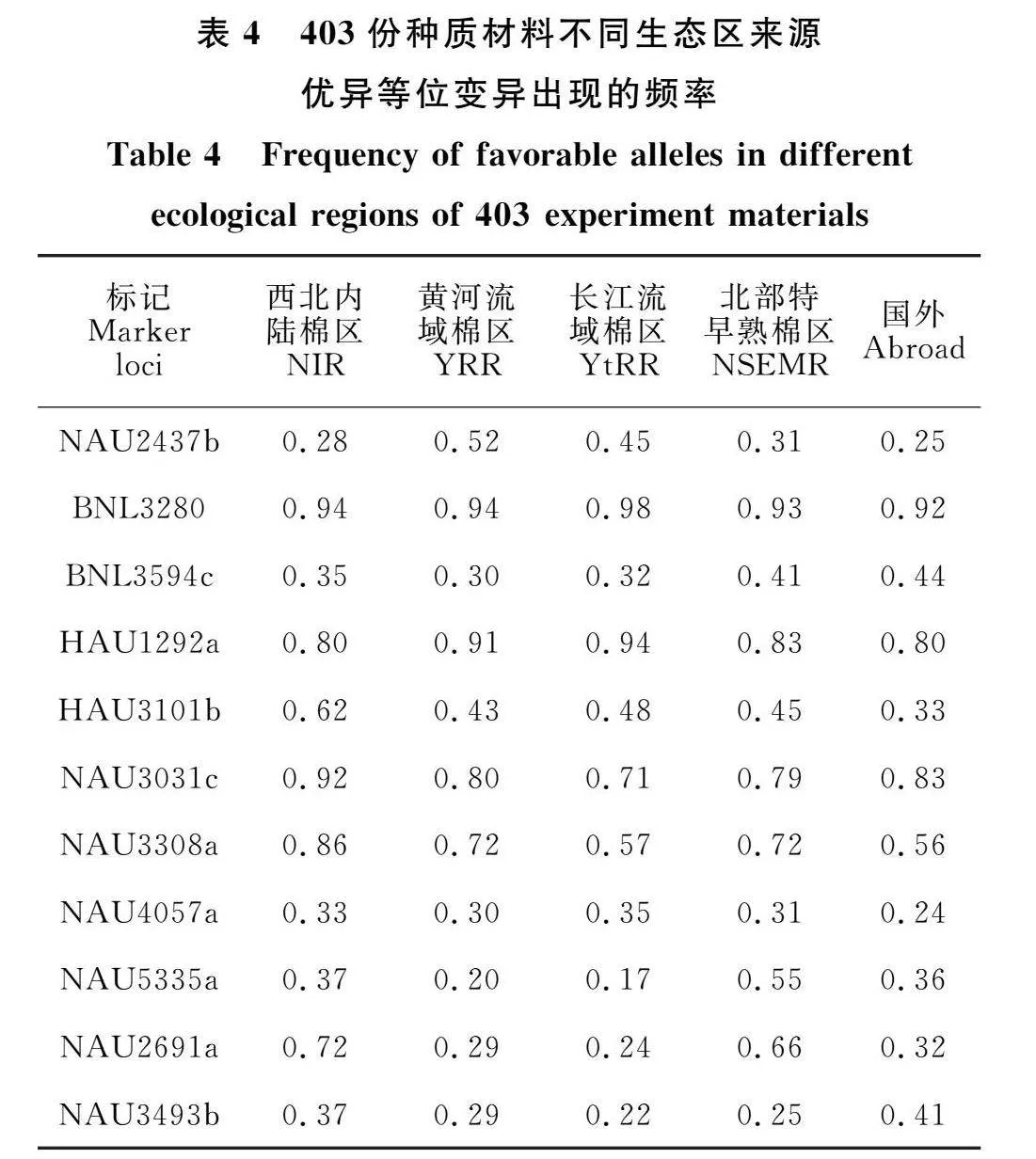
为明确优异等位变异的地域分布情况,对403份种质材料按照不同生态区来源比较优异等位变异出现的频率。结果表明(表4),在每个关联位点含有的优异等位变异材料个数中,其中有4个关联位点(HAU3101b、NAU3031c、NAU3308a和NAU2691a)来源于西北内陆棉区的材料出现频率最多,有3个关联位点(BNL3280、HAU1292a和NAU4057a)来源于长江流域棉区的材料出现频率最多,有两个关联位点(BNL3594c和NAU3493b)来源于国外棉区的材料出现频率最多,有一个关联位点(NAU2437b)来源于黄河流域棉区的材料出现频率最多,有一个关联位点(NAU5335a)来源于北部特早熟棉区的材料出现频率最多;进一步分析各生态区材料在每个关联位点中含有优异等位变异的抗黄萎病病指的平均表型效应发现(图2),来源于黄河流域棉区的材料具有较低的平均表型效应,其次为来源于长江流域棉区的材料,而来源于国外棉区的材料具有较高的平均表型效应。
3 讨" 论
棉花黄萎病属于土传维管束系统性病害,且抗病遗传机制较为复杂。因此,对鉴定材料的精准表型鉴定尤为重要。对于棉花抗黄萎病性的鉴定方法,当前主要有田间病圃鉴定、营养钵苗期鉴定和黄萎病菌毒素等鉴定方法[27]。从选育抗黄萎病品种角度而言,田间病圃鉴定法更能直接有效的鉴定出各材料的抗病性,是目前较为常用的方法。本研究利用本研究所不同环境的田间育种病圃对403份材料进行抗黄萎病性鉴定,并设置不同重复,且在鉴定过程中通过设置合适的感病对照品种,利用相对病情指数来划分棉花品种的抗病性,可进一步有效降低环境变异的影响[28],提高鉴定的精准性,为后续关联分析提供较为精准的表型数据。
国内外研究学者通过构建不同分离群体和利用自然群体,应用各类型分子标记发掘定位了多个与抗黄萎病性状相关的QTLs和关联位点。甄瑞等[10]以高抗黄萎病的海岛棉品种和高感黄萎病的陆地棉品种构建了一个海陆杂交F2群体,利用SSR分子标记,采用极端混合分析法发现一个与抗黄萎病性紧密连锁的标记;王省芬等[11]对甄瑞等定位到标记在不同群体中进行进一步验证,并进行测序发现该片段为含有TG重复且长211bp的片段,可用于抗黄萎病性状的分子辅助育种研究;Li等[20]对299个棉花材料进行简化基因组测序结合关联分析,鉴定出17个SNP与黄萎病抗性有关,其中A10上的1个SNP表现为多环境稳定,并鉴定到1个基因CG02可能是与抗病相关的候选基因。本研究以3个环境的黄萎病抗性相对病情指数为表型值进行关联分析,结果发现能够在两个以上环境中检测到的位点有11个(其中有2个位点能同时在3个环境中检测到),有1个位点BNL3280与前人通过连锁分析方法得到QTL具有相同的关联性状[9],其余10个标记位点可能为新开发的标记位点。这些标记可为进一步理解棉花抗黄萎病性状的遗传机理提供理论依据,并可为后期棉花抗黄萎病分子标记辅助选择育种提供标记来源;另外,本研究发掘出含有优异等位变异的典型材料7份,其中来源于黄河流域棉区的‘鲁棉研28’同时含有9个优异等位变异,进一步结合不同生态区来源种质资源优异等位变异出现的频率,发现来源于黄河流域棉区的材料与来源其他棉区的材料相比具有较低的平均表型效应,说明来源于黄河流域棉区的材料具有较强的抗黄萎病性,并可作为优良亲本资源或供体材料用于西北内陆棉区抗黄萎病棉花新品种选育研究。
参考文献 Reference:
[1] 许宗弘.棉花枯黄萎病研究现状及展望[J].知识经济,2010,16:113.
[2]李国英,杜树春,李 子,等.南疆棉区棉花黄萎病发生趋势和品种抗病研究[J].新疆农业科学,2015,52(9):1640-1647.
[3]马 存,简桂良,孙文姬.我国棉花抗黄萎病育种现状、问题及对策[J].中国农业科学,1997,30(2):58-64.
[4]王红梅,张献龙,李运海,等.陆地棉黄萎病抗性遗传分析.棉花学报,2004,16(2):84-88.
[5]齐俊生,马 存.海岛棉品种抗黄萎病遗传规律初步研究[J].棉花学报,2000,12(4):169-171.
[6]房卫平,许守明,孙玉堂,等.棉花黄萎病的RAPD标记[J].河南农业科学,200 30(9):11-13.
[7]高玉千,聂以春,张献龙,等.棉花抗黄萎病基因的QTL定位[J].棉花学报,2003,15(2):73-78.
[8]杜威世,杜雄明,马峙英.棉花黄萎病抗性基因SSR标记研究[J].西北农林科技大学学报(自然科学版),2004,32(3):20-24.
[9]王红梅,张献龙,贺道华,等.陆地棉对黄萎病抗性的分子标记研究[J].植物病理学报,2005,35(4):333-339.
[10] 甄 瑞,王省芬,马峙英,等.海岛棉抗黄萎病基因SSR标记研究[J].棉花学报,2006," 18(5):269-272.
[11]王省芬,甄 瑞,马峙英,等.海岛棉品种抗黄萎病基因SSR标记的验证及克隆[J].植物遗传资源学报,2007," 8(2):149-152.
[12]蒋 锋,赵 君,周 雷,等.陆地棉抗黄萎病基因的分子标记定位[J].中国科学C辑:生命科学,2009,39(9):849-861.
[13]NING Z Y,ZHAO R,CHEN H,et al.Molecular tagging of a major quantitative trait locus for broad-spectrum resistance to Verticillium wilt in upland cotton cultivar prema[J].Crop Science,2013,53:2304-2312.
[14]LI C Q,LIU G S,ZHAO H H,et al.Marker-assisted selection of verticillium wilt resistance in progeny populations of upland cottonderived from mass selection-mass crossing[J].Euphytica,2013,191:469-480.
[15]FANG H,ZHOU H P,SANOGO S,et al.Quantitative trait locus analysisof verticillium wilt resistance in an introgressed recombinant inbredpopulation of upland cotton[J].Molecular Breeding,2014,33:709-720.
[16]SHI Y Z,ZHANG B C,LIU A Y,et al.Quantitative trait loci analysis of Verticillium wilt resistance in interspecific backcross populations of Gossypium hirsutum × Gossypium barbadense[J].BMC Genomics,2016,17:877.
[17]ZHAO J,LIU J G,XU J W,et al.Quantitative trait locus mapping and candidate gene analysis for Verticillium wilt resistance using Gossypium barbadense chromosomal segment introgressed line[J].Frontiers in Plant Sciencene,2018,9:682.
[18]ZHAO Y L,WANG H M,CHEN W,et al.Genetic structure,linkage disequilibrium and association mapping of verticillium wilt resistance in elite cotton (Gossypium hirsutum L.) germplasm population[J].PLoS One,2014,9:e86308.
[19]FANG L,WANG Q,HU Y,et al.Genomic analyses in cotton identify signatures of selection and loci associated with fiber quality and yield traits[J].Nature Genetics,2017,49 (7):1089-1098.
[20]LI T G,MA X F,LI N Y,et al.Genome-wide association study discovered candidate genes of Verticillium wilt resistance in upland cotton (Gossypium hirsutum L.)[J].Plant Biotechnology Journal,2017,15:1520-1532.
[21]赵云雷,王红梅,陈 伟,等.基于优异等位基因的棉花抗黄萎病性状的分子鉴定[J].中国农业科学,2017,50(2):216-227.
[22]DONG C G,WANG J,YU Y,et al.Association mapping and favorable QTL alleles for fiber quality traitsin upland cotton (Gossypium hirsutum L.)[J].Journal of Genetics,2018,97:1-12.
[23]朱荷琴,吴征彬,邹 奎,等.国家棉花品种区域试验抗枯黄萎病鉴定方法[J].中国棉花,2007,34(11):9-24.
[24]PATERSON A H,BRUBAKER C L,WENDEL J F.A rapid method for extraction of cotton (Gossypium spp.) genomic DNA suitable for RFLP and PCR analysis[J].Plant Molecular Biology Reporter,1993,11:122-127.
[25]张 军,武耀廷,郭旺珍,等.棉花微卫星标记的PAGE/银染快速检测[J].棉花学报,2000,12:267-269.
[26]王 娟,董承光,刘 丽,等.棉花适宜机采相关性状的SSR标记关联分析及优异等位基因挖掘[J].作物学报,2017,43(7):954-966.
[27]房慧勇,马峙英.棉花抗黄萎病机制及抗病性鉴定研究进展[J].河北农业科学,2002,6(2):1-7.
[28]朱荷琴,冯自力,李志芳,等.蛭石沙土无底纸钵定量蘸菌液法鉴定棉花品种(系)的抗黄萎病性[J].中国棉花,2010,37(12):15-17.
Association Analysis of" SSR Markers with Verticillium
Wilt Resistance Traits in Upland Cotton
WANG Juan ZHEN Zhihong2,MA Xiaomei ZHOU Xiaofeng WANG Xin
TIAN Qin AI Nijiang2,CHEN Hong1 and DONG Chengguang1
(1.Cotton Research Institute,Xinjiang Academy of Agricultural and Reclamation Sciences,Shihezi" Xinjiang
832000,China;" 2.Shihezi Academy of Agricultural Sciences,Shihezi" Xinjiang 832000,China)
Abstract A genome-wide association study (GWAS) was conducted to identify marker loci,elite alleles and typical accessions associated with Verticillium wilt resistance in upland cotton.These findings provide a theoretical basis for molecular breeding to enhance disease resistance.In this study,403 upland cotton lines were analyzed using 201 pairs of polymorphic SSR markers covering the whole genome.A mixed linear model (MLM) was applied to three environmental datasets.The average Verticillium wilt disease index was 53.45,with an average coefficient of variation of 36.85%,skewness of -0.46,and kurtosis of -0.31.A total of 394 allelic variation loci were detected,among which 11 loci were consistently identified in two environments,and two loci (NAU2437b and NAU3493b) wereidentified in all three environments.Seven typical accessions containing elite alleles were discovered," among whichLuxianyan 28" contained nine elite alleles.Materials from the Yellow River Region exhibited lower average phenotypic effects,indicating potential for targeted breeding in this area.
Key words Upland cotton;Verticillium wilt; SSR association analysis; Elite allele
Received" 2023-08-02 Returned 2024-03-26
Foundation item The Science and Technology Innovation Talent Program of The Xinjiang Production and Construction Corps (No.2022CB003-04); The Science and Technology Project of Sixth Division of Xinjiang Production and Construction Corps (No.2205); Key Field Science and Technology Development Program of Eighth Division of Xinjiang Production and Construction Corps (No.2023NY10).
First author WANG Juan,female,associate research fellow.Research area:cotton genetics and breeding.E-mail:cottonwj@126.com
Correspondingauthor" DONG Chengguang,male,research fellow.Research area:cotton genetics and breeding.E-mail:dcg318@163.com
CHEN Hong,female,research fellow.Research area:cotton genetics and breeding.E-mail:xjchenh990122@163.com
(责任编辑:成 敏 Responsible editor:CHENG" Min)

