小型猪微卫星标记的遗传多样性




摘要:挖掘五指山猪、巴马猪、近交陆川猪和藏香猪中全基因组SSR,比较分析4个猪种中SSR分布特性并进行功能性SSR注释,为小型猪种质资源的保存及利用提供分子标记。选取4个品种猪各15头,分别构建全基因组SSR文库并进行测序,结合生物信息学分析方法,统计不同类型SSR(双、三、四、五、六核苷酸)的数量、SSR重复单元的分布模式、SSR的长度多态性并对功能性SSR进行注释,比较分析4个小型猪品种中全基因组的SSR特性。对猪参考基因组和4个猪种全基因组SSR统计发现,五指山猪中SSR的数量最多,巴马猪和近交陆川猪其次,藏香猪中最少,且每个品种中双、三、四核苷酸SSR的数量占比最多,与猪参考基因组中不同类型SSR的占比结果相似。SSR重复单元的分布模式显示,4个猪种中双、五核苷酸SSR中重复单元的分布模式相同,藏香猪中三、四核苷酸SSR中重复单元的分布模式与其他3个猪种不同,而六核苷酸SSR中重复单元的分布模式在4个猪种中完全不同。SSR的长度多态性分析表明,五指山猪、巴马猪、近交陆川猪和藏香猪中分别有19 957、14 099、20 671、14 120个长度多态性gt;1的SSR定位到猪参考基因组,其中,有2 518个SSR共同存在于4个品种中,而特异存在于4个猪种中的SSR分别有 5 173、2 802、5 969、4 463个。此外,对SSR进行注释发现,多态性SSR可能影响FGF23、MYF6、IGF1R和LEPROT等与猪的生长发育相关基因的编码区和调控区。本试验首次构建猪全基因组SSR文库并测序,在全基因组水平上比较分析4个小型猪品种的SSR,揭示了不同小型猪品种间SSR重复单元的分布模式存在差异,并对品种间共有及特异的SSR进行挖掘和注释,发现了可能与小型猪的生长发育相关具有长度多态性的SSR。本研究挖掘得到的SSR补充了猪基因组遗传变异信息和小型猪分子标记,建立的SSR分析方法可为不同物种、品种间SSR分析及鉴定工作提供参考。
关键词:微卫星;重复单元;长度多态性;小型猪;分子标记
中图分类号:S828.2" 文献标志码:A
文章编号:1002-1302(2024)23-0174-07
涂尾龙,张莺莺,黄" 济,等. 小型猪微卫星标记的遗传多样性[J]. 江苏农业科学,2024,52(23):174-181.
doi:10.15889/j.issn.1002-1302.2024.23.024
收稿日期:2023-09-19
基金项目:上海市科技兴农技术创新项目(编号:X2022-02-08-00-12-F01106)。
作者简介:涂尾龙(1983—),男,江西抚州人,博士研究生,高级畜牧师,主要从事动物遗传育种研究。E-mail:tuweilong@saas.sh.cn。
通信作者:王洪洋,博士,副研究员,主要从事动物遗传育种研究。E-mail:wanghongyang@saas.sh.cn。
简单序列重复(simple sequence repeat,SSR)是由2~6个碱基组成的重复单元以串联的方式形成,其具有广泛存在性和高度多态性,而位于基因上(编码区和非编码区)的多态性SSR具有很强的功能性[1]。此外,小型猪具有独特的生长发育规律,其体型小的特点可用于骨骼、肌肉发育等研究,而本研究中的巴马猪、藏香猪又分别具有肉质鲜美、抗逆性强等特点,因此对于小型猪基因组SSR的挖掘可为猪的经济性状研究提供条件。由此可见,对全基因组SSR的研究可为猪的遗传育种研究提供分子标记,进而应用于群体遗传多样性及进化等。此外,功能性SSR的分析对猪生长发育等性状的研究至关重要。目前,大部分关于SSR的研究仅限于应用单个或多个SSR进行群体遗传多样性及种间鉴定方面[2-5],而其在猪中的应用往往需要借鉴于其他物种中的SSR序列[6-7],由于不同物种间的SSR具有很大的多态性差异,常导致试验复杂性和结果的不准确性。随着组学技术的发展,使基于表达序列标签(EST)和高通量测序数据挖掘SSR成为可能,如石甜甜等对144个EST-SSR标记进行糜子遗传资源评估[8],魏丹丹等采用磁珠富集法建立SSR文库并挖掘序列,结合EST-SSR分子标记验证了可以稳定扩增的SSR引物[9]。Liu等首次分析了猪参考基因组(Sus scrofa 10.2)中SSR的特性,并结合公共数据库中的102个重测序个体(源于国内外17个猪种)数据进行SSR分型,发现了与疾病发生相关的功能性SSR位点[10],但该研究中的SSR源于对猪参考基因组的扫描,而多态性SSR来自于多个品种的个体进行分析,并不能全面反映猪基因组层面的SSR特性。基于SSR在分子标记方面的应用和功能性SSR的重要性,以及猪基因组SSR的不全面性,本研究拟从全基因组水平对4个小型猪的SSR进行全面挖掘和比较分析。利用选择杂交法构建4个猪种的SSR文库,结合生物信息学比较分析4个猪种中SSR的分布模式及差异,对SSR进行注释并分析功能性SSR。
1" 材料与方法
1.1" 试验材料
试验材料包括170头小型猪的耳组织样品,来源于上海市农业科学院封闭繁育20年的近交陆川猪(LC)53头、上海交通大学的巴马猪(BM)16头、南方医科大学动物科技学院的藏香猪(ZX)50头和海南省农业科学院畜牧兽医研究所的五指山猪(WZS)51头。耳组织采集后保存于装有75%乙醇的EP管中,带回实验室置于-80 ℃冰箱保存备用。试验时间为2019年6—12月及2020年6—12月。
1.2" 基因组DNA提取
采用酚三氯甲烷方法对所有样品进行基因组DNA提取,分别利用NanoDrop 2000、1% 琼脂糖凝胶电泳、凝胶成像(Newbio Gi-1)检测DNA浓度、D值及DNA质量。
1.3" 微卫星及其引物的筛选
基于4个品种小型猪的全基因组微卫星(simple sequence repeat,SSR)测序数据,获得 2 518 个品种间共有的SSR,从中选取11个位于基因上的SSR进行引物设计及多态性片段检测,用于评估4个品种小型猪的遗传多样性。其中,正向引物的5′端连接荧光标记,本研究共用到3种荧光标记,6-羧基荧光素(6-carboxyfluorescein,6-FAM)、六氯荧光素(hexachloro fluorescein,HEX)和羧基四甲基罗丹明(carboxytetramethylrhodamine,TAMRA)分别显示蓝光、绿光和橙色荧光,反向引物为普通引物,均由上海派森诺生物科技股份有限公司合成。
1.4" 荧光引物PCR扩增
PCR反应体系(20 μL)包括30 ng/μL的模板DNA 1 μL、Mg2+含量25 mmol/L 10×buffer 2 μL、10 mmol/L 的dNTP 0.5 μL、5 U/L的Taq聚合酶0.5 μL、1 mmol/L的正向荧光引物和反向普通引物各 1 μL 及ddH2O 14 μL。PCR扩增在ABI-2720扩增仪(美国应用生物系统公司)上进行,程序为 94 ℃ 4 min;94 ℃ 30 s,不同退火温度退火30 s,72 ℃ 30 s,35个循环;72 ℃ 7 min;4 ℃保存。PCR产物经1.5%琼脂糖凝胶电泳检测,若条带清晰,无明显引物二聚体和杂带,可判定为扩增到目的片段。
1.5" 毛细管电泳及等位基因分型
毛细管电泳可以精确地区分SSR片段的大小。取10 μL荧光PCR产物,加入冷的70%乙醇至终体积50 μL,振荡充分混匀后3 700 r/min、4 ℃离心 30 min,静置15 min待乙醇挥发干净后加入GS-500LIZ分子内标和HiDi,瞬离样品至PCR仪进行95 ℃ 4 min变性后,于ABI 3730XL(美国应用生物系统公司)中进行毛细管电泳,利用GeneMapper软件对电泳结果进行微卫星片段大小和信号值的分析,确定每个微卫星位点的等位基因型。
1.6" 数据统计
采用Cervus 3.0.7软件计算每个SSR的等位基因数(Na)、等位基因频率、基因型频率、期望杂合度(He)、观察杂合度(Ho)、多态信息含量(PIC),同时检验4个品种猪的Hardy-Weinberg遗传平衡状态。采用PopGen 1.32进行4个品种间遗传相似性及遗传距离分析。采用MEGA 7.0.26构建Neighbour-Joining聚类图及Structure 2.2分析群体遗传结构。
2" 结果与分析
2.1" 4个品种小型猪SSR的获得
笔者所在课题组前期建立4个品种小型猪的全基因组SSR文库并进行高通量测序,发现4个品种中共有的多态性SSR位点有2 518个,对其进行注释发现大部分SSR位于基因间区(63.0%~65.4%),而位于基因区域的SSR中,有80、357和436个SSR分别位于5′UTR、3′UTR和编码区域。对位于基因区域上的SSR,在不同染色体上随机选取11个进行4个品种小型猪的遗传多样性分析。11个SSR在基因组上的位置及相应引物信息见表1。
2.2" 毛细管电泳检测SSR的目的片段
图1仅列出2个SSR在相应个体中的检测结果,其中,A图是ZX的35号个体在SYW7上的等位基因峰值,其2个峰值表明该等位基因是杂合子,其片段大小分别为208、232 bp;B图是WZS的1号个体在SYW9上的等位基因峰值,仅有1个峰值,表明该等位基因是纯合子,其片段大小是164 bp。
2.3" 等位基因数及品种间等位基因
采用11对SSR引物对170头个体进行荧光PCR,获得等位基因数和等位基因大小的统计结果,由表2可知, 在4个品种小型猪的170头个体中检
测到122个等位基因,每个位点检测到的等位基因数为8~17个,平均为11.1个;其中,SYW5和SWY8的等位基因数最多(17个),SYW10、SYW13和SWY19的等位基因数最少(8个)。此外,11个SSR在4个品种中扩增到的等位基因数不同,WZS、BM、LC和ZX中的等位基因数分别为76、66、76、81个,平均等位基因数分别为6.909 1、6.000 0、6.909 1、7.363 6个。
在4个品种猪中,分别计算每个SSR的最小等位基因频率(minor allele frequency,MAF),保留MAFgt;0.01的等位基因,通过计算每个品种内的等位基因频率和基因型频率挖掘种间特有的等位基因和基因型。由表3可知,在11个SSR座位中,检测到WZS特有等位基因和基因型分别有4、6个,BM特有等位基因和基因型分别有3、11个,LC特有等位基因和基因型均有9个,ZX特有等位基因和基因型分别有13、10个。以SYW5座位为例,在4个品种中共检测到17个等位基因,其中,172 bp的等位基因为BM特有,122、134、160 bp的等位基因为LC特有,138、144、146 bp 的等位基因为ZX特有,由148 bp组成的纯合基因型在WZS中特有,由 130 bp 和158 bp组成的杂合基因型、132 bp和 158 bp 组成的杂合基因型在BM中特有;其余SSR座位中品种特有的等位基因及基因型见表3。
2.4" 基于SSR分析小型猪的遗传多样性
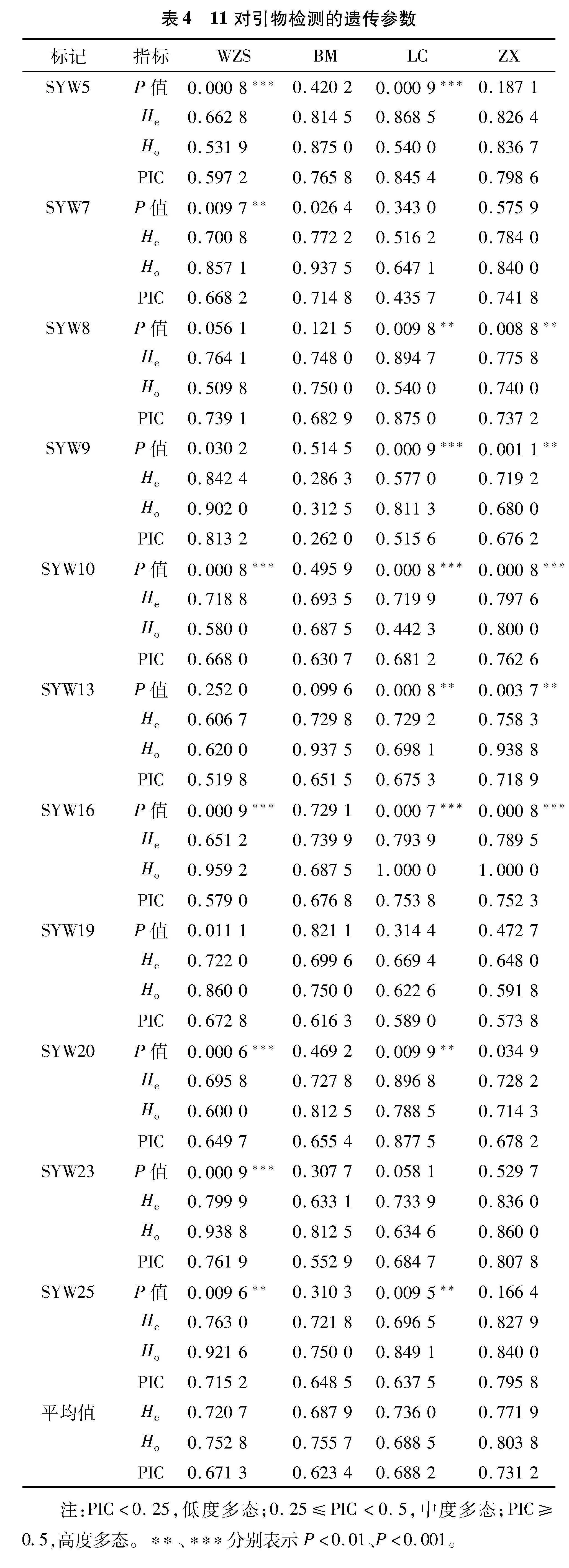
利用Cervus 3.0.7软件计算4个品种猪在11个SSR座位的杂合度、PIC和Hardy-Weinberg遗传平衡状态,进而分析4个品种猪的遗传多样性。由表4可知,在11个SSR座位中,4个群体的平均期望杂合度在0.687 9~0.771 9之间,SYW9座位最小为0.286 3;平均观察杂合度在0.688 5~0.803 8间,SYW9座位最小为0.3 125。在WZS、BM和ZX中,平均观察杂合度均高于平均期望杂合度,而LC的平均观察杂合度低于平均期望杂合度。
WZS在SYW 5、7、10、16、20、23和25座位均处于Hardy-Weinberg不平衡状态(Plt;0.01);BM在所有SSR座位上均处于Hardy-Weinberg平衡状态(Pgt;0.01);LC在SYW5、8、9、10、13、16、20和25座位均处于Hardy-Weinberg不平衡状态(Plt;0.01);ZX在SYW8、9、10、13和16座位均处于 Hardy-Weinberg不平衡状态(Plt;0.01)。
11个SSR座位的PIC值为0.262 0(SYW9)~0.877 5(SYW20),WZS和ZX在11个SSR座位上均表现为高度多态(PICgt;0.5);BM在SYW9座位上表现为中度多态(PIC=0.262 0),其余10个座位上为高度多态(PICgt;0.5);LC在SYW7座位上表现为中度多态(PIC=0.435 7),其余10个座位上为高度多态(PICgt;0.5)。表明4个品种猪均具有较丰富的遗传多样性,而ZX的平均PIC最高(0.731 2),BM的平均PIC最低(0.623 4),说明ZX的遗传多样性最高,LC和WZS次之,BM的遗传多样性最低。
2.5" 4个品种小型猪的遗传距离及系统进化树
由表5可知,4个品种猪的遗传一致度为 0.951 3~0.968 7(平均为0.962 1),遗传距离为0.031 8~0.049 9(平均为0.038 7)。WZS与LC的遗传一致度最高,遗传距离最小;BM与LC的遗传一致度最低,遗传距离最大。
由图2可知,WZS与LC聚类到一个分支,再与藏香猪聚类到一个分支,说明WZS与LC的亲缘关系较近。
3" 讨论与结论
3.1" 全基因组SSR分析方法的确定
本研究参考MISA软件的最佳分析参数[11-13],以猪参考基因组(Sus scrofa 11.1)为例进行全基因组范围SSR分析方法的初探, 通过比较SSR中重复单元的分布趋势证实本研究分析方法的可靠性。Liu等对猪参考基因组(Sus scrofa 10.2)的研究表明,Di-SSR中AC/GT重复单元占比最多,其次为AT/TA、AG/CT,最少为CG/GC,占比分别为45.91%、30.09%、23.64%和0.36%;Tri-SSR中AAC/GTT所占比例(30.46%)最多,其次为AAT/ATT(26.91%)、AAG/CTT(11.82%),最少为ACG/CGT(0.13%);Tetra-SSR中重复比例最多的3个是AAAT/ATTT(27.57%)、AAAG/CTTT(14.86%)和AAAC/GTTT(17.91%)[14],本研究结果与以上结果相似,进一步证实本研究分析方法的可行性,保证了其应用于后续4个猪种中全基因组SSR的分析的可靠性。
3.2" 基于SSR分型的种间遗传多样性研究
利用SSR的分型可进行物种的遗传多样性、品种的资源鉴定与分类、亲缘关系与系统发育的构建及动物标记辅助选育等方面的研究。但基于现有的研究报道发现,不同物种间的SSR具有很大的多态性差异。何瑜林等利用貂、鱼中的SSR进行猪品种的鉴定,其分别选取的34、55条SSR引物中仅有少数几条可用于品种的鉴定[15-16]。此外,随着基因组重测序技术的发展,Liu等以猪参考基因组中扫描的SSR为基础,并结合国内外家猪及野猪的个体基因组重测序数据进行SSR的分型,首次从全基因组层面进行猪SSR的分析[14]。由于猪的参考基因组(Sus scrofa 10.2)源于国外的杜洛克猪,以此挖掘得到的SSR在我国家猪中进行分型具有品种的片面性,导致在猪参考基因组中挖掘得到的1 620 469个SSR中,仅有16 527个在测序个体中表现为高度多态性而作为遗传标记。因此,利用种间的SSR信息进行品种的遗传多样性、品种鉴定等研究并不能很好地反映出研究对象的真实情况,在进行相应的研究过程中可能会费时费力。
为真实反映4个小型猪品种中的SSR特性,本研究直接针对4个待测小型猪品种进行全基因组SSR文库构建,通过8种生物素标记的探针杂交全基因组含有SSR的序列,获得待测个体中更为准确的SSR,并依据其长度进行基因分型,在WZS、BM、LC及ZX中分别发现60 020、70 886、63 968、42 400个具有多态性的SSR(SSLP≥2),丰富了小型猪基因组中的SSR信息并可作为小型猪的遗传标记而进一步应用。
3.3" SSR类型及组成的特点
据研究报道,在绝大多数真核生物中单核苷酸组成的SSR占有最多的比例,其次为双核苷酸组成的SSR[17-19]。本研究在4个小型猪品种中发现,SSR总数在WZS中最多,而在ZX中最少,但双、三、四、五、六核苷酸SSR的含量呈现由多至少的变化。此外,4个猪种中不同类型SSR重复单元的分布模式却存在差异,Hexa-SSR中重复单元在4个猪种中均表现不同的分布模式,而ZX中Tri-和Tetra-SSR重复单元的分布模式不同于其他3个猪种。因此,推测依据SSR中重复单元的分布模式可以用于区分4个小型猪品种,结合SSR的SSLP进行群体验证,可应用于种间的鉴定。
现有研究认为,基因组重测序的建库及测序过程中GC序列的偏好性可能是导致不同类型SSR中含有GC的重复单元(CG、CCG等)较少的原因[20],但本研究利用的寡核苷酸探针兼顾了4种核苷酸,包含(ACG)8及(AGG)8探针等对基因组的SSR序列进行杂交,保证更为全面地富集全基因组中SSR;此外,基于猪参考基因组的研究结果,均发现CG/GC、ACG/CGT等重复单元组成的SSR占据很少的比例,这结果与玉米等物种中的研究结果[21-24]相似。以上表明,GC重复单元的SSR占据少部分且具有较低的多态性,其可能是因为GC富集的序列常与功能性相关[25]。
本研究首次从全基因组水平进行4个小型猪种SSR的比较,分析了SSR重复单元的分布模式,并发现在4个猪种中,双、三、四、五及六核苷酸组成的SSR中重复单元的分布模式截然不同,借此可用于区分和鉴别4个猪种。对品种间共有及特异的SSR进行挖掘和注释,发现了具有长度多态性的SSR可能通过影响相关基因的编码区或调控区进而影响基因功能,如FGF23、MYF6、IGF1R和LEPROT等基因,提示多态性SSR可能与小型猪的生长发育相关。
参考文献:
[1]Li Y C,Korol A B,Fahima T,et al. Microsatellites within genes:structure,function,and evolution[J]. Molecular Biology and Evolution,2004,21(6):991-1007.
[2]俞春英,杨燕燕,周建华,等. 3个品种兔微卫星标记的遗传多样性研究[J]. 中国畜牧兽医,2018,45(12):3486-3496.
[3]许雯雯,龙松华,邱财生,等. 95份甘蔗栽培材料的SSR引物筛选及遗传多样性分析[J]. 西南农业学报,2018,31(12):2498-2505.
[4]王瑞云,刘笑瑜,王海岗,等. 用高基元微卫星标记分析中国糜子遗传多样性[J]. 中国农业科学,2017,50(20):3848-3859.
[5]师沛琼,杨茂发,吕召云,等. 贵州省烟青虫遗传多样性[J]. 中国农业科学,2014,47(9):1836-1846.
[6]Levine A J,Brivanlou A H. GDF3,a BMP inhibitor,regulates cell fate in stem cells and early embryos[J]. Development,2006,133(2):209-216.
[7]Just F,Reyer H,Muráni E,et al. Genetic variants of major genes contributing to phosphate and calcium homeostasis and their association with serum parameters in pigs[J]. Journal of Applied Genetics,2018,59(3):325-333.
[8]石甜甜,何杰丽,高志军,等. 利用EST-SSR评估糜子资源遗传差异[J]. 中国农业科学,2019,52(22):4100-4109.
[9]魏丹丹,刘" 燕,杜" 洋,等. 柑橘全爪螨微卫星位点鉴定与信息分析[J]. 中国农业科学,2016,49(2):282-293.
[10]Liu C C,Liu Y,Zhang X Y,et al. Characterization of porcine simple sequence repeat variation on a population scale with genome resequencing data[J]. Scientific Reports,2017,7:2376.
[11]Wang L J,Zhang G M,Lin F,et al. Expression of the insulin-like growth factor system in skeletal muscle during embryonic and postnatal development in the first filial generation pigs from Erhualian and Yorkshire reciprocal crosses[J]. General and Comparative Endocrinology,2011,173(1):56-62.
[12]King K,Moody A,Fisher S A,et al. Genetic variation in the IGSF6 gene and lack of association with inflammatory bowel disease[J]. European Journal of Immunogenetics,2003,30(3):187-190.
[13]Wu X W,Shi W B,Cao X. Multiplicity of BMP signaling in skeletal development[J]. Annals of the New York Academy of Sciences,2007,1116(1):29-49.
[14]Liu C C,Liu Y,Zhang X Y,et al. Characterization of porcine simple sequence repeat variation on a population scale with genome resequencing data[J]. Scientific Reports,2017,7(1):2376.
[15]何瑜林,黄" 丽,封" 毅,等. ISSR分子标记鉴定陆川猪及其肉制品[J]. 肉类研究,2014,28(10):10-14.
[16]金玉兰,李婉丽,李" 松,等. ISSR分子标记鉴定巴马香猪[J]. 四川动物,2016,35(4):569-573.
[17]Sharma P C,Grover A,Kahl G. Mining microsatellites in eukaryotic genomes[J]. Trends in Biotechnology,2007,25(11):490-498.
[18]Murat C,Riccioni C,Belfiori B,et al. Distribution and localization of microsatellites in the Perigord black truffle genome and identification of new molecular markers[J]. Fungal Genetics and Biology,2011,48(6):592-601.
[19]Qian J,Xu H B,Song J Y,et al. Genome-wide analysis of simple
sequence repeats in the model medicinal mushroom Ganoderma lucidum[J]. Gene,2013,512(2):331-336.
[20]Xu J,Liu L,Xu Y B,et al. Development and characterization of simple sequence repeat markers providing genome-wide coverage and high resolution in maize[J]. DNA Research,2013,20(5):497-509.
[21]Xiao J,Zhao J,Liu M J,et al. Genome-wide characterization of simple sequence repeat (SSR) loci in Chinese jujube and jujube SSR primer transferability[J]. PLoS One,2015,10(5):e0127812.
[22]Wang X W,Yang S,Chen Y D,et al. Comparative genome-wide characterization leading to simple sequence repeat marker development for Nicotiana[J]. BMC Genomics,2018,19(1):500.
[23]Cui Y F,Yan Y Q,Liu D,et al. Platelet endothelial aggregation receptor-1 (PEAR1) is involved in C2C12 myoblast differentiation[J]. Experimental Cell Research,2018,366(2):199-204.
[24]Kazarian E,Son H,Sapao P,et al. SPAG17 is required for male germ cell differentiation and fertility[J]. International Journal of Molecular Sciences,2018,19(4):1252.
[25]Benjamini Y,Speed T P. Summarizing and correcting the GC content bias in high-throughput sequencing[J]. Nucleic Acids Research,2012,40(10):e72.

