西藏牦牛源牛支原体T10株全基因组测序及其序列分析




摘 要: 旨在进一步完善牛支原体全基因组数据库,阐明其生物学功能和遗传进化关系,对西藏牦牛源牛支原体T10株进行了全基因组测序及其序列分析。使用Nanopore和Illumina PE150测序平台对T10株进行全基因组序列测定,利用生物信息学对其进行基因组特征分析和利用基因系统进化树与国内外分离株进行遗传进化关系比对。基因测序结果显示,T10株全基因组大小为987 812 bp,GC含量为29.27%,预测到822个编码基因,编码基因总长度为709 129 bp;通过功能数据库注释结果显示,注释到与膜转运蛋白相关基因22个,碳水化合物酶相关基因7个、糖基转移酶类相关基因4个,分泌蛋白基因相关43个、T3SS效应蛋白相关基因51个,病原与宿主互作相关基因28个,细菌毒力相关基因14个,抗性相关基因10个;通过比较基因组学分析结果显示,T10与08M、CQ-W70、Hubei-1、Ningxia-1和PG45均存在氨基酸突变和基因片段的插入或缺失,以及基因家族数量的差异,其中基因家族数量差异在8~32个。通过遗传进化关系分析结果显示:T10与HB0801、16M、Hubei-1、CQ-W70、NM2012、Ningxia-1、08M、JF4278、KG4397和PG45均在同一分支,但与PG1、PG2处于不同分支,其中与HB0801亲缘关系最近,与PG45亲缘关系较远。本研究不仅完善了牛支原体全基因组数据库,详细阐述了与致病性相关基因,还充分阐明了T10株以及与国内外其他牛支原体之间的遗传进化关系,明确了T10株基本基因组信息以及与国内外菌株亲缘关系,为后续进行致病机制以及疫苗研究提供理论依据。
关键词: 牦牛;牛支原体;全基因组测序;基因功能注释;比较基因组
中图分类号:S852.62
文献标志码:A""" 文章编号:0366-6964(2024)05-2154-14
收稿日期:2023-09-21
基金项目:西藏自治区重点研发计划项目(XZ202001ZY0046N);财政部和农业农村部“国家现代农业产业技术体系”项目(CARS-37);西藏农牧学院研究生教育创新计划项目(YJS2023-20)
作者简介:罗 婷(1999-),女,河北邢台人,硕士生,主要从事动物病原及分子生物学的研究,E-mail: win2021shan@163.com
*通信作者:牛家强,主要从事动物病原及分子生物学的研究,E-mail: lznjq@163.com
Whole Genome Sequencing and Sequence Analysis on T10 of Mycoplasma bovis Strain
from Yaks in Xizang
LUO" Ting, HAN" Zhu, XU" Yefen, CAI" Lin, SUOLANG" Sizhu, XU" Jinhua,
NIU" Jiaqiang*
(
Provincial Key Laboratory of Xizang Plateau Animal Epidemic Disease Research, College of
Animal Science, Xizang Agriculture and Animal Husbandry University,
Linzhi 860000," China)
Abstract: This study aims to further improve the complete genome database of Mycoplasma bovis, elucidate its biological characteristics and genetic evolution relationship, the whole genome sequencing and sequence analysis of Mycoplasma bovis T10 strain derived from Xizang yaks were conducted. The entire genome of T10 strain was sequenced using the Nanopore and Illumina PE150 sequencing platforms, and the genomic characteristics were analyzed using bioinformatics. Genetic phylogenetic trees were used to compare genetic evolution relationships with domestic and foreign isolates. The gene sequencing results showed that the total genome size of T10 strain was 987 812 bp, with a GC content of 29.27%. It was predicted that 822 coding genes were present, with a total length of 709 129 bp; According to the annotation results of the functional database, 22 genes related to membrane transport proteins, 7 genes related to carbohydrate enzymes, 4 genes related to glycosyltransferases, 43 genes related to secretion proteins, 51 genes related to T3SS effector proteins, 28 genes related to pathogen host interaction, 14 genes related to bacterial virulence, and 10 genes related to resistance were annotated; Through comparative genomic analysis, it was found that T10 and 08M, CQ-W70, Hubei-1, Ningxia-1, and PG45 all exhibit amino acid mutations and gene fragment insertions or deletions, as well as differences in the number of gene families, with a difference in the number of gene families ranging from 8 to 32. The results of genetic evolution analysis show that T10 is in the same branch as HB0801, 16M, Hubei-1, CQ-W70, NM2012, Ningxia-1, 08M, JF4278, KG4397, and PG45, but in different branches from PG1 and PG2. Among them, T10 has the closest genetic relationship with HB0801 and the farther genetic relationship with PG45. This study not only improved the complete genome database of Mycoplasma bovis and elaborated on the genes related to pathogenicity, but also fully elucidated the genetic evolution relationship between T10 strain and other Mycoplasma bovis strains at home and abroad. It clarified the basic genome information of T10 strain and its genetic relationship with domestic and foreign strains, providing a theoretical basis for future research on pathogenic mechanism vaccines.
Key words: yak; Mycoplasma bovis; whole-genome sequencing; gene function annotation; comparative genomics
*Corresponding author:" NIU Jiaqiang, E-mail: lznjq@163.com
牛支原体(Mycoplasma bovis,M. bovis)是影响养牛业健康发展的重要病原体之一,可通过呼吸道、生殖道、消化道、近距离接触等途径进行传播,引起犊牛肺炎、角膜结膜炎、中耳炎、关节炎、奶牛乳腺炎和生殖道炎等多种疾病[1]。牛支原体单纯感染一般不表现临床症状,但会降低牛的机体抵抗力,临床中常与多杀性巴氏杆菌(Pasteurella multocida, Pm)、溶血性曼氏杆菌(Mannheimia haemolytica, Mh)、牛呼吸道合胞病毒(bovine respiratory syncytial virus, BRSV)、牛传染性鼻气管炎病毒(infectious bovine rhiontracheitis virus, IBRV)等病原体发生混合感染或继发感染[2],可引发牛呼吸道疾病综合征(bovine respiratory disease complex, BRDC)。自1961年Hale[3]首次从美国患乳腺炎的病牛中分离出牛支原体以来,牛支原体病开始在美洲及丹麦、荷兰、瑞士等欧洲国家相继出现[4-7]。直到2008年石磊[8]和辛九庆[9]对牛呼吸道疾病主要病原确诊为牛支原体以来,该病相继在重庆、宁夏、内蒙、新疆等地均有发现[10-13]。
近年来,众多学者对西藏牦牛感染牛支原体状况进行了一系列研究[14],尤其在2017—2019年间,牛家强团队对我国牦牛主产区感染牛支原体情况进行了较为系统研究,结果表明,牛支原体感染较为普遍[15],从阳性牛群中成功分离到10株牛支原体(暂命名为西藏牦牛源牛支原体)[16],对其进行了培养特性和生长特性的研究,发现分离株在添加10%马血清的培养基更适合生长,培养24~36 h达到对数生长期[17]。并对分离株进行了药物敏感性及其耐药机制的研究,发现分离株对氟喹诺酮类和氨基糖苷类抗生素存在不同程度的耐药,存在两个及两个以上基因位点的氨基酸同时发生突变,则会导致耐药菌株的出现[18]。也进行了鸡胚、家兔和牦牛的致病性试验,发现试验动物均出现了典型的临床症状和病理变化[19-21]。通过小鼠免疫原性研究,发现分离株可引起强烈的免疫应答[19]。经家兔肺脏组织转录组学研究,发现试验组与对照组miRNA基因数量及功能均存在差异[20]。
作者开展本研究旨在进一步追溯其传染来源,全面掌握T10株基因组基本特征,明确遗传进化关系,了解与国内外其他牛支原体之间的遗传多样性,从而完善牛支原体基因数据库,为牛支原体病致病机制与疫苗研究提供基础依据。
1 材料与方法
1.1 菌株来源
本试验测序用菌株为本团队成员前期经分离鉴定所获得的牦牛源牛支原体T10株,-80℃保存。其他菌株08M(GenBank:CP019639.1)、16M(GenBank:CP038861.1)、CQ-W70(GenBank:CP005933.1)、Hubei-1(GenBank:CP002513.1)、HB0801(GenBank:CP002058.1)、NM2012(GenBank:CP011348.1)、Ningxia-1(GenBank:CP023663.1)、PG1(GenBank:BX293980.2)、PG2(GenBank:CU179680.1)、KG4397(GenBank:AP019558.1)、JF4278(GenBank:LT578453.1)和PG45(GenBank:CP002188.1)的基因组序列均来源于NCBI。
1.2 菌株基因组总DNA的提取
将T10株在液体培养基中[16]按1∶10的比例传代培养3代,稳定生长后,再按照1∶100的比例进行扩大培养至500 mL。采用煮沸离心法提取牛支原体基因组总DNA,送至诺禾致源生物有限公司进行牛支原体全基因组测序及其序列分析。
1.3 样品合格性检测
采用SDS或STE法对样本基因组DNA进行提取,再利用琼脂糖凝胶电泳对样品进行纯度和完整性检测,用Qubit进行定量检测。
1.4 基因组的序列测定与分析
合格样品进行Nanopore和Illumina PE150测序,测序后原始数据按照以下步骤进行分析:原始数据过滤后得到分析数据,进行基因组组装,使用GeneMarkS[22](Version4.17)、RepeatMasker[23](Versionopen-4.0.5)、TRF[24](Tandem Repeats Finder, Version 4.07b)、tRNAscan-SE[25]、rRNAmmer[26]、IslandPath-DIOMB[27]、phiSpy[28]软件分别预测基因组的编码基因、非编码RNA、基因岛、重复序列和前噬菌体,进行基因组组分分析。使用GO(Gene Ontology)[29]、KEGG(Kyoto Encyclopedia of Genes and Genomes)[30]、COG(Cluster of Orthologous Groups of proteins)[31]数据库对基因组功能进行注释,再使用TCDB(Transporter Classification Database)[32]、CAZY(Carbohydrate-Active enZYmes Database)[33]、PHI(Pathogen Host Interactions Database)[34]和VFDB(Virulence Factors of Pathogenic Bacteria)[35]数据库对基因组中耐药基因进行预测分析。使用CRISPR[36]digger(Version 1.0)软件对T10基因组进行基因圈图制作,并进行基因组可视化分析。
1.5 比较基因组学分析
采用MUM mer[37](Version 3.22)和LASTZ[38](Version 1.02.00)分别进行SNP、Indel和SV注释与统计。采用OrthoFinder[39](Version 2.5.4b)软件对样本的蛋白质序列进行基因家族聚类分析。采用Tree best(Version 1.9.2)(Neighbor-Joining, NJ)或PHYML(Maximum likelihood, ML)(Version v3.0)软件构建物种遗传进化树,使用iTOL对构建的遗传进化树进行可视化分析[40]。
2 结 果
2.1 T10株基因组基本特征分析
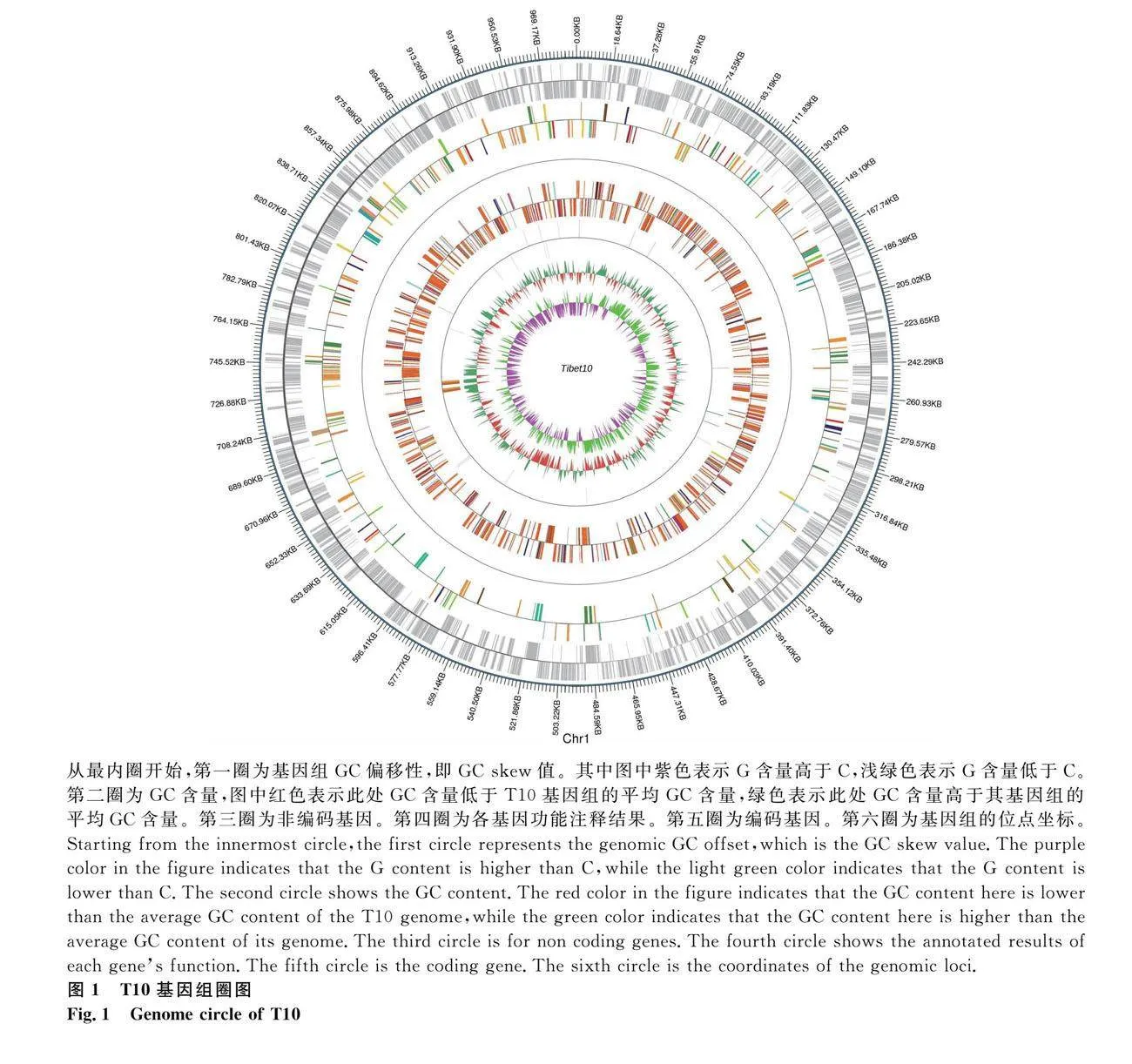
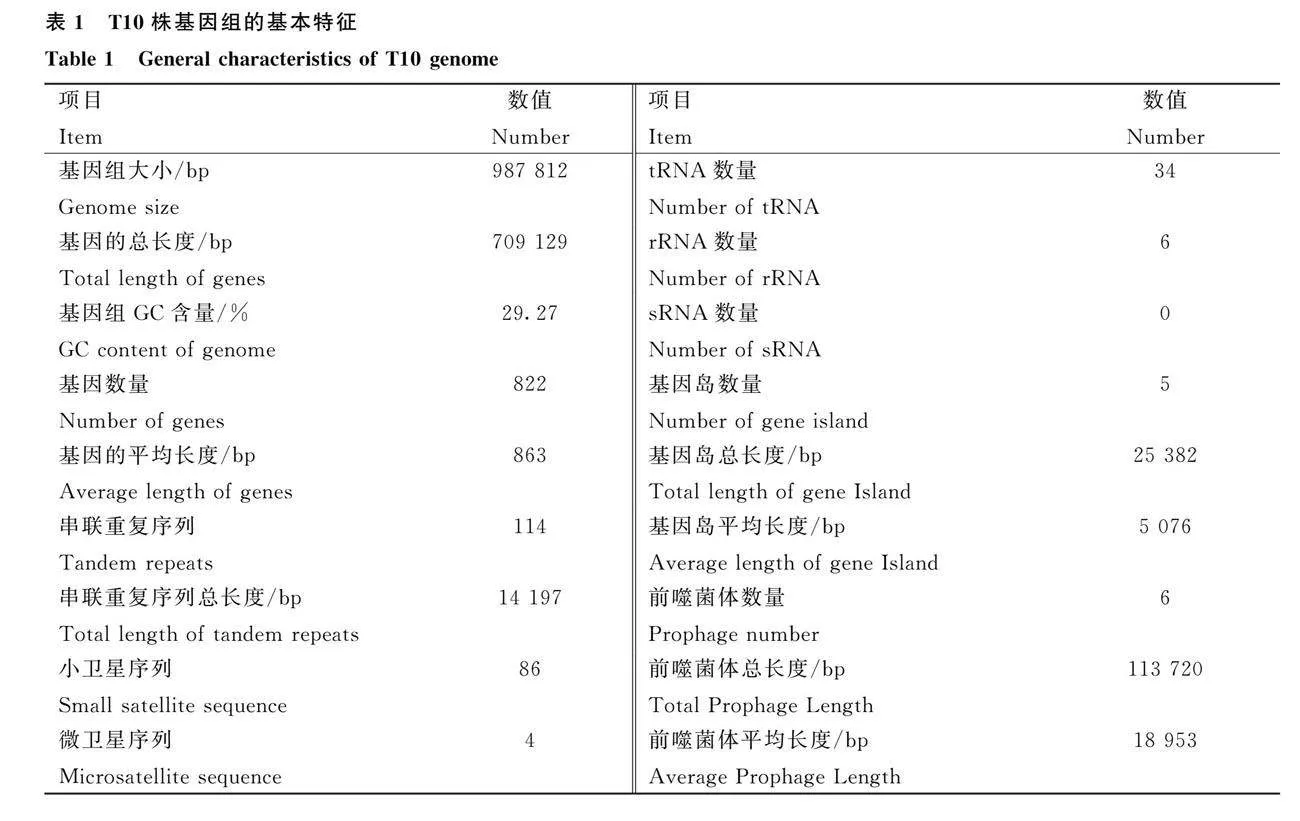
T10株基因组大小为987 812 bp,基因组GC含量为29.27%,预测到编码基因有822个,串联重复序列有114个,tRNA基因有34个,rRNA基因有6个,还预测到T10存在5个基因岛和6个前噬菌体(表1),并且绘制了T10的基因组圈图(图1)。T10全基因组数据已上传至NCBI,登录号为CP062195.1。
2.2 基因功能注释分析
2.2.1 GO数据库注释结果
GO数据库注释分为三大类,分别是细胞组分、分子功能和生物过程。在GO分析中,在细胞组分中,与细胞和细胞部件相关的基因最多,各有304个。在分子功能中,与催化活性有关的基因最多,共有532个;与结合相关的基因有438个。在生物过程中,与代谢过程相关的基因最多,有562个;与细胞过程相关的基因有546个(图2A)。
2.2.2 KEGG数据库注释结果
在KEGG分析中,在细胞过程、环境信息处理、遗传信息处理、人类疾病、新陈代谢和生物体系统的26个通路中均得到基因注释结果。其中在细胞功能中,与原核细胞群落相关基因有11个。在环境信息处理功能中预测到与膜传输通路相关基因数有23个。并且在遗传信息处理功能中,与翻译有关的基因数目最多,共有60个基因。还在人类疾病功能中,与癌症相关基因数有9个。另外,在新陈代谢功能碳水化合物代谢通路相关的基因有33个;与核苷酸代谢通路相关的基因有29个;与复制和修复相关的基因有27个;与能量代谢通路相关的基因有26个。除此之外,在生物体系统中,与内分泌和老化系统相关基因有3个(图2B)。
2.2.3 COG数据库注释结果
在COG蛋白数据库分析中,与翻译,核糖体结构功能相关的基因最多,共有97个;与复制、重组和修复相关的基因有30个;与碳水化合物运输和代谢相关的基因有26个;与核苷酸转运与代谢相关的基因有21个(图2C)。
2.3 特定基因功能注释
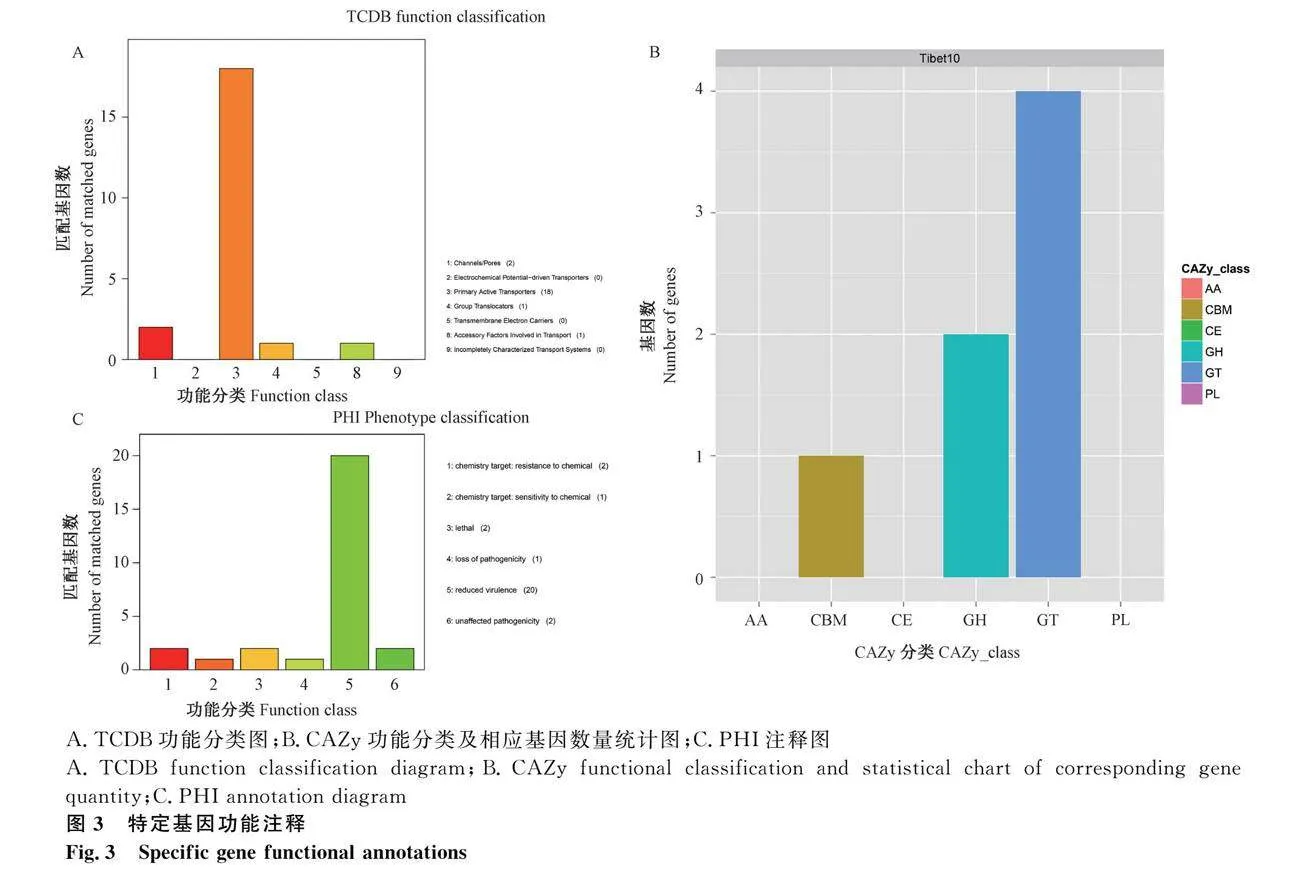
2.3.1 TCDB数据库注释结果
在TCDB数据库分析中,共注释到22个与膜转运蛋白相关的基因,其中第一级统计结果中初级活性转运体基因有18个;通道/孔基因2个;群转运体及转运辅助因子相关基因各1个(图3A)。
2.3.2 CAZy数据库注释结果
在CAZy数据库分析中,共注释到7个与碳水化合物酶相关的基因。其中有4个糖基转移酶类的基因,2个糖苷水解酶的基因和1个与碳水化合物结合组件相关的基因(图3B)。
2.3.3 分泌蛋白及T3SS效应蛋白预测结果
在分泌蛋白分析中,共预测到具有信号肽结构的蛋白58个,具有跨膜结构的蛋白240个,综合预测为分泌蛋白有43个。在822个编码基因中,预测到T3SS效应蛋白基因51个。
2.3.4 PHI数据库分析结果
在PHI分析中,共注释到28个病原与宿主互作相关的基因,其中存在20个导致病原体致病能力减弱的基因;2个抗药基因;2个致死因子;2个对病原体致病性没有影响的基因;1个药物敏感基因;1个导致病原体致病能力丧失的基因(图3C)。
2.3.5 VFDB数据库分析结果
在VFDB分析中,预测T10存在EF-Tu、LOS、ClpE、MOMP、Lipoate protein ligase A1等14个潜在毒力基因。其中EF-Tu和LOS基因与黏附功能有关。
2.3.6 CARD数据库分析结果
在CARD分析中,注释到gyrB、macB、bcrA、sav1866、tetB、tetA、lmrC、lmrD、efrA和efrB10个抗性基因。
2.4 比较基因组分析
2.4.1 牛支原体的基本特征比较分析
本研究将T10与GenBank公布的7个牛支原体菌株进行了基本特征信息比较,T10基本特征与国际参考株PG45相似,在rRNA数量中与CQ-W70和Hubei-1存在差异(表2)。
2.4.2 SNP注释与统计
通过SNP注释与统计,发现T10与08M、CQ-W70、Hubei-1、Ningxia-1和PG45均存在SNP变异。T10与08M相比,基因区内非同义突变有49个SNP位点;与CQ-W70相比,在基因区内非同义突变有62个SNP位点;与Hubei-1相比,在基因区内非同义突变有55个SNP位点;与Ningxia-1相比,在基因区内非同义突变有70个SNP位点;与PG45相比,在基因区内非同义突变有3 090个SNP位点,分别导致相应的氨基酸发生改变(表3)。
2.4.3 InDel注释与统计
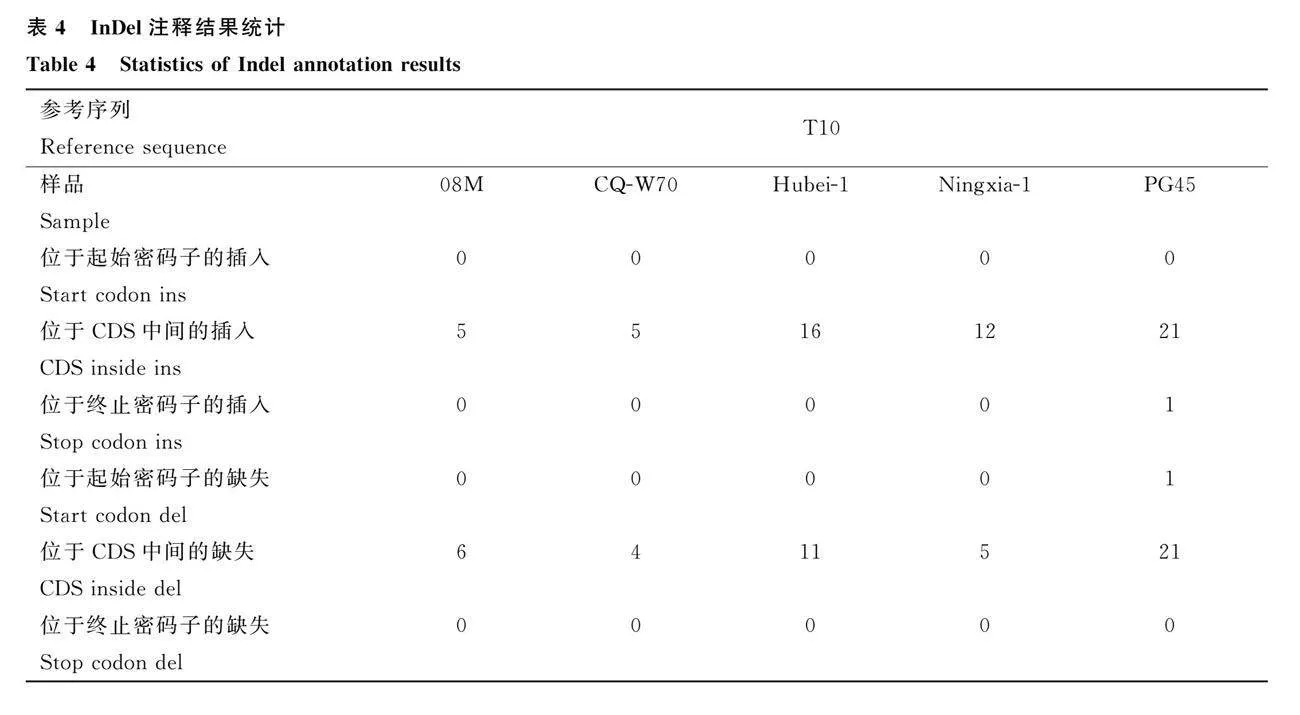
InDel是指基因组中小片段的插入和缺失序列。通过InDel注释与统计,发现T10与08M、CQ-W70、Hubei-1、Ningxia-1均存在位于CDS中间的插入或缺失,其中Hubei-1的插入缺失数量较多。还与PG45在位于CDS中间的插入或缺失存在21个插入或缺失序列,并且还在位于终止密码子的插入和起始密码子的缺失各1个序列(表4)。
2.4.4 SV(Structural Variation)注释与统计
结构性变异(Structural Variation,SV)指基因组水平上大片段的插入、缺失、倒置、易位等序列。T10与08M、Hubei-1在结构上存在大片段的插入/缺失区域;与CQ-W70、Ningxia-1存在大片段的插入/缺失和易位兼倒置区域;与PG45存在大片段的插入/缺失,还存在易位、倒置和易位兼倒置区域(图4)。
2.4.5 基因家族分析
通过基因家族分析,发现T10有822个编码基因,有757个基因家族,有812个基因家族中的基因。T10存在10个未参与聚类的基因,并且T10与08M、CQ-W70、Hubei-1、Ningxia-1和PG45中均存在未参与聚类的家族基因,但PG45在未被聚类到任何家族的基因方面的数目最多。除此之外,未发现T10存在特有基因家族,而Hubei-1存在1个特有基因家族,3个特有基因家族中含有的基因(表5)。
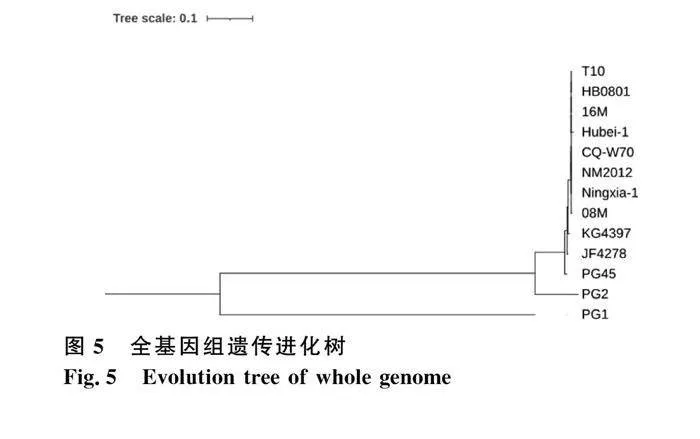
2.4.6 遗传进化分析
遗传进化树是具有共同祖先的各物种相互间演化关系的树。T10的基因组序列与HB0801、16M、Hubei-1、CQ-W70、NM2012、Ningxia-1、08M、JF4278、KG4397和PG45均处于同一分支上,且与HB0801亲缘关系最近,与PG1和PG2处于不同的分支(图5)。
3 讨 论
本研究对T10株进行了全基因组序列测定,对该基因组的基本特征进行了分析,并与国内外菌株进行了比较基因组分析。发现T10基因组由987 812 bp的碱基组成,其中GC%含量为29.27%,T10基因组大小比PG45[41]减少了15 592 bp,与NM2012[42]最相似;GC%含量比PG45减少了0.04%,与08M[43]的GC%含量一致。预测到T10株存在非编码RNA包括34个tRNA,6个rRNA,其中包括5S rRNA、16S rRNA、23S rRNA各2个,与PG45预测结果一致;基因岛与病原机理、生物体的适应性等多种生物功能相关,T10预测到5个基因岛;前噬菌体能增强菌体的黏附性,协助菌体逃避宿主的防御,与菌体毒力有关,T10预测到6个前噬菌体。
牛支原体常寄生在牛体内,它需要通过影响牛的代谢过程和翻译来获取营养、维持自身生存和实现其生物学功能。在基因功能注释分析中,T10主要在代谢过程和翻译相关基因的富集程度最高。因此,牛支原体主要通过代谢、翻译相关途径来满足其生存需求。研究表明糖基转移酶可以介导甘油糖脂的合成,而甘油糖脂是支原体膜的结构成分,对膜的性质和稳定性起着重要作用[44]。糖苷水解酶可以水解各种含糖化合物中的糖苷键,在生物体的糖基化方面发挥重要作用。在CAZy数据库中,T10注释到4个糖基转移酶类基因,2个糖苷水解酶基因。因此糖基转移酶和糖苷水解酶对牛支原体的代谢活动有重要作用。
牛支原体是一种导致牛呼吸系统感染的病原体,它感染时会通过多种不同的基因来改变宿主细胞的功能,以利于自身的生存和繁殖[45]。本研究通过PHI分析,预测T10具有GAPDH、secA、lplA1、pdhB、glyA、FbaA等20个导致致病机制减弱的基因,2个GyrA抗药基因和导致病原体致病能力丧失的nifS基因。有研究表明甘油醛-3-磷酸脱氢酶(GAPDH)除了在糖酵解中的作用外,还与病原体的毒力有关,并且由于其细胞表面的结构特点,是定殖在致病性和非致病性正常菌群的组织黏附素[46]。Perez-Casal等[47]分离了牛支原体编码GAPDH的gap基因,并表明用牛支原体定殖的牛能够对GAPDH产生免疫反应,为后续开发有效的牛支原体疫苗提供方向。GyrA基因表达DNA促旋酶A亚基,在DNA复制、重组和转录中具有重要作用,本团队在研究氟喹诺酮类抗生素耐药机制中,将GyrA作为该类药物的靶位基因进行检测[18]。nifS是一种磷酸吡哆醛结合酶,催化L-半胱氨酸脱硫产生L-丙氨酸和硫[48],nifS已被证实参与幽门螺杆菌的重要毒力特征的调节,其失活对幽门螺杆菌的生长具有重要影响[49]。但还未在牛支原体中得到证实,因此后续可以对该基因进行验证是否会导致病原体致病能力丧失。通过VFDB分析,预测T10存在EF-Tu、LOS、ClpE、MOMP、Lipoate protein ligase A1等14个潜在毒力基因,其中EF-Tu是具有黏附因子的特征,并诱导促炎[50];LOS基因参与免疫系统逃避,附着于上皮组织,是促炎反应的重要介质[51]。在HB0801中预测到一个潜在毒力基因,即VSP Y1。T10株与HB0801[52]株相比,其潜在毒力因子种类和数量不同,但是具体对动物的致病性还需要通过临床试验进一步验证。在CARD分析中,注释到gyrB、macB、tetB、tetA、lmrC、lmrD等10个抗性基因,其中tetB、tetA基因对四环素具有抗性;MacB基因对大环内酯类抗生素具有抗性;GyrB基因对氟喹诺酮类抗生素具有抗性;lmrC、lmrD基因对林可霉素具有抗性。前期本团队已对氟喹诺酮类和大环内酯类抗生素进行药物敏感性验证,药物敏感性结果显示T10对这两类药物均存在耐药。可能与临床不规范使用抗生素有关,也可能是其他菌株耐药基因的平行转移导致的,因此这对该菌株以后的临床治疗具有指导意义。
为了探究T10的来源,选取了国内4个不同地区的菌株分别为08M、CQ-W70、Hubei-1、Ningxia-1和国际参考株PG45进行了比较基因组分析。基因组可塑性是通过三种水平基因转移过程产生的,主要是通过转化、转导和接合三种方式进行水平基因转移。单核苷酸多态性(single nucleotide polymorphism, SNP)和InDel的变异是影响基因功能和表达的调控机制。SNP是在基因组水平上由单个核苷酸的变异所引起的DNA序列多态性,包括单个碱基的转换、颠换等。学者Qi等[52]对HB0801和Hubei-1的SNP位点进行分析,发现共存在122个SNP位点。在SNP数据库注释发现,T10株与Hubei-1[53]在基因区发生的单个核苷酸的变异有66个位点,与PG45[41]在基因区发生的单个核苷酸的变异数量最多;与08M、CQ-W70、Hubei-1、Ningxia-1和PG45相比,在基因区内发生非同义突变各有49、62、55、70和3 090个SNP位点。发生非同义突变会引起蛋白质的形态、功能和酶催化活性发生改变。InDel是基因组中小片段的插入和缺失序列。预测到T10株与08M、CQ-W70、Hubei-1、Ningxia-1和PG45均在位于CDS中间的插入或缺失序列。另外,还与PG45存在位于终止密码子的插入和起始密码子的缺失序列。这些基因组中小片段插入或缺失会影响蛋白质的稳定性和功能,对后续研究基因表达和蛋白质功能具有重要意义。基因家族是衡量家族内具有相似的结构和功能的一组基因。T10与08M、CQ-W70、Hubei-1、Ningxia-1和PG45在所有基因家族个数比较相近,与Hubei-1的基因家族个数最为接近,由此可见,T10与Hubei-1具有共同来源,另外T10还发现存在10个未被聚类到的基因,其中5个基因未被注释到,其他5个基因分别与假定蛋白、脂蛋白、锌依赖性醇脱氢酶等功能有关。脂蛋白是牛支原体膜上一种能够调节宿主的免疫系统,使得免疫细胞凋亡或者死亡的蛋白质[54]。由此可见,脂蛋白与其感染性有关。锌依赖性醇脱氢酶(Zinc-dependent alcohol dehydrogenase)是生物体内参与氧化还原的催化剂,存在两个形状不同的活性位点,其决定了酶的底物范围和选择性,但未见文献报道该酶与感染性相关,若需了解其感染性可后续进行验证[55]。通过遗传进化树分析发现T10与08M、CQ-W70、Hubei-1、Ningxia-1和PG45均在同一分支且亲缘关系很近,这些探究为追溯T10株来源提供研究方向。
4 结 论
本研究分析了牛支原体T10株的完整基因组信息,并与国内外其他牛支原体进行了比较,从而进一步完善了牛支原体的基因数据库。阐明了T10株的生物学特性与遗传进化关系,其中,对T10株的致病性和耐药性进行了详细说明,为今后开展牛支原体病的相关研究提供基础依据。
参考文献(References):
[1] MAUNSELL F P, WOOLUMS A R, FRANCOZ D, et al. Mycoplasma bovis infections in cattle[J]. J Vet Intern Med, 2011, 25(4):772-783.
[2] 武 琪, 张玉娟, 刘 桐, 等. 牛支原体黏附素及黏附素结合蛋白研究进展[J]. 畜牧兽医学报, 2023, 54(8):3206-3216.
WU Q, ZHANG Y J, LIU T, et al. Research progress of Mycoplasma bovis adhesins and adhesin-binding proteins[J]. Acta Veterinaria et Zootechnica Sinica, 2023, 54(8):3206-3216. (in Chinese)
[3] HALE H H, HELMBOLDT C F, PLASTRIDGE W N, et al. Bovine mastitis caused by a Mycoplasma species[J]. Cornell Vet, 1962, 52:582-591.
[4] KIRK J H, GLENN K, RUIZ L, et al. Epidemiologic analysis of Mycoplasma spp isolated from bulk-tank milk samples obtained from dairy herds that were members of a milk cooperative[J]. J Am Vet Med Assoc, 1997, 211(8):1036-1038.
[5] FEENSTRA A, BISGAARD MADSEN E, FRIIS N F, et al. A field study of Mycoplasma bovis infection in cattle[J]. J Vet Med, 1991, 38(1-10):195-202.
[6] TER LAAK E A, NOORDERGRAAF J H, DIELTJES R P J W. Prevalence of mycoplasmas in the respiratory tracts of pneumonic calves[J]. J Vet Med Ser B, 1992, 39(1-10):553-562.
[7] POUMARAT F, LE GRAND D, PHILIPPE S, et al. Efficacy of spectinomycin against Mycoplasma bovis induced pneumonia in conventionally reared calves[J]. Vet Microbiol, 2001, 80(1):23-35.
[8] 石 磊, 龚 瑞, 尹争艳, 等. 肉牛传染性牛支原体肺炎流行的初步诊断[J]. 华中农业大学学报, 2008, 27(4):572.
SHI L, GONG R, YIN Z Y, et al. Preliminary diagnosis of cattle infectious Mycoplasma bovis pneumonia[J]. Journal of Huazhong Agricultural University, 2008, 27(4):572. (in Chinese)
[9] 辛九庆, 李 媛, 郭 丹, 等. 国内首次从患肺炎的犊牛肺脏中分离到牛支原体[J]. 中国预防兽医学报, 2008, 30(9):661-664.
XIN J Q, LI Y, GUO D, et al. First isolation of Mycoplasma bovis from calf lung with pneumoniae in China[J]. Chinese Journal of Preventive Veterinary Medicine, 2008, 30(9):661-664. (in Chinese)
[10] 冉智光, 谢建华, 骆 璐, 等. 我国部分地区牛支原体肺炎和关节炎的病原体诊断[J]. 中国预防兽医学报, 2010, 32(1):40-43.
RAN Z G, XIE J H, LUO L, et al. Etiological diagnosis of Mycoplasma bovis pneumonia and arthritis in some provinces in China[J]. Chinese Journal of Preventive Veterinary Medicine, 2010, 32(1):40-43. (in Chinese)
[11] 郭澍强, 蒋 万, 罗海峰, 等. 银川地区某奶牛场牛支原体病的诊断[J]. 中国预防兽医学报, 2015, 37(3):203-206.
GUO S Q, JIANG W, LUO H F, et al. Diagnosis of Mycoplasma bovis in a cattle farm of Yinchuan[J]. Chinese Journal of Preventive Veterinary Medicine, 2015, 37(3):203-206. (in Chinese)
[12] 高航飞. 牛支原体NM2012株生物学特性研究[D]. 呼和浩特:内蒙古农业大学, 2017.
GAO H F. Studies on the biological characteristics of Mycoplasma bovis NM2012 strain[D]. Hohhot:Inner Mongolia Agricultural University, 2017. (in Chinese)
[13] 姚永进, 剡根强, 王静梅, 等. 致犊牛肺炎和关节炎牛支原体新疆株的分离与鉴定[J]. 中国畜牧兽医, 2011, 38(12):76-79.
YAO Y J, YAN G Q, WANG J M, et al. Isolation and identification of Mycoplasma bovis Xinjiang strain inducing pneumonia and arthritis of calf[J]. China Animal Husbandry amp; Veterinary Medicine, 2011, 38(12):76-79. (in Chinese)
[14] 李 媛, 闫 磊, 刘 桐, 等. 中国西部和北部边境4省区牛支原体血清流行病学调查及分析[J]. 中国预防兽医学报, 2021, 43(7):717-721.
LI Y, YAN L, LIU T, et al. Seroprevalence and analysis of Mycoplasma bovis infection in four provinces of western and northern China[J]. Chinese Journal of Preventive Veterinary Medicine, 2021, 43(7):717-721. (in Chinese)
[15] NIU J Q, LI K W, PAN H C, et al. Epidemiological survey of Mycoplasma bovis in yaks on the Qinghai Tibetan Plateau, China[J]. Biomed Res Int, 2021, 2021:6646664.
[16] NIU J Q, WANG D J, YAN M S, et al. Isolation, identification and biological characteristics of Mycoplasma bovis in yaks[J]. Microb Pathog, 2021, 150:104691.
[17] 王冬经. 牦牛牛支原体病流行病学调查及其病原生物学特性研究[D]. 林芝:西藏农牧学院, 2020.
WANG D J. Epidemiological investigation of yak mycoplasma disease and study on the biological characteristics of its pathogen[D]. Linzhi:Xizang Institute of Agriculture and Animal Husbandry, 2020. (in Chinese)
[18] NIU J, YAN M, XU J, et al. The resistance mechanism of Mycoplasma bovis from Yaks in Tibet to fluoroquinolones and aminoglycosides[J]. Front Vet Sci, 2022, 9:840981.
[19] 严明帅. 牛支原体西藏分离株生物学特性研究[D]. 林芝:西藏农牧学院, 2021.
YAN M S. Study on biological characteristics of Mycoplasma bovis Xizang isolate[D]. Linzhi:Xizang Institute of Agriculture and Animal Husbandry, 2021. (in Chinese)
[20] 牛家强. 西藏牦牛源牛支原体生物学特性及感染兔肺脏转录组学研究[D]. 武汉:华中农业大学, 2021.
NIU J Q. Biological characteristics of isolated Mycoplasma bovis from yaks in Tibet and transcriptomics of lungs in infected rabbits[D]. Wuhan:Huazhong Agricultural University, 2021. (in Chinese)
[21] 徐晋花. 西藏牦牛源牛支原体比较基因组学及其致病性研究[D]. 林芝:西藏农牧学院, 2023.
XU J H. Comparative genomics and pathogenicity of Mycoplasma bovis isolated from yak in Tibet[D]. Linzhi:Xizang Institute of Agriculture and Animal Husbandry, 2023. (in Chinese)
[22] BESEMER J, LOMSADZE A, BORODOVSKY M. GeneMarkS:a self-training method for prediction of gene starts in microbial genomes. Implications for finding sequence motifs in regulatory regions[J]. Nucleic Acids Res, 2001, 29(12):2607-2618.
[23] SAHA S, BRIDGES S, MAGBANUA Z V, et al. Empirical comparison of ab initio repeat finding programs[J]. Nucleic Acids Res, 2008, 36(7):2284-2294.
[24] BENSON G. Tandem repeats finder:a program to analyze DNA sequences[J]. Nucleic Acids Res, 1999, 27(2):573-580.
[25] LOWE T M, EDDY S R. tRNAscan-SE:a program for improved detection of transfer RNA genes in genomic sequence[J]. Nucleic Acids Res, 1997, 25(5):955-964.
[26] LAGESEN K, HALLIN P, RDLAND E A, et al. RNAmmer:consistent and rapid annotation of ribosomal RNA genes[J]. Nucleic Acids Res, 2007, 35(9):3100-3108.
[27] HSIAO W, WAN I, JONES S J, et al. IslandPath:aiding detection of genomic islands in prokaryotes[J]. Bioinformatics, 2003, 19(3):418-420.
[28] ZHOU Y, LIANG Y J, LYNCH K H, et al. PHAST:a fast phage search tool[J]. Nucleic Acids Res, 2011, 39(Web server issue 2):W347-W532.
[29] ASHBURNER M, BALL C A, BLAKE J A, et al. Gene Ontology:tool for the unification of biology[J]. Nat Genet, 2000, 25(1):25-29.
[30] KANEHISA M, GOTO S, KAWASHIMA S, et al. The KEGG resource for deciphering the genome[J]. Nucleic Acids Res, 2004, 32(Datebase issue 1):D277-D280.
[31] GALPERIN M Y, MAKAROVA K S, WOLF Y I, et al. Expanded microbial genome coverage and improved protein family annotation in the COG database[J]. Nucleic Acids Res, 2015, 43(Datebase issue 1):D261-D269.
[32] SAIER M H Jr, REDDY V S, TAMANG D G, et al. The transporter classification database[J]. Nucleic Acids Res, 2014, 42(Datebase issue 1):D251-D258.
[33] CANTAREL B L, COUTINHO P M, RANCUREL C, et al. The carbohydrate-active EnZymes database (CAZy):an expert resource for Glycogenomics[J]. Nucleic Acids Res, 2009, 37(Datebase issue 1):D233-D238.
[34] URBAN M, PANT R, RAGHUNATH A, et al. The Pathogen-Host Interactions database (PHI-base):additions and future developments[J]. Nucleic Acids Res, 2015, 43(Datebase issue 1):D645-D655.
[35] CHEN L H, XIONG Z H, SUN L L, et al. VFDB 2012 update:toward the genetic diversity and molecular evolution of bacterial virulence factors[J]. Nucleic Acids Res, 2012, 40(Datebase issue 1):D641-D645.
[36] GRISSA I, VERGNAUD G, POURCEL C. CRISPRFinder:a web tool to identify clustered regularly interspaced short palindromic repeats[J]. Nucleic Acids Res, 2007, 35(Web server issue 2):W52-W57.
[37] KURTZ S, PHILLIPPY A, DELCHER A L, et al. Versatile and open software for comparing large genomes[J]. Genome Biol, 2004, 5(2):R12.
[38] HARRIS R S. Improved pairwise alignment of genomic DNA[D]. University Park:Pennsylvania State University, 2007.
[39] EMMS D M, KELLY S. OrthoFinder:phylogenetic orthology inference for comparative genomics[J]. Genome Biol, 2019, 20(1):238.
[40] LETUNIC I, BORK P. Interactive Tree Of Life (iTOL) v5:an online tool for phylogenetic tree display and annotation[J]. Nucleic Acids Res, 2021, 49(W1):W293-W296.
[41] WISE K S, CALCUTT M J, FOECKING M F, et al. Complete genome sequence of Mycoplasma bovis type strain PG45 (ATCC 25523)[J]. Infect Immun, 2011, 79(2):982-983.
[42] 王艳杰, 李平安, 吴太平, 等. 牛支原体分离株全基因组序列测定及初步分析[J]. 动物医学进展, 2014, 35(8):44-49.
WANG Y J, LI P A, WU T P, et al. Complete genome sequence and preliminary analysis of Mycoplasma bovis isolated strain[J]. Progress in Veterinary Medicine, 2014, 35(8):44-49. (in Chinese)
[43] CHEN S L, HAO H F, ZHAO P, et al. Complete genome sequence of Mycoplasma bovis strain 08M[J]. Genome Announc, 2017, 5(19):e00324-17.
[44] ROMERO-GARCA J, FRANCISCO C, BIARNS X, et al. Structure-function features of a Mycoplasma glycolipid synthase derived from structural data integration, molecular simulations, and mutational analysis[J]. PLoS One, 2013, 8(12):e81990.
[45] SIROVER M A. On the functional diversity of glyceraldehyde-3-phosphate dehydrogenase:biochemical mechanisms and regulatory control[J]. Biochim Biophys Acta (BBA)-Gen Subj, 2011, 1810(8):741-751.
[46] VAN DER MERWE J, PRYSLIAK T, GERDTS V, et al. Protein chimeras containing the Mycoplasma bovis GAPDH protein and bovine host-defence peptides retain the properties of the individual components[J]. Microb Pathog, 2011, 50(6):269-277.
[47] PEREZ-CASAL J, PRYSLIAK T. Detection of antibodies against the Mycoplasma bovis glyceraldehyde-3-phosphate dehydrogenase protein in beef cattle[J]. Microb Pathog, 2007, 43(5-6):189-197.
[48] ZHENG L, WHITE R H, CASH V L, et al. Mechanism for the desulfurization of L-cysteine catalyzed by the nifS gene product[J]. Biochemistry, 1994, 33(15):4714-4720.
[49] PFLOCK M, BATHON M, SCHR J, et al. The orphan response regulator HP1021 of Helicobacter pylori regulates transcription of a gene cluster presumably involved in acetone metabolism[J]. J Bacteriol, 2007, 189(6):2339-2349.
[50] DALLO S F, KANNAN T R, BLAYLOCK M W, et al. Elongation factor Tu and E1 β subunit of pyruvate dehydrogenase complex act as fibronectin binding proteins in Mycoplasma pneumoniae[J]. Mol Microbiol, 2002, 46(4):1041-1051.
[51] WANG H, LIU L, CAO Q, et al. Haemophilus parasuis α-2, 3-sialyltransferase-mediated lipooligosaccharide sialylation contributes to bacterial pathogenicity[J]. Virulence, 2018, 9(1):1247-1262.
[52] QI J J, GUO A Z, CUI P, et al. Comparative geno-plasticity analysis of Mycoplasma bovis HB0801 (Chinese Isolate)[J]. PLoS One, 2012, 7(5):e38239.
[53] LI Y, ZHENG H J, LIU Y, et al. The complete genome sequence of Mycoplasma bovis strain Hubei-1[J]. PLoS One, 2011, 6(6):e20999.
[54] 季文恒, 储岳峰, 赵 萍, 等. 牛支原体逃避宿主免疫的研究进展[J]. 畜牧兽医学报, 2017, 48(3):393-402.
JI W H, CHU Y F, ZHAO P, et al. Advances in the research of immune evasion by Mycoplasma bovis[J]. Acta Veterinaria et Zootechnica Sinica, 2017, 48(3):393-402. (in Chinese)
[55] MARIA-SOLANO M A, ROMERO-RIVERA A, OSUNA S. Exploring the reversal of enantioselectivity on a zinc-dependent alcohol dehydrogenase[J]. Org Biomol Chem, 2017, 15(19):4122-4129.
(编辑 "白永平)

