猪圆环病毒3型Rep蛋白的原核表达及酶活性分析

摘 要: Rep蛋白对猪圆环病毒(porcine circovirus,PCV)的复制具有重要作用。为分析PCV3 Rep蛋白的核酸内切酶活性、ATPase活性和解旋酶活性,作者使用镍亲和层析法纯化相应的重组蛋白,通过优化反应条件,建立Rep蛋白酶活性的测定方法,并探究Rep蛋白关键活性位点。结果表明,Rep蛋白在体外具有核酸内切酶活性、ATPase活性和解旋酶活性。当蛋白浓度为1.6 μmol·L-1,Mg2+浓度为1.0 mmol·L-1,37 ℃反应75 min时其ATPase活性达到最佳。当蛋白浓度为7 μmol·L-1,Mg2+浓度为12.5 mmol·L-1,37 ℃反应60 min时其解旋酶活性达到最佳。Y89为Rep蛋白核酸内切酶活性的关键位点,K173、D210、N250为ATPase活性和解旋酶活性的关键位点。本研究建立了PCV3 Rep蛋白酶活性分析的方法,初步揭示了该蛋白的主要功能位点,为Rep蛋白的功能研究以及基于Rep蛋白活性的抗病毒药物研究奠定了基础。
关键词: 猪圆环病毒3;Rep蛋白;核酸内切酶活性;ATPase活性;解旋酶活性
中图分类号:S852.659.2
文献标志码:A""" 文章编号:0366-6964(2024)05-2061-11
收稿日期:2023-07-20
基金项目:国家自然科学基金项目(31972658)
作者简介:韩 阳(1999-),女,安徽天长人,硕士生,主要从事猪圆环病毒3型蛋白功能的研究,E-mail:hanyang@webmail.hzau.edu.cn
*通信作者:宋云峰,主要从事动物病毒学以及动物传染病学研究,E-mail:syf@mail.hzau.edu.cn
Prokaryotic Expression and Protein Activity of Porcine Circovirus Type 3 Rep
HAN" Yang, GUAN" Shuaiyin, LI" Zhen, ZHOU Saisai, YUAN" Honggen, SONG" Yunfeng*
(College of Veterinary Medicine,Huazhong Agricultural University, Wuhan 430070, China)
Abstract:" Rep protein plays an important role in porcine circovirus replication. This study aimed to analyze the endonuclease activity,ATPase activity and helicase activity of porcine circovirus type 3(PCV3)Rep protein. The recombinant proteins were purified by Ni2+ affinity column chromatography. We established an enzymatic activity assay of Rep protein by optimizing the reaction conditions,and the key active site of Rep protein was explored. Our results confirmed that Rep has endonuclease activity、ATPase activity and helicase activity in vitro. Rep protein exhibited optimal ATPase activity in reaction with 1.6 μmol·L-1 Rep and 1 mmol·L-1 Mg2+ at 37 ℃ for 75 min. It exhibited optimal ATPase activity in reaction with 7 μmol·L-1 Rep and 12.5 mmol·L-1 Mg2+ at 37 ℃ for 75 min. Y89 of PCV3 Rep was essential for its endonuclease activity. And K173, D210, N250 sites were essential for its ATPase and helicase activities. In this paper, we developed a method for analysis of the enzyme activity and preliminarily revealed the main functional sites of PCV3 Rep protein,which laid a foundation for the functional study of PCV3 Rep protein and the development of antiviral drugs based on the activity of Rep protein.
Key words: porcine circovirus type 3; Rep; endonuclease activity; ATPase activity; helicase activity
*Corresponding author:" SONG Yunfeng,E-mail:syf@mail.hzau.edu.cn
猪圆环病毒3型(porcine circovirus type 3,PCV3)属于圆环病毒科圆环病毒属,于2016年从具有猪皮炎肾病综合征的患病母猪和其流产胎儿中首次发现[1]。有研究表明,PCV3感染主要与呼吸系统疾病、生殖系统疾病、胃肠道疾病、神经系统疾病以及多系统炎症有关[2]。临床上,PCV3与猪圆环病毒2型(PCV2)、猪繁殖与呼吸综合征病毒(PRRSV)、猪流行性腹泻病毒(PEDV)和猪丁型冠状病毒(PDCoV)存在混合感染[3-4]。PCV3可在全球范围内传播,在欧洲[5]、亚洲[6]、北美洲[1]、南美洲[7]均有报道。我国已有超过20个省份报道存在PCV3感染的情况,且阳性率呈不断上升的趋势,多以继发混合感染为主[8-9]。目前,尚无PCV3的商品化疫苗,导致临床防控PCV3的难度较大,病毒的蔓延呈逐年上涨趋势,给世界养猪业造成了潜在的威胁。
PCV3为单股环状DNA病毒,基因组长约2 000 bp,主要包含3个开放阅读框(open reading frames,ORFs),其中ORF1是圆环病毒基因组最保守的区域,编码复制起始的Rep和Rep’蛋白[1]。Rep蛋白的N端包含病毒基因组滚环复制过程相关的标志性氨基酸基序即RC-Ⅰ(FTLN)、RC-Ⅱ(HLQ)、RC-Ⅲ(YxxK)。FTLN具有结构作用并可能参与单链DNA(ssDNA)的结合;HLQ的作用是协调二价离子;YxxK具有核酸内切酶活性,可在复制起始区九核苷酸序列引入一切口并共价结合到DNA的5′端[10-11]。有研究证实,PCV1的Rep蛋白具有核酸内切酶活性和ATPase活性,能够切割并连接病毒ssDNA并在病毒复制的起始和终止过程中发挥重要作用[12-13]。在PCV2中Rep蛋白被证实具有良好的核酸内切酶活性、ATPase活性和解旋酶活性[14-16]。而关于PCV3 Rep蛋白酶活性的研究目前尚未报道,PCV3与PCV1和PCV2 Rep蛋白的氨基酸相似性均低于50%[17],这是否会引起PCV3 Rep蛋白活性的差异也尚不清楚。因此,本研究表达并纯化了PCV3 Rep蛋白,通过分析该蛋白核酸内切酶活性、ATPase活性和解旋酶活性,以期为PCV3 Rep蛋白功能的研究提供参考。
1 材料与方法
1.1 材料
PCV3阳性病料为本实验室PCR检测为阳性并保存。pET-28a载体、pcold-TF载体、TF蛋白由本实验室保存。DNA提取试剂盒购自上海惠凌生物技术有限公司、2×F8 FastLong PCR MasterMix、质粒小量提取试剂盒购自北京艾德莱生物科技有限公司。大肠杆菌感受态细胞DH5α与BL21(DE3)购自北京擎科生物科技有限公司。限制性内切酶BamHⅠ、NdeⅠ、SalⅠ、T4 DNA 连接酶、核酸Marker购自大连宝生物公司,核酸荧光染料购自TOYOBO公司。氨苄青霉素(Amp)、卡那霉素(Kana)、异丙基-β-D-硫代半乳糖苷(IPTG)购自Biosharp公司,琼脂糖、胰蛋白胨、酵母浸出物、十二烷基硫酸钠、咪唑、乙二胺四乙酸(EDTA)等试剂购自国药集团化学试剂有限公司,Ni Sepharose 6 Fast Flow购自GE Healthcare。His标签单克隆抗体、鼠源IgG购自武汉三鹰生物技术有限公司。
1.2 重组质粒及其突变体的构建及鉴定
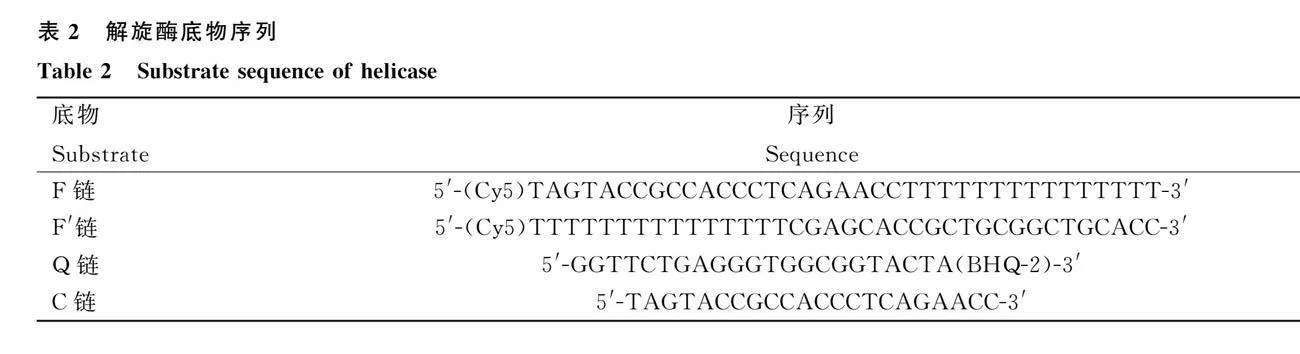
以临床PCV3阳性病料DNA为模板,参考PCV3 KH25/2017毒株(GenBank登录号:MK934766)的ORF1序列,采用表1中的引物对rep全长基因及截短基因repN(1-101 aa)进行PCR扩增。反应体系:25 μL 2×F8 Mix,上下游引物各2 μL,模板2 μL,19 μL ddH2O。PCR反应条件:95℃ 5min;95℃ 30 s,58℃ 30 s,72℃ 15 s,共35个循环;72℃ 5 min。PCR产物用1%琼脂糖凝胶电泳检测后进行胶回收。将rep全长基因和pcold-TF载体经BamHⅠ、SalⅠ双酶切后进行连接,构建原核表达质粒pcoldTF-Rep。截短基因repN和pET-28a载体经NdeⅠ、BamHⅠ双酶切后进行连接,构建原核表达质粒pET28a-RepN。利用repN基因的上游引物pET28a-RepN-F和突变引物RepN Y89F-R扩增repN基因点突变序列,经NdeⅠ、BamHⅠ双酶切后连接到pET-28a载体,命名为pET28a-RepN Y89F。再以pcoldTF-Rep为模板,通过Overlap PCR技术分别扩增rep基因K173A、D210A、N250A突变体全长。将突变的全长片段经BamHⅠ、SalⅠ双酶切后连接到pcold-TF载体上,分别命名为pcoldTF-Rep K173A、pcoldTF-Rep D210A、pcoldTF-Rep N250A。以上质粒经酶切鉴定正确后送至北京擎科生物技术有限公司进行测序。
1.3 重组蛋白的原核表达与纯化
将重组质粒转化BL21(DE3)感受态细胞,挑取单菌落于含有抗性的LB液体培养基中37℃培养过夜。按照1∶100的比例接种菌液到LB液体培养基中,37℃、180 r·min-1摇床振荡培养至菌液OD600 nm为0.6~0.8时加入终浓度为1 mmol·L-1的IPTG,于16℃,180 r·min-1摇床上培养16 h。收集菌体超声破碎,离心后取上清过亲和层析柱,250 mmol·L-1咪唑洗脱目的蛋白,使用超滤管浓缩后进行SDS-PAGE电泳,电泳结束后将蛋白转印至NC膜上,以His标签抗体作为一抗,HRP标记的羊抗鼠IgG作为二抗,采用Western blot鉴定获取目的蛋白信号。
1.4 Rep蛋白核酸内切酶活性检测
1.4.1 核酸内切酶活性反应体系的建立
在北京擎科生物技术有限公司合成含有PCV3茎环结构保守的ssDNA底物P15(TAGTATT^ACCCGGCA),切割位点用^表示。蛋白切割底物后,与切割位点后8个核苷酸的5′磷酸二酯键共价结合,导致在变性凝胶中电泳迁移率变慢。将RepN蛋白与P15进行切割试验,分析核酸内切酶活性。反应体系中含有20 μmol·L-1蛋白,5 μmol·L-1 P15,20 mmol·L-1 Tris-HCl(pH 8.0),0.1 mol·L-1 NaCl,2.5 mmol·L-1 MgCl2或MnCl2。37℃孵育30 min后加入5×Loading buffer终止反应,95℃煮沸10 min,通过SDS-PAGE电泳及考马斯亮蓝染色观察切割效果。
1.4.2 底物浓度对核酸内切酶活性的影响
将底物P15稀释至终浓度为:0、1、2、3、4、5、6 μmol·L-1,37℃孵育30 min,SDS-PAGE电泳后进行考马斯亮蓝染色。
1.4.3 时间对核酸内切酶活性的影响
分别在0、15、30、45、60、75、90 min时向反应体系中加入5×Loading buffer终止反应,进行SDS-PAGE电泳并用考马斯亮蓝染色。
1.4.4 二价金属离子及其浓度对核酸内切酶活性的影响
有研究表明,内切酶依赖于金属离子Mg2+,Mn2+的存在,故本试验探究Mg2+或Mn2+的参与对核酸内切酶活性的影响,于ssDNA底物之前加入30 mmol·L-1 EDTA以消除二价金属离子的干扰。向反应体系中分别加入0、0.25、0.5、1.0、1.5、2.0、2.5、5 mmol·L-1的Mg2+或Mn2+,37℃反应30 min,SDS-PAGE电泳后进行考马斯亮蓝染色。
1.4.5 底物类型对核酸内切酶活性的影响
对底物P15的核苷酸进行突变,依次与RepN蛋白进行切割反应。37℃反应30 min,SDS-PAGE电泳后进行考马斯亮蓝染色。
1.4.6 核酸内切酶活性位点的探究
对Rep蛋白核酸内切酶活性的关键氨基酸进行突变,纯化突变体蛋白。在切割反应体系中分别加入RepN和RepN Y89F蛋白,37℃条件下孵育30 min,SDS-PAGE电泳后进行考马斯亮蓝染色。
1.5 Rep蛋白ATPase活性检测
1.5.1 ATPase活性反应体系的建立
本试验使用Kinase Glo Luminescent Kinase Assay Platform试剂盒测定Rep蛋白的ATPase活性。该试剂盒采用化学发光的方法,通过定量激酶反应后检测体系中剩余ATP的含量来反映目的蛋白的活性,所检测的发光信号与ATP含量和蛋白活性成负相关。反应体系如下:20 mmol·L-1 Tris-HCl(pH 8.0),50 mmol·L-1 NaCl,2 mmol·L-1 MgCl2,100 μmol·L-1 ATP以及1 μmol·L-1的蛋白。
1.5.2 Rep蛋白浓度对ATPase活性的影响
在反应体系中加入0、0.2、0.4、0.6、0.8、1.0、1.2、1.4、1.6、1.8、2.0 μmol·L-1的Rep蛋白和TF标签蛋白,37℃反应60 min后,加入等体积的化学发光试剂,震荡混匀,多功能酶标仪检测发光值。
1.5.3 时间对ATPase活性的影响
确定最佳蛋白浓度后,将反应体系置于37℃分别孵育0、15、30、45、60、75、90 min后检测化学发光值。
1.5.4 二价金属离子及其浓度对ATPase活性的影响
向最佳蛋白浓度的反应体系中分别加入2 mmol·L-1的二价金属离子(Mg2+、Mn2+、Ca2+、Cu2+、Zn2+),37℃孵育最佳时间后测定化学发光值。
将Mg2+ 梯度稀释至0、0.1、0.3、0.5、0.7、1.0、1.5、2、3、4、5 mmol·L-1 ,相同条件下测定化学发光值。
1.5.5 ATPase活性位点的探究
在ATP水解反应体系中分别加入最佳浓度的Rep蛋白,以及突变体蛋白K173A、D210A、N250A,以不加蛋白的样品为空白对照,37℃孵育最佳时间,检测化学发光值。
1.6 Rep蛋白解旋酶活性检测
1.6.1 解旋酶活性反应体系的建立
本研究采用荧光共振能量转移(fluorescence resonance energy transfer, FRET)的方法检测蛋白的解旋酶活性。解旋酶底物由上海生工生物工程公司合成(表2),将荧光底物和淬灭底物以1∶1.5比例混合,设置双链退火程序:95 ℃ 20 min,60 ℃ 10 min,37 ℃ 10 min,20 ℃ 10 min,通过缓慢降温的方式使其形成双链DNA底物。解旋反应体系:200 nmol·L-1荧光底物(F或F′链)、240 nmol·L-1 淬灭底物(Q链)、3.6 μmol·L-1捕获链(C链)、10 mmol·L-1 Mg2+、0.5 mmol·L-1 ATP和5 μmol·L-1蛋白,混匀后用多功能酶标仪检测荧光值。荧光值越高,表明解旋酶活性越好。
1.6.2 Rep蛋白解旋方向的判定
将合成的3′端突出荧光底物F链和5′端突出荧光底物F′链,分别与Q链退火形成双链,于384孔配制解旋反应体系,进行解旋活性的测定,判断其解旋活性。
1.6.3 Rep蛋白浓度对解旋酶活性的影响
确定Rep蛋白解旋方向后,选择相应的荧光底物进行后续试验。设置Rep蛋白试验组和TF标签蛋白对照组,进行0、1、2、3、4、5、6、7、8 μmol·L-1的蛋白浓度梯度试验,37 ℃孵育60 min,检测荧光值。
1.6.4 时间对解旋酶活性的影响
选择最佳蛋白浓度,配制反应体系,37℃条件下进行解旋反应,每15 min检测一次荧光值,观察荧光值变化趋势。
1.6.5 二价金属离子及其浓度对解旋酶活性的影响
在解旋反应体系中加入最佳浓度的Rep蛋白,依次与10 mmol·L-1的二价金属离子(Mg2+、Mn2+、Ca2+、Cu2+、Zn2+)于37℃反应最佳时间后检测荧光值。
将Mg2+ 浓度稀释至0、2.5、5、7.5、10、12.5、15、17.5、20 mmol·L-1 ,相同条件下测定荧光值。
1.6.6 解旋酶活性位点的探究
在最佳解旋反应体系下,检测野生型Rep蛋白与其突变体K173A、D210A、N250A蛋白解旋活性的差异,同时设置无蛋白对照组,37℃条件下孵育最佳时间后检测荧光值。
2 结 果
2.1 重组质粒及其突变体的构建及鉴定
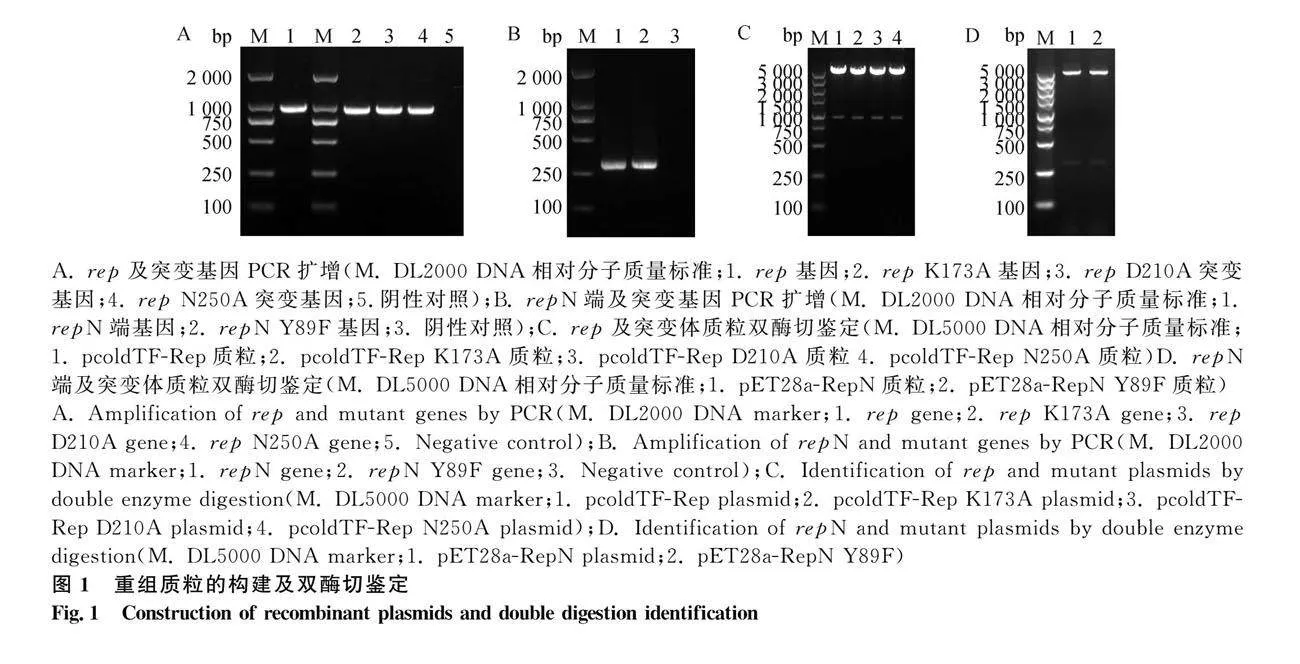
利用特异性引物扩增PCV3 rep全长及其截短基因repN,PCR产物经1%琼脂糖凝胶电泳,分别可观察到约891 bp(图1A)和303 bp(图1B)的特异性条带,与预期片段大小一致。将rep基因构建到pcold-TF载体,repN基因构建到pET-28a载体。选择Rep蛋白第173位赖氨酸(Lys)、第210位天冬氨酸(Asp)、第250位天冬酰胺(Asn)进行定点突变,以rep基因为模板进行Overlap PCR扩增,获得突变后rep基因全长,大小为891 bp(图1A)。将三个突变后的rep基因连接至表达载体pcold-TF。选择RepN蛋白第89位酪氨酸(Tyr)进行突变,利用引物pET28a-RepN-F和突变引物repN Y89F-R扩增截短基因repN点突变序列(图1B),连接至pET-28a载体。重组质粒双酶切后得到的两条片段大小均符合预期(图1C、D),进一步通过测序证实插入基因的位置、方向以及读码框均正确,突变体也均构建成功。
2.2 重组蛋白的原核表达与纯化
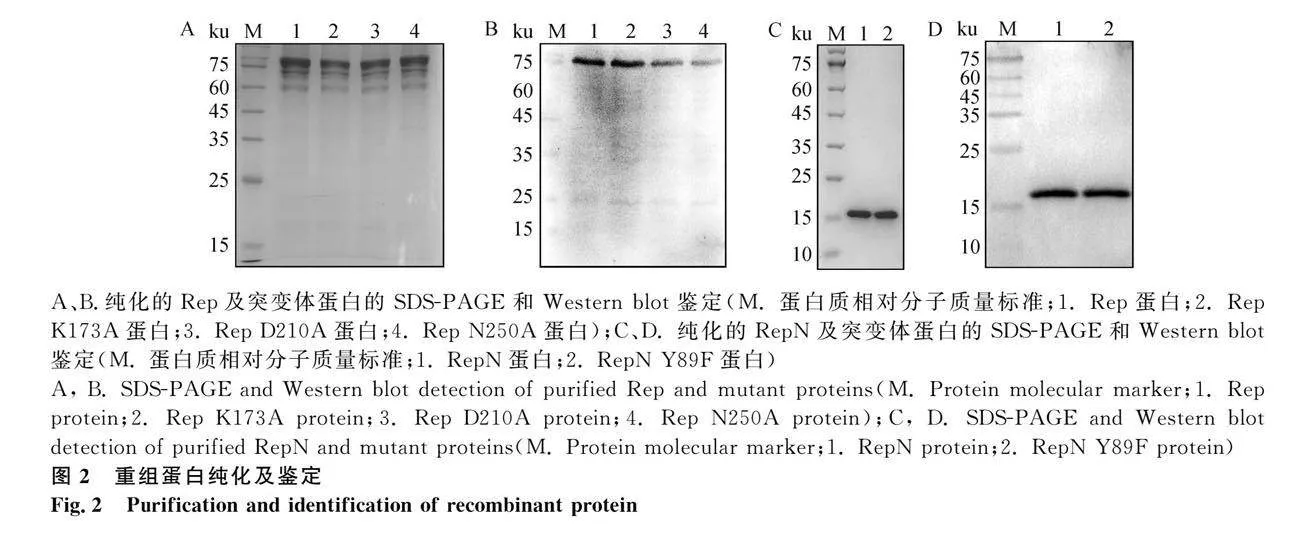
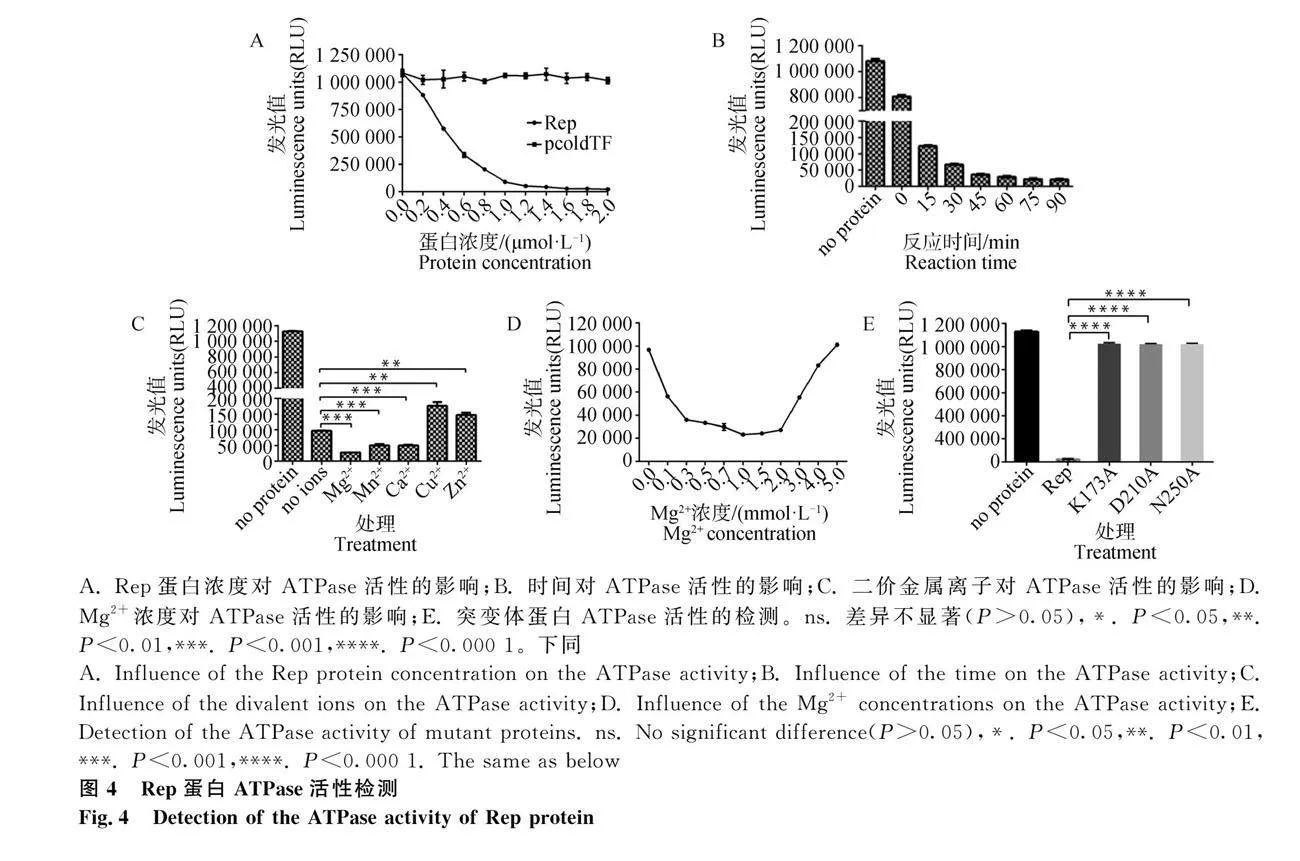
将鉴定正确的重组质粒转化BL21(DE3),对成功表达在上清的重组蛋白经镍层析柱进行纯化。利用SDS-PAGE及Western blot检测纯化后的蛋白,结果显示,目的蛋白与His标签抗体有良好的结合性,Rep、Rep K173A、Rep D210A、Rep N250A蛋白大小约为86.6 ku(图2A、2B),目的蛋白RepN、RepN Y89F大小约为15.9 ku(图2C、D),均与预期蛋白大小相符,表明成功纯化得到目的蛋白。
2.3 Rep蛋白核酸内切酶活性检测
2.3.1 底物浓度对核酸内切酶活性的影响
底物浓度对Rep蛋白核酸内切酶活性的影响见图3A。结果显示,随着底物浓度的增加,形成的共价化合物增多,其核酸内切酶活性升高,说明Rep蛋白以底物浓度依赖的方式进行切割。在底物浓度为6 μmol·L-1时,20 μmol·L-1的Rep蛋白能够发挥最佳的核酸内切酶活性。
2.3.2 时间对核酸内切酶活性的影响
不同时间对Rep蛋白核酸内切酶活性的影响见图3B。结果发现,Rep在37℃反应15min即可完成对底物的切割,之后再增加反应时间对其核酸内切酶活性效率影响不明显。
2.3.3 二价金属离子及其浓度对核酸内切酶活性的影响
如图3C、D所示,随着Mg2+和Mn2+浓度升高,Rep蛋白核酸内切酶活性不变。加入离子螯合剂EDTA时,核酸内切酶活性消失,表明Mg2+和Mn2+的存在对Rep蛋白发挥核酸内切酶活性具有决定作用,但浓度对其影响不大。
2.3.4 底物序列对核酸内切酶活性的影响
底物序列对Rep蛋白发挥核酸内切酶活性的影响如图3E所示,Rep蛋白不能切割P15-9,而其余位点不影响Rep蛋白的切割活性。结果表明Rep蛋白识别的底物序列为5′-TAGTATTAC-3′,其中C9是关键位点。
2.3.5 核酸内切酶活性位点的探究
将Rep蛋白和突变体Y89F蛋白进行核酸内切酶活性验证,如图3F所示,Y89F突变体不切割底物P15,不具有核酸内切酶活性。上述结果表明Y89位点对Rep蛋白的核酸内切酶活性具有重要意义。
2.4 Rep蛋白ATPase活性检测
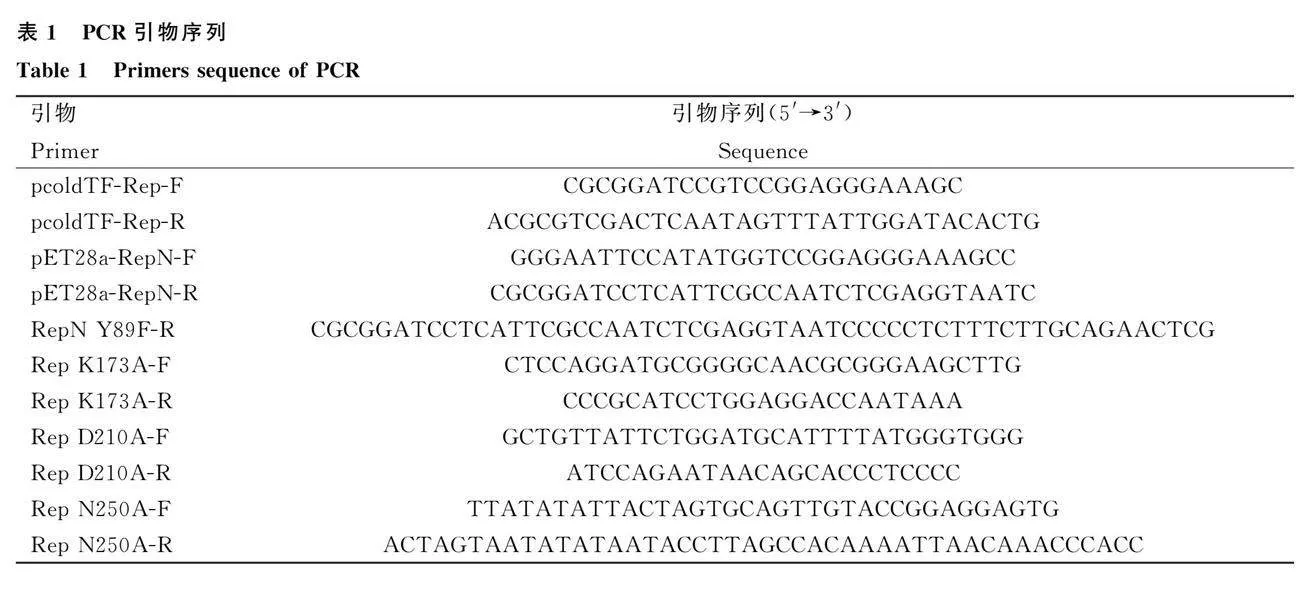
2.4.1 Rep蛋白浓度对ATPase活性的影响
Rep蛋白浓度与ATP水解反应之间的关系如图4A所示,不同浓度的TF蛋白组发光值均和无蛋白组接近,ATP未被水解。而Rep蛋白组具有较好的ATPase活性,且随着蛋白浓度升高,ATP的水解量逐渐增加。当蛋白浓度到达1.6 μmol·L-1时,发光值趋于稳定,说明ATP基本水解完全。因此,确定Rep蛋白ATPase活性最佳的蛋白浓度为1.6 μmol·L-1。
2.4.2 时间对ATPase活性的影响
不同时间对Rep蛋白ATPase活性的影响见图3B。结果表明,随着反应的进行,ATP逐渐水解完全。在0~60 min发光值下降趋势明显,75 min之后趋于稳定。因此,选择75 min作为Rep蛋白ATPase活性测定的最佳反应时间。
2.4.3 二价金属离子及其浓度对ATPase活性的影响
二价金属离子对Rep蛋白ATPase 活性的影响如图4C所示,试验组与不加金属离子的对照组相比差异显著。Mg2+、Mn2+、Ca2+对Rep蛋白ATPase活性有促进作用,Cu2+和Zn2+对其则产生抑制作用。Rep蛋白对二价金属离子的利用率由高到低依次为Mg2+、Ca2+、Mn2+、Zn2+、Cu2+。
Mg2+是细胞中ATPase广泛利用的离子,而ATPase活性也严格依赖Mg2+,作者进一步探究Mg2+对Rep蛋白ATPase活性的影响。结果显示,Mg2+浓度在0~1.0 mmol·L-1对ATPase活性的促进作用增强,且在1.0 mmol·L-1时促进作用达到峰值。当Mg2+浓度高于1.0 mmol·L-1之后开始出现抑制作用(图4D)。因此,能使Rep蛋白活性达到最佳的Mg2+反应浓度为1.0 mmol·L-1。
2.4.4 ATPase活性位点的探究
为探究Rep蛋白ATPase活性的关键位点,在相同条件下检测野生型蛋白和突变体蛋白的ATP水解能力,结果发现加入突变体蛋白K173A、D210A、N250A的试验组与未加蛋白的对照组发光值相近,ATP均未被水解。经t检验分析,Plt;0.000 1,差异显著,说明突变体蛋白丧失了ATPase活性(图4E),因此K173A、D210A、N250A为Rep蛋白ATPase活性的关键位点。
2.5 Rep蛋白解旋酶活性检测
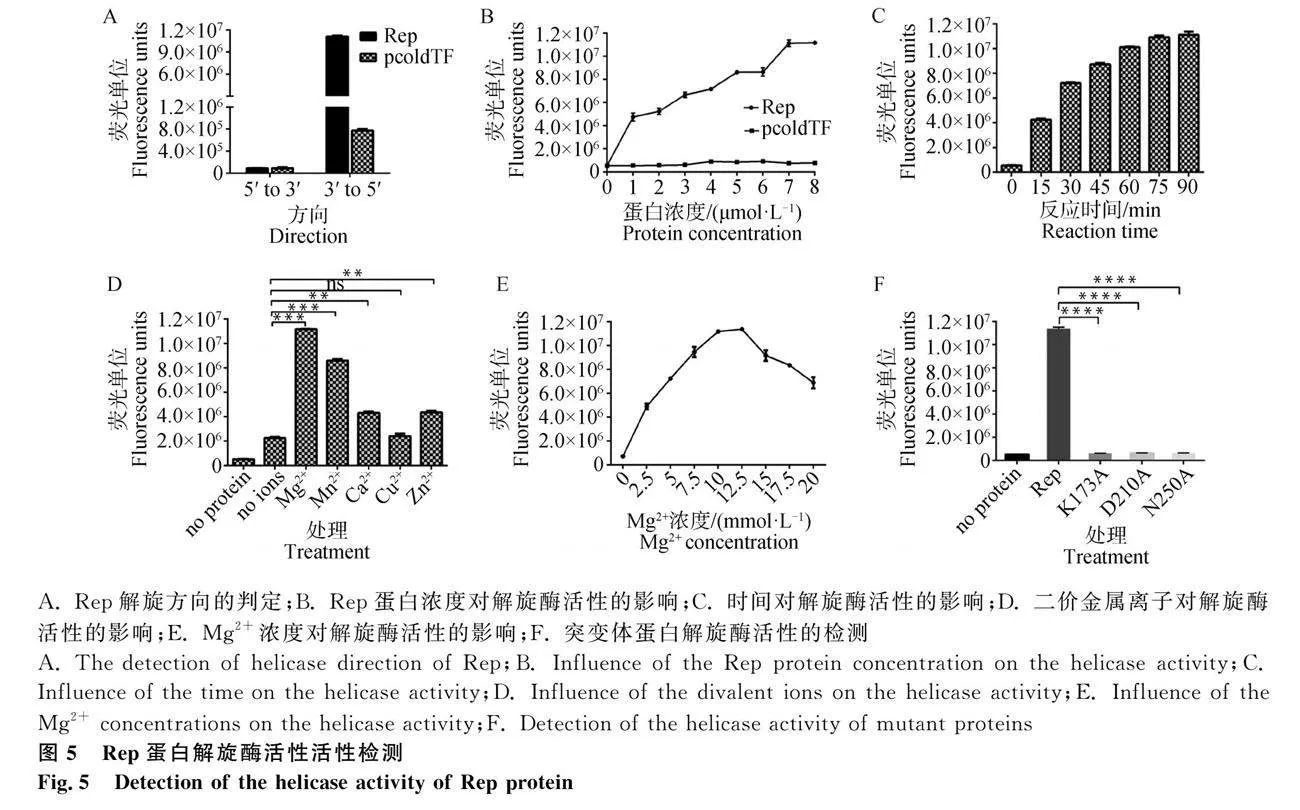
2.5.1 Rep解旋方向的判定
为研究Rep蛋白是否具有解旋方向性,分别用3′端突出荧光底物和5′端突出荧光底物检测其解旋活性。上述结果显示,Rep蛋白对3′端突出荧光底物具有解旋活性,而对5′端突出荧光底物则不具有解旋活性(图5A)。TF蛋白作为对照组,对3′和5′端突出荧光底物均不存在解旋活性。由此可判定Rep蛋白具有3′至5′的解旋方向性。
2.5.2 Rep蛋白浓度对解旋酶活性的影响
Rep蛋白浓度对解旋酶活性的影响如图5B所示。TF蛋白组的荧光值较低,与未加蛋白时数值相近,且随着浓度升高,其荧光值几乎不变,说明TF蛋白不具有解旋活性。Rep蛋白解旋反应速率随蛋白浓度增加而增强,在蛋白浓度达到7 μmol·L-1后其解旋活性增长缓慢,故确定最佳蛋白浓度为7 μmol·L-1。
2.5.3 时间对解旋酶活性的影响
在解旋反应体系中加入ATP起始反应,在0~90 min每间隔15 min检测一次荧光值,绘制解旋反应时间曲线。结果如图5C所示,在前60 min内,荧光值增长较快,60 min后荧光值趋于稳定。因此,确定Rep蛋白解旋酶活性测定的最佳反应时间为60 min。
2.5.4 二价金属离子及其浓度对解旋酶活性的影响
以3′端突出为解旋底物,研究二价金属离子对于解旋酶活性的影响。试验结果如图5D所示,Mg2+、Mn2+、Ca2+、Zn2+组的荧光值显著高于无金属离子组,Cu2+组与无金属离子组差异不显著。整体而言,Ca2+、Zn2+对Rep蛋白解旋活性存在微弱的促进作用,Mg2+、Mn2+对Rep蛋白的解旋活性促进效果明显,并且在加入Mg2+时Rep蛋白解旋能力最强。
为进一步确认Mg2+对Rep蛋白的解旋功能的影响,作者设置了Mg2+浓度梯度。结果显示,Rep蛋白在Mg2+浓度为12.5 mmol·L-1时,其解旋酶活性达到最高,继续增加Mg2+浓度则对其解旋酶活性产生一定的抑制作用(图5E)。
2.5.5 解旋酶活性位点的探究
在解旋反应体系中分别加入7 μmol·L-1的Rep蛋白和突变体蛋白K173A、D210A、N250A,于37℃条件下反应60 min。结果如图5F所示,与野生型蛋白相比,三种突变蛋白的解旋活性均显著性降低,表明K173、D210、N250是Rep蛋白的关键活性位点。
3 讨 论
Rep蛋白是参与病毒滚环复制的关键蛋白,通过识别并切割病毒基因组的茎环结构起始病毒的复制,其N端是一个内切酶结构域,负责ssDNA的切割和连接;中心是一个低聚结构域,负责六聚体化;在C端是一个ATP酶结构域,通过水解ATP供能,解开双链DNA(dsDNA)[18]。Mankertz和Hillenbrand[19]发现PCV1 Rep蛋白保守基序中的位点发生突变可有效抑制病毒的复制。在PCV2中,Rep蛋白与宿主蛋白PARP1相互作用促进了病毒基因组的复制,当该蛋白因自然突变导致C端截短后能够降低病毒的复制和感染性[20-21]。PCV3 Rep蛋白通过上调IFN-β和IFN-γ的表达,调控宿主先天免疫反应[22]。可见Rep蛋白在病毒复制和免疫逃逸中发挥重要作用,但目前对于PCV3 Rep蛋白的研究相对有限。本研究采用原核表达系统制备获得了可溶性的PCV3 Rep重组蛋白,并在体外验证了核酸内切酶活性、ATPase活性和解旋酶活性,为抗病毒药物的筛选提供了理想靶标。
连凯琪等[23]使用原核表达系统表达的PCV3 Rep蛋白主要以包涵体形式存在,不利于蛋白纯化。本研究通过截短获得了可溶性的Rep蛋白N端蛋白(1—101 aa),使用TF(trigger factor)标签获得了可溶性的Rep全长蛋白,降低了蛋白的纯化难度。TF是一种原核的核糖体结合伴侣蛋白,能够促进新生肽链的共翻译折叠,具有良好的促溶效果[24-25]。考虑到标签蛋白较大,对目的蛋白的活性研究可能存在影响,本试验设置了TF蛋白对照组,检测并验证了TF标签蛋白不具有ATPase活性和解旋酶活性。
PCV1和PCV2 Rep蛋白在金属离子的存在下具有良好的核酸内切酶活性,在复制起始区处具有切割及连接作用[12-15]。本试验使用带TF标签的Rep全长蛋白未检测到明显的核酸内切酶活性,因此截短了Rep N端蛋白进行核酸内切酶活性分析。研究结果发现,PCV3 Rep蛋白在体外也具有核酸内切酶活性,表明该蛋白能作为滚环复制的启动蛋白发挥作用并参与PCV3基因组的复制起始。核酸内切酶活性严格依赖金属离子的存在,PCV2 Rep蛋白在Mn2+的作用下活性最佳,然而在PCV3和PCV1 Rep蛋白中Mg2+和Mn2+对该活性的影响差异不大[13-14]。在PCV1和PCV2中均证实了Rep蛋白通过RC-III基序中酪氨酸羟基的亲核攻击作用完成对复制起始区九聚核苷酸序列[5′-(T/A)AGTATTAC-3′]的切割[12,18]。本研究构建了PCV3 RepN Y89F突变体,第89位酪氨酸突变后核酸内切酶活性丧失,这证实PCV3 Rep蛋白RC-III中的酪氨酸是核酸内切酶活性的关键残基。
解旋酶(helicase)是一种能够水解双链DNA和RNA底物的蛋白酶,同时也是核酸依赖的ATP水解酶[26]。Shu等[27]发现盐酸胍能够抑制埃博拉病毒VP35蛋白的NTPase活性和解旋酶活性,从而抑制埃博拉病毒(Ebola virus,EV)的复制和转录。本实验室前期筛选了NS3解旋酶抑制剂,有效抑制了日本脑炎病毒(Japanese encephalitis virus,JEV)的复制[28],证明解旋酶对病毒复制有重要作用。周旋[29]发现PCV2 Rep蛋白在体外具有ATPase活性和解旋酶活性,其ATPase活性的最佳金属离子为2.5 mmol·L-1的Mg2+,解旋酶活性的最佳金属离子为15 mmol·L-1的Mn2+。本试验通过优化条件发现,PCV3 Rep蛋白在蛋白浓度为1.6 μmol·L-1,Mg2+浓度为1 mmol·L-1,37℃反应75 min时其ATPase活性达到最佳。进一步检测PCV3 Rep蛋白的解旋活性,结果显示Rep蛋白能够以dsDNA为底物进行3′端至5′端的解旋反应,其最适蛋白浓度为7 μmol·L-1,最佳金属离子为12.5 mmol·L-1的Mg2+,最适反应时间为60 min。对比发现PCV3 Rep蛋白与PCV2 Rep蛋白的解旋方向是一致的,但最佳反应条件存在差异[28]。本研究在探究Mg2+浓度对蛋白ATPase活性和解旋酶活性的影响时,发现过高的Mg2+浓度会在一定程度上抑制Rep蛋白的活性,这一结果与王小童[30]对呼吸道合胞病毒P蛋白解旋活性的测定以及Shu等[27]对埃博拉病毒VP35蛋白ATPase活性测定的趋势相似,说明Mg2+需要在合适的浓度范围下才能最好的发挥作用。Tarasova等[18]研究证明,PCV2 Rep突变体的ATPase活性和解旋酶活性,相比野生型Rep均明显减弱。本试验通过序列比对,将PCV3 Rep的以上位点进行突变,得到K173A、D210A、N250A突变体蛋白,并对其酶活性进行研究。结果显示,PCV3 Rep突变体的ATPase活性和解旋酶活性也均受到抑制,这表明PCV2和PCV3 Rep蛋白的氨基酸同源性虽然较低,但是关键酶活位点是保守的[17-18]。因此,作者推测PCV3 Rep蛋白能够为抑制剂的筛选提供试验基础。
4 结 论
本研究表达并纯化出PCV3 Rep蛋白,首次验证了PCV3 Rep蛋白的核酸内切酶活性、ATPase活性和解旋酶活性,建立了Rep蛋白酶活性的方法并且找到了对应酶活性的关键位点,为PCV3复制机制提供了参考,为抗病毒药物的研发提供了靶标。
参考文献(References):
[1] PALINSKI R, PIEYRO P, SHANG P C, et al. A novel porcine circovirus distantly related to known circoviruses is associated with porcine dermatitis and nephropathy syndrome and reproductive failure[J]. J Virol, 2017, 91(1):e01879-16.
[2] KROEGER M, TEMEEYASEN G, PIEYRO P E. Five years of porcine circovirus 3:what have we learned about the clinical disease, immune pathogenesis, and diagnosis[J]. Virus Res, 2022, 314:198764.
[3] CHEN G H, MAI K J, ZHOU L, et al. Detection and genome sequencing of porcine circovirus 3 in neonatal pigs with congenital tremors in South China[J]. Transbound Emerg Dis, 2017, 64(6):1650-1654.
[4] NGUYEN N H, DO TIEN D, NGUYEN T Q, et al. Identification and whole-genome characterization of a novel Porcine circovirus 3 subtype b strain from swine populations in Vietnam[J]. Virus Genes, 2021, 57(4):385-389.
[5] FRANZO G, CORTEY M, DE CASTRO A M M G, et al. Genetic characterisation of Porcine circovirus type 2 (PCV2) strains from feral pigs in the Brazilian Pantanal:an opportunity to reconstruct the history of PCV2 evolution[J]. Vet Microbiol, 2015, 178(1-2):158-162.
[6] SHEN H, LIU X, ZHANG P, et al. Genome characterization of a porcine circovirus type 3 in South China[J]. Transbound Emerg Dis, 2018, 65(1):264-266.
[7] TOCHETTO C, LIMA D A, VARELA A P M, et al. Full-Genome Sequence of Porcine Circovirus type 3 recovered from serum of sows with stillbirths in Brazil[J]. Transbound Emerg Dis, 2018, 65(1):5-9.
[8] QI S S, SU M J, GUO D H, et al. Molecular detection and phylogenetic analysis of porcine circovirus type 3 in 21 Provinces of China during 2015-2017[J]. Transbound Emerg Dis, 2019, 66(2):1004-1015.
[9] CHEN S, ZHANG L Y, LI X, et al. Recent progress on epidemiology and pathobiology of porcine circovirus 3[J]. Viruses, 2021, 13(10):1944.
[10] CHEUNG A K. Porcine circovirus:transcription and DNA replication[J]. Virus Res, 2012, 164(1-2):46-53.
[11] WAWRZYNIAK P, PUCIENNICZAK G, BARTOSIK D. The different faces of rolling-circle replication and its multifunctional initiator proteins[J]. Front Microbiol, 2017, 8:2353.
[12] STEINFELDT T, FINSTERBUSCH T, MANKERTZ A. Functional analysis of cis- and trans-acting replication factors of porcine circovirus type 1[J]. J Virol, 2007, 81(11):5696-5704.
[13] STEINFELDT T, FINSTERBUSCH T, MANKERTZ A. Demonstration of nicking/joining activity at the origin of DNA replication associated with the rep and rep’ proteins of porcine circovirus type 1[J]. J Virol, 2006, 80(13):6225-6234.
[14] VEGA-ROCHA S, BYEON I J L, GRONENBORN B, et al. Solution structure, divalent metal and DNA binding of the endonuclease domain from the replication initiation protein from porcine circovirus 2[J]. J Mol Biol, 2007, 367(2):473-487.
[15] TOMPKINS K J, HOUTTI M, LITZAU L A, et al. Molecular underpinnings of ssDNA specificity by Rep HUH-endonucleases and implications for HUH-tag multiplexing and engineering[J]. Nucleic Acids Res, 2021, 49(2):1046-1064.
[16] LUO G, ZHU X F, LV Y, et al. Crystal structure of the dimerized N terminus of porcine circovirus type 2 replicase protein reveals a novel antiviral interface[J]. J Virol, 2018, 92(18):e00724-18.
[17] OUYANG T, NIU G Y, LIU X H, et al. Recent progress on porcine circovirus type 3[J]. Infect Genet Evol, 2019, 73:227-233.
[18] TARASOVA E, DHINDWAL S, POPP M, et al. Mechanism of DNA interaction and translocation by the replicase of a circular rep-encoding single-stranded DNA virus[J]. mBio, 2021, 12(4):e0076321.
[19] MANKERTZ A, HILLENBRAND B. Replication of porcine circovirus type 1 requires two proteins encoded by the viral rep gene[J]. Virology, 2001, 279(2):429-438.
[20] 贾含笑, 袁红根, 李 振, 等. PCV2复制起始区DNA互作蛋白的筛选及PARP1蛋白对病毒复制的调控[J]. 畜牧兽医学报, 2022, 53(10):3511-3521.
JIA H X, YUAN H G, LI Z, et al. Screening of proteins interacting with PCV2 replication initiation region and study on the regulation of virus replication by PARP1 protein[J]. Acta Veterinaria et Zootechnica Sinica, 2022, 53(10):3511-3521. (in Chinese)
[21] HU Y, CAI X, ZHAN Y, et al. Truncated Rep protein of porcine circovirus 2 (PCV2) caused by a naturally occurring mutation reduced virus replication in PK15 cells[J]. BMC Vet Res, 2019, 15(1):248.
[22] 李 萌. PCV3 Cap和Rep诱导相关细胞因子mRNA转录及IL-10表达机制的初步研究[D]. 长沙:湖南农业大学, 2020.
LI M. PCV3 Cap and Rep protein induce related cytokine mRNA transcription and IL-10 expression mechanism[D]. Changsha:Hunan Agricultural University, 2020. (in Chinese)
[23] 连凯琪, 周玲玲, 张明亮, 等. 猪圆环病毒3型Rep蛋白的表达与多克隆抗体制备[J]. 中国兽医学报, 2019, 39(11):2107-2111.
LIAN K Q, ZHOU L L, ZHANG M L, et al. Prokaryotic expression of Rep protein of porcine circovirus type 3 and preparation of polyclonal antibodies against Rep protein[J]. Chinese Journal of Veterinary Science, 2019, 39(11):2107-2111. (in Chinese)
[24] GUPTA R, LAKSHMIPATHY S K, CHANG H C, et al. Trigger factor lacking the PPIase domain can enhance the folding of eukaryotic multi-domain proteins in Escherichia coli[J]. FEBS Lett, 2010, 584(16):3620-3624.
[25] KAISER C M, CHANG H C, AGASHE V R, et al. Real-time observation of trigger factor function on translating ribosomes[J]. Nature, 2006, 444(7118):455-460.
[26] FRICK D N. Helicases as antiviral drug targets[J]. Drug News Perspect, 2003, 16(6):355-362.
[27] SHU T, GAN T Y, BAI P, et al. Ebola virus VP35 has novel NTPase and helicase-like activities[J]. Nucleic Acids Res, 2019, 47(11):5837-5851.
[28] 方金娥. 日本乙型脑炎病毒NS3解旋酶活性研究及抑制剂筛选[D]. 武汉:华中农业大学, 2016.
FANG J E. Activity of Japanese encephalitis virus NS3 helicase and inhibitors screening[D]. Wuhan:Huazhong Agricultural University, 2016. (in Chinese)
[29] 周 旋. 猪圆环病毒2型Rep蛋白的ATP酶和解旋酶活性的研究[D]. 武汉:华中农业大学, 2014.
ZHOU X. Study on the ATPase and helicase activity of PCV2 Rep[D]. Wuhan:Huazhong Agricultural University, 2014. (in Chinese)
[30] 王小童. 呼吸道合胞病毒P蛋白NTPase活性与解旋活性研究[D]. 武汉:中国科学院大学(中国科学院武汉病毒研究所), 2021.
WANG X T. Respiratory syncytial virus P has NTPase and helicase-like activities[D]. Wuhan: University of Chinese Academy of Sciences (Wuhan Institute of Virology, Chinese Academy of Sciences), 2021. (in Chinese)
(编辑 白永平)

