蔷薇科花楸属白毛系的系统发生分析




摘要:【目的】 明晰花楸属白毛系(Sorbus ser. Folgnerianae)是否为单系,重建该系3种棕脉花楸(S. dunnii)、石灰花楸(S. folgneri)和江南花楸(S. hemsleyi)的系统发生关系。【方法】 通过标本查阅和野外观察比较白毛系的叶、花、果特征;基于新增的白毛系种和大果花楸(S. megalocarpa,冠萼组Sect. Aria)4种5个样本的质体基因组序列特征、重复序列和序列变异,结合花楸属已发表物种的数据,以蔷薇科其他属代表种和Barbeya oleoides为外类群,分析白毛系的系统发生关系。【结果】白毛系3种在花柱数目、花药颜色、果实形态及花萼是否宿存等方面差异显著,易于鉴别。5个样本的质体基因组具有相似的结构、基因含量和组成。质体基因组长度为159 898~160 755 bp,GC含量为36.4%~36.6%。均注释到113个Unique基因(79个蛋白编码基因、30个tRNA基因、4个rRNA基因),且IR区具扩展长度不等的rps19Ψ和ycf1Ψ 2个假基因。检测到的48~54条SSRs、36~49条散在重复序列和20个高分化度的非编码区可为花楸属物种的群体遗传学、谱系地理学和系统发生研究提供分子标记。基于ML/BI树的系统发生分析结果显示花楸属为复系,6个组各自形成单系;白毛系的3种虽然聚为一组,但水榆花楸(S. alnifolia,直脉系Ser. Alnifoliae)同棕脉花楸、石灰花楸的亲缘关系较江南花楸更近。【结论】白毛系不为单系,形态特征和质体基因组分析为理解白毛系的系统发生提供了有效途径。
关键词:花楸属;形态特征;质体基因组;系统发生
中图分类号:S718.4"""""" 文献标志码:A开放科学(资源服务)标识码(OSID):
文章编号:1000-2006(2024)04-0025-12
Phylogenetic analysis of" Sorbus ser. Folgnerianae (Rosaceae)
MA Jianhui, CHEN Xin*, GENG Liyang, TANG Chenqian, WEI Xueyan
(Co-Innovation Center for Sustainable Forestry in Southern China, College of Life Sciences, Nanjing Forestry University, Nanjing 210037, China)
Abstract: 【Objective】To test"" whether Sorbus ser. Folgnerianae is a monophyletic group" and to reconstruct the phylogenetic relationship among" three species, S. dunnii, S. folgneri and S. hemsleyi.【Method】Morphological characteristics of leaves, flowers and fruits of Ser. Folgnerianae species were compared through specimen examination and field observation. Phylogenetic relationships within Ser. Folgnerianae were analyzed based on" the comparison of the plastid genomes, repeat sequences, sequence variations of the five plastid genomes newly sequenced including three Ser. Folgnerianae species and S. megalocarpa from Sect. Aria, together with other plastid genomes available in this" genus, using representatives of related genera in Rosaceae and Barbeya oleoides (Barbeyaceae) as the outgroups.【Result】Species of Ser. Folgnerianae can be easily distinguished from each other in the number of styles, color of anthers, fruit morphology and the persistence of calyx. Plastid genomes of five samples have a similar structure, gene content and organization. This sizes of plastid genomes range from 159 898 to 160 755 bp, with the" GC contain range between 36.4% and 36.6%. All plastid genomes contain 113 unique genes (79 protein-coding genes, 30 tRNA genes and four ribosomal RNA genes). The IR region has two pseudogenes, rps19Ψand ycf1Ψ, with different extension lengths. 48-54 simple sequence repeats (SSRs), 36-49 long repeats sequences (LRSs) and 20 highly variable regions in the noncoding regions are identified as the most promising potentially variable makers for population genetics, species delimitation and phylogenetic studies. Phylogenetic analyses under ML/BI indicated that Sorbus is polyphyletic and the six sections within it are all monophyletic. Although, three sampled species of Ser. Folgnerianae are clustered in one group, S. Alnifolia of Ser. Alnifolia is more closely related to S. dunnii and S. folgneri than S. hemsleyi. 【Conclusion】Sorbus ser. Folgnerianae is not monophyletic. Morphological characteristics and plastid genome analysis are" effective in understanding the phylogenetic relationship in Ser. Folgnerianae.
Keywords:Sorbus; morphology; plastid genome; phylogeny
花楸属(Sorbus)是蔷薇科(Rosaceae)极具开发价值的属,叶、花、果兼赏,部分种的果实还可制作果酱、果糕、酿酒和入药[1-2]。该属有100[3]~260 种[4],以中国为多样性分布中心。花楸属植物有单叶和复叶两种,常分为6组:冠萼组(Sect. Aria)、落萼组(Sect. Micromeles)、Sect. Chamaemespilus、Sect. Torminalis、Sect. Cormus和复叶组(Sect. Sorbus)[5-6],前4组为单叶,后2组为复叶。一些分类学者将这些组提升到亚属[4, 7]或属的地位[8-9]。
俞德浚等[10]依据单复叶、花萼宿存或脱落等形态特征将中国产花楸属植物分为冠萼组、落萼组和复叶组3个组。白毛系(Ser. Folgnerianae)是落萼组的4个系之一,包含棕脉花楸(S. dunnii)、石灰花楸(S. folgneri)和江南花楸(S. hemsleyi)3种,均为中国特有种。一直以来,分类学者对白毛系植物的系统关系认知存在较大分歧。Gabrielian[6]在其西亚和喜马拉雅地区花楸属植物的分类研究中,将单叶类群花楸分为5组13亚组,白毛系3种被置于冠萼组的2个亚组:石灰花楸和江南花楸置于白毛亚组(Subsect. Folgnerianae),棕脉花楸则置于康藏亚组(Subsect. Thibeticae)。Phipps等[4]在蔷薇科苹果亚科的植物名录整理中,将单叶花楸分为4个亚属,江南花楸置于白花楸亚属(Subg. Aria,即冠萼组),棕脉花楸和石灰花楸置于水榆亚属(Subg. Micromeles,即落萼组)。Aldasoro等[7]在最新的世界单叶花楸属植物分类修订中,将单叶花楸分为白花楸和Torminalis 2个亚属,白毛系的3种均置于白花楸亚属,但石灰花楸置于直脉组(Sect. Alnifoliae),棕脉花楸和江南花楸置于康藏组(Sect. Thibeticae)。
花楸属的分子系统学研究目前多基于少量片段[11-15]或质体基因组开展,但仅涉及少数种类[16-18]。质体基因组属单亲遗传,且相对保守,随着测序技术的成熟和测序成本的降低,越来越多类群的质体基因组被报道,使之成为物种系统发生研究的有效技术手段[19-21]。本研究拟基于白毛系3种植物的质体基因组信息,结合已报道的属内及近缘属物种基因组数据,综合形态学特征,探讨白毛系的系统发生,以期为该系乃至花楸属的系统发生认知提供参考,同时促进对白毛系植物的资源认知和开发利用。
1 材料与方法
1.1 形态特征比较
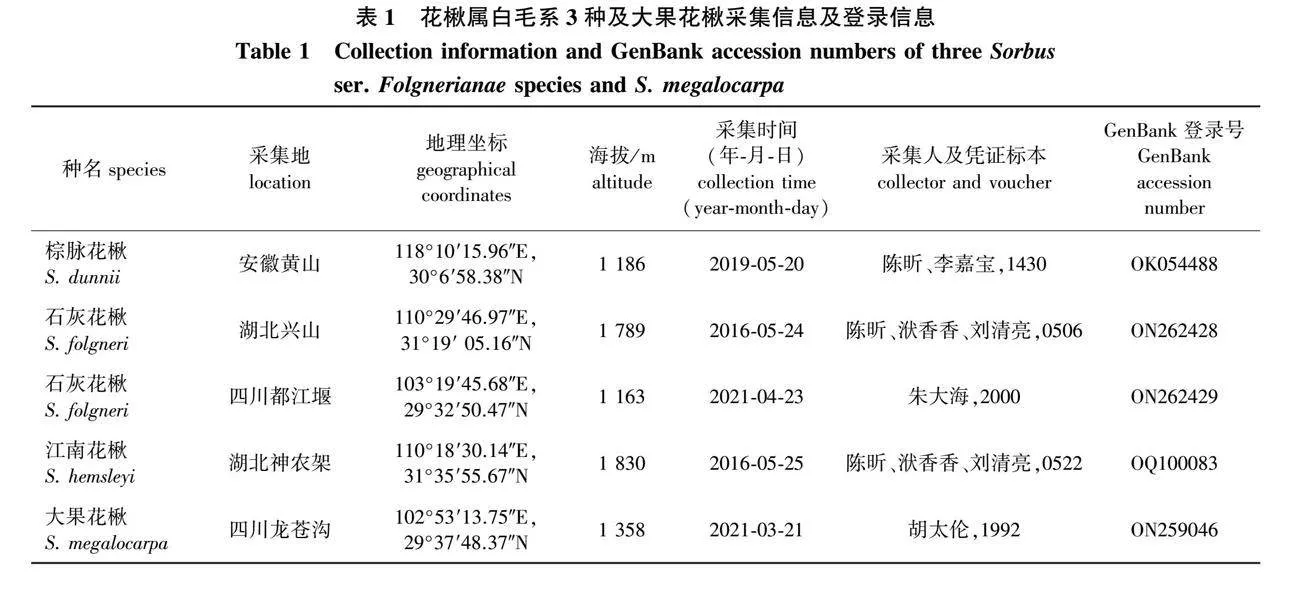
以白毛系3种棕脉花楸、石灰花楸和江南花楸为研究对象,采集其野生居群的花期和果期标本并拍摄照片,比较其形态特征。凭证标本存放于南京林业大学树木标本馆(NF),采集信息见表1。
1.2 质体基因组分析
1.2.1 实验材料
以白毛系3种花楸为研究对象,以冠萼组的大果花楸(S. megalocarpa)为对照,采集4种花楸野生居群生长健壮、无病虫害植株的新鲜叶片,硅胶干燥后,-80" ℃ 低温保存备用。石灰花楸采集了来自2个居群的样本,棕脉花楸、江南花楸和大果花楸各1个居群样本,采集信息和GenBank登录号见表1。
1.2.2 DNA提取、检测与测序
采用多糖多酚植物基因组DNA提取试剂盒(天根生化科技公司, 北京)提取总DNA,使用 Qubit核酸蛋白定量仪检测DNA质量和纯度,检测合格后利用超声波将其片段化,而后进行片段纯化、末端修复、3′端加A、连接测序接头等操作,再用琼脂糖凝胶电泳选择片段大小,PCR扩增构建测序文库,最后将合格文库在DNBSEQ平台进行高通量测序。
1.2.3 基因组组装与注释
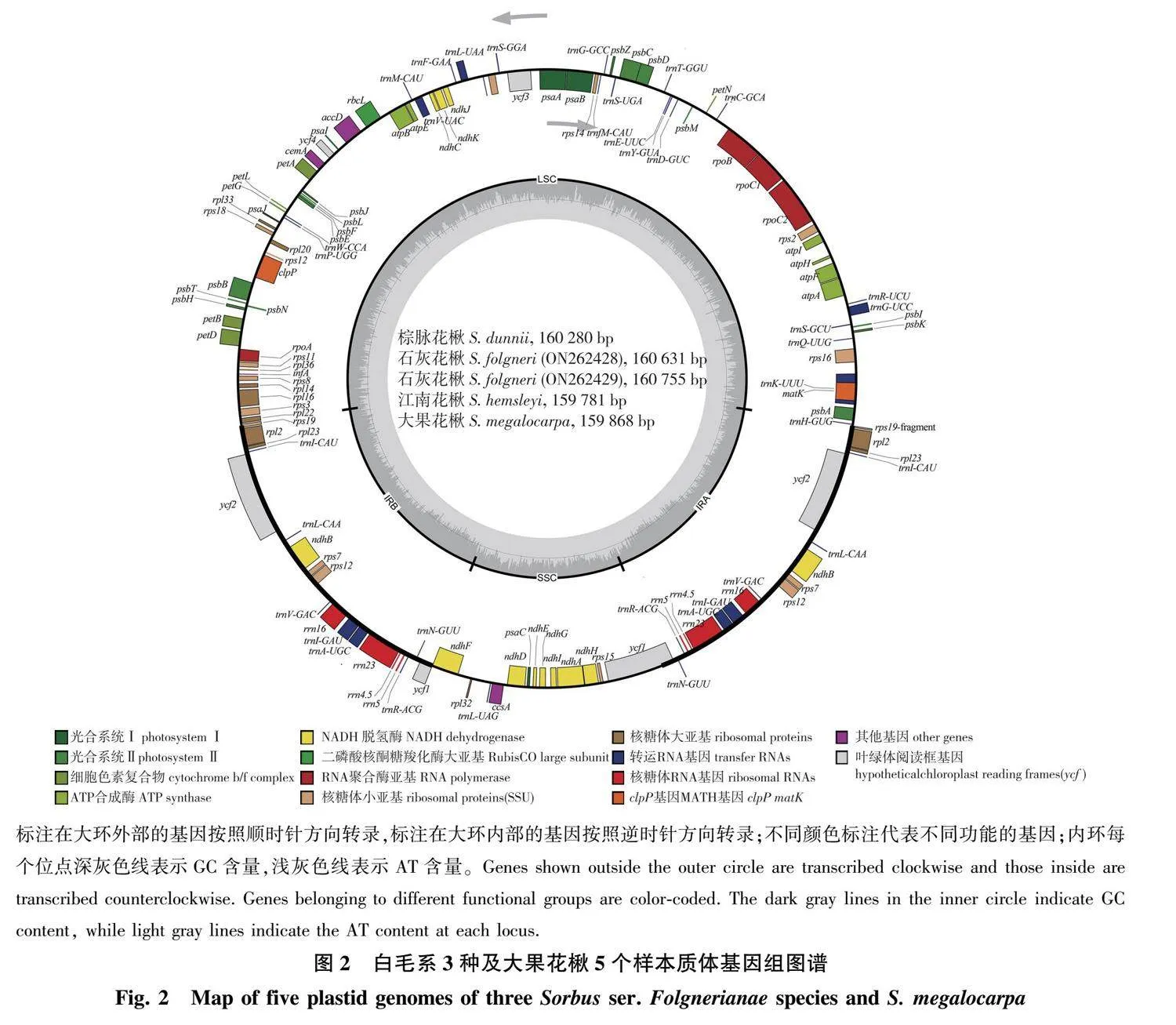
以Sorbus torminalis(NC_033975.1)为参考序列,使用Get Organelle1.7.5.3程序[22]对原始数据进行组装,参数修改为适合花楸属的数值(wordsize为103;k=75、85、95、105、115、127)。将组装好的序列利用Bandage 0.8.1[23]进行可视化处理并过滤。以天山花楸(S. tianschanica; ON049666)为参照,使用PGA[24] 和Geneious 11.0.3[25]完成质体基因组注释。通过Geneious 11.0.3修正注释序列获得最终结果,提交至GenBank。使用OGDRAW(https://chlorobox.mpimp-golm.mpg.de/OGDraw.html)[26]绘制质体基因组图谱。
1.2.4 SSRs与散在重复序列分析
使用MISA(https://pgrc.ipk-gatersleben.de/misa/)[27]检测5个样本以及从NCBI中下载的石灰花楸(MK161058)的SSRs数量和位置分布,单核苷酸、二核苷酸、三核苷酸、四核苷酸、五核苷酸、六核苷酸重复次数依次设置为12、6、4、3、3、3。使用REPuter(https://bibiserv.cebitec.uni-bie lefeld.de/reputer/)[28]分析重复序列,包括正向(forward repeats)、反向(reverse repeats)、回文(palindromic repeats)和互补序列(complement repeats),设置最小重复大小为30 bp,Hamming距离3,序列同源性90%,e-value值均小于10-5。
1.2.5 序列分化分析
使用mVISTA(http://genome.lbl.gov/vista/mvista/instructions.shtml)中的Shuffle-LAGAN程序[29],以康藏花楸(Sect. Aria)质体基因组序列为参照,对5个样本以及NCBI中下载的花楸属6个组8种:石灰花楸(MK161058)、水榆花楸(S. alnifolia;MZ145061.1;Sect. Micromeles)、S. aria(NC_045418.1;Sect. Aria)、S. chamaemespilus(NC_045419.1;Sect. Chamaemespilus)、S. domestica(KY419956;Sect. Cormus)、卷边花楸(S. insignis;NC_051947.1;Sect. Sorbus)、鼠李叶花楸(S. rhamnoides;KY419962;Sect. Micromeles)、S. torminalis(NC_033975.1;Sect. Torminalis)共14个质体基因组序列进行比对,检验序列差异性。使用IRscope软件(https: //irscope.shinyapps.io/irapp/)[30]分析上述14个序列、苹果亚科(Maloideae)的湖北海棠(Malus hupehensis)和李亚科(Prunoideae)的桃(Prunus persica;OK545754.1)共16个序列的IR边界收缩和扩张情况。
1.3 系统发生分析
使用 MAFFT7.037[31],以Barbeya oleoildes(Barbeyaceae)为外类群,以本实验的5个样本数据为基础,从NCBI中下载花楸属6个组、苹果亚科其他属、蔷薇科其他亚科代表种共46个质体基因组数据,进行序列比对。采用最大似然法(ML)和贝叶斯法(BI)构建系统发生树,进行系统进化分析。使用RAxML 8.0.0(m=GTR+GAMMA)[32]完成ML分析;将MrModel test 3.7[33]构建的模型导入MrBayes 3.2.2[34]完成BI分析。ML和BI的分析结果用FigTree 1.4.3(http://tree.bio.ed.ac.uk/software/figtree/)进行可视化处理。
2 结果与分析
2.1 形态特征分析
白毛系的棕脉花楸、石灰花楸和江南花楸均为落叶乔木,叶均为卵形至椭圆状卵形,侧脉均直达叶边齿端,叶背密被毛。但棕脉花楸和石灰花楸叶背密被白色毛,而江南花楸密被灰白色毛。3种花楸在叶柄、叶背中脉和侧脉毛被上差异显著:棕脉花楸均被棕色毛(图1a)、石灰花楸均被白色毛(图1b)、江南花楸则光滑无毛(图1c)。从花序及花特征上看,棕毛花楸花序被棕色毛(图1d),石灰花楸(图1e)和江南花楸(图1f)花序被白色毛;棕毛花楸和石灰花楸花柱2枚、花药黄白色,而江南花楸花柱3~4枚、花药紫红色。果实特征也易于区分,棕脉花楸(图1g)和石灰花楸(图1h)花部均脱落、果实顶端留有圆穴,但前者果实近球形、后者果实卵球形;江南花楸果实形状同棕脉相似,但其花萼、雄蕊、花柱宿存(图1i)。
2.2 质体基因组特征比较分析
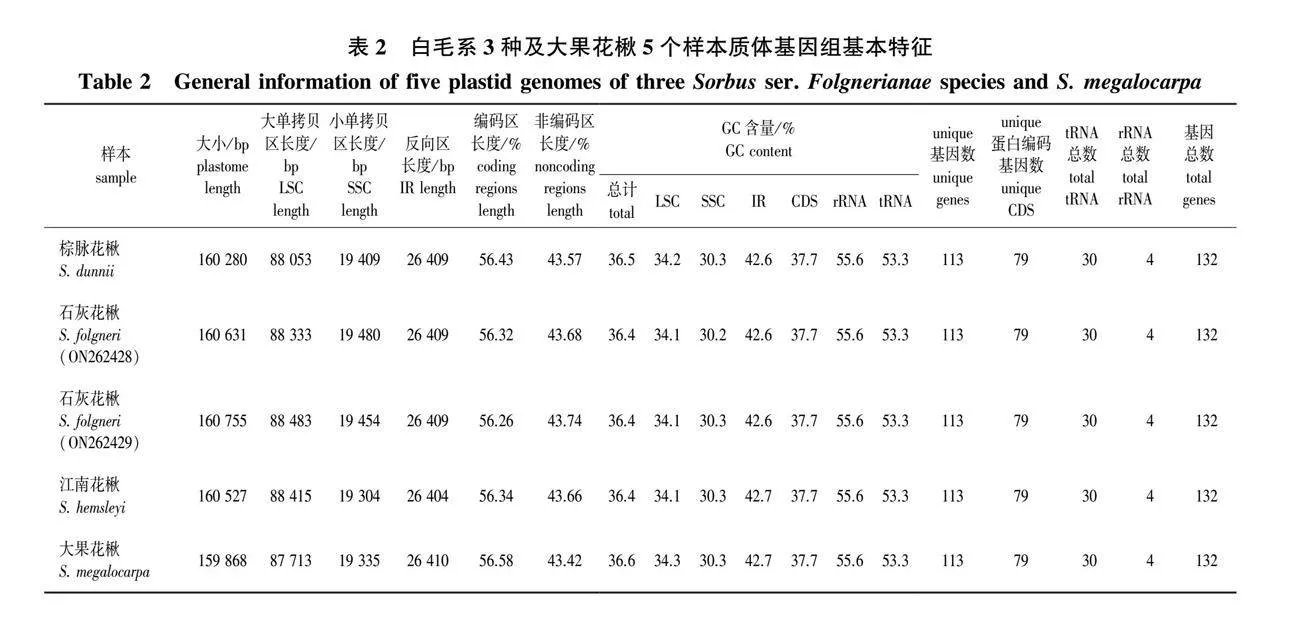
2.2.1 基因组基本特征
白毛系棕脉花楸、石灰花楸(两个体)、江南花楸及大果花楸4种共5个样本的质体基因组特征比较见图2和表2。5个样本的质体基因组全长介于159 868(大果花楸)~160 755 bp(石灰花楸-ON262429)。石灰花楸的两个体相差124 bp,均较另3种长。5样本均具有双链环状四分体结构,包含一个大单拷贝区(LSC)、一个小单拷贝区(SSC)和两个反向重复区(IRa/IRb)。LSC、SSC、IR区长度分别为87 713~88 483 bp、19 304~19 480 bp和26 404~26 410 bp。5个样本的编码区长度占比为56.26%~56.58%,非编码区长度占比为43.42%~43.74%。GC含量差异不大,石灰花楸和江南花楸最小,2个种均为36.4%,棕脉花楸为36.5%,大果花楸为36.6%;LSC和SSC区的种间差异也仅为0.2和0.1个百分点;5个样本CDS、rRNA、tRNA 3个区的GC含量则完全相同,分别为37.7%、55.6%和53.3%。
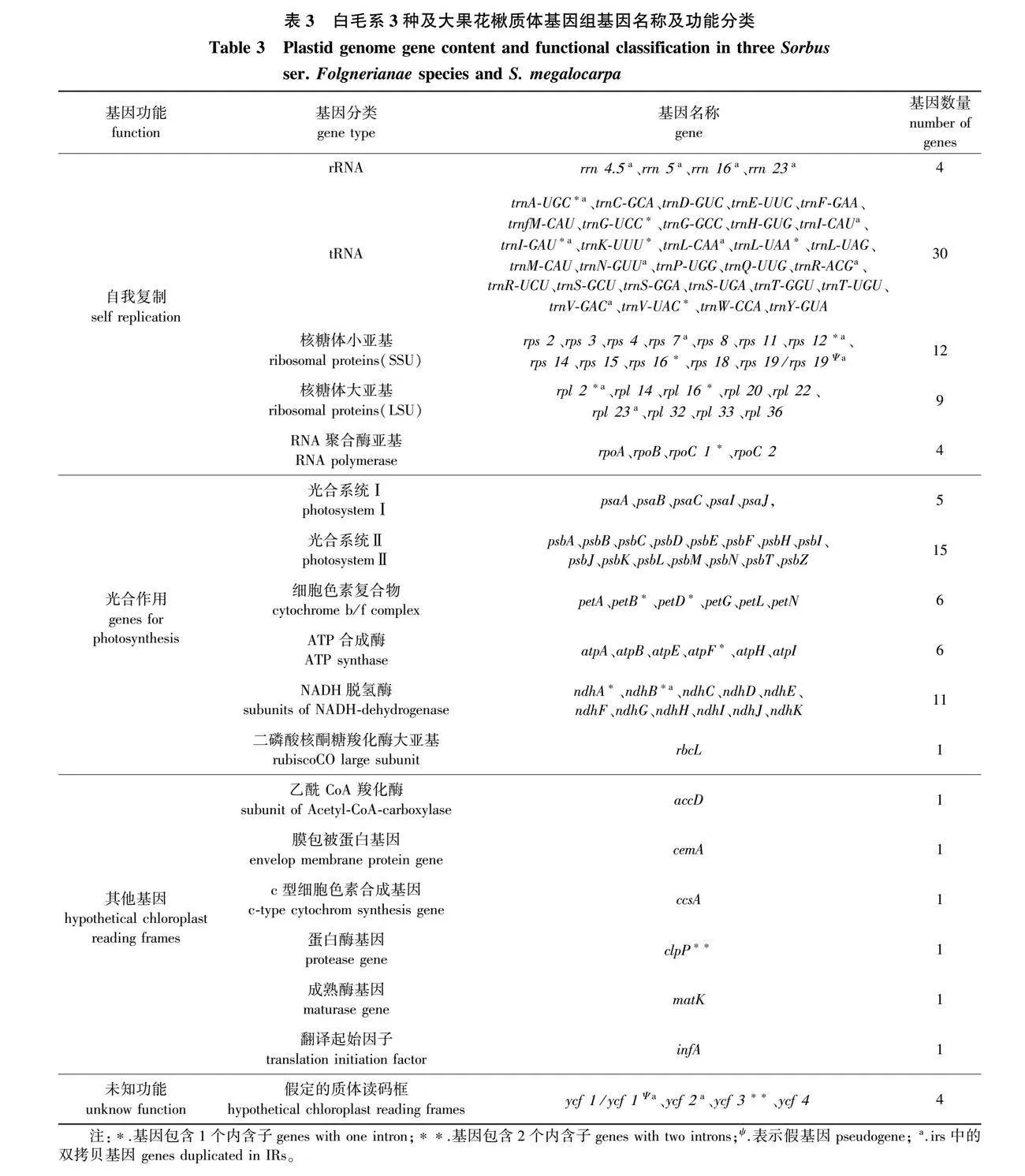
5个样本均注释到113个unique基因,包括蛋白编码基因79个、tRNA基因30个、rRNA基因4个。19个基因在IR区复制,基因总数达132。有18个基因含有内含子,其中16个基因(trnA-UGC、trnG-UCC、trnI-GAU、trnK-UUU、trnL-UAA、trnV-UAC、ndhA、ndhB、petB、petD、rpl16、rps12、rpoC1、rps16、rpl2、atpF)含有1个内含子,2 个基因(clpP和ycf3)含有2个内含子,含内含子的基因中有6个为tRNA基因。根据基因功能可将基因分为 3 类,其中与光合作用相关的基因44个,与自我复制相关的基因59个,其他编码蛋白质的基因6个以及未知功能基因4个(表3)。
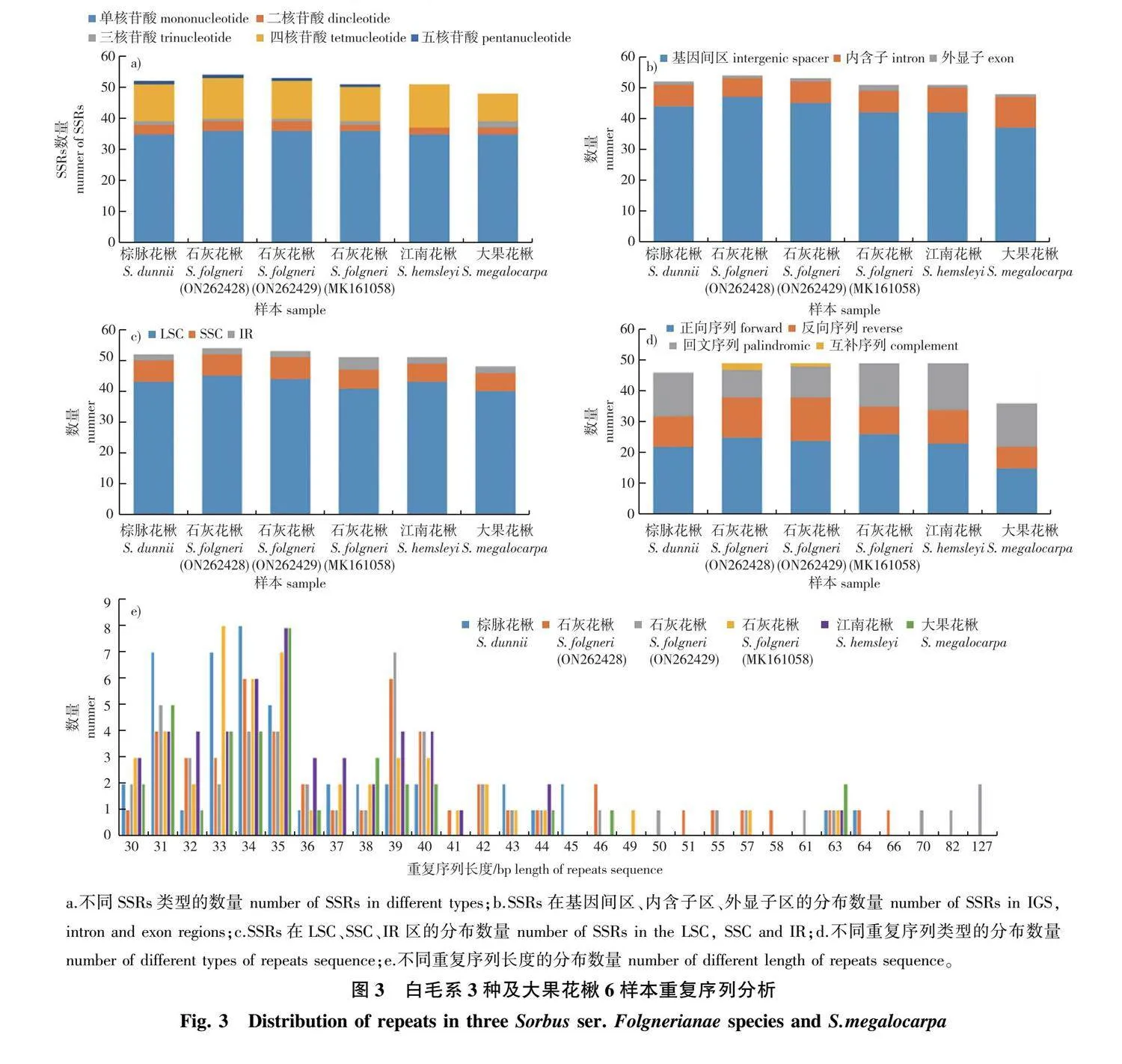
2.2.2 SSRs和散在重复序列分析
基于实验的5个样本及石灰花楸(MK161058)的MISA分析结果见图3a—3c。6个序列的SSRs数量为48(大果花楸)~54(石灰花楸-ON262428)。SSRs以单核苷酸为主,占比为66.67%~72.92%,且偏向使用A/T碱基。单、四核苷酸数量远高于二、三、五核苷酸,均不含六核苷酸(图3a)。棕脉花楸、江南花楸和大果花楸单核苷酸均为35,石灰花楸3个样本均为36;四核苷酸数目为9~14,二核苷酸为2或3,江南花楸不含三核苷酸,其他样本为1或2,江南花楸和大果花楸不含五核苷酸,其他样本均有1个五核苷酸。基因间区、外显子区和内含子区均有SSRs分布,且基因间区的SSRs占比最高,为77.08%~87.04%(图3b)。LSC区的SSRs占比在80.39%~84.31%之间,远高于SSC区(11.76%~13.46%)和IRs区(3.70%~7.84%)(图3c)。
基于REPuter检查结果见图3d—3e。6个质体基因组的散在重复序列数量为36(大果花楸)~49(石灰花楸、江南花楸)。正向、反向、回文、互补4种序列的数量变异区间分别为:15~26、7~14、9~15和1~2,正向序列最普遍(图3d)。重复序列长度变化为30~127 bp,主要位于30~40 bp(图3e)。6个质体基因组的ycf1、trnS-GUC、trnS-GGA编码区和clpP、ycf3和ndhA的内含子上均有重复序列分布。此外,白毛系4个测定样本的rpl32编码区、大果花楸的trnK-UUU和atpF的内含子上、除大果花楸以外5个样本的trnG-UCC 的内含子上也有分布。
2.2.3 基因组序列变异分析
白毛系3种与同花楸属另9种植物共14个序列不同区域的比较显示:编码区较非编码区、IR区较LSC/SSC 区更为保守(图4)。20个非编码区表现出高分化度,其中17个位于基因间区,分别是:trnK-rps16、trnR-atpA、atpF-atpH、trnC-petN、petN-psbM、psbM-trnD、trnT-psbD、psbZ-trnG、trnT-trnL、ndhC-trnV、trnM-atpE、accD-psaI、clpP-psbB、ndhF-rpl32、rpl32-trnL、ndhG-ndhI、rps19-trnH;3个位于内含子区:分别为clpP、rpl16、ndhA。除了ndhF-rpl32、rpl32-trnL、ndhG-ndhI和ndhA位于SSC区,其他非编码区均位于LSC区。
白毛系同落萼组其他种、花楸属另5个组、苹果亚科的湖北海棠以及李亚科的桃共14种植物16个序列IR区边界的收缩和扩展比较见图5。除石灰花楸(MK161058)外,所有序列的LSC/IRb边界均位于rps19基因内,且rps19的5′末端均不同程度扩张进入IRb中,从而在IRa区产生了1个不同大小的rps19假基因(rps19Ψ)。rps19Ψ长度介于120~182 bp,桃的最长,鼠李叶花楸和大果花楸为152 bp,棕脉花楸、石灰花楸2个样本及水榆花楸的长度均为142 bp,江南花楸为146 bp,其他序列均为120 bp。所有序列的SSC/IRa边界均位于ycf1基因内,ycf1的5′末端位于IRa内,导致了IRb中ycf1Ψ假基因(ycf1Ψ)的产生。ycf1Ψ长度介于1 003~2 573 bp,桃的最短,石灰花楸(MK161058)的最长,水榆花楸为1 221 bp,S. torminalis为1 092 bp,湖北海棠为1 074 bp,其余序列均为1 083 bp。
2.3 系统发生分析
以Barbeya oleoides(Barbeyaceae)为外类群,基于白毛系3种植物、花楸属其他组以及蔷薇科其他属共46个分类群的质体基因组数据,构建了ML和BI系统发生树。两种树的拓扑结构一致,在此仅展示了ML树的结果,并在树上标注了BI树的支持率(图6)。由系统发生树(图6)可见:花楸属为复系,属下6组中的4组:Chamaemespilus、Cormus、复叶组和Torminalis形成了高支持率的单系,但冠萼组与落萼组未形成单系,白毛系亦未形成单系。白毛系的棕脉花楸、石灰花楸与落萼组直脉系(Ser. Alnifoliae)的水榆花楸形成姐妹群,而后与江南花楸聚为一组。冠萼组大果系(Ser. Megalocarpae)的大果花楸和落萼组直脉系的鼠李叶花楸形成姐妹类群,而后与冠萼组康藏系(Ser. Thibeticae)的康藏花楸聚为一组。
3 讨 论
花楸属植物的质体基因组在序列结构、基因组成、GC含量等方面具有高度保守性,未出现基因重排或缺失现象,但仍然存在一些具有区分度的显著序列变异,同本属前期研究结果相似[17-18, 35]。石灰花楸两个样本的基因组长度均较另4种的长,由于重复序列的数量和分布的差异造成了其种内基因组长度的差异高于种间。IR边界的收缩和扩展在一些类群的演化研究中具有重要意义[36-37],同Byrsonima属的物种相似[38],花楸属rps19和ycf1基因向IR区扩展长度不等,产生了rps19Ψ和ycf1Ψ 两个长度不一的假基因,且ycf1Ψ在JSB处的重叠长度在种间亦有一定变化。同蔷薇科[39-41]及多数被子植物序列进化式样一致[42-43],白毛系及其他花楸属植物的非编码区较编码区、LSC/SSC 区较IR区分化度更高。本实验检测出的20个高变区不仅可以用于该属条形码的开发,也可为对该属的系统进化研究提供快捷途径。SSRs在基因间区占比较高、散在重复序列中正向序列最普遍,同陕甘花楸(S. koehneana)和四川花楸(S. setschwanensis)相似[18, 35]。本研究检测到的48~54条SSRs、36~49条散在重复序列可用于该属物种的群体遗传学和谱系地理学研究。
基于质体基因组的系统发生分析显示花楸属为复系,同Campbell等[11]、Potter等[12]、Sun等[44]、Ulaszewski等[17]的研究结果一致。本研究中,花楸属Chamaemespilus、Cormus、复叶组和Torminalis 4个组形成了单系;但中国产冠萼组花楸同复叶组的亲缘关系较近,而同欧洲产冠萼组模式种S. aria的亲缘关系较远;中国产冠萼组和落萼组虽然聚为1支,但各自未形成单系,同Li等[45]基于ITS的研究结果一致。本实验中,源自湖北兴山和四川都江堰的2个石灰花楸样本同源自江西庐山的石灰花楸(MK161058)[46] 聚在了一起,可见基于质体基因组的系统树构建在花楸属植物系统发生关系理解上的可靠性。棕脉花楸、石灰花楸和江南花楸基于质体基因组的系统发生关系不支持俞德浚等[10]、Gabrielian[6]和Aldasoro等[7]的分类处理。从形态上看,俞德浚等[10]将3种花楸置于一个系的前提条件“落萼”并不存在,因为江南花楸果期花萼宿存。
花楸属是蔷薇科分类最为困难的属之一,属下分类一直存有争议,本研究中落萼组和冠萼组未形成单系,白毛系和直脉系亦未形成单系,分类学者基于形态和分子的系统研究尚未达成一致,因而花楸属种的系统关系确立需要基于更广泛的属内物种、同一物种不同居群的取样,需要更综合的形态及分子数据。
参考文献(reference):
[1]俞德浚, 陆玲娣. 绣线菊属, 牛筋条属, 栒子属, 花楸属, 木瓜海棠属中国植物志 第36卷[M]. 北京: 科学出版社, 1974: 283-344. YU D J, LU L D. Spiraea, Dichotomanthes, Cotoneaster, Sorbus, Chaenomeles. Flora Republicae Popularis, Sinica: Vol. 36[M]. Beijing: Science Press, 1974: 283-344.
[2]KAI K,TU L J,SUN H J,et al.Extraction and purification of anthocyanins from Sorbus pohuashanensis fruits[J].Curr Top Nutraceutical Res,2020,18(4):319-324.DOI: 10.37290/ctnr2641-452x.18:319-324.
[3]LU L T, SPONGBERG S A.Sorbus Linnaeus[M]// WU Z Y, RAVEN P H, HONG D Y. Flora of China: Vol. 9. Beijing: Science Press, 2003: 144-170.
[4]PHIPPS J B,ROBERTSON K R,SMITH P G,et al.A checklist of the subfamily maloideae (Rosaceae)[J].Can J Bot,1990,68(10):2209-2269.DOI: 10.1139/b90-288.
[5]DON G.A General history of the dichlamydeous plants:Vol.2[M]. London: J G and F Rivington, 1832: 875. https://biodiversitylibrary.org/page/340686.
[6]GABRIELIAN E. The genus" Sorbus L. in western Asia and the Himalayas[M]. Yerevan: Academy of Sciences of the Armenian SSR, 1978: 264+62.
[7]ALDASORO J J,AEDO C,GARMENDIA F M,et al.Revision of Sorbus subgenera Aria and Torminaria (Rosaceae-maloideae)[J].Syst Bot Monogr,2004,69:1.DOI: 10.2307/25027918.
[8]DECAISNE M J. Mémoirs sur la famille des Pomacées[J]. Nouv arch Mus hist nat, 1874, 10: 113-192.
[9]SPANCH E. Histoire naturelle des végétaux: phanerogames Ⅱ[M]. Paris: Librairie Encyclopédique de Roret, 1834:1-160.
[10]俞德浚, 关克俭. 中国蔷薇科植物分类之研究(一)[J]. 植物分类学报, 1963, 8(3): 202-234.YU D J, GUAN K J. Taxa nova Rosacearum sinicarum(Ⅰ)[J]. J Syst Evol, 1963, 8(3): 202-234.
[11]CAMPBELL C S,EVANS R C,MORGAN D R,et al.Phylogeny of subtribe Pyrinae (formerly the Maloideae,Rosaceae):limited resolution of a complex evolutionary history[J].Plant Syst Evol,2007,266:119-145.DOI: 10.1007/s00606-007-0545-y.
[12]POTTER D,ERIKSSON T,EVANS R C,et al.Phylogeny and classification of Rosaceae[J].Plant Syst Evol,2007,266:5-43.DOI: 10.1007/s00606-007-0539-9.
[13]LO E Y Y,DONOGHUE M J.Expanded phylogenetic and dating analyses of the apples and their relatives (Pyreae,Rosaceae)[J].Mol Phylogenetics Evol,2012,63(2):230-243.DOI: 10.1016/j.ympev.2011.10.005.
[14]ZHANG S D,JIN J J,CHEN S Y,et al.Diversification of Rosaceae since the Late Cretaceous based on plastid phylogenomics[J].New Phytol,2017,214(3):1355-1367.DOI: 10.1111/nph.14461.
[15]LI M,OHI-TOMA T,GAO Y D,et al.Molecular phylogenetics and historical biogeography of Sorbus sensu stricto (Rosaceae)[J].Mol Phylogenetics Evol,2017,111:76-86.DOI: 10.1016/j.ympev.2017.03.018.
[16]LIU B B,HONG D Y,ZHOU S L,et al.Phylogenomic analyses of the Photinia complex support the recognition of a new genus Phippsiomeles and the resurrection of a redefined Stranvaesia in Maleae (Rosaceae)[J].J Syst Evol,2019,57(6):678-694.DOI: 10.1111/jse.12542.
[17]ULASZEWSKI B,JANKOWSKA-WRBLEWSKA S,SWILO K,et al.Phylogeny of maleae (Rosaceae) based on complete chloroplast genomes supports the distinction of Aria,Chamaemespilus and Torminalis as separate Genera,different from Sorbus sp.[J].Plants (Basel),2021,10(11):2534.DOI: 10.3390/plants10112534.
[18]汤晨茜,仇志欣,檀超,等.陕甘花楸叶绿体基因组及其与爪瓣花楸的系统关系[J].园艺学报,2022,49(3):641-654.TANG C Q,QIU Z X,TAN C,et al.Sorbus koehneana(Rosaceae):its complete chloroplast genome and phylogenetic relationship with S.unguiculata[J].Acta Hortic Sin,2022,49(3):641-654.DOI: 10.16420/j.issn.0513-353x.2021-0040.
[19]HUANG H,SHI C,LIU Y,et al.Thirteen Camellia chloroplast genome sequences determined by high-throughput sequencing:genome structure and phylogenetic relationships[J].BMC Evol Biol,2014,14:151.DOI: 10.1186/1471-2148-14-151.
[20]BARRETT C F,BAKER W J,COMER J R,et al.Plastid genomes reveal support for deep phylogenetic relationships and extensive rate variation among palms and other commelinid monocots[J].New Phytol,2016,209(2):855-870.DOI: 10.1111/nph.13617.
[21]BROCK J R,MANDKOV T,MCKAIN M,et al.Chloroplast phylogenomics in Camelina (Brassicaceae) reveals multiple origins of polyploid species and the maternal lineage of C.sativa[J].Hortic Res,2022,9:uhab050.DOI: 10.1093/hr/uhab050.
[22]JIN J J,YU W B,YANG J B,et al.Get Organelle:a fast and versatile toolkit for accurate de novo assembly of organelle genomes[J].Genome Biol,2020,21(1):241.DOI: 10.1186/s13059-020-02154-5.
[23]WICK R R,SCHULTZ M B,ZOBEL J,et al.Bandage:interactive visualization of de novo genome assemblies[J].Bioinformatics,2015,31(20):3350-3352.DOI: 10.1093/bioinformatics/btv383.
[24]QU X J,MOORE M J,LI D Z,et al.PGA:a software package for rapid,accurate,and flexible batch annotation of plastomes[J].Plant Methods,2019,15:50.DOI: 10.1186/s13007-019-0435-7.
[25]KEARSE M,MOIR R,WILSON A,et al.Geneious basic:an integrated and extendable desktop software platform for the organization and analysis of sequence data[J].Bioinformatics,2012,28(12):1647-1649.DOI: 10.1093/bioinformatics/bts199.
[26]GREINER S,LEHWARK P,BOCK R.Organellar GenomeDRAW (OGDRAW) version 1.3.1:expanded toolkit for the graphical visualization of organellar genomes[J].Nucleic Acids Res,2019,47(W1):59-64.DOI: 10.1093/nar/gkz238.
[27]BEIER S,THIEL T,MNCH T,et al.MISA-web:a web server for microsatellite prediction[J].Bioinformatics,2017,33(16):2583-2585.DOI: 10.1093/bioinformatics/btx198.
[28]KURTZ S,CHOUDHURI J V,OHLEBUSCH E,et al.REPuter:the manifold applications of repeat analysis on a genomic scale[J].Nucleic Acids Res,2001,29(22):4633-4642.DOI: 10.1093/nar/29.22.4633.
[29]MAYOR C,BRUDNO M,SCHWARTZ J R,et al.VISTA:visualizing global DNA sequence alignments of arbitrary length[J].Bioinformatics,2000,16(11):1046-1047.DOI: 10.1093/bioinformatics/16.11.1046.
[30]AMIRYOUSEFI A,HYVNEN J,POCZAI P.IRscope:an online program to visualize the junction sites of chloroplast genomes[J].Bioinformatics,2018,34(17):3030-3031.DOI: 10.1093/bioinformatics/bty220.
[31]KATOH K,STANDLEY D M.MAFFT multiple sequence alignment software version 7:improvements in performance and usability[J].Mol Biol Evol,2013,30(4):772-780.DOI: 10.1093/molbev/mst010.
[32]STAMATAKIS A.RAxML version 8:a tool for phylogenetic analysis and post-analysis of large phylogenies[J].Bioinformatics,2014,30(9):1312-1313.DOI: 10.1093/bioinformatics/btu033.
[33]POSADA D,CRANDALL K A.MODELTEST:testing the model of DNA substitution[J].Bioinformatics,1998,14(9):817-818.DOI: 10.1093/bioinformatics/14.9.817.
[34]RONQUIST F,TESLENKO M,van der MARK P,et al.MrBayes 3.2:efficient Bayesian phylogenetic inference and model choice across a large model space[J].Syst Biol,2012,61(3):539-542.DOI: 10.1093/sysbio/sys029.
[35]WANG Q,NIU Z Y,LI J B,et al.The complete chloroplast genome sequence of the Chinese endemic species Sorbus setschwanensis (Rosaceae) and its phylogenetic analysis[J].Nord J Bot,2020,38(2):e02532.DOI: 10.1111/njb.02532.
[36]李倩,郭其强,高超,等.贵州威宁红花油茶的叶绿体基因组特征分析[J].园艺学报,2020,47(4):779-787.LI Q,GUO Q Q,GAO C,et al.Characterization of complete chloroplast genome of Camellia weiningensis in Weining,Guizhou Province[J].Acta Hortic Sin,2020,47(4):779-787.DOI: 10.16420/j.issn.0513-353x.2019-0410.
[37]赵儒楠,褚晓洁,刘维,等.鹅耳枥属树种叶绿体基因组结构及变异分析[J].南京林业大学学报(自然科学版),2021,45(2):25-34.ZHAO R N,CHU X J,LIU W,et al.Structure and variation analyses of chloroplast genomes in Carpinus[J].J Nanjing For Univ (Nat Sci Ed),2021,45(2):25-34.DOI: 10.12302/j.issn.1000-2006.202009007.
[38]MENEZES A P A,RESENDE-MOREIRA L C,BUZATTI R S O,et al.Chloroplast genomes of Byrsonima species (Malpighiaceae):comparative analysis and screening of high divergence sequences[J].Sci Rep,2018,8(1):2210.DOI: 10.1038/s41598-018-20189-4.
[39]KIMURA T,IKETANI H,KOTOBUKI K,et al.Genetic characterization of pear varieties revealed by chloroplast DNA sequences[J].J Hortic Sci Biotechnol,2003,78(2):241-247.DOI: 10.1080/14620316.2003.11511612.
[40]ROH M S,CHEONG E J,CHOI I Y,et al.Characterization of wild Prunus yedoensis analyzed by inter-simple sequence repeat and chloroplast DNA[J].Sci Hortic,2007,114(2):121-128.DOI: 10.1016/j.scienta.2007.06.005.
[41]YAZBEK M,OH S-H.Peaches and almonds:phylogeny of Prunus subg. amygdalus (Rosaceae) based on DNA sequences and morphology[J].Plant Syst Evol,2013,299(8):1403-1418.DOI: 10.1007/s00606-013-0802-1.
[42]THODE V A,LOHMANN L G.Comparative chloroplast genomics at low taxonomic levels:a case study using Amphilophium (Bignonieae,Bignoniaceae)[J].Front Plant Sci,2019,10:796.DOI: 10.3389/fpls.2019.00796.
[43]WANG Y H,WANG S,LIU Y L,et al.Chloroplast genome variation and phylogenetic relationships of Atractylodes species[J].BMC Genomics,2021,22(1):103.DOI: 10.1186/s12864-021-07394-8.
[44]SUN J H,SHI S,LI J L,et al.Phylogeny of maleae (Rosaceae) based on multiple chloroplast regions:implications to genera circumscription[J].Biomed Res Int,2018,2018:7627191.DOI: 10.1155/2018/7627191.
[45]LI Q Y,GUO W,LIAO W B,et al.Generic limits of Pyrinae:insights from nuclear ribosomal DNA sequences[J].Bot Stud,2012,53(1):151-164.
[46]QIU J,CHEN L,YI X G,et al.The complete chloroplast genome of Sorbus folgneri (C.K.Schneid.) Rehder (Rosaceae)[J].Mitochondrial DNA B,2019,4(1):728-729.DOI: 10.1080/23802359.2018.1558127.
(责任编辑 吴祝华)
收稿日期Received:2022-05-02""" 修回日期Accepted:2022-06-10
基金项目:江苏省自然科学基金项目(BK20141472);江苏高校优势学科建设工程资助项目(PAPD)。
第一作者:马建慧(2556429572@qq.com)。
*通信作者:陈昕(chenxinzhou@njfu.edu.cn),教授。
引文格式:马建慧,陈昕,耿礼阳,等. 蔷薇科花楸属白毛系的系统发生分析[J]. 南京林业大学学报(自然科学版),2024,48(4):25-36.
MA J H, CHEN X, GENG L Y, et al. Phylogenetic analysis of" Sorbus ser. Folgnerianae (Rosaceae)[J]. Journal of Nanjing Forestry University (Natural Sciences Edition),2024,48(4):25-36.
DOI:10.12302/j.issn.1000-2006.202205003.

