根际微生境中生化过程的原位可视化研究进展


摘要: 根际及近根际区域(根际微生境) 进行着活跃的物质交换和能量流动,大部分陆地生态系统中碳、氧和矿质元素等关键生源要素循环的生化过程都发生在根际微生境中。根际微生境空间动态过程的可视化对于准确捕捉和量化根际界面物质迁移转化过程和功能至关重要。过去几十年中,采用二维成像方法进行营养元素、酶和微生物等多种生化过程的同步原位可视化研究取得了重要进展。本文系统总结了根际化学环境、微生物环境和代谢物原位可视化研究的最新方法与研究进展,讨论了多参数成像技术在根际微生境研究中的应用前景,并对根际生化过程原位可视化研究的未来发展方向及亟待解决的问题进行了展望。为进一步提高根际原位可视化技术的研发与应用,深入解析微生境中生化过程与机制,需要在以下几方面重点展开研究:1)开展操作简单、耗时少、成本低的可视化技术开发,并进行多参数及多手段耦合的兼容性验证;2)从样品制备和数据整合等方面,开发根际三维及四维可视化方法,探索根际微生境的时空动态可视化;3)研发便携式仪器,开展根际原位可视化技术的田间应用。
关键词: 根际微生境; 多参数成像技术; 原位可视化
植物−土壤相互作用是地球关键带中最重要的生物与环境互作过程之一,根际是植物−土壤−微生物互作的核心界面,决定着自然生态系统的多功能性和农业生态系统的可持续性和生产力[1−2]。自Hiltner提出“根际”的概念之后,其被广泛应用于土壤学、植物学和微生物学等学科研究中。根际中进行着活跃的物质交换和能量流动,直接影响着陆地生态系统中关键要素如碳、氧和矿质元素的循环,而大部分生化过程实际上都发生在根际微区中。根际微生境下,根系、土壤、根系分泌物和生物体之间强烈的相互作用驱动了根际生物化学过程[3−5]。
随着多组学技术与机器学习的迅猛发展,学界在元素生物地球化学循环、根际代谢物介导的植物−微生物互作和微生物群落组装模式等根际生化过程研究方面取得了显著成果[6−8]。然而,受限于原位可视化技术,根际微生境的多种生物化学过程很大程度上仍处于“黑箱”状态。与破坏性方法相比,可视化技术得到的根际范围准确性可提高3~5 倍[9]。因此,根际微域空间动态过程的可视化对于捕捉和量化根际界面物质迁移转化过程和功能至关重要[9]。由于根际多组分的复杂性,为了实现不同生化参数的可视化,需要开发基于多尺度(亚微米~厘米) 的实验设计和测试技术[4]。然而,由于各方面条件的限制,目前大多数根际可视化研究都在半人工实验装置中模拟田间条件,而对根际微生境生化过程的可视化研究仍处于起步阶段[4]。可见,目前对根际的实际情况特别是根−土界面的多要素空间分布特征只有模糊的了解,在根际微区可视化方面研究仍存在巨大挑战。
过去十年,前人已从不同方面回顾了根际微生境相关研究进展。例如,Oburger 等[4]系统综述了根系分泌物的收集、植物−微生物互作及根际过程可视化的最新方法与技术。Védère 等[10]总结了土壤微生境中微生物及其活动的成像方法。近期,Li 等[11]重点综述了土壤/沉积物研究中常用的微尺度采样和二维成像方法,归纳了其研究进展和应用成果,并从研发方向和应用领域等方面对土壤、沉积物微尺度研究技术进行了展望。然而,已有的研究主要从总结单一技术的特性角度出发,针对根际特定生化过程的可视化方法研究进展尚缺乏系统性的总结。此外,依赖于断层扫描技术,根系系统结构和水力特性可视化已被广泛揭示[12],而根际化学环境、微生物环境和根际代谢物等核心生物化学过程原位可视化的研究进展鲜有关注。因此,本文在强调根际微生境的范围与特性的基础上,旨在总结近年来在根际微生境生化过程非破坏性成像方向的进展,简要归纳了不同生化参数的主流成像技术及优势、存在的问题,最后展望了根际微生境原位可视化的未来努力方向,以期为根际过程研究提供参考。
1 根际微生境的范围与功能
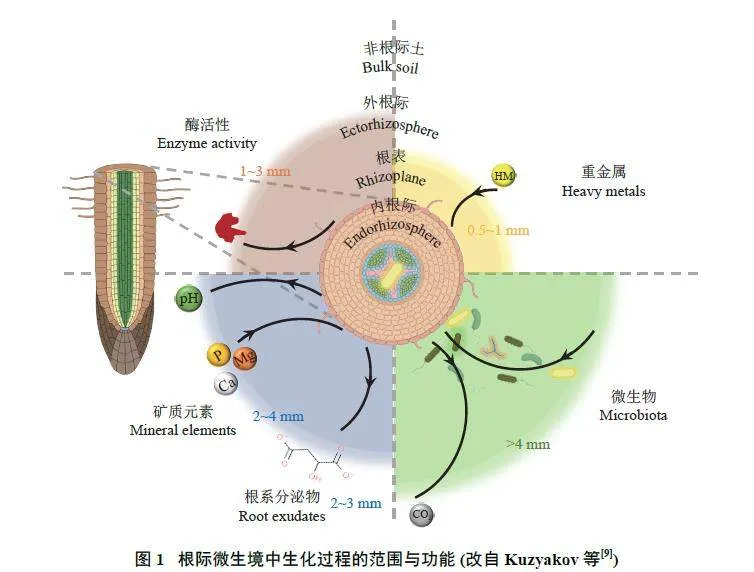
土壤中生物化学过程主要发生在根际微域,且会被周围巨大的土体“稀释”,这使得根际成为土壤乃至陆地生态系统中最重要的热点区域[9]。“根际”通常指受植物根系影响的土壤,也可扩展为根−土界面,细分为外根际(ectorhizosphere)、根表(rhizoplane)和内根际(endorhizosphere) 3 个微生境[4, 13]。与内根际相比,外根际和根表微生境直接与土壤接触,且由于受到根系的影响,其生物化学性质呈现高度的异质性和复杂性。此外,根际微生境存在以下特点:1) 根际的隐蔽性;2) 常规破坏式采样方法易造成界面间功能和空间联系消失;3) 根际边界范围和形状不清晰;4) 不同生化参数的变化方式迥异;5) 时空波动造成难以捕捉根际瞬时状态[14−15]。上述特性致使获取根际微生境不同生化参数的准确范围困难重重。
Kuzyakov 等[9]根据已有文献,梳理了部分根际生化过程参数的边界范围和变化特征。其中,微量营养元素和重金属的活动范围最小,通常为0.5~1 mm,呈现出从外根际向根表迁移并逐渐耗竭的现象,这是由于部分元素向根表的扩散速率低于根系吸收速率所致[16]。土壤酶分子量较大,造成难以扩散,因此其活性的根际范围约为1~3 mm,呈现由根表向外根际活性减弱的趋势[17]。同位素成像发现,根系分泌物的根际范围仅为2~3 mm[18]。Ca2+、Mg2+、NH4+及PO43−等养分和pH 的根际范围为2~4 mm[19]。只有部分参数的根际范围gt;4 mm,如O2、CO2、水和微生物,其中CO2 可扩散至距根表12 mm,这是由于根和根际微生物的呼吸作用造成的[20]。值得注意的是,微生物的根际范围是根系分泌物的2 倍。可见,微生物可捕获部分根系分泌物,根际对微生物的影响大于根系分泌物所能扩散的距离[9]。由上述研究可知,根际各个生化参数并非独立存在,而是处于剧烈的相互作用中(图1)。正因如此,精细刻画不同参数的准确范围,实现根际微生境生化过程的原位可视化尤为迫切。
2 原位可视化在根际生化过程中的应用
2.1 根际化学环境可视化
2.1.1 pH、O2 和CO2
pH、O2 和CO2 是影响根际生化过程及其功能的关键要素。早在20 世纪80 年代,有学者就进行了根际pH 值可视化的探索,将含有溴甲酚紫(pH 4.5~7.5) 或溴甲酚绿(pH 3.8~5.4)染料的琼脂粘附在根箱可拆卸侧的根土界面上,通过荧光激发、相机拍摄研究根际pH 的微区分布特征[21−22]。在此之后,基于荧光化学光学感应膜整合成像技术快速发展,成像对象逐渐扩展到O2、CO2、H2S、Fe2+、Mn2+、NH4+和温度等参数[23−26]。近年来,模块化成像系统 (VisiSens,PreSens Precision SensingGmbH,德国)[20]和平面光极分析仪 [Easysensor PO2100/1100,中科智感(南京) 环境科技有限公司][26]等商业化产品的出现,使得根际pH 值的可视化更加便捷高效。此外,依托于平面光极技术(PO),利用不同荧光化学光学感应膜实现了pH、O2 和CO2 等参数的同步定量可视化研究[27]。上述手段可通过在室内模拟及田间试验构建根土界面,测定pH、O2 和CO2 等参数在根际微域的时空分布特征,从而揭示根际微生境酸化、泌氧及CO2 排放等生物化学过程与功能,具有原位非破坏、实时性、高分辨率及用户友好的特性[26]。
2.1.2 矿质营养元素
矿质元素的根际空间分布特征表征目前主要采用薄膜扩散梯度(DGT)。该技术最初主要用于水体污染物和微量元素的有效性测定[28],近年来被广泛应用于土壤养分和污染物的生物有效性评价[11, 29−31]。DGT 主要由滤膜、扩散层和固定层组成,通过模拟根系吸收并耦合激光剥蚀电感耦合等离子体质谱仪(LA-ICP-MS) 获取元素的二维空间分布[32]。利用这一技术,可获取包括铁(Fe)、钙(Ca)、铜(Cu)、锰(Mn)、锌(Zn)、钼(Mo)、镁(Mg)、钾(K)、硫(S) 和磷(P) 等大部分矿质元素的根际空间分布特征[29]。例如,有研究利用DGT 成像,发现部分植物根表表现出P 耗竭特征,据此证实根系表面吸收P 速率高于P 从土壤向根表的扩散速率[19, 30, 33]。此外,利用纳米二次离子质谱技术(NanoSIMS) 亦可高分辨率绘制部分矿质元素(如P、Si、S、Fe 等) 的根际空间分布图,可在亚微米尺度上揭示植物−矿质元素−微生物的根际互作机理[4]。
2.1.3 污染物
与矿质营养元素类似,重金属等污染物的根际二维分布特征表征亦主要采用DGT 开展。Kreuzeder 等[34]首次采用DGT 同时得到了根际阳离子(Cu、Mn 和Zn) 和阴离子[P 和砷(As)] 的高分辨空间分布图像。基于DGT 元素分布表征和通量计算,发现水稻根尖O2 富集与酸化是造成根际土壤As、铅(Pb) 和Fe (Ⅱ) 活化的重要原因[35]。类似的,锑(Sb, Ⅲ) 在水稻根际沿根轴及根尖区域呈明显活化过程,且峰值出现在根表区域[36]。相反的,Smolders等[37]首次发现镉(Cd) 在玉米根际呈现耗竭现象,这主要是由于Cd 的吸收受扩散控制所致。镍(Ni) 超富集植物Odontarrhena serpyliffolia 和非富集植物Holcus lanatus 根际均显示Ni 耗竭现象,但富集型植物耗竭区范围为非富集型的4 倍[38]。Corzo 等[39]利用同步辐射微X 射线荧光光谱和X 射线吸收近边缘结构成像技术,研究了As 在甘蓝蕨和蜈蚣草根际−根界面的原位分布,发现As 可从富砷土壤中的根系向对照土壤中的根系迁移。
对于有机污染物而言,由于缺乏有效吸附有机污染物的膜材料,其根际空间分布至今仍未见报道,仅有少量研究发现了新兴有机污染物在植物根系内的分布特征。譬如,Wang 等[40]利用解吸电喷雾电离质谱(DESI-MS) 发现,全氟辛烷磺酸(PFOS) 和全氟辛酸(PFOA) 在美人蕉和再力花、风车草及芦苇根的表皮、皮层和维管束中有分布。近期,Chen 等[41]发现,全氟聚醚类(HFPO) 替代物和PFOA 主要累积在生菜根的表皮细胞和皮层中,而且在皮层中的含量远高于相邻表皮细胞。随着吸附膜材料研发的进步,有机污染物的根际空间分布将被逐步揭示。
2.2 根际微生物环境可视化
可视化对于深入揭示微生物与土壤−植物根际互作至关重要。早期的切片−体式显微镜方法存在难以区分目标微生物的缺陷[42]。荧光染料与表观荧光显微镜的结合,可以从背景中区分目标微生物体,进而实现细菌和真菌的空间定位和量化。与之相比,扫描和透射电子显微镜(SEM 和TEM) 的分辨率(0.5~1 nm) 要高得多,但所需的土壤固定、脱水、包埋和切片等步骤繁琐[10, 43]。近年来,荧光原位杂交(FISH)、纳米二次离子质谱技术(NanoSIMS)[ 4 4 ]和FISH-NanoSIMS 等技术逐渐应用到微生物可视化研究中。近期,Cao 等[45]开发了一种可容错编码的序贯荧光原位杂交(SEER-FISH) 技术,该方法可识别复杂群落中的不同微生物物种,在单细胞尺度上原位解析微生物物种之间以及微生物−宿主之间的相互作用。然而,现有研究主要采用室内培养基条件,微生物的根际原位可视化仍鲜有报道。此外,微生物可视化方法需要使用的部分仪器(如NanoSIMS)可用性较差,这限制了微生物可视化方法的进一步开发。
2.3 根际代谢物可视化
2.3.1 根系分泌物
根系分泌物能够改变根际土壤理化性质,是造成根际土和非根际土之间根本区别的主要原因[46]。根系分泌物的原位高分辨率成像方法开发尤其艰难,这是由于根系分泌物组成复杂,根际生物与非生物因子交织所致。大多数根系分泌物的采集方法为破坏式,且不同采样方法结果差异较大[14−15, 47],无法提供样品中目标分析物的准确空间分布。目前为止,根系分泌物的成像仍主要依赖于同位素标记[48−49],但其效果受多种因素影响,如根中1 4C 活性、根半径、根表到成像屏幕的距离、土壤中根系分泌物的数量等因素强烈影响成像信号的分布[50]。最为关键的是,同位素成像至今无法获得某一特定根系分泌物的根际空间分布特征,这极大地限制其广泛应用。
近年来,针对根系分泌物可视化研究的探索并未停止。Hoang 等[51]采用优化的荧光显色法获得了根际葡萄糖的分布图像,也有研究利用基质辅助激光解吸电离质谱(MALDI-MS) 获得地钱苹果酸分泌物的空间分布[52],但上述研究采用的人工基质与实际的根−土界面相差甚远。Tiziani 等[53]采用DGT-氢氧化锆结合凝胶,结合离子色谱表征了白羽扇豆根系分泌物柠檬酸的原位空间分布,但该方法过程较为复杂、容易交叉污染,分辨率较低。Veličković等[54]首次利用疏水性聚偏二氟乙烯(PVDF) 蛋白印迹膜和MALDI-MS 对根际代谢物进行了原位空间分布研究,发现了不同代谢物的空间分布特征。近期,Liu等[55]利用亲水性PVDF 膜耦合DESI-MS 分析,实现了根系分泌物的原位定量成像,具有操作简单、分辨率高、干扰少等特点。尽管已有方法仍存在分辨率低的局限性,但这无疑为根系分泌物的可视化带来了新的思路。上述方法与平面光极、原位酶谱等技术的耦合有望实现根系分泌物、矿质元素、pH、O2 和CO2 的同步空间可视化。
2.3.2 微生物代谢物
微生物代谢物的可视化可为重要生物活性分子的发现、微生物菌落的代谢交流等过程提供直观证据[56]。有学者耦合荧光原位杂交(FISH) 显微镜技术和高分辨率大气压基质辅助激光解吸/电离质谱技术,在同一组织切片上获得了宿主和共生体代谢物的空间分布[57−58],实现单细胞水平的空间代谢组学检测。此外,二次离子质谱(SIMS)亦被报道可用于单细胞水平下的微生物代谢物可视化[57]。可见,质谱成像已成为发现微生物重要代谢物的重要平台。不仅如此,微生物之间的化学对话亦获得可视化。通过质谱成像技术,Moree 等[59]观察到铜绿假单胞菌的吩嗪代谢物可被烟曲霉转化,造成毒性增强并诱导真菌产生铁载体。然而,上述有关微生物代谢物的可视化研究均基于培养基培养,原位根际土壤中微生物代谢物的空间分布特征仍鲜有报道。近期,有方法原位可视化获得了根际代谢物的空间分布,目标代谢物从小分子扩展到高分子量代谢物[54−55],但无法将根系分泌物和微生物代谢物区分开来。未来借助上述手段的改进,有望实现土壤微生物代谢物的原位高分辨可视化。
2.3.3 酶活性
根际酶活性的可视化目前主要依赖于原位酶谱技术。将含有特异性荧光标记底物吸附膜贴附在根土界面表面,底物与土壤酶反应生成的荧光产物信号可转换为酶活性及其空间分布信息[48, 60−61]。原位酶谱技术在十年间发展迅猛,目前主要应用于土壤C、N、P 和S 循环相关的水解酶和氧化酶[17]。借助其非损伤、原位的特点和空间分辨率较高的优势,原位酶谱技术克服了传统土壤酶学技术的不足,为揭示土壤微观生态过程及土壤−根系−微生物相互作用的微生境过程提供了直观的证据[60]。已有数篇文献综述了原位酶谱的特点与应用前景[17, 60, 62]。然而,原位酶谱尚缺乏统一的技术规程,仍存在操作方法混乱和应用范围较窄等问题[60]。
3 原位可视化多手段的耦合
多参数的同步原位可视化是解析根际生化过程的必由之路。近年来,原位可视化多手段的耦合逐渐引起学界的广泛关注,并在土壤学、植物营养、农业资源与环境等多学科领域取得重要进展(表1)。Wei 等[49]联合平面光极、酶谱和14C 成像进行pH、酶活性和根系分泌物3 种生化参数的同步可视化分析,量化并定位了水稻根表铁膜对根际微氧区域有机质稳定的贡献,进而评估了其对水稻土全球碳固存的作用。Liu 等[ 5 5 ]将DGT-LA-ICP-MS 与DESIMS成像分析相结合,表明DGT 与DESI 成像分析相结合在技术上是可行的,可以亚毫米分辨率同步定量测定元素和根系分泌物的根际空间分布。Li 等[29]利用平面光极(PO) 和薄膜扩散梯度(DGT) 等高分辨采样和分析技术,发现苦草根系泌氧和根际酸化改变了沉积物中微量金属的生物有效性,对Fe、As、Co 等元素的生物有效性起到抑制作用,而对Mn、Ni、Sb 和Cu 的生物有效性起到促进作用。由此可见,原位可视化多手段的耦合可在微观尺度上提高现有根际多参数的范围准确度。然而,目前的研究仅限于同步观测2~3 个生化过程指标,多参数的根际可视化亟需开展多手段的耦合。
多手段联用所面临的限制因子,是双方或多方的结合是否相互影响以及方法的兼容性如何。譬如,Hummel 等[30]通过DGT 与原位酶谱联用,发现狭叶羽扇豆根际存在磷损耗与磷酸酶富集现象。然而,原位酶谱所需的4-甲基伞形酮酰磷酸酯中含有微量磷,这可能影响DGT 测定磷的准确性,因此建议DGT 取样应先于酶谱分析,从而减小二者的相互影响。Védère 等[10]建议,在进行多种手段耦合时,首先进行不需要特定准备的方法(如X 射线或断层扫描),其次是平面探针或凝胶法(如酶谱或平面光极),然后是需要制备薄片的方法,最后是对物体表面造成不可逆地损坏的方法(如SEM、LA-ICP-MS)。因此,在进行多种方法联用时,有必要针对特定方法的样品制备需求,选定多手段联用的测试程序,从而最大限度减少方法间的相互影响,提高方法间的兼容性,以利于方法的推广。
4 展望
获取根际微生境生化过程的原位可视化证据,可促进我们对土壤热点区域发生的物质转化与能量流动的深入理解,有助于填补土壤“黑箱”的诸多研究空白。尽管近年来在根际过程的成像上取得了较多的重要进展(图2),然而在根际时空动态可视化、多参数成像技术及可视化的田间应用等方面仍然存在较多的不足,未来需从以下3 个方面展开更深入的研究:
1) 根际微生境多参数的同步可视化。在根际中,多种生化过程往往同时发生,且在空间上具有重叠特征。例如,缺磷胁迫下,植物根际pH、有效磷、根系分泌物、磷酸酶活性和微生物群落结构均发生响应,但上述多种参数的同步空间分布特征仍不清晰。已有关于根际可视化的研究大多仅观测2~3 个生化过程参数,亟需开展多手段的耦合,实现多参数的根际可视化。因此,技术开发仍然是多参数同步可视化的重要前提,特别是操作简单、耗时少、成本低的方法开发尤为迫切。此外,不同方法之间的兼容性和组合优化尚需开展更多的验证工作。
2) 根际微生境的时空动态可视化。时间动态是全面认识根际微生境中生物地球化学过程的关键,但已有的根际可视化研究仍较少涉及时间动力学,这主要是由于在构建时间序列时所需的样品制备以及分析繁琐所致,而分析仪器耗时较长且费用昂贵是限制其发展的另一重要因子。此外,除X 射线计算机断层扫描等技术外,大部分可视化技术仅能获取二维空间分布图。因此,在拓展二维空间可视化参数的同时,应积极开发根际三维乃至四维可视化方法。未来可从三维可视化技术开发、样品制备和数据整合方法等方面进行深入探究。
3) 根际原位可视化技术的田间应用。由于根际微生境空间分辨率的要求、田间条件的不可预测性,以及缺少便携式仪器,已有研究主要在实验室内模拟田间条件下,基于根箱或微根窗培养等方法构建根−土界面,探究根际生化参数的空间分布特征。然而,模拟获得的根−土界面可能与田间条件在根系构型、化学和微生物环境等诸多方面有较大差异。因此,亟需开展根际可视化技术在田间的应用验证。
参 考 文 献:
[ 1 ]Mariotte P, Mehrabi Z, Bezemer T M, et al. Plant–soil feedback:Bridging natural and agricultural sciences[J]. Trends in Ecology andEvolution, 2018, 33(2): 129−142.
[ 2 ]Zhang K, Rengel Z, Zhang F S, et al. Rhizosphere engineering forsustainable crop production: Entropy-based insights[J]. Trends inPlant Science, 2023, 28(4): 390−398.
[ 3 ]Keiluweit M, Bougoure J J, Nico P S, et al. Mineral protection of soilcarbon counteracted by root exudates[J]. Nature Climate Change,2015, 5(6): 588−595.
[ 4 ]Oburger E, Schmidt H. New methods to unravel rhizosphere processes[J]. Trends in Plant Science, 2016, 21(3): 243−255.
[ 5 ]Bicharanloo B, Bagheri Shirvan M, Keitel C, Dijkstra F A.Rhizodeposition mediates the effect of nitrogen and phosphorousavailability on microbial carbon use efficiency and turnover rate[J].Soil Biology and Biochemistry, 2020, 142: 107705.
[ 6 ]Edwards J, Johnson C, Santos-Medellín C, et al. Structure,variation, and assembly of the root-associated microbiomes of rice[J]. Proceedings of the National Academy of Sciences of theUnited States of America, 2015, 112(8): E911−E920.
[ 7 ]Crowther T W, Van Den Hoogen J, Wan J, et al. The global soilcommunity and its influence on biogeochemistry[J]. Science, 2019,365: eaav0550.
[ 8"]Wen T, Xie P H, Liu H W, et al. Tapping the rhizosphere metabolitesfor the prebiotic control of soil-borne bacterial wilt disease[J]. NatureCommunications, 2023, 14(1): 4497.
[ 9 ]Kuzyakov Y, Razavi B S. Rhizosphere size and shape: Temporaldynamics and spatial stationarity[J]. Soil Biology and Biochemistry,2019, 135: 343−360.
[10]Védère C, Vieublé Gonod L, Nunan N, Chenu C. Opportunitiesand limits in imaging microorganisms and their activities in soilmicrohabitats[J]. Soil Biology and Biochemistry, 2022, 174: 108807.
[11]Li C, Ding S M, Chen M S, et al. Visualizing biogeochemicalheterogeneity in soils and sediments: A review of advancedmicro-scale sampling and imaging methods[J]. Critical Reviewsin Environmental Science and Technology, 2023, 53(12): 1229−1253.
[12]Helliwell J R, Sturrock C J, Grayling K M, et al. Applications ofX-ray computed tomography for examining biophysical interactionsand structural development in soil systems: A review[J]. EuropeanJournal of Soil Science, 2013, 64(3): 279−297.
[13]申建波, 白洋, 韦中, 等. 根际生命共同体: 协调资源、环境和粮食安全的学术思路与交叉创新[J]. 土壤学报, 2021, 58(4): 805−813.
Shen J B, Bai Y, Wei Z, et al. Rhizobiont: An interdisciplinaryinnovation and perspective for harmonizing resources, environment,and food security[J]. Acta Pedologica Sinica, 2021, 58(4): 805−813.
[14]Oburger E, Jones D L. Sampling root exudates-mission impossible?[J]. Rhizosphere, 2018, 6: 116−133.
[15]Williams A, Langridge H, Straathof A L, et al. Comparing rootexudate collection techniques: An improved hybrid method[J]. SoilBiology and Biochemistry, 2021, 161: 108391.
[16]Hoefer C, Santner J, Puschenreiter M, Wenzel W W. Localized metalsolubilization in the rhizosphere of Salix smithiana upon sulfurapplication[J]. Environmental Science and Technology, 2015, 49(7):4522−4529.
[17]Bilyera N, Kuzyakov Y. Soil zymography: A decade of rapiddevelopment in microbial hotspot imaging[J]. Soil Biology andBiochemistry, 2024, 189: 109264.
[18]Holz M, Zarebanadkouki M, Kaestner A, et al. Rhizodepositionunder drought is controlled by root growth rate and rhizosphere watercontent[J]. Plant and Soil, 2018, 423(1): 429−442.
[19]Kreuzeder A, Santner J, Scharsching V, et al. In situ observation oflocalized, sub-mm scale changes of phosphorus biogeochemistry inthe rhizosphere[J]. Plant and Soil, 2018, 424(1): 573−589.
[20]Koop-Jakobsen K, Mueller P, Meier R J, et al. Plant-sedimentinteractions in salt marshes: An optode imaging study of O2, pH, andCO2 gradients in the rhizosphere[J]. Frontiers of Plant Science, 2018,9: 290993.
[21]Marschner H, Römheld V. In vivo measurement of root-induced pHchanges at the soil-root interface: Effect of plant species and nitrogensource[J]. Zeitschrift Filer Pflanzenphysiologie, 1983, 111(3): 241−251.
[22]Häussling M, Leisen E, Marschner H, Römheld V. An improvedmethod for non-destructive measurements of the pH at the root-soilinterface (rhizosphere)[J]. Journal of Plant Physiology, 1985, 117(4):371−375.
[23]Glud R N, Ramsing N B, Gundersen J K, Klimant I. Planar optrodes:A new tool for fine scale measurements of two-dimensional O2distribution in benthic communities[J]. Marine Ecology ProgressSeries, 1996, 140: 217−226.
[24]Larsen M, Santner J, Oburger E, et al. O2 dynamics in the rhizosphereof young rice plants (Oryza sativa L.) as studied by planar optodes[J].Plant and Soil, 2015, 390(1): 279−292.
[25]Blossfeld S, Schreiber C M, Liebsch G, et al. Quantitative imaging ofrhizosphere pH and CO2 dynamics with planar optodes[J]. Annals ofBotany, 2013, 112(2): 267−276.
[26]Li C, Ding S M, Yang L Y, et al. Planar optode: A two-dimensionalimaging technique for studying spatial-temporal dynamics of solutesin sediment and soil[J]. Earth-Science Reviews, 2019, 197: 102916.
[27]Borisov S M, Seifner R, Klimant I. A novel planar optical sensorfor simultaneous monitoring of oxygen, carbon dioxide, pH andtemperature[J]. Analytical and Bioanalytical Chemistry, 2011,400(8): 2463−2474.
[28]Davison W, Zhang H. In situ speciation measurements of tracecomponents in natural waters using thin-film gels[J]. Nature, 1994,367: 546−548.
[29]Li C, Ding S M, Chen M S, et al. Mechanistic insights into tracemetal mobilization at the micro-scale in the rhizosphere of Vallisneriaspiralis[J]. Science of the Total Environment, 2021, 806: 150735.
[30]Hummel C, Boitt G, Santner J, et al. Co-occurring increasedphosphatase activity and labile P depletion in the rhizosphere ofLupinus angustifolius assessed with a novel, combined 2D-imagingapproach[J]. Soil Biology and Biochemistry, 2021, 153: 107963.
[31]Fang W, Williams P N, Zhang H, et al. Combining multiple highresolutionin situ techniques to understand phosphorous availabilityaround rice roots[J]. Environmental Science and Technology, 2021,55(19): 13082−13092.
[32]Guan D X, He S X, Li G, et al. Application of diffusive gradients inthin-films technique for speciation, bioavailability, modeling andmapping of nutrients and contaminants in soils[J]. Critical Reviews inEnvironmental Science and Technology, 2021, 52(7): 3035−3079.
[33]Santner J, Zhang H, Leitner D, et al. High-resolution chemicalimaging of labile phosphorus in the rhizosphere of Brassica napus L.cultivars[J]. Environmental and Experimental Botany, 2012, 77:219−226.
[34]Kreuzeder A, Santner J, Prohaska T, Wenzel W W. Gel forsimultaneous chemical imaging of anionic and cationic solutesusing diffusive gradients in thin films[J]. Analytical Chemistry,2013, 85(24): 12028−12036.
[35]Williams P N, Santner J, Larsen M, et al. Localized flux maxima ofarsenic, lead, and iron around root apices in flooded lowland rice[J].Environmental Science and Technology, 2014, 48(15): 8498−8506.
[36]Fang W, Shi X Y, Yang D X, et al. In situ selective measurementbased on diffusive gradients in thin-films technique with mercaptofunctionalizedmesoporous silica for high-resolution imaging of SbIIIin soil[J]. Analytical Chemistry, 2020, 95(2): 3581−3588.
[37]Smolders E, Wagner S, Prohaska T, et al. Sub-millimeter distributionof labile trace element fluxes in the rhizosphere explains differentialeffects of soil liming on cadmium and zinc uptake in maize[J].Science of the Total Environment, 2020, 738: 140311.
[38]Álvarez-López V, Puschenreiter M, Santner J, et al. Evidence fornickel mobilisation in rhizosphere soils of Ni hyperaccumulatorOdontarrhena serpyllifolia[J]. Plant and Soil, 2021, 464: 89−107.
[39]Corzo Remigio A, Harris H H, Paterson D J, et al. Chemicaltransformations of arsenic in the rhizosphere–root interface ofPityrogramma calomelanos and Pteris vittata[J]. Metallomics,2023, 15(8): mfad047.
[40]Wang T T, Ying G G, Shi W J, et al. Uptake and translocationof perfluorooctanoic acid (PFOA) and perfluorooctanesulfonicacid (PFOS) by wetland plants: Tissue- and cell-level distributionvisualization with desorption electrospray ionization massspectrometry (DESI-MS) and transmission electron microscopyequipped with energy-dispersive spectroscopy (TEM-EDS)[J].Environmental Science and Technology, 2020, 54(10): 6009−6020.
[41]Chen L, Chen D J, Zhou S Y, et al. New insights into theaccumulation, transport, and distribution mechanisms ofhexafluoropropylene oxide homologues, important alternativesto perfluorooctanoic acid, in lettuce (Lactuca sativa L.)[J].Environmental Science and Technology, 2023, 57(26): 9702−9712.
[42]Tippkötter R, Ritz K. Evaluation of polyester, epoxy and acrylicresins for suitability in preparation of soil thin sections for in situbiological studies[J]. Geoderma, 1996, 69(1): 31−57.
[43]Vidal A, Remusat L, Watteau F, et al. Incorporation of 13C labelledshoot residues in Lumbricus terrestris casts: A combination oftransmission electron microscopy and nanoscale secondary ion mass spectrometry[J]. Soil Biology and Biochemistry, 2016, 93: 8−16.
[44]Li Q, Chang J J, Li L F, et al. Research progress of nano-scalesecondary ion mass spectrometry (NanoSIMS) in soil science:Evolution, applications, and challenges[J]. Science of the TotalEnvironment, 2023, 905: 167257.
[45]Cao Z H, Zuo W L, Wang L X, et al. Spatial profiling of microbialcommunities by sequential FISH with error-robust encoding[J].Nature Communications, 2023, 14(1): 1477.
[46]Badri D V, Vivanco J M. Regulation and function of root exudates[J].Plant, Cell and Environment, 2009, 32(6): 666−681.
[47]White R A, Rivas-Ubach A, Borkum M I, et al. The state ofrhizospheric science in the era of multi-omics: A practical guide toomics technologies[J]. Rhizosphere, 2017, 3: 212−221.
[48]Spohn M, Kuzyakov Y. Distribution of microbial- and root-derivedphosphatase activities in the rhizosphere depending on P availabilityand C allocation – Coupling soil zymography with 14C imaging[J].Soil Biology and Biochemistry, 2013, 67: 106−113.
[49]Wei L, Zhu Z K, Razavi B S, et al. Visualization and quantificationof carbon “rusty sink” by rice root iron plaque: Mechanisms, functions,and global implications[J]. Global Change Biology, 2022, 28(22):6711−6727.
[50]Holz M, Zarebanadkouki M, Carminati A, Kuzyakov Y. Visualizationand quantification of root exudation using 14C imaging: Challengesand uncertainties[J]. Plant and Soil, 2019, 437: 473−485.
[51]Hoang D T T, Rashtbari M, Anh L T, et al. Mutualistic interactionbetween arbuscular mycorrhiza fungi and soybean roots enhancesdrought resistant through regulating glucose exudation and rhizosphereexpansion[J]. Soil Biology and Biochemistry, 2022, 171: 108728.
[52]Gomez-Zepeda D, Frausto M, Nájera-González H-R, et al. Massspectrometry-based quantification and spatial localization of smallorganic acid exudates in plant roots under phosphorus deficiency andaluminum toxicity[J]. The Plant Journal, 2021, 106(6): 1791−1806.
[53]Tiziani R, Puschenreiter M, Smolders E, et al. Millimeter-resolutionmapping of citrate exuded from soil grown roots using a novel, lowinvasivesampling technique[J]. Journal of Experimental Botany,2021, 72(10): 3513−3525.
[54] Veličković D, Lin V S, Rivas A, et al. An approach for broad molecular imaging of the root-soil interface via indirect matrixassistedlaser desorption/ionization mass spectrometry[J]. SoilBiology and Biochemistry, 2020, 146: 107804.
[55]Liu C, Ding T X, Van Der Ent A, et al. A novel method for in situimaging of root exudates and labile elements reveals phosphorusdeficiency-induced mobilization of rare earth elements in therhizosphere of Phytolacca americana[J]. Plant and Soil, 2024,495(1): 13−26.
[56]Watrous J D, Dorrestein P C. Imaging mass spectrometry inmicrobiology[J]. Nature Reviews Microbiology, 2011, 9(9): 683−694.
[57]Geier B, Sogin E M, Michellod D, et al. Spatial metabolomics of insitu host–microbe interactions at the micrometre scale[J]. NatureMicrobiology, 2020, 5(3): 498−510.
[58]Bourceau P, Geier B, Suerdieck V, et al. Visualization of metabolitesand microbes at high spatial resolution using MALDI mass spectrometryimaging and in situ fluorescence labeling[J]. Nature Protocols, 2023,18(10): 3050−3079.
[59]Moree W J, Phelan V V, Wu C H, et al. Interkingdom metabolictransformations captured by microbial imaging mass spectrometry[J].Proceedings of the National Academy of Sciences of the United Statesof America, 2012, 109(34): 13811−13816.
[60]郭澍, 许佳扬, 魏晓梦, 等. 原位酶谱技术在土壤酶活性研究中的应用进展[J]. 生态学报, 2022, 42(3): 862−871.
Guo S, Xu J Y, Wei X M, et al. A review on the applications of insitu zymography in soil enzyme activity research[J]. Acta EcologicaSinica, 2022, 42(3): 862−871.
[61]Razavi B S, Zarebanadkouki M, Blagodatskaya E, Kuzyakov Y.Rhizosphere shape of lentil and maize: Spatial distribution of enzymeactivities[J]. Soil Biology and Biochemistry, 2016, 96: 229−237.
[62]Razavi B S, Zhang X C, Bilyera N, et al. Soil zymography: Simpleand reliable? Review of current knowledge and optimization of themethod[J]. Rhizosphere, 2019, 11: 100161.
[63]Ma X M, Mason-Jones K, Liu Y, et al. Coupling zymography withpH mapping reveals a shift in lupine phosphorus acquisition strategydriven by cluster roots[J]. Soil Biology and Biochemistry, 2019, 135:420−428.
作者简介:
顾文杰,博士,广东省农业科学院农业资源与环境研究所所长,三级研究员,博士生导师,国家农业环境微生物种质资源库(广东)主任,担任农业农村部南方植物营养与肥料重点实验室主任、广东省耕地保育与资源循环利用重点实验室主任、中国植物营养与肥料学会副理事长、广东省土壤学会副理事长。长期致力于微生物−植物互作与废弃物资源化利用等研究。近年主持国家自然科学基金项目、国家重点研发项目子课题、广东省自然科学基金、广东省科技计划重点项目及产学研项目等30 余项国家及省市级项目,发表论文50 余篇,获授权专利20 件,参编专著3 本。研究成果分别获教育部科技成果1 项、广东省科技进步奖二等奖2 项、广东省农业技术推广奖一等奖1 项和二等奖3 项。
刘 冲,博士,广东省农业科学院农业资源与环境研究所助理研究员,主要从事植物−微生物根际互作、土壤−植物系统微量元素行为等研究,主持国家自然科学基金青年基金、广东省自然科学基金面上项目等项目,作为主要成员参与国家重点研发计划、国家自然科学基金等多项国家、省部级项目。以第一作者在国内外重要学术刊物上发表学术论文10 余篇。获广东省农业技术推广奖二等奖1 项。
基金项目:广东省农业科学院农业资源与环境研究所科技条件平台创新项目(ZHS2023-01);国家自然科学基金项目(42307410);广东省基础与应用基础研究基金项目(2024A1515011125);广东省重点领域研发计划项目(2023B0202010027);“十四五”广东省农业科技创新九大主攻方向“揭榜挂帅”项目(2023SDZG08);广东省科技计划项目(2021B1212050020);十四五新兴学科团队项目(202121TD)。

