氮磷养分介导的植物–微生物互作研究进展
摘要: 氮、磷是植物生长发育所必需的大量营养元素,植物对氮磷养分的高效吸收利用与外界环境密切相关,同时氮磷养分又会影响植物的环境适应性。为此,植物如何整合适应不同的外部环境,特别是生物环境,以实现氮磷养分的高效吸收利用,是植物营养研究领域的前沿新热点。本文对近年来氮磷养分介导的植物−微生物互作研究进展进行综述,主要涉及氮磷养分介导的有益和有害生物互作研究,阐释了氮磷核心转录调控因子NLPs 和PHRs 在调控植物−微生物互作过程中的重要作用,总结了植物体内磷素感受蛋白SPXs 在调控植物−微生物互作过程中的功能多样性。此外,本文还对未来氮磷养分介导的植物−微生物互作研究的关键点进行了展望。
关键词: 氮; 磷; 植物微生物互作; 有益生物; 有害生物
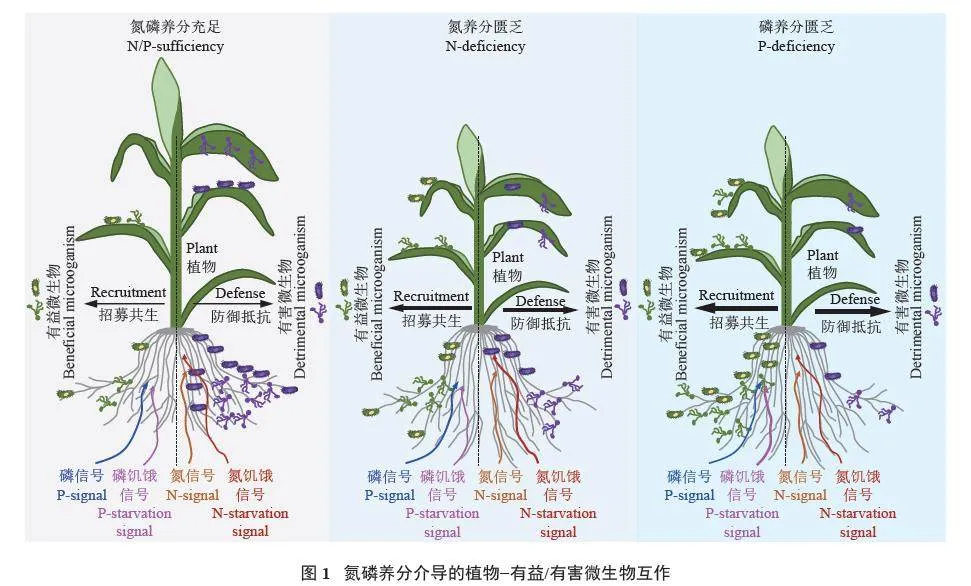
氮(nitrogen,N) 和磷(phosphorus,P) 是植物生长发育所必需的大量营养元素。对农作物而言,土壤逆境、养分状况、气候条件等诸多因素都会对其产量和品质产生重要影响,而氮磷养分在土壤中的生物有效性是诸多影响因素中的关键因子之一[1−2]。因此,植物氮磷养分的高效吸收利用是植物营养领域关注的重要科学问题。在复杂的生态环境中,植物氮磷养分的吸收利用不仅会受到土壤氮磷养分状态的影响,还会受到其它不同环境因素的影响,如不同的生物环境(有益微生物、病害、虫害等)。近年来,随着分子生物学研究的深入及基因测序技术的发展,越来越多的研究发现植物生长的生物环境与其氮磷养分的高效吸收利用密切相关,与之对应的氮磷养分的供给状态又会改变植物对生物环境的适应性。鉴于此,本文综述了近年来氮磷养分介导的植物生物互作研究进展,包括有益微生物与植物互作及有害微生物与植物互作研究(图1)。此外,本文还对未来氮磷养分介导的植物生物互作关键点进行了展望,并提出了土壤氮磷养分高效利用的生物改良新路径。
1 氮磷养分对植物与有益微生物互作的影响
植物与其它有益微生物相互作用主要建立的是互惠共生关系,通过物质交换满足各自所需。植物的氮磷养分状态对相关互作过程有重要影响。目前研究比较多的是氮磷养分对豆科植物与固氮菌之间共生互作过程的调控机制。根瘤菌与植物根系互作主要涉及以下几个过程:豆科植物分泌黄酮类物质到土壤招募固氮菌,同时固氮菌分泌结瘤因子(LCOs/NFs) 到根际,此后植物根系表皮细胞质膜上的结瘤因子受体蛋白感知并传递结瘤信号启动根瘤的生长发育并固氮[3−5]。当土壤环境中的硝酸盐缺乏时,豆科植物中根瘤菌的生物固氮过程会受到促进;相反,当土壤环境中的硝酸盐充足时,固氮菌诱导的根瘤形成过程会受到显著抑制[6−8]。在氮养分介导的豆科植物−根瘤菌互作过程中,硝酸根诱导的转录抑制子NLPs 是调控生物固氮的重要因子[8]。硝酸根通过诱导NLPs 蛋白磷酸化并进入细胞核抑制下游结瘤基因的表达,来抑制根系结瘤。研究发现,当百脉根中的LjNLP4 基因或苜蓿中的MtNLP1/3/4/5 基因功能活性受到抑制时,即使外界硝酸盐含量很高时也能产生大量根瘤;而当LjNLP4 和MtNLP1 基因表达时,高硝酸盐浓度会极大地抑制根瘤的形成[9]。
与硝酸盐类似,磷酸盐在根瘤菌与植物的互作过程中也发挥重要作用。根瘤菌诱导的根瘤生长需要充足的无机磷来维持。低磷胁迫通过抑制根瘤菌中结瘤因子的合成及固氮酶活性,或者通过抑制根瘤生长来影响根瘤菌的生物固氮[10−13]。此外,低磷胁迫还可以通过抑制光合作用来限制宿主植物向根瘤菌供给碳源,从而抑制根瘤的生长[14]。在磷养分介导的豆科植物−根瘤菌互作分子调控网络中,磷养分的核心调控因子PHOSPHATE STARVATION RESPONSE(PHR)、PHR-like (PHL) 和质膜上的无机磷转运蛋白PHT1 发挥着重要作用[10, 15]。在大豆中,GmPHR1 和GmPHT1;11 在根瘤组织不同细胞中表达,以调控根瘤中无机磷的供给,从而影响根瘤的形成和生长。进一步的研究发现,增强GmPHT1;11 基因的表达可以显著提高根瘤重量,但根瘤数目会减少[15]。此外,大豆中的磷素感受蛋白SPXs 对根瘤的生长调控也发挥关键作用,最近的研究发现增强大豆根瘤组织特异表达的GmSPX5 基因能显著促进根瘤的生长[16]。另外有研究表明,低磷胁迫可以通过诱导miR2111的表达,从而抑制结瘤因子TML 的表达,最终抑制根瘤产生和生长[17]。
氮磷养分除了在调控植物−根瘤菌互作过程中发挥作用以外,还在调控植物与丛枝菌根真菌(AM) 的互作过程中发挥关键作用,其中磷养分发挥主导作用[18]。按照目前的研究认知有超过80% 的植物能与AM 建立互作共生关系,宿主植物为AM 提供碳源,而AM 向植物提供氮磷等矿质营养和水分[19−20]。植物−AM 共生体系的建成主要涉及以下几个过程:植物合成的独脚金内酯(SL) 分泌到根际诱导AM 孢子萌发;萌发孢子产生的AM 菌丝分泌菌根因子(Mycfactor);根系表皮细胞质膜上的菌根因子受体蛋白识别菌根因子并启动植物体内的信号响应,使菌丝能进入植物根系;此后菌丝进入皮层细胞形成植物−AM的共生体,并展开物质交换。在植物和AM 的互作过程中,磷养分发挥着非常关键的作用,磷匮乏时会促进AM−植物共生体的形成,而磷充足时则会抑制AM 在植物根系的定殖[21−23]。目前,对磷养分依赖的植物−AM 共生体系建成调控的分子机制已经有比较深入的了解,其中由磷养分信号激活转录调控因子PHRs 和磷信号抑制因子SPXs 组成的磷饥饿应答调控系统发挥着关键作用[24]。当磷养分匮乏时,PHRs可以激活磷养分依赖的独脚金内酯生物合成过程,并将独脚金内酯分泌到根际,促进AM 孢子萌发,相反,当磷养分充足时该过程受到抑制[25]。在水稻中,低磷胁迫可以通过激活PHRs 来诱导转录因子NSP1/2 的转录,NSP1/2 则进一步直接激活独脚金内酯合成路径关键酶编码基因D10、D17、D27 等的表达,以此增加独脚金内酯合成量[26]。此外,研究发现大量AM 诱导的基因含有PHRs 的结合基序P1BS 元件[27],进一步的研究表明这些基因大部分都是PHRs直接调控的下游基因[24, 28]。因此,当PHRs 基因突变时,AM 的定殖和生长过程都会受到显著的抑制[24, 28−29]。当SPXs 基因突变时,对AM 的定殖和生长的影响在不同物种中有所不同。水稻spx1/2/3/5 四重突变体和番茄spx1 突变体表现出较高的根系定殖率,增强这些基因的表达则导致AM 在根系中的定殖率下降[24, 29]。而在豆科紫花苜蓿中,在含丛枝的细胞中特异增强表达SPX1/3 或突变这两个基因都减少了AM 的定殖[30]。这些研究结果暗示,不同物种SPX 基因在AM 的定殖和生长调控中发挥不同甚至是相反的功能。
氮对AM 的定殖和生长都有影响,但是影响有限,没有磷养分显著[18, 31]。研究发现,高硝酸盐(NO3–)对AM 共生的抑制作用强于铵盐(NH4+)[32]。与无机磷对AM 定殖的抑制相比,植物氮养分状态对AM 定殖的影响则比较复杂,而且其影响程度总是与植物无机磷的状态相关。尽管达到抑制AM 的确切氮浓度因植物种类和实验设计的差异而有所不同,一般情况下低氮和中等氮供应会增加菌根定殖水平,高氮则会减少菌根定殖[33−34]。高氮抑制AM 共生的主要机制可能是由于氮诱导的土壤酸化,因为土壤酸化会显著抑制AM 的定殖率[35]。此外,由于氮和磷之间存在密切的分子信号互作,硝酸盐可以激活植物对磷酸盐的获取和利用[36−37],这说明植物氮磷供给状况对AM 共生的影响也是相互关联的。一方面,植株氮素对AM 共生的影响受植株无机磷状态的影响,当无机磷有限时,供应氮会显著提高菌根定殖水平[38],在磷素充足而氮匮乏条件下,植株中氮浓度和氮磷比都低,导致AM 定殖量增加,相比之下,在氮含量和氮磷比都高的植株中,AM 定殖水平显著降低[39]。另一方面,在高磷供应下观察到的AM定殖抑制也取决于植株的氮状态。研究发现,氮饥饿胁迫可以部分解除高无机磷对AM 定殖的抑制作用[40]。因此,低无机磷促进菌根共生,而氮对菌根共生的调节依赖于植物无机磷状态。
氮磷养分除了对根瘤菌和丛枝菌根真菌的定殖和生长有重要影响外,还会影响其它很多有益真菌或细菌与植物的共生过程。尽管目前的研究发现,毛霉菌属根系内生菌的共生和木本植物外生菌根真菌的共生对植物获取无机磷营养也有重要贡献[41−43],但人们对这些相关的分子调控机制仍然不清楚 [44]。还没有证据表明植物磷饥饿分子调控系统能参与调节根系内生菌或外生菌根真菌的功能,但其他真菌可以通过转运磷来满足没有AM 共生的植物的磷需求,这些植物可以依靠其他真菌(除AM 真菌外) 分泌有机酸、产生丛生根或者与其他菌的相互作用来加强磷的吸收[45−46]。此外,研究发现解磷菌在增加植物磷养分获取能力,提高环境适应性,提高作物产量等方面都发挥着积极作用[47]。但是有关植物的磷饥饿的分子调控系统如何影响或调控不同类型解磷菌的定殖仍然研究的比较少,未来将会是亟需突破的关键点。
2 氮磷养分信号对植物与有害微生物互作的调控
氮磷养分系统除了介导植物与有益微生物的互作外,还影响植物与病原微生物之间的互作。植物的目标是隔离养分防止被病原体获取,但病原体的目标可能是操纵植物使其能够获得宿主组织中的养分,以实现病原微生物的增殖和繁衍[48−49]。早在20世纪60 年代,研究就发现大量施用氮肥会增加稻瘟病菌的侵染率;相反少量施用氮肥可以降低侵染率,但同时会导致水稻的产量急剧下降[50]。在换了试验时间和地域后,不同的研究团队也开展了类似试验,并得出了相似的结论[51−53];同时,研究也发现稻瘟病易感型植株总氮和氨基酸态氮的积累量高于抗病型植株[53]。与之相类似,在磷养分−稻瘟病生理互作关系的研究中发现,大量磷肥的施用也会加剧稻瘟病害发生的风险[54]。这些研究暗示氮磷养分在调控有害微生物与植物互作的免疫应答中发挥着重要作用。
近年来,有关氮磷养分介导的植物免疫应答分子调控研究也取得了部分进展。其中氮养分对植物免疫应答的分子调控研究报道相对较少,而对磷养分介导的植物免疫应答分子调控研究相对较多。尽管氮养分对植物免疫应答的分子调控研究报道相对有限,但还是开展了大量氮养分介导的植物−有害微生物互作生理研究[55]。研究发现,植物氮养分通过植物体内初级和次级代谢来调节细胞结构和组成,大量的氮养分供应会减少植物表面的蜡质层,减少或延迟细胞壁木质素沉积等[56−57],这些结构缺陷将有利于病原微生物和草食性昆虫对植物的侵袭[58]。此外,氮养分可以通过调控一些酶的活性、代谢物含量或植物抗氧化系统来影响植物的免疫抗性[55]。由于植物氮养分吸收形态和代谢利用的复杂性,导致氮养分对植物−有害微生物互作的影响因氮养分形态的不同而不同。研究发现,硝态氮可以通过增强硝酸还原酶的活性来促进一氧化氮(NO) 的产生,而铵态氮则会抑制一氧化氮合成[59]。由于一氧化氮是植物免疫应答中的重要信号物质,其信号路径能调控大量致病基因的表达,也能调控过敏反应引起的细胞程序性死亡,还能促进植物抗毒素和水杨酸的合成[60−62],因此这些研究说明硝态氮正调控植物对病原微生物的免疫防御。然而铵态氮主要通过调控氨基酸代谢来影响病原体的发育或防御相关基因的表达[63],最新的研究也发现铵态氮转运蛋白AMT 1.1 负调控植物的免疫防御[64]。上述研究说明,氮养分介导的植物免疫应答调控具有非常高的复杂性和多样性。
对于磷养分介导的植物免疫应答调控虽然十分复杂,但主要是围绕磷养分核心调控系统P H R -SPX 展开。在拟南芥的研究中发现,当用合成菌群对phr 突变体进行处理时,植物免疫相关的基因在突变体中明显变化,包括水杨酸信号上调和茉莉酸信号下调的基因,这导致拟南芥中phr1 phl1 双突变对细菌的抗性更强[65]。然而,一项研究发现phr1 突变增加了植物对一种致病性卵霉菌肉桂疫霉的易感性[66]。与此对应,水稻中的研究发现增强PHRs 活性能显著提高水稻对稻瘟病菌的抗性[67−68]。此外,研究表明拟南芥中的PHR1 可以直接激活 RALFs,从而干扰植物免疫防御系统的激活[69−70]。这些研究说明,磷饥饿的分子调控系统介导的免疫应答是一个非常复杂的调控系统,面对不同的微生物,植物可能采取截然不同的应对策略。最近也有研究发现,拟南芥磷高积累的突变体nla 和pho2 表现出对真菌病原体抗性比野生型植株更高的现象[71]。与此类似,研究发现多聚磷酸肌醇激酶编码基因IPK1 和IPTK1 功能失活时,导致无机磷积累增加,对细菌病原体的抵抗力也增强[72]。与此不同,当拟南芥中磷吸收蛋白编码基因PHT1;1 和PHT1;4 突变时,植株的磷含量降低,同时对致病细菌青枯菌的抗性增强[73]。这说明植物体内磷含量高低不是决定植物免疫应答强度的唯一路径,磷信号的转导也应该发挥关键作用。
3 研究展望
植物氮磷养分利用能力除了受植物自身遗传特性和物理化学环境影响外,还受到生物环境的影响。生物环境如何影响植物氮磷养分的吸收利用效率及氮磷养分如何影响植物的生物环境适应性将持续为多领域交叉研究的热点和难点。尽管目前对植物−氮磷养分−生物互作的研究取得了明显的进步,未来还需持续深入探讨以下问题:1) 氮磷养分应答调控系统如何帮助植物整合免疫信号、共生信号及养分信号;2) 氮磷养分应答调控系统如何根据氮磷养分状态来区分和应对不同的生物信号(包括有益或有害);3) 如何通过对作物进行精准的养分管理,来帮助作物更好地应对不同的病虫害,或者帮助作物高效地与不同有益微生物进行互作;4) 氮养分分子调控网络中的关键调控因子是否及如何参与氮依赖的植物生物互作过程;5) 如何利用遗传改良的方式实现氮磷养分高效与抗病高效的协同改良。
参 考 文 献:
[ 1 ]Chiou T J, Lin S I. Signaling network in sensing phosphate availabilityin plants[J]. Annual Review of Plant Biology, 2011, 62(1): 185−206.
[ 2 ]Xuan W, Beeckman T, Xu G H. Plant nitrogen nutrition: Sensing andsignaling[J]. Current Opinion in Plant Biology, 2017, 39: 57−65.
[ 3 ]Yang J, Lan L Y, Jin Y, et al. Mechanisms underlying legumerhizobiumsymbioses[J]. Journal of Integrative Plant Biology, 2022,64(2): 244−267.
[ 4 ]Suzaki T, Kawaguchi M. Root nodulation: A developmental programinvolving cell fate conversion triggered by symbiotic bacterial infection[J]. Current Opinion in Plant Biology, 2014, 21: 16−22.
[ 5 ]Poole P, Ramachandran V, Terpolilli J. Rhizobia: From saprophytesto endosymbionts[J]. Nature Reviews Microbiology, 2018, 16(5): 291−303.
[ 6 ]Nishida H, Suzaki T. Nitrate-mediated control of root nodule symbiosis[J]. Current Opinion in Plant Biology, 2018, 44: 129−136.
[ 7 ] Nishida H, Tanaka S, Handa Y, et al. A NIN-LIKE PROTEIN mediates nitrate-induced control of root nodule symbiosis in Lotusjaponicus[J]. Nature Communications, 2018, 9(1): 499.
[ 8 ]Soyano T, Shimoda Y, Hayashi M. NODULE INCEPTIONantagonistically regulates gene expression with nitrate in Lotusjaponicus[J]. Plant and Cell Physiology, 2015, 56(2): 368−376.
[ 9 ]Marchive C, Roudier F, Castaings L, et al. Nuclear retention of thetranscription factor NLP7 orchestrates the early response to nitrate inplants[J]. Nature Communications, 2013, 4(1): 1713.
[10]Nussaume L, Kanno S, Javot H, et al. Phosphate import in plants:Focus on the PHT1 transporters[J]. Frontiers in Plant Science, 2011,2: 83.
[11]Sulieman S, Tran L S P. Phosphorus homeostasis in legume nodulesas an adaptive strategy to phosphorus deficiency[J]. Plant Science,2015, 239: 36−43.
[12]Liese R, Schulze J, Cabeza R A. Nitrate application"or P deficiencyinduce a decline in Medicago truncatula N2-fixation by similarchanges in the nodule transcriptome[J]. Scientific Reports, 2017,7(1): 46264.
[13]Ma Y L, Chen R J. Nitrogen and phosphorus signaling and transportduring legume-rhizobium symbiosis[J]. Frontiers in Plant Science,2021, 12: 683601.
[14]Hernández G, Valdés-López O, Ramírez M, et al. Global changes inthe transcript and metabolic profiles during symbiotic nitrogenfixation in phosphorus stressed common bean plants[J]. PlantPhysiology, 2009, 151(3): 1221−1238.
[15]Lu M Y, Cheng Z Y, Zhang X M, et al. Spatial divergence of PHRPHT1modules maintains phosphorus homeostasis in soybeannodules[J]. Plant Physiology, 2020, 184(1): 236−250.
[16]Zhuang Q L, Xue Y B, Yao Z F, et al. Phosphate starvationresponsive GmSPX5 mediates nodule growth through interactionwith GmNF-YC4 in soybean (Glycine max)[J]. The Plant Journal,2021, 108(5): 1422−1438.
[17]Tsikou D, Yan Z, Holt D B, et al. Systemic control of legumesusceptibility to rhizobial infection by a mobile microRNA[J].Science, 2018, 362: 233−236.
[18]Rui W J, Mao Z P, Li Z F. The roles of phosphorus and nitrogennutrient transporters in the arbuscular mycorrhizal symbiosis[J].International Journal of Molecular Sciences, 2022, 23(19): 11027.
[19]Wang W X, Shi J C, Xie Q J, et al. Nutrient exchange and regulationin arbuscular mycorrhizal symbiosis[J]. Molecular Plant, 2017, 10(9):1147−1158.
[20]Wipf D, Krajinski F, van Tuinen D, et al. Trading on the arbuscularmycorrhiza market: From arbuscules to common mycorrhizal networks[J]. New Phytologist, 2019, 223(3): 1127−1142.
[21]Graham J H, Leonard R T, Menge J A. Membrane-mediated decreasein root exudation responsible for phosphorus inhibition of vesiculararbuscularmycorrhiza formation[J]. Plant Physiology, 1981, 68(3):548−552.
[22]Thomson B D, Robson A D, Abbott L K. Effects of phosphorus onthe formation of mycorrhizas by Gigaspora calospora and Glomusfasciculatum in relation to root carbohydrates[J]. New Phytologist,1986, 103(3): 751−765.
[23] Elias K S, Safir G R. Hyphal elongation of Glomus fasciculatus in response to root exudates[J]. Applied and Environmental Microbiology,1987, 53(8): 1928−1933.
[24]Shi J C, Zhao B Y, Zheng S, et al. A phosphate starvation responsecenterednetwork regulates mycorrhizal symbiosis[J]. Cell, 2021,184(22): 5527−5540.
[25]Paries M, Gutjahr C. The good, the bad, and the phosphate: Regulationof beneficial and detrimental plant–microbe interactions by the plantphosphate status[J]. New Phytologist, 2023, 239(1): 29−46.
[26]Yuan K, Zhang H, Yu C J, et al. Low phosphorus promotes NSP1-NSP2 heterodimerization to enhance strigolactone biosynthesis andregulate shoot and root architecture in rice[J]. Molecular Plant, 2023,16(11): 1811−1831.
[27]Lota F, Wegmüller S, Buer B, et al. The cis-acting CTTC-P1BSmodule is indicative for gene function of LjVTI12, a Qb-SNAREprotein gene that is required for arbuscule formation in Lotus japonicus[J]. The Plant Journal, 2013, 74(2): 280−293.
[28]Das D, Paries M, Hobecker K, et al. PHOSPHATE STARVATIONRESPONSE transcription factors enable arbuscular mycorrhizasymbiosis[J]. Nature Communications, 2022, 13(1): 477.
[29]Liao D H, Sun C, Liang H Y, et al. SlSPX1–SlPHR complexesmediate the suppression of arbuscular mycorrhizal symbiosis byphosphate repletion in tomato[J]. The Plant Cell, 2022, 34(10):4045−4065.
[30]Wang P, Snijders R, Kohlen W, et al. Medicago SPX1 and SPX3regulate phosphate homeostasis, mycorrhizal colonization, andarbuscule degradation[J]. The Plant Cell, 2021, 33(11): 3470−3486.
[31]Shi J C, Wang X L, Wang E T. Mycorrhizal symbiosis in plantgrowth and stress adaptation: From genes to ecosystems[J]. AnnualReview of Plant Biology, 2023, 74(1): 569−607.
[32]Nagy R, Karandashov V, Chague W, et al. The characterization ofnovel mycorrhiza-specific phosphate transporters from Lycopersiconesculentum and Solanum tuberosum uncovers functional redundancyin symbiotic phosphate transport in solanaceous species[J]. The PlantJournal, 2005, 42(2): 236−250.
[33]Mashiguchi K, Seto Y, Yamaguchi S. Strigolactone biosynthesis,transport and perception[J]. The Plant Journal, 2021, 105(2): 335−350.
[34]Govindarajulu M, Pfeffer P E, Jin H R, et al. Nitrogen transfer in thearbuscular mycorrhizal symbiosis[J]. Nature, 2005, 435: 819−823.
[35]Guether M, Neuhäuser, B, Balestrini R, et al. A mycorrhizal-specificammonium transporter from Lotus japonicus acquires nitrogenreleased by arbuscular mycorrhizal fungi[J]. Plant Physiology, 2009,150(1): 73−83.
[36]Walder F, Brulé D, Koegel S, et al. Plant phosphorus acquisition in acommon mycorrhizal network: Regulation of phosphate transportergenes of the Pht1 family in sorghum and flax[J]. New Phytologist,2015, 205(4): 1632−1645.
[37]Kojima S, Konishi N, Beier M P, et al. NADH-dependent glutamatesynthase participated in ammonium assimilation in Arabidopsisroot[J]. Plant Signaling amp; Behavior, 2014, 9(8): e29402.
[38]Volpe V, Giovannetti M, Sun X G, et al. The phosphate transportersLjPT4 and MtPT4 mediate early root responses to phosphate status innonmycorrhizal roots[J]. Plant, Cell amp; Environment, 2016, 39(3):660–671.
[39]Gaude N, Bortfeld S, Duensing N, et al. Arbuscule-containing andnon-colonized cortical cells of mycorrhizal roots undergo extensiveand specific reprogramming during arbuscular mycorrhizal development[J]. The Plant Journal, 2012, 69(3): 510−528.
[40]Filiz E, Akbudak M A. Ammonium transporter 1 (AMT1) genefamily in tomato (Solanum lycopersicum L. ): Bioinformatics,physiological and expression analyses under drought and saltstresses[J]. Genomics, 2020, 112(5): 3773−3782.
[41]Hoysted G A, Field K J, Sinanaj B, et al. Direct nitrogen, phosphorusand carbon exchanges between Mucoromycotina ‘fine root endophyte’fungi and a flowering plant in novel monoxenic cultures[J]. NewPhytologist, 2023, 238(1): 70−79.
[42]Bücking H, Heyser W. Uptake and transfer of nutrients inectomycorrhizal associations: Interactions between photosynthesisand phosphate nutrition[J]. Mycorrhiza, 2003, 13(2): 59−68.
[43]Cairney J W G. Ectomycorrhizal fungi: The symbiotic route to theroot for phosphorus in forest soils[J]. Plant and Soil, 2011, 344:51–71.
[44]Garcia K, Delaux P M, Cope K R, Ané J M. Molecular signalsrequired for the establishment and maintenance of ectomycorrhizalsymbioses[J]. New Phytologist, 2015, 208(1): 79−87.
[45]Werner G D A, Cornelissen J H C, Cornwell W K, et al. Symbiontswitching and alternative resource acquisition strategies drivemutualism breakdown[J]. Proceedings of the National Academy ofSciences, 2018, 115(20): 5229−5234.
[46]Almario J, Fabiańska I, Saridis G, Bucher M. Unearthing theplant–microbequid pro quo in root associations with beneficialfungi[J]. New Phytologist, 2022, 234(6): 1967−1976.
[47]Cheng Y Y, Narayanan M, Shi X J, et al. Phosphate-solubilizingbacteria: Their agroecological function and optimistic application forenhancing agro-productivity[J]. Science of the Total Environment,2023, 901: 166468.
[48]Radtke A L, O’Riordan M X D. Intracellular innate resistance tobacterial pathogens[J]. Cellular Microbiology, 2006, 8(11): 1720−1729.
[49]Hood M I, Skaar E P. Nutritional immunity: Transition metals at thepathogen-host interface[J]. Nature Reviews Microbiology, 2012,10(8): 525−537.
[50]Rao A A. Nitrogenous manuring in relation to blast disease of rice[J].Proceedings/Indian Academy of Sciences, 1964, 59(3): 173−184.
[51]Matsuyama N, Dimond A E. Effect of nitrogenous fertilizer onbiochemical processes that could affect lesion size of rice blast[J].Phytopathology, 1973, 63(9): 1202−1203.
[52]Amin K S, Venkatarao G. Rice blast control by nitrogen management[J]. Journal of Phytopathology, 1979, 96(2): 140−145.
[53]Long D H, Lee F N, TeBeest D O. Effect of nitrogen fertilization ondisease progress of rice blast on susceptible and resistant cultivars[J].Plant Disease, 2000, 84(4): 403−409.
[54]Campos-Soriano L, Bundó M, Bach-Pages M, et al. Phosphate excessincreases susceptibility to pathogen infection in rice[J]. MolecularPlant Pathology, 2020, 21(4): 555−570.
[55] Sun Y M, Wang M, Mur L A J, et al. Unravelling the roles of nitrogen nutrition in plant disease defences[J]. International Journalof Molecular Sciences, 2020, 21(2): 572.
[56]Plavcová L, Hacke U G, Almeida-Rodriguez A M, et al. Geneexpression patterns underlying changes in xylem structure andfunction in response to increased nitrogen availability in hybridpoplar[J]. Plant, Cell amp; Environment, 2013, 36(1): 186–199.
[57]Camargo E L O, Nascimento L C, Soler M, et al. Contrastingnitrogen fertilization treatments impact xylem gene expressionand secondary cell wall lignification in Eucalyptus[J]. BMC PlantBiology, 2014, 14: 256.
[58]Kováčik J, Klejdus B, Bačkor M, Repčák M. Phenylalanineammonia-lyase activity and phenolic compounds accumulation innitrogen-deficient Matricaria chamomilla leaf rosettes[J]. PlantScience, 2007, 172(2): 393−399.
[59]Modolo L V, Augusto O, Almeida I M G, et al. Nitrite as the majorsource of nitric oxide production by Arabidopsis thalianain responseto Pseudomonas syringae[J]. FEBS Letters, 2005, 579(17): 3814−3820.
[60]Mur L A J, Santosa I E, Laarhoven L J J, et al. Laser photoacousticdetection allows in planta detection of nitric oxide in tobaccofollowing challenge with avirulent and virulent Pseudomonassyringae pathovars[J]. Plant Physiology, 2005, 138(3): 1247−1258.
[61]Chen J, Vandelle E, Bellin D, Delledonne M. Detection and functionof nitric oxide during the hypersensitive response in Arabidopsisthaliana: Where there’s a will there’s a way[J]. Nitric Oxide, 2014,43: 81−88.
[62]Modolo L V, Cunha F Q, Braga M R, Salgado L. Nitric oxidesynthase-mediated phytoalexin accumulation in soybean cotyledonsin response to the Diaporthe phaseolorum f. sp. meridionalis elicitor[J]. Plant Physiology, 2002, 130(3): 1288−1297.
[63]Seifi H S, Van Bockhaven J, Angenon G, Höfte M. Glutamatemetabolism in plant disease and defense: Friend or foe?[J]. MolecularPlant-Microbe Interactions, 2013, 26(5): 475−485.
[64] Pastor V, Gamir J, Camañes G, et al. Disruption of the ammonium transporter AMT1.1 alters basal defenses generating resistance againstPseudomonas syringae and Plectosphaerella cucumerina[J].Frontiers in Plant Science, 2014, 5: 231.
[65]Castrillo G, Teixeira P J P L, Paredes S H, et al. Root microbiotadrive direct integration of phosphate stress and immunity[J]. Nature,2017, 543: 513−518.
[66]Eshraghi L, Anderson J P, Aryamanesh N, et al. Suppression of theauxin response pathway enhances susceptibility to Phytophthoracinnamomi while phosphite-mediated resistance stimulates the auxinsignalling pathway[J]. BMC Plant Biology, 2014, 14: 68.
[67]He Y Q, Zhao Y, Hu J T, et al. The OsBZR1–OsSPX1/2 modulefine-tunes the growth–immunity trade-off in adaptation to phosphateavailability in rice[J]. Molecular Plant, 2024, 17(2): 258−276.
[68]Kong Y Z, Wang G, Chen X, et al. OsPHR2 modulates phosphatestarvation-induced OsMYC2 signalling and resistance to Xanthomonasoryzae pv. oryzae[J]. Plant, Cell amp; Environment, 2021, 44(10): 3432-3444.
[69]Tang J, Wu D S, Li X X, et al. Plant immunity suppression viaPHR1-RALF-FERONIA shapes the root microbiome to alleviatephosphate starvation[J]. EMBO Journal, 2022, 41(6): e109102.
[70]Stegmann M, Monaghan J, Smakowska-Luzan E, et al. The receptorkinase FER is a RALF-regulated scaffold controlling plant immunesignaling[J]. Science, 2017, 355: 287−289.
[71]Val-Torregrosa B, Bundo M, Mallavarapu M D, et al. Loss-offunctionof NITROGEN LIMITATION ADAPTATION confersdisease resistance in Arabidopsis by modulating hormone signalingand camalexin content[J]. Plant Science, 2022, 323: 111374.
[72]Gulabani H, Goswami K, Walia Y, et al. Arabidopsis inositolpolyphosphate kinases IPK1 and ITPK1 modulate crosstalk betweenSA-dependent immunity and phosphate-starvation responses[J]. PlantCell Reports, 2022, 41(2): 347−363.
[73]Dindas J, DeFalco T A, Yu G, et al. Direct inhibition of phosphatetransport by immune signaling in Arabidopsis[J]. Current Biology,2022, 32(2): 488−495.
作者简介:
阮文渊,博士,中国农业科学院农业资源与农业区划研究所研究员,博士生导师。担任中国植物营养与肥料学会第十届植物营养分子生理专业委员会副主任委员、国际合作工作委员会和青年工作委员会委员。入选国家“优青”、中国农业科学院“领军人才”、“青年英才”等人才支持计划。主要从事作物高效利用土壤氮磷养分的分子调控机理研究,相关成果发表在Nature Plants、Molecular Plant、Plant Cell、PNAS 等国际学术期刊。主持国家自然科学基金青年项目、面上项目、优秀青年科学基金项目、国家重点研发计划子课题等科研项目,获中国农业科学院大北农人才奖和中国农业科学院杰出科技创新奖。
基金项目:国家自然科学基金优秀青年基金项目(32222078);国家自然科学基金面上项目(32272810);中国农业科学院创新工程项目。

