大泰竹叶绿体全基因组及系统进化分析
李傅达 黄欣 韦波 毛纯 秦波 黄红宝 孙开道 周启华


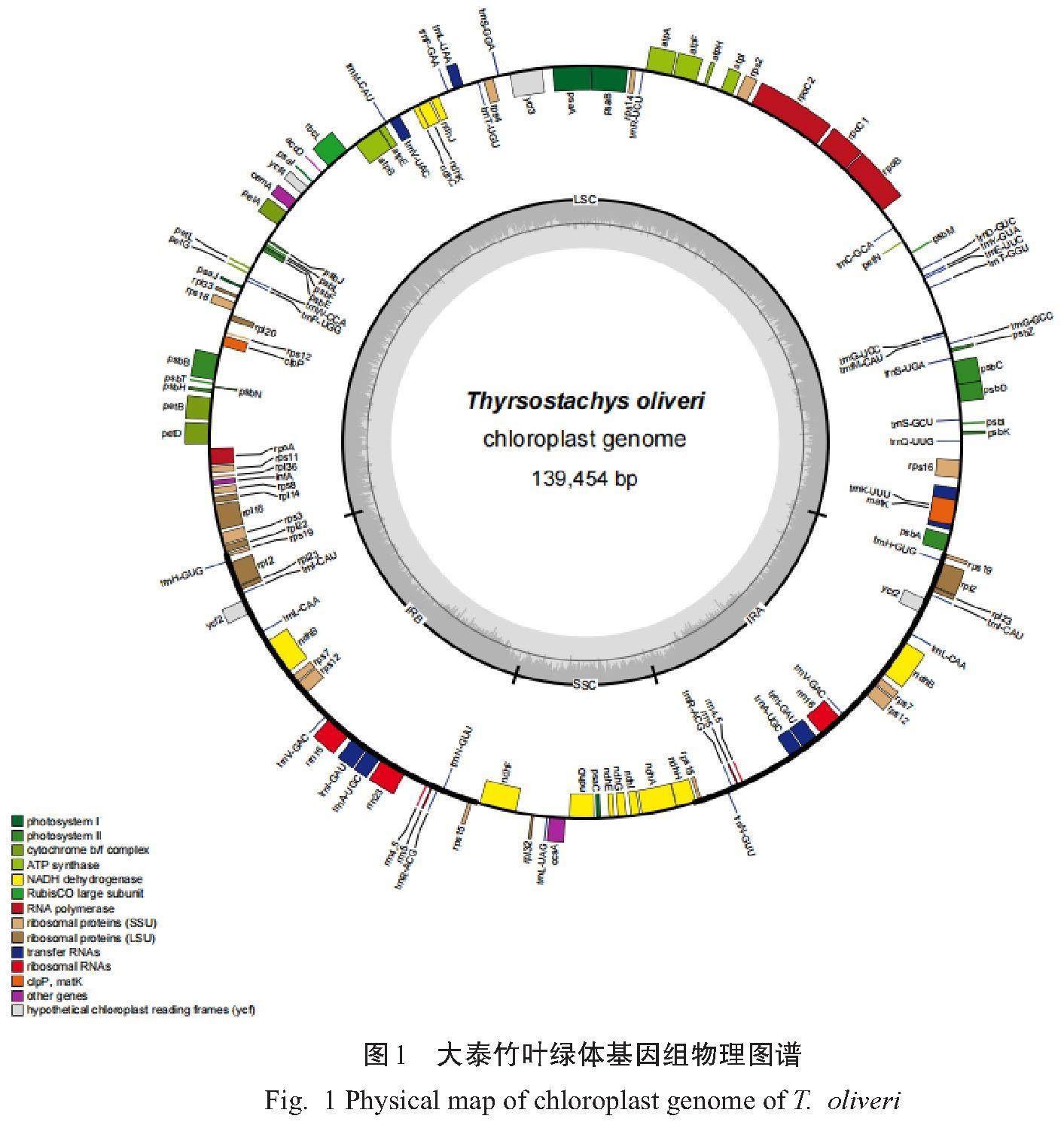
摘要:大泰竹(Thyrsostachys oliveri)是一种在东亚地区广泛分布的竹子,具有重要的生态和经济价值。为获取大泰竹的叶绿体分子生物学特性及明确其系统发育位置,本研究采用高通量测序技术对其叶绿体基因组进行了测序和组装,获得了完整的大泰竹叶绿体基因组序列,并对其进行基因组特征解析和系统发育分析。结果表明:(1)大泰竹的叶绿体基因组为一个典型的双环状结构,全长为139,454 bp,LSC区域的长度为82,986 bp,SSC区域的长度为12,878 bp,IR区域的长度为21,795 bp,全基因组的GC含量为38.90%,共编码了131个基因;(2)共检测到123个SSR位点,其中单核苷酸SSR最多,为102个,占总数的82.93%;(3)系统发育分析表明,大泰竹为禾本科竹亚科中的一个明确分支,与牡竹属(Dendrocalamus)的麻竹(D. latiflorus)和大叶慈(D. farinosus)亲缘关系较近。研究结果明确了大泰竹在竹类植物中的系统发育位置,同时可为竹类分类和鉴定提供新的分子依据。
关键词:大泰竹;叶绿体基因组;系统发育;SSR
中图分类号:S792.99 文献标志码:A
Chloroplast Whole Genome and Phylogenetic
Analysis of Thyrsostachys oliveri
Abstract: Thyrsostachys oliveri is a bamboo widely distributed in East Asia, with important ecological and economic value. To deepen our comprehension of the molecular biology and evolutionary placement of T. oliveri, the research employed advanced high-throughput sequencing techniques to meticulously sequence and piece together the chloroplast genome. This effort culminated in the acquisition of a fully intact chloroplast genome sequence for T. oliveri, which was then subjected to an exhaustive analysis of its genomic features and phylogenetic analysis. The results showed that: (1) The chloroplast genome of T. oliveri is a typical double ring structure, with a total length of 139454 bp, an LSC region length of 82986 bp, an SSC region length of 12878 bp, and an IR region length of 21795 bp. The GC content of the entire genome is 38.90%, encoding a total of 131 genes; (2) A total of 123 SSRs were detected, with a maximum of 102 single nucleotide SSRs, accounting for approximately 82.93%; (3) The phylogenetic tree shows that T. oliveri belongs to a clear branch of the Bambusoideae in the Poaceae, and is closely related to Dendrocalambus latiflorus and D. farinosus. The research results were of great significance for understanding the phylogenetic of T. oliveri in bamboo plants and provided new basis for bamboo identification.
Keywords: Thyrsostachys oliveri; chloroplast genome; phylogeny; SSR
大泰竹(Thyrsostachys oliveri)属禾本科(Poaceae)竹亚科,自然分布区主要在东南亚,在中国主要分布在云南。大泰竹被广泛用作建筑原材料[1],也被用作观赏竹[2]。大泰竹叶小枝高,木质素含量高,具有优异的物理和机械性能[3],且其α-纤维素含量高于其他竹材,因而还可以作为生物精炼原料的来源之一[4]。传统上,竹子的识别和分类主要基于其生物学形态特征,如竹茎形态、叶片结构和分枝模式。然而,由于物种之间的微妙差异以及趋同进化等因素,仅依靠形态特征进行准确识别可能变得复杂且不可靠。近年来高通量测序技术成熟,为阐明物种间亲缘关系远近及物种识别提供直接分子水平证据,同时还能揭示它们的演化历史,这为竹资源的分类及科学利用提供了可靠的分析依据。
大泰竹作为一种多用途竹种,其系统进化关系尚未明确。本研究利用高通量测序技术,对大泰竹叶绿体基因组进行测序,通过分析其序列基础特征,构建系统进化树以明确其系统进化位置,为大泰竹的物种鉴定和开发利用提供依据,并进一步补充完善竹类的叶绿体基因组信息库。
1 材料与方法
1.1 试验材料
试验材料为大泰竹(Thyrsostachys oliveri),植株位于广西南宁市E 108.21°,N 20.55°。2022年6月采集其新鲜嫩叶,放入干燥硅胶中保存备用。同时,剪取其枝叶制作凭证标本(标号为TO2022060801)保存于广西壮族自治区林业科学研究院标本馆。
1.2 试验方法
1.2.1 基因组DNA提取及测序
取适量的大泰竹叶片,使用改良的CTAB法[5]提取总DNA,对其浓度和质量进一步检测,符合送测要求后,利用Illumina Hiseq测序技术对样品总DNA进行paired-end测序。
1.2.2 叶绿体基因组组装和注释
测序完成后,首先使用NGS QC Toolkit软件[6]对原始测序序列(Raw reads)进行质量控制,去除接头和低质量序列,得到高质量的待分析序列(Clean reads);利用NGSQC[6]对测序数据进行质控,使用SPAdes软件[7]、Bowtie 2[8]完成叶绿体基因组的从头组装;使用DOGMA[9]和CpGAVAS[10]在线工具预测叶绿体基因组中的蛋白编码基因、转移RNA(tRNA)基因和核糖体RNA(rRNA)基因;使用PGA[11]对这些基因进行功能注释,并使用OGDRAW[12]在线软件绘制大泰竹叶绿体基因组的物理图谱。
1.2.3 叶绿体基因组SSR特征分析
利用MISA在线软件[13]鉴定大泰竹叶绿体基因组中的SSR位点,搜索重复参数依次设置为:1/8、2/5、3/4、4/3、5/3、6/3,位点间最小长度设置为100 bp,其余参数使用默认值。
1.2.4 系统发育分析
利用NCBI数据库下载总计19个物种的叶绿体基因组,所有物种均来自禾本科,包含牡竹属(Dendrocalamus)等11个属,共19个种,物种拉丁名及叶绿体基因组登录号详见表1。从每个物种的叶绿体基因组中挑选75个共有的同源蛋白基因,使用MAFFT 7.037软件[14]和FFT-NS-2策略进行序列比对。接着,使用PhyloSuite软件ModelFinder选择TPM3+F+I+G4模型[15]。最后,利用IQtree 2.0软件[16]基于最大似然法(ML方法)构建系统发育树,bootstrap值为1000。
2 结果与分析
2.1 叶绿体基因组基本特征
通过高通量测序和叶绿体基因组物理图谱构建的结果显示(表2和图1),大泰竹叶绿体基因组为一个典型的双链闭合环状结构,长度为139, 454 bp,平均测序深度为260X,其中IR(Inverted repeats)长度为21, 795 bp,LSC(Large single copy)长度为82, 986 bp,SSC(Small single copy)长度为12, 878 bp,全基因组GC含量为38.90%。而且,共编码131个基因,包括蛋白编码基因86个、rRNA基因7个、38个tRNA基因(表3)。获得的大泰竹叶绿体基因组已上传NCBI数据库,序列号为 NC_068070.1。
2.2 SSR特征分析
MISA在线软件的分析结果显示(表4),在大泰竹叶绿体基因组中共检测到123个SSR位点;其中,单核苷酸SSR数量最多,共102个,占比为82.93%,包括A/T重复(98个)、C/G重复(4个);二核苷酸SSR数量有4个,包括AG/CT重复(1个)和AT/AT重复(3个);三核苷酸SSR数量为4个,包括AAG/CTT重复(2个)、AAT/ATT重复(1个)、AGC/CTG重复(1个);四核苷酸SSR数量为13个,具有6种不同的重复类型;在大泰竹叶绿体基因组中未发现五核苷酸和六核苷酸SSR。
2.3 系统发育分析
基于19个禾本科物种叶绿体基因组共有的75个同源蛋白,构建最大似然(ML)系统发育树。系统发育树结果显示(图2),大泰竹为禾本科(Poaceae)竹亚科(Bambusoideae)的一个明确分支,大泰竹叶绿体基因组(NC_068070.1)与参试的牡竹属(Dendrocalamus)麻竹Dendrocalamus latiflorus,NC_013088.1)和大叶慈(D. farinosus,NC_063702.1)亲缘关系较近。
3 讨论与结论
泰竹属包含大泰竹和泰竹两个物种,本研究测序的大泰竹叶绿体基因组大小为139, 454 bp,与同属的泰竹叶绿体基因组(139, 522 bp)[17]大小相比相差68 bp,其GC含量一致;大泰竹编码基因131个,比泰竹(139个)少了8个编码基因。
SSR分子标记数量多、多态性高,在鉴别物种、研究物种遗传多样性、构建遗传图谱方面有着较高应用价值。本研究在大泰竹叶绿体基因组中共发现123个SSR位点,与亲缘关系较近的大慈竹(D. farinosus)相比SSR多出56个[18],在大泰竹最主要重复类型为单核苷酸序列重复(98个),而在大慈竹中单核苷酸重复仅包含17个,较高的遗传多样性表明了大泰竹能够适应更广泛的环境,该结果为后期竹亚科的分子生物学研究提供了较为丰富的SSR标记。
前人的研究结果表明,泰竹属与牡竹属(Dendrocalamus)、滇竹属(Gigantochloa)、梨藤竹属(Melocalamus)、慈竹属(Neosinocalamus)和锐药竹属(Oxytenanthera)为姐妹群[19, 20]。本研究中大泰竹与牡竹属植物聚为一支,这与前人研究结果一致。然而,本研究发现同为泰竹属的大泰竹和泰竹两个物种并未聚在一起,推测与其形态特征差异有关,大泰竹叶色、叶型、杆型等与泰竹相似,但大泰竹杆高、直径、叶长叶宽等均明显大于泰竹[21]。同时,传统意义上的分类学,主要依靠植物的形态学特征来探讨和揭示物种的生物学特征和其在生态系统中的位置,而分子聚类则基于遗传信息来揭示物种间的亲缘关系。本研究大泰竹和泰竹在分子聚类中的分离,也表明了形态学特征可能不足以反应所有遗传差异,也可能是由于形态学特征的趋同演化、遗传变异的非均匀分布,或者是杂交事件、网状进化等所导致的。通过整合形态学和分子数据,可以更准确地描述物种的边界,并为保护和管理竹类植物资源提供科学依据。
参考文献
[1] CHAOWANA K, WISADSATORN S, CHAOWANA P. Bamboo as a sustainable building material—Culm characteristics and properties[J]. Sustainability, 2021, 13(13): 7376.
[2] BANIK R L. Silviculture of South Asian Priority Bamboos[M]. Singapore: Springer Singapore, 2016: 261-276.
[3] ZHANG Z H, RAO F, WANG Y J. Morphological, chemical, and physical-mechanical properties of a clumping bamboo (Thyrsostachys oliveri) for construction applications[J]. Polymers, 2022, 14(17): 3681.
[4] HOSSAIN M J, GHOSH R K, DAS A K, et al. Investigation of the potentiality of five bamboo species in biorefinery through analysis of chemical profiles[J]. Journal of Wood Chemistry and Technology, 2022, 42(3): 204-210.
[5] 樊守金, 郭秀秀. 植物叶绿体基因组研究及应用进展[J]. 山东师范大学学报(自然科学版), 2022, 37(1): 22-31.
[6] PATEL R K, JAIN M. NGS QC toolkit: A toolkit for quality control of next generation sequencing data[J]. PLoS One, 2012, 7(2): e30619.
[7] BANKEVICH A, NURK S, ANTIPOV D, et al. SPAdes: A new genome assembly algorithm and its applications to single-cell sequencing[J]. Journal of Computational Biology: a Journal of Computational Molecular Cell Biology, 2012, 19(5): 455-477.
[8] LANGMEAD B. Bowtie: An accelerated, memory-efficient short read aligner [J]. Nature Methods, 2010, 7(4): 335-336.
[9] WYMAN S K, JANSEN R K, BOORE J L. Automatic annotation of organellar genomes with DOGMA[J]. Bioinformatics, 2004, 20(17): 3252-3255.
[10] LIU C, SHI L C, ZHU Y J, et al. CpGAVAS, an integrated web server for the annotation, visualization, analysis, and GenBank submission of completely sequenced chloroplast genome sequences[J]. BMC Genomics, 2012, 13: 715.
[11] QU X J, MOORE M J, LI D Z, et al. PGA: A software package for rapid, accurate, and flexible batch annotation of plastomes[J]. Plant Methods, 2019, 15: 50.
[12] GREINER S, LEHWARK P, BOCK R. OrganellarGenomeDRAW (OGDRAW) version 1.3.1: Expanded toolkit for the graphical visualization of organellar genomes[J]. Nucleic Acids Research, 2019, 47(W1): W59-W64.
[13] BEIER S, THIEL T, M?NCH T, et al. MISA-web: A web server for microsatellite prediction[J]. Bioinformatics, 2017, 33(16): 2583-2585.
[14] KATOH K, STANDLEY D M. A simple method to control over-alignment in the MAFFT multiple sequence alignment program[J]. Bioinformatics, 2016, 32(13): 1933-1942.
[15] KALYAANAMOORTHY S, MINH B Q, WONG T K F, et al. ModelFinder: Fast model selection for accurate phylogenetic estimates[J]. Nature Methods, 2017, 14(6): 587-589.
[16] MINH B Q, SCHMIDT H A, CHERNOMOR O, et al. IQ-TREE 2: New models and efficient methods for phylogenetic inference in the genomic era[J]. Molecular Biology and Evolution, 2020, 37(5): 1530-1534.
[17] TANG S L, XIE J H, CAI J Z. The complete plastid genome of Thyrsostachys siamensis (Poaceae, Bambusoideae)[J]. Mitochondrial DNA. Part B, Resources, 2021, 6(6): 1781-1783.
[18] YANG H Q, YANG J B, PENG Z H, et al. A molecular phylogenetic and fruit evolutionary analysis of the major groups of the paleotropical woody bamboos (Gramineae: Bambusoideae) based on nuclear ITS, GBSSI gene and plastid trnL-F DNA sequences[J]. Molecular Phylogenetics and Evolution, 2008, 48(3): 809-824.
[19] PEI J L, WANG Y, ZHUO J, et al. Complete chloroplast genome features of Dendrocalamusfarinosus and its comparison and evolutionary analysis with other bambusoideae species[J]. Genes, 2022, 13(9): 1519.
[20] YANG J B, YANG H Q, LI D Z, et al. Phylogeny of Bambusa and its allies (Poaceae: Bambusoideae) inferred from nuclear GBSSI gene and plastid psbA-trnH, rpl32-trnL and rps16 intron DNA sequences[J]. TAXON, 2010, 59(4): 1102-1110.
[21] 陈松河. 泰竹生物学特性研究初报[J]. 竹子研究汇刊, 2006, 25(2): 28-30, 34.

