玉米ZmZIM家族基因鉴定及其对氮素的响应特征
孙扬名 张明亮 葛敏 邬奇 赵涵
摘要:為明确玉米ZmZIM家族基因结构、位置、编码蛋白质性质及其对氮素的响应特征,本研究利用TBtools、MEGA X等软件分析玉米ZmZIM家族基因的结构、染色体位置、顺式作用元件及系统发育关系及其编码蛋白质的理化性质、保守结构域及基序,结合玉米不同发育时期不同器官的转录组数据及充足氮与低氮水平下四叶一心期玉米地上部转录组数据解析玉米ZmZIM家族基因的表达模式及差异。结果表明:从玉米全基因组中共鉴定到32个玉米ZmZIM基因,主要分布于1号、2号、5号和7号染色体, 8号和10号染色体上无ZmZIM基因。32个ZmZIM基因可划分为4个亚类,其编码蛋白质由134~467个氨基酸残基构成,均为亲水性蛋白质且全部定位在细胞核中。32个ZmZIM基因启动子区域顺式作用元件主要有调控元件、光信号响应元件、激素信号响应元件、胁迫响应元件、生长发育元件及蛋白质结合位点等6大类。不同发育时期,ZmZIM基因在玉米不同器官中存在差异性表达;在充足氮与低氮处理下,随着处理时间的增加,玉米植株地上部12个ZmZIM基因无表达或相对表达量较低,6个ZmZIM基因相对表达量较高且稳定,其余的14个ZmZIM基因的相对表达量差异较大;ZmZIM5、ZmZIM16、和ZmZIM31 3个基因的相对表达量普遍高于其他基因。充足氮条件下,ZmZIM8、ZmZIM15、ZmZIM20、ZmZIM24、ZmZIM29和ZmZIM31基因的相对表达量普遍高于低氮条件。本研究结果为玉米氮高效吸收利用基因筛选和利用奠定基础。
关键词:玉米;ZIM转录因子;基因家族分析;氮响应
中图分类号:S513文献标识码:A文章编号:1000-4440(2024)04-0577-14
Identification of ZmZIM family genes and their response to nitrogen in maize
SUN Yang-ming1,2,ZHANG Ming-liang2,GE Min2,WU Qi2,ZHAO Han2
(1.College of Agriculture, Nanjing Agricultural University, Nanjing 210095, China;2.Institute of Germplasm Resources and Biotechnology, Jiangsu Academy of Agricultural Sciences, Nanjing 210014, China)
Abstract:In order to clarify the structure, location, encoded protein properties and response characteristics of maize ZmZIM family genes to nitrogen, this study used TBtools, MEGA X and other softwares to analyze the structure, chromosome location, cis-acting elements and phylogenetic relationships of maize ZmZIM family genes and the physical and chemical properties, conserved domain and motifs of their encoded proteins. The expression patterns and differences of maize ZmZIM family genes were analyzed by combining the transcriptome data of different organs at different development stages of maize and the transcriptome data of maize shoots at four-leaf and one-heart stage under sufficient nitrogen and low nitrogen levels. The results showed that a total of 32 ZmZIM genes were identified from the whole genome of maize, which were mainly distributed on chromosomes 1, 2, 5 and 7, and no ZmZIM genes were found on chromosomes 8 and 10. The 32 ZmZIM genes could be divided into four subclasses, and the encoded proteins were composed of 134-467 amino acid residues, all of which were hydrophilic proteins and located in the nucleus. The cis-acting elements in the promoter region of 32 ZmZIM genes were mainly divided into six categories: regulatory elements, light signal response elements, hormone signal response elements, stress response elements, growth and development elements and protein binding sites. At different developmental stages, ZmZIM gene was differentially expressed in different organs of maize. Under sufficient nitrogen and low nitrogen treatments, with the increase of treatment time, the 12 ZmZIM genes in the aboveground part of maize plants had no expression or low relative expression, the relative expression of six ZmZIM genes was high and stable, and the relative expression of the remaining 14 ZmZIM genes varied greatly. The relative expression levels of ZmZIM5, ZmZIM16, and ZmZIM31 were generally higher than other genes. Under sufficient nitrogen conditions, the relative expression levels of ZmZIM8, ZmZIM15, ZmZIM20, ZmZIM24, ZmZIM29 and ZmZIM31 were generally higher than those under low nitrogen conditions. The results of this study lay a foundation for the screening and utilization of nitrogen efficient absorption and utilization genes in maize.
Key words:maize;ZIM transcription factors;gene family analysis;nitrogen response
ZIM(Zinc-finger protein expressed in Inflorescence Meristem)转录因子含有C2C2-GATA锌指结构(CX2CX20CX2C,其中X代表任意氨基酸),由Nishii等[1]从拟南芥花序分生组织中发现,因此被命名为ZIM。随后GATA锌指结构在其他蛋白质中也被发现,相应的蛋白质被归入植物特异性转录因子GATA家族[2-3]。ZIM蛋白除了含锌指结构域外,还包含一段由36个氨基酸残基构成的结构域(T[I/L]F[F/Y]XG,其中X代表任意氨基酸),但该结构域在各数据库中使用的基序略有不同。White[4]报道了拟南芥中另外两个分别由AT4G14713和AT4G14720基因编码的PEAPOD1蛋白和PEAPOD2蛋白,含有TIFY及一段特殊结构域。与ZIM蛋白不同的是,PEAPOD蛋白不存在GATA结构域,因此将其归类为C2C2-GATA家族并不准确,Vanholme等[5]认为应将其重新命名并归类为TIFY家族。在TIFY超转录因子家族中仅含有TIFY结构域的蛋白划分为TIFY亚族,含有TIFY结构域和Jas结构域的划分为JAZ亚族,同时含有TIFY、GATA和CCT结构域的划分为ZML亚族,在N端包含一段PEAPOD蛋白且序列中含有TIFY结构域的划分为PPD亚族[4,6-9]。本研究沿用以ZIM命名的蛋白质进行研究。玉米是世界上最主要的粮食作物之一,在工业、农业、食品等行业都起着重要的作用[10-11],但生产中由于氮肥的过度施用,导致玉米的氮肥利用效率较低[12]。ZIM家族在植物生长发育、非生物胁迫(高盐、低温和干旱)应对及茉莉酸信号通路调控等多种反应中发挥重要作用[13-14]。因此研究玉米ZIM基因家族的特征、表达模式,解析玉米ZIM基因家族在氮素响应中的分子机制对选育氮高效玉米品种具有重要意义。
自2000年Nishii等[1]从拟南芥中首次鉴定到AtZIM1基因以来,水稻[15]、葡萄[16]、二穗短柄草[13]、大豆[17]、棉花[18-19]、小麦[20]、西瓜[21]、木薯[22]、番茄[6]、杜仲[14]、柑橘[23]等植物中的ZIM基因继续得到了鉴定。同时ZIM家族基因的功能也得到了广泛研究。拟南芥JAZ蛋白可与AtMYB21和AtMYB24的R2R3结构域结合导致拟南芥雄性不育,接收到茉莉酸信号后COI1结合JAZ蛋白并被泛素化降解使AtMYB21和AtMYB24激活下游基因表达调控雄蕊发育[24];苹果MdABI4蛋白与ICE1和JAZ蛋白互作形成JAZ-ABI4-ICE1-CBF模块调控ABA(脱落酸)信号介导的苹果耐冷性[25]; SlJAZ10和SlJAZ11蛋白不仅可通过茉莉酸信号来抑制番茄叶片衰老,还可通过协同电信号、钙离子信号等调控番茄再生[26];丙烯可诱导MaTIFY1基因表达,加快香蕉果实成熟[27];小麦TaZIM-A1基因的表达量有明显的昼夜节律,且过表达TaZIM-A1基因会下调部分关键开花调控基因的表达量进而导致小麦开花时间的推迟[28];盐胁迫下过表达TdTIFY11a基因可使小麦在高渗透压条件下仍保持较高的成活率及发芽率[29],同样PnJAZ1蛋白赋予核桃种子较高的萌发率及耐盐性[30];TIFY家族基因积极参与毛果杨对高盐、极端温度、干旱等非生物胁迫的调控过程[31]。
虽然ZIM家族基因及其功能在多种植物中得到了研究,但玉米ZIM家族基因的鉴定及氮素响应特征研究还鲜见报道。本研究基于玉米V5版本的基因组数据,利用生物信息学及TBtools软件鉴定玉米ZmZIM基因家族成员,从基因的结构、染色体位置、系统进化关系、启动区域顺式作用元件及表达模式及其编码蛋白质的理化性质、蛋白质结构、保守基序等角度解析ZmZIM基因家族的特性,并通过充足氮及低氮水平下玉米地上部的转录组测序,进一步分析玉米ZmZIM家族基因的表达量差异,旨在为ZmZIM家族基因对氮素的响应机制挖掘及氮高效利用玉米种质选育提供依据。
1材料与方法
1.1玉米ZmZIM家族基因的鉴定及其在染色体上的分布
从MaizeGDB网站和EnsemblPlant数据库分别下载玉米B73_RefGen_V5版本ZIM家族蛋白质氨基酸序列和全部蛋白质氨基酸序列,利用TBtools提取ZIM家族的蛋白质氨基酸序列,通过氨基酸序列比对后保留序列相同的蛋白质氨基酸序列,并在NCBI网站下载玉米ZIM家族蛋白质氨基酸序列,将两种方式获得的ZIM家族蛋白质氨基酸序列再次进行比对,选择两次比对结果中氨基酸序列均一致的蛋白质氨基酸序列作为玉米ZIM家族蛋白质氨基酸序列用于后续分析。使用TBtools软件的Gene Location Visualize from GXF模块分析玉米ZmZIM家族基因在染色体上的位置[32]。
1.2玉米ZmZIM基因编码蛋白质的理化性质分析及亚细胞定位
采用TBtools软件中Protein Parameter Calc程序进行ZIM家族基因编码蛋白质的氨基酸数目、理论等电点(PI)、不稳定指数、脂肪系数、平均亲水系数等理化性质分析。根据蛋白质氨基酸序列采用Cell-PLoc2.0网站进行玉米ZIM蛋白的亚细胞定位。
1.3基因结构分析及蛋白质结构分析
利用TBtools软件结合保守结构域数据库(CDD)和基因编码蛋白质氨基酸序列进行玉米ZIM家族保守结构域预测。设置保守基序(Motif)最大数值为20,采用TBtools软件的Simple MEME Wrapper模块进行玉米ZIM家族蛋白质保守基序预测。根据ZmZIM家族基因的编号(ID)下载对应的基因序列,利用TBtools軟件的Gene Structures View模块对下载的ZmZIM基因序列进行基因非编码区(UTR)及编码序列(CDS)分析。
1.4启动子顺式作用元件分析
首先利用TBtools软件的GXF Sequences Extract模块提取玉米基因组全部基因CDS上游2 000 bp的序列,随后使用TBtools中Fasta Extract模块提取ZmZIM家族基因CDS上游2 000 bp序列,再利用PlantCARE在线网站预测启动子区域的顺式作用元件,对各个基因中存在的无过多生物学意义的元件如TATA-box、CAAT-box等元件进行删减后,保留注释文件中具有抗逆、光响应、激素响应等功能的顺式作用元件,利用TBtools中Basic BioSequence View模块对玉米ZmZIM家族基因启动子区域顺式作用元件可视化。
1.5系统发育树分析
采用MEGA-X软件的MUSCLE模块对玉米ZmZIM蛋白进行多序列比对[33],设置Bookstrap值为1 000,利用邻接法(Neighbor-joining,NJ)构建系统发育进化树,并利用iTOL在线网站进行进化树的可视化。
1.6表达模式分析
以玉米自交系B73为试验材料,先在育苗盘中育苗,待发芽后选取长势相近的个体移栽至人工搅拌均匀的基质中,每3 d补水1次,保证基质水分适宜,期间适当施加尿素及复合肥保证植株正常生长。在玉米生长发育的不同阶段对不同组织取样并进行转录组测序,每样品3个重复。利用FastQC v11.9[34]和Fastp软件进行测序结果的质量控制,按照默认参数去除重复和低质量的read,得到高质量序列(Clean data)。利用Hisat2 v2.2.1[35]将Clean data比对到玉米参考基因组(B73_RefGen_v5),然后利用Samtools v1.19[36]软件将SAM文件转化成BAM文件并排序,参考基因组数据和注释信息下载自MaizeGDB數据库。提取玉米ZmZIM家族基因的FPKM值,统计获得ZmZIM家族基因的相对表达量。利用TBtools软件进行不同生长发育阶段不同组织ZmZIM家族基因表达量的热图(Heat map)制作与分析。
1.7ZmZIM家族基因对氮素的响应
以玉米自交系B73为试验材料,于2023年3月进行水培处理。种子发芽后移入含2 mmol/L KNO3的Hoagland培养液中,每3 d更换1次培养液,待植株长至四叶一心时移入充足氮(50 mmol/L KNO3)和低氮(05 mmol/L KNO3+45 mmol/L KCl)的水培液中进行不同氮素水平培育,并于处理后0 h、05 h、10 h、15 h、20 h、30 h、40 h、60 h、80 h时进行地上部取样,取得样品采用植物总RNA提取试剂盒(南京诺唯赞医疗科技有限公司产品)提取RNA后,送北京贝瑞和康生物技术有限公司进行转录组测序,测序结果参照材料与方法16中的方法进行质量控制与表达量分析,进而得到不同氮素水平下ZmZIM家族基因表达量的热图。
2结果与分析
2.1玉米ZIM家族基因的鉴定及分析
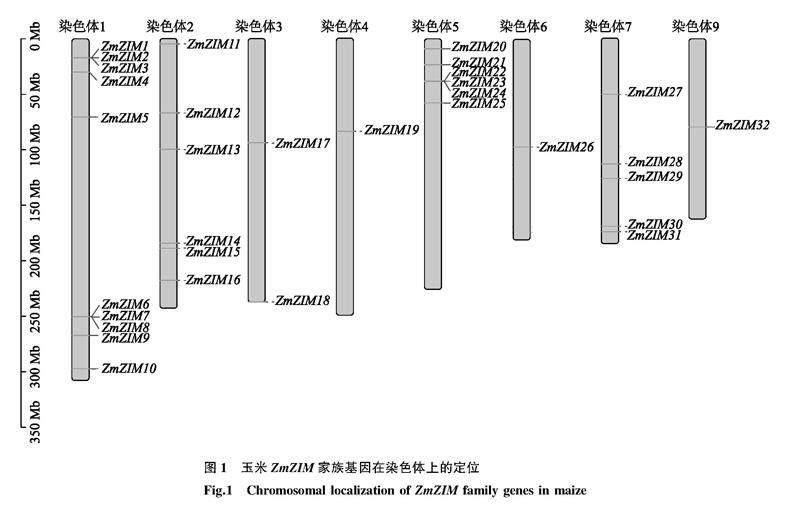
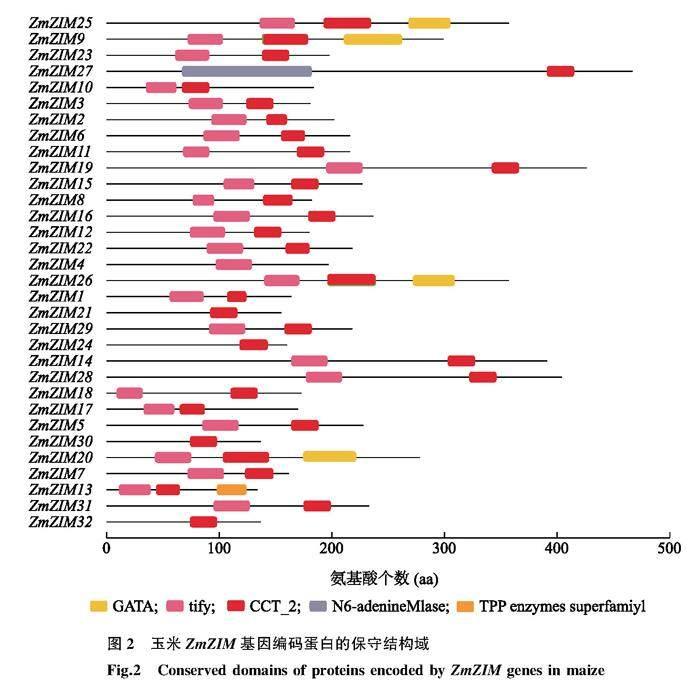
根据MaizeGDB网站及NCBI网站比对后共鉴定出32个玉米ZIM基因。将获得的32个ZIM基因按照其在染色体分布位置依次命名为ZmZIM1~ZmZIM32。32个ZIM基因不均匀分布在玉米8条染色体上(图1)。其中1号染色体含有10个ZmZIM基因,2号、5号染色体各分布6个ZmZIM基因,3号、7号染色体分别分布2个和5个ZmZIM基因,4号、6号、9号染色体各分布1个ZmZIM基因。值得注意的是ZmZIM基因有时在染色体上成簇分布,每簇包含3个ZmZIM基因,如1号染色体含有2个ZmZIM基因簇(ZmZIM1、ZmZIM2、ZmZIM3和ZmZIM6、ZmZIM7、ZmZIM8),5号染色体含有1个ZmZIM基因簇(ZmZIM22、ZmZIM23、ZmZIM24)。32个ZmZIM基因的编码蛋白质共鉴定出3个典型的特征结构域(图2),其中含TIFY结构域的共27个,含CCT结构域的共31个,仅ZmZIM4基因的编码蛋白质不含有CCT结构域,同时含锌指结构域GATA、TIFY结构域和CCT结构域的编码蛋白质共4个(ZmZIM9、ZmZIM20、ZmZIM25及ZmZIM26)。
上述32个ZmZIM蛋白由134~467个氨基酸残基构成,相对分子质量为14 161.23~52 501.39,理论等电点(PI)为4.60~1018,其中等电点大于70的蛋白质有23个,表明大多数ZmZIM蛋白偏碱性,32个ZmZIM蛋白均为亲水性蛋白(表1)。亚细胞定位预测结果显示,32个蛋白质均定位在细胞核中。
2.2玉米ZmZIM家族基因结构分析及编码的蛋白质结构域预测
32个ZmZIM基因中ZmZIM13、ZmZIM17和ZmZIM27没有非编码区(UTR),其余29个ZmZIM基因均含1~2个UTR及多个编码序列(CDS)。ZmZIM14、ZmZIM19、ZmZIM25、ZmZIM26、ZmZIM28、ZmZIM20和ZmZIM9含有不少于7个CDS(图3),这些基因形成剪切体的潜在能力较高。ZIM31和ZIM5具有较为相似的结构,表明这2个基因的进化过程可能较为相似。
32个ZmZIM基因编码的蛋白质均含有多个保守基序(Motif)(图4)。其中,Motif1、Motif3、Motif4出现次数较多,表明ZmZIM家族基因可能具有较为相似的功能,一些基因编码的蛋白质独有特定的Motif说明这些基因可能有特殊的功能。基因编码的蛋白质保守基序的多样性与基因编码的蛋白质的保守结构域多样化基本一致。
2.3玉米ZIM家族基因启动子顺式作用元件
32个ZmZIM基因启动子区域包含的顺式作用元件主要有6大类:调控元件、光信号响应元件、激素信号响应元件、胁迫响应元件、生长发育元件及蛋白质结合位点等(图5)。其中,光信号响应元件和激素信号响应元件数量最多,分别出现453次及341次,且每个ZmZIM中均有1个以上的光信号响应元件。这表明ZmZIM家族可能调控多种信号通路,从而对玉米生长发育及多种胁迫作出应答。同时,ZmZIM家族可能受光信号通路的影响从而存在表达的周期性。胁迫响应元件出现141次。上述结果说明ZmZIM家族可能在调控玉米胁迫应答、激素调节和光信号响应中发挥重要作用。
2.4玉米ZIM蛋白系统发育树分析
玉米ZmZIM家族蛋白质系统发育树显示32个ZmZIM蛋白可以划分为S1、S2、S3、S4 4个亚类。其中,S4亚类包含19个基因的编码蛋白质,S1亚类包含3个基因的编码蛋白(ZmZIM14、ZmZIM19、ZmZIM28),S2和S3亚类各含有5个基因的编码蛋白质,分别是ZmZIM9、ZmZIM12、ZmZIM20、ZmZIM25、ZmZIM26和ZmZIM11、ZmZIM18、ZmZIM21、ZmZIM30、ZmZIM32(图6)。
2.5玉米ZIM家族基因表达模式分析
不同生长发育时期,玉米自交系B73不同组织ZmZIM基因的表达模式如图7所示。从图中可以看出,32个ZmZIM基因的表达差异显著。15个基因(ZmZIM1、ZmZIM2、ZmZIM4、ZmZIM9、ZmZIM10、ZmZIM12、ZmZIM13、ZmZIM14、ZmZIM17、ZmZIM18、ZmZIM22、ZmZIM23、ZmZIM26、ZmZIM27、ZmZIM32)在玉米生长过程中多个组织中几乎不表达或低表达,ZmZIM21、ZmZIM25和ZmZIM30在玉米生長过程中相对表达量基本稳定,其余14个基因在玉米生长过程中相对表达量存在较大差异。在第9叶片展开期第8叶中ZmZIM8、ZmZIM15、ZmZIM24、ZmZIM28和ZmZIM29等基因的相对表达量较高,在第14叶片展开期第13叶中ZmZIM7、ZmZIM11、ZmZIM19、ZmZIM29、ZmZIM30和ZmZIM31等基因的相对表达量较高。
2.6玉米ZIM家族基因对氮素的响应
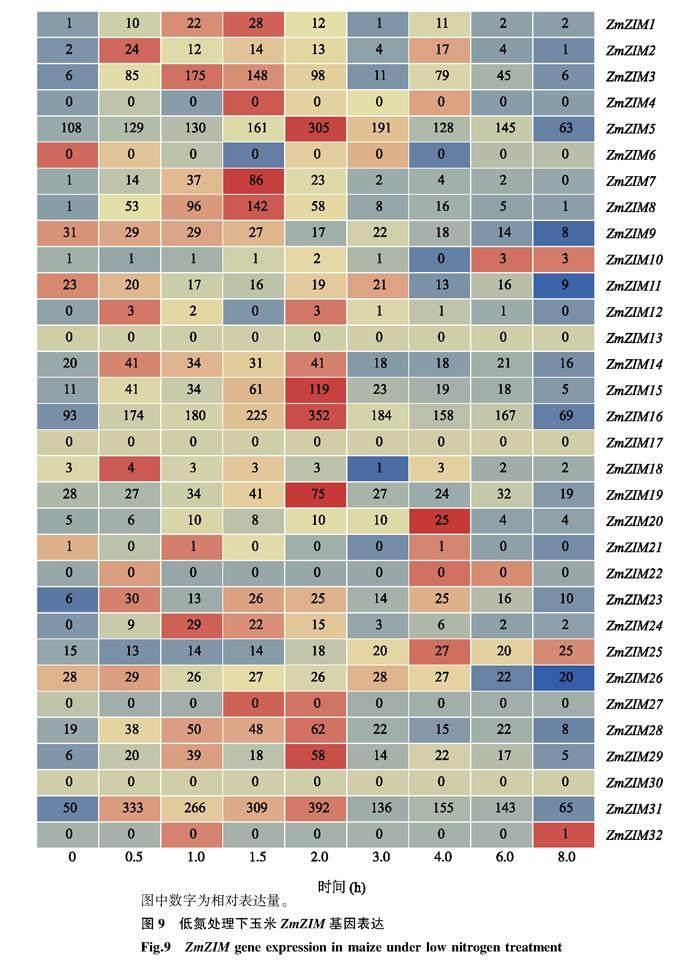
充足氮和低氮处理后,玉米自交系B73地上部ZmZIM基因表达量的变化分别如图8和图9所示。从图中可以看出,2种氮素水平下,随着氮素处理时间的增加,32个ZmZIM基因的相对表达量出现不同的变化特征:12个ZmZIM基因(ZmZIM4、ZmZIM6、ZmZIM10、ZmZIM12、ZmZIM13、ZmZIM17、ZmZIM18、ZmZIM21、ZmZIM22、ZmZIM27、ZmZIM30和ZmZIM32)在2种氮素水平下均不表达或表达量极低;ZmZIM9、ZmZIM11、ZmZIM19、ZmZIM20、ZmZIM25和ZmZIM26 6个ZmZIM基因相对表达量较高,且变化比较稳定,其余14个ZmZIM基因的相对表达量差异较大。与处理初始相比,充足氮处理8 h后ZmZIM1、ZmZIM3、ZmZIM8、ZmZIM29和ZmZIM31基因的相对表达量提高了4倍以上。2种氮素水平下,ZmZIM5、ZmZIM16和ZmZIM31基因的相对表达量普遍高于其他基因。充足氮处理下,ZmZIM8、ZmZIM15、ZmZIM20、ZmZIM24、ZmZIM29和ZmZIM31基因的相对表达量普遍高于低氮处理,这表明该家族基因对氮素变化的响应较为明显。因此,可以认为玉米ZmZIM基因在氮素响应调控中确实发挥了重要功能。
3讨论
转录因子参与生物体众多生理生化过程的调控,从基因组及转录组水平入手解析转录因子家族结构和功能有助于明确转录因子基因家族的特性[37-39]。ZIM蛋白是植物特有的一类转录因子,在植物应对非生物胁迫、光响应、激素信号传导等方面起着重要的作用。本研究通过挖掘ZmZIM家族基因的染色体位置、结构、顺式作用元件、表达模式及其对氮素的响应特征,初步明确了ZmZIM基因的功能及对氮素的响应机制。
本研究基于最新版本玉米基因组信息鉴定出玉米全基因组中含有32个ZmZIM基因,对前人研究结果进行了补充与完善[40]。玉米ZmZIM基因在染色体上的分布较为分散,在1号染色体及5号染色体有成簇密集分布的现象,这可能与基因的重复、互补功能相关。根据ZmZIM家族成员结构相似程度及系统进化关系将32个玉米ZmZIM基因分为4个亚类。本研究中对该家族成员的分类标准和范畴与其他学者研究有所不同。其他学者[5]侧重TIFY家族的分类 ,而本研究则是重点关注蛋白质中是否存在TIFY结构域、CCT结构域及GATA锌指结构域而进行分类。尽管本研究中ZmZIM4蛋白仅含TIFY结构域,但多次验证后本研究认为该蛋白质仍属于ZIM家族,这为ZIM家族的进化分析提供了基础。
Shikata等[3]研究结果表明长日照条件下(16 h光照/8 h黑暗)过表达AtZIM可使拟南芥细胞增大进而导致拟南芥下胚轴及叶柄伸长,短日照条件下(8 h光照/16 h黑暗)过表达AtZIM则使拟南芥叶片变小,这说明光周期能影响到该家族基因的表达,进而调控植株形态。本研究中ZmZIM基因启动子区域顺式作用元件预测结果显示,ZmZIM基因中光信号响应元件数量最多,每个ZmZIM基因至少含有1个胁迫响应元件及激素相应元件,这表明该家族在响应外源激素及非生物胁迫的过程中发挥重要功能。此外,ZmZIM家族基因在玉米叶片中表达量普遍较高,且在不同生长发育期表达量有显著差异,推测ZmZIM基因可能与其他基因互作来调控玉米叶片的发育。
在充足氮及低氮环境下,随着氮素处理时间的增加,12个ZmZIM基因不表达或表达量极低,ZmZIM9、ZmZIM11、ZmZIM19、ZmZIM20、ZmZIM25和ZmZIM26等6个ZmZIM基因相对表达量较高,且变化比较稳定,其余14个ZmZIM基因的相对表达量差异较大;ZmZIM5、ZmZIM16、和ZmZIM31等3个基因的相对表达量普遍高于其他基因。充足氮条件下,ZmZIM8、ZmZIM15、ZmZIM20、ZmZIM24、ZmZIM29和ZmZIM31等基因的相对表达量普遍高于低氮条件下相对表达量。上述结果说明ZmZIM基因可积极响应氮素条件的变化,一些成员在低氮条件下高表达以增强玉米对外界氮素的吸收、同化及转运用来维持植株自身的生长,而另一部分成员在充足氮素条件下高表达一方面促进氮素的同化及转运,另一方面通过其生长发育响应元件与其他基因互作来调控玉米的生长发育来维持玉米植株碳氮平衡。因此,可以认为玉米ZmZIM家族基因在氮代謝过程中可能发挥重要作用,在未来的研究中可通过ZmZIM基因的过表达试验、敲除突变体试验及共表达网络分析,从表型及遗传角度进一步验证和解析ZmZIM基因的功能,明确ZmZIM基因在玉米氮代谢中的作用。
参考文献:
[1]NISHII A, TAKEMURA M, FUJITA H, et al. Characterization of a novel gene encoding a putative single zinc-finger protein, ZIM, expressed during the reproductive phase in Arabidopsis thaliana[J]. Biosci Biotechnol Biochem,2000,64(7):1402-1409.
[2]REYES J C, MURO-PASTOR M I, FLORENCIO F J. The GATA family of transcription factors in Arabidopsis and rice[J]. Plant Physiol,2004,134(4):1718-1732.
[3]SHIKATA M, MATSUDA Y, ANDO K, et al. Characterization of Arabidopsis ZIM, a member of a novel plant-specific GATA factor gene family[J]. Journal of Experimental Botany,2004,55(397):631-639.
[4]WHITE D W. PEAPOD regulates lamina size and curvature in Arabidopsis[J]. Proceedings of the National Academy of Sciences,2006,103(35):13238-13243.
[5]VANHOLME B, GRUNEWALD W, BATEMAN A, et al. The tify family previously known as ZIM[J]. Trends Plant Sci,2007,12(6):239-244.
[6]CHINI A, BEN-ROMDHANE W, HASSAIRI A, et al. Identification of TIFY/JAZ family genes in Solanum lycopersicum and their regulation in response to abiotic stresses[J]. PLoS One,2017,12(6):e177381.
[7]CAI Q, YUAN Z, CHEN M, et al. Jasmonic acid regulates spikelet development in rice[J]. Nat Commun,2014,5:3476.
[8]TIAN J, CAO L, CHEN X, et al. The OsJAZ1 degron modulates jasmonate signaling sensitivity during rice development[J]. Development,2019,146(4):dev173419.
[9]YAN Y, STOLZ S, CHETELAT A, et al. A downstream mediator in the growth repression limb of the jasmonate pathway[J]. Plant Cell,2007,19(8):2470-2483.
[10]CUI T T, HE K H, CHANG L G, et al. QTL mapping for leaf area in maize(Zea mays L.) under multi-environments[J]. Journal of Integrative Agriculture,2017,16(4):800-808.
[11]JU M, ZHOU Z, MU C, et al. Dissecting the genetic architecture of Fusarium verticillioides seed rot resistance in maize by combining QTL mapping and genome-wide association analysis[J]. Sci Rep,2017,7:46446.
[12]AHMED M, RAUF M, MUKHTAR Z, et al. Excessive use of nitrogenous fertilizers: an unawareness causing serious threats to environment and human health[J]. Environ Sci Pollut Res,2017,24:26983-26987.
[13]ZHANG L, YOU J, CHAN Z. Identification and characterization of TIFY family genes in Brachypodium distachyon[J]. J Plant Res,2015,128(6):995-1005.
[14]刘俊,陈玉龙,刘燕,等. 杜仲TIFY转录因子鉴定与表达分析[J]. 中国实验方剂学杂志,2021,27(19):165-174.
[15]YE H, DU H, TANG N, et al. Identification and expression profiling analysis of TIFY family genes involved in stress and phytohormone responses in rice[J]. Plant Mol Biol,2009,71(3):291-305.
[16]ZHANG Y, GAO M, SINGER S D, et al. Genome-wide identification and analysis of the TIFY gene family in grape[J]. PLoS One,2012,7(9):e44465.
[17]ZHU D, BAI X, LUO X, et al. Identification of wild soybean (Glycine soja) TIFY family genes and their expression profiling analysis under bicarbonate stress[J]. Plant Cell Rep,2013,32(2):263-272.
[18]ZHAO G, SONG Y, WANG C, et al. Genome-wide identification and functional analysis of the TIFY gene family in response to drought in cotton[J]. Mol Genet Genomics,2016,291(6):2173-2187.
[19]SUN Q, WANG G, ZHANG X, et al. Genome-wide identification of the TIFY gene family in three cultivated Gossypium species and the expression of JAZ genes[J]. Sci Rep,2017,7:42418.
[20]XIE S, CUI L, LEI X, et al. The TIFY gene family in wheat and its progenitors:genome-wide identification, evolution and expression analysis[J]. Curr Genomics,2019,20(5):371-388.
[21]YANG Y, AHAMMED G J, WAN C, et al. Comprehensive analysis of TIFY transcription factors and their expression profiles under jasmonic acid and abiotic stresses in watermelon[J]. Int J Genomics,2019,2019:6813086.
[22]ZHENG L, WAN Q, WANG H, et al. Genome-wide identification and expression of TIFY family in cassava (Manihot esculenta Crantz)[J]. Front Plant Sci,2022,13:1017840.
[23]張沪,肖翠,王贵元,等. 柑橘TIFY基因结构特征及响应低温表达分析[J]. 中国南方果树,2020,49(2):34-39.
[24]SONG S, QI T, HUANG H, et al. The Jasmonate-ZIM domain proteins interact with the R2R3-MYB transcription factors MYB21 and MYB24 to affect Jasmonate-regulated stamen development in Arabidopsis[J]. Plant Cell,2011,23(3):1000-1013.
[25]AN J P, XU R R, LIU X, et al. Abscisic acid insensitive 4 interacts with ICE1 and JAZ proteins to regulate ABA signaling-mediated cold tolerance in apple[J]. J Exp Bot,2022,73(3):980-997.
[26]TANG B, TAN T, CHEN Y, et al. SlJAZ10 and SlJAZ11 mediate dark-induced leaf senescence and regeneration[J]. PLoS Genet,2022,18(7):e1010285.
[27]罗冬兰,巴良杰,陈建业,等. 香蕉MaTIFY1转录因子特性及其在成熟过程中基因表达分析[J]. 园艺学报,2017,44(1):43-52.
[28]LIU H, LI T, WANG Y, et al. TaZIM-A1 negatively regulates flowering time in common wheat (Triticum aestivum L.)[J]. J Integr Plant Biol,2019,61(3):359-376.
[29]EBEL C, BENFEKI A, HANIN M, et al. Characterization of wheat (Triticum aestivum) TIFY family and role of Triticum Durum TdTIFY11a in salt stress tolerance[J]. PLoS One,2018,13(7):e200566.
[30]LIU S, ZHANG P, LI C, et al. The moss jasmonate ZIM-domain protein PnJAZ1 confers salinity tolerance via crosstalk with the abscisic acid signalling pathway[J]. Plant Sci,2019,280:1-11.
[31]WANG Y, PAN F, CHEN D, et al. Genome-wide identification and analysis of the Populus trichocarpa TIFY gene family[J]. Plant Physiol Biochem,2017,115:360-371.
[32]CHEN C, CHEN H, ZHANG Y, et al. TBtools:an integrative toolkit developed for interactive analyses of big biological data[J]. Mol Plant,2020,13(8):1194-1202.
[33]KUMAR S, STECHER G, LI M, et al. MEGA X:molecular evolutionary genetics analysis across computing platforms[J]. Mol Biol Evol,2018,35(6):1547-1549.
[34]BOLGER A M, LOHSE M, USADEL B. Trimmomatic: a flexible trimmer for Illumina sequence data[J]. Bioinformatics,2014,30(15):2114-2120.
[35]KIM D, LANGMEAD B, SALZBERG S L. HISAT: a fast spliced aligner with low memory requirements[J]. Nat Methods,2015,12(4):357-360.
[36]LI H, HANDSAKER B, WYSOKER A, et al. The sequence alignment/map format and SAMtools[J]. Bioinformatics,2009,25(16):2078-2079.
[37]RIECHMANN J L, HEARD J, MARTIN G, et al. Arabidopsis transcription factors: genome-wide comparative analysis among eukaryotes[J]. Science,2000,290(5499):2105-2110.
[38]QU L J, ZHU Y X. Transcription factor families in Arabidopsis: major progress and outstanding issues for future research[J]. Curr Opin Plant Biol,2006,9(5):544-549.
[39]LIU L, WHITE M J, MACRAE T H. Transcription factors and their genes in higher plants functional domains, evolution and regulation[J]. Eur J Biochem,1999,262(2):247-257.
[40]ZHANG Z, LI X, YU R, et al. Isolation, structural analysis, and expression characteristics of the maize TIFY gene family[J]. Mol Genet Genomics,2015,290(5):1849-1858.
(責任编辑:石春林)
收稿日期:2023-05-09
基金项目:国家自然科学基金项目(32272133);江苏省种业振兴“揭榜挂帅”项目[JBGS(2021)012]
作者简介:孙扬名(1998-),男,吉林长春人,博士研究生,主要从事玉米氮素吸收及高效利用研究。(Tel)18151665609;(E-mail)Sunyangming9808@163.com
通讯作者:赵涵,(E-mail)zhaohan@jaas.ac.cn

