长期饥饿胁迫下厚壳贻贝的生理生化响应及性腺转录组
孙闻婧, 陈传悦,2, 梁泽玮, 廖 智,严小军,2, 张晓林*
(1. 浙江海洋大学海洋科学与技术学院,浙江 舟山 316022;2. 宁波大学海洋学院,浙江 宁波 315211)
厚壳贻贝(Mytilus coruscus)是我国重要的海产贝类养殖品种,个体大,肉肥味美,营养价值高[1]。其主要分布于我国黄、渤海和东海沿岸,以浙江省资源量最大。位于浙江北部的嵊泗县枸杞岛是我国最大的厚壳贻贝养殖区,该海域厚壳贻贝养殖面积约1 500 hm2,产量高达20 余万t[2-3]。然而,随着日益增长的市场需求和经济利益的驱使,为了提高产量,厚壳贻贝的养殖密度不断增加,海域资源无法满足贻贝快速生长的需求,再加上季节变化等自然因素的影响,养殖海区经常出现食物严重缺乏的现象,导致厚壳贻贝遭受短期或长期饥饿的胁迫[4-5]。已有相关研究显示厚壳贻贝在饥饿胁迫下足丝蛋白合成减少,导致附着强度减弱[6],此外,长期饥饿胁迫也会导致肥满度下降,空壳率增加,肉质降低,严重影响贻贝养殖产业的经济效益和高品质发展。因此,探究厚壳贻贝饥饿下的生理生化响应和分子适应机制对合理规划贻贝养殖密度,有效利用海区饵料资源,促进厚壳贻贝养殖业的健康高效发展提供参考。
有研究发现,饥饿胁迫对水生生物的生长发育和代谢均有一定影响,饥饿会引起水生生物的卵细胞和精子数量降低,严重影响性腺的发育和成熟,如日本大鲍(Haliotis gigantea)在摄食水平较低的情况下,随着性腺成熟度的降低,卵子数目也会显著下降[7]。雄性斑马鱼(Danio rerio)的性腺组织结构在饥饿胁迫下严重受损,精子发生受阻,精原细胞和精母细胞比例增加,成熟精子比例显著降低[8]。此外,饥饿也会导致动物组织的生理状态的改变,如光倒刺鲃(Spinibarbus hollandi)在饥饿胁迫下细胞分裂处于完全停滞状态,尽可能地维持当下的状态而减少过多的能量损耗[9]。虹鳟(Oncorhynchus mykiss)肝脏中的己糖激酶活性在饥饿14 d 后显著降低[10]。草鱼(Ctenopharyngodon idella)在长期饥饿下其丙酮酸激酶和磷酸果糖激酶的活性均极显著下降[11]。异育银鲫(Carassius auratus gibelio)在饥饿期间与葡萄糖异生有关的酶(PEPCK、FBPase 和G6Pase)的表达量均上调[12]。目前关于饥饿胁迫对水生生物的影响研究主要集中在鱼类,而对具有重要经济价值贝类的研究较缺乏。因此,探究饥饿胁迫下贻贝的代谢变化对于解析贝类等软体动物对饥饿胁迫的生理响应和调控机制具有积极的指导意义。
性腺组织是贻贝生殖发育最为关键的器官,也是贻贝体内最大的可食用部分,性腺的肥满度直接关系到贻贝的品质和繁育。贻贝短期饥饿下的代谢变化和分子响应已有研究。本实验室前期研究发现,贻贝在短期饥饿下首先通过消耗自身储存的葡萄糖提供能量,然后激活细胞自噬途径加强能量的回收能力,从而提高贻贝体内平衡和稳定[4]。长期饥饿胁迫下的生存代谢和分子调控机制尚不清楚。因此,探究长期饥饿胁迫对贻贝代谢和基因表达调控的影响有利于揭示其饥饿胁迫下的分子适应机制,以提供有效的应对饥饿策略,从而提高贻贝产品的品质和经济价值。
本研究以厚壳贻贝为对象,在实验室条件下进行长达90 d 的饥饿处理,处理结束后首先统计厚壳贻贝在饥饿90 d 的存活率,然后取其性腺组织进行能量代谢相关酶[己糖激酶(HK)、磷酸果糖激酶(PFK)、琥珀酸脱氢酶(SDH)、苹果酸脱氢酶(MDH)和磷酸烯醇式丙酮酸羧激酶(PEPCK)等] 活性的测定,以明确饥饿状态下其能量代谢的变化,最后通过Illumina Hiseq X10 高通量测序技术分析饥饿处理下性腺组织的转录组变化,以期揭示厚壳贻贝饥饿胁迫下的基因表达差异及其调控通路的改变,阐明其饥饿胁迫下的分子调控机制。本实验的结果可为厚壳贻贝适应饥饿胁迫的能量分配机制与生理对策提供参考数据,同时也为合理有效地规划贻贝养殖密度、促进贻贝产业的高质量发展提供思路。
1 材料与方法
1.1 样本采集与处理
成年2 龄厚壳贻贝采集自舟山市嵊泗县枸杞岛养殖基地,体长(10±2) cm,于充分曝气的人工海水中暂养1 周,控制温度为(18±2) °C,盐度28±1,每天换水1 次。贻贝随机分为两组:饥饿组和对照组,每组200 只。对照组每日正常喂养螺旋藻2 次,换水1 次,饥饿组在实验期间不进行任何投喂,仅每日换水1 次[13]。实验90 d 后参考Liu 等[14]采用混样的方法消除个体间的差异进行取样,饥饿组和对照组各取6 个性腺组织样品,每2 个样品等量混合为一管,液氮速冻后置于-80°C 冰箱保存备用,每组3 个生物学重复。本研究获得了浙江海洋大学动物伦理委员会批准(审批号:2023030),实验过程中操作人员严格遵守浙江海洋大学动物伦理委员会伦理规范,并按照浙江海洋大学动物伦理委员会制定的规章制度执行。
1.2 存活率的计算
观察贻贝贝壳是否张开,若发现贝壳张开,用手指触碰贻贝无闭合反应,判定为已经死亡。为准确记录实验过程中饥饿组和对照组贻贝的存活率,本次使用样本数两组各200 只。按下式计算贻贝存活率(RS,%):
式中,n1为初始贻贝数(只),n2为死亡数(只)[15]。
1.3 酶活性测定
取饥饿组和对照组贻贝性腺组织,加入预冷的PBS 缓冲液(0.01 mol/L,pH 7.4),经冰浴匀浆后,4 °C 1 000×g离心10 min,取上清液用于酶活性检测,使用BCA 试剂盒(TaKaRa,货号:T9300A)测定其中的蛋白浓度。HK、SDH、PFK、MDH和PEPCK 活性均采用南京建成生物工程研究所试剂盒测定,具体测定方法参照试剂盒说明书进行。
1.4 总RNA 提取、文库构建和测序
从-80 °C 的冰箱中取出保存的性腺组织,剪碎,研钵提前预冷,加入液氮和组织一并研磨成粉末状。根据Total RNA Extractor (Trizol)试剂盒(上海生工生物工程股份有限公司)使用说明,对所取性腺样品进行总RNA 提取。用超微量核酸蛋白定量仪检测RNA 浓度,琼脂糖凝胶检测RNA完整性以及基因组污染情况。并且通过Agilent 2100 bioanalyzer 对RNA 样品进行质控,检测RNA 完整性。
由北京诺禾致源科技股份有限公司完成cDNA 文库构建以及测序,建库用试剂盒为NEBNext®Ultra™ RNA Library Prep Kit for Illumina®。文库构建完成后,先使用Qubit 2.0 Fluorometer 软件进行初步定量,稀释文库至1.5 ng/μL,随后使用Agilent 2 100 bioanalyzer 对文库的insert size 进行检测,insert size 符合预期后,qRT-PCR 对文库有效浓度进行准确定量(文库有效浓度高于2 nmol/L),以保证文库质量。库检合格后,把不同文库按照有效浓度及目标下机数据量的需求pooling 后进行Illumina 测序。测序平台使用Illumina Hiseq ×10 (Illumina,美国),S4 套组组件。测序片段被高通量测序仪测得的图像数据经CASAVA 碱基识别转化为序列数据(reads),文件为fastq 格式,其中主要包含测序片段的序列信息以及其对应的测序质量信息。
1.5 转录组数据预处理和注释
测序获得的原始数据去除带接头的、无法确定碱基信息的以及低质量的reads,以保证数据分析的质量和可靠性。使用HISAT2 软件将clean reads 与厚壳贻贝参考基因组进行快速精确比对,获取Reads 在参考基因组上的定位信息。参考基因和基因组注释文件下载自NCBI 网站(https: //www.ncbi.nlm.nih.gov/assembly/GCA_011752425.2/)。所获得的clean reads 用StringTie 软件进行组装拼接。将贻贝参考基因组转录本序列分别与GO、KEGG、KO 和PPI 等数据库进行比对注释,从而全面获得注释信息。
1.6 差异表达基因
使用subread 软件中的Feature Counts 工具对基因进行定量分析。根据基因比对在参考基因组上的位置信息,从而统计每个基因(包括新预测基因)从起始到终止范围内覆盖的reads 数。分别过滤掉比对质量值低于10 的reads,非成对比对上的reads,比对到基因组多个区域的reads。通过DESeq2 软 件 在P<0.05 且|log2(Fold Change)|≥1、Padj≤0.05 条件下进行饥饿胁迫的差异显著性分析[16]。采用Cluster Profiler 软件对差异基因集进行GO 功能富集分析和KEGG 通路富集分析,以Padj<0.05 作为显著性富集的阈值。
1.7 实时荧光定量PCR 验证
随机选取14 个显著差异表达的基因进行荧光定量PCR 验证,包括5 个上调和9 个下调基因。荧光定量PCR 引物设计和合成均由上海生工生物工程股份有限公司完成。基因名称和引物序列见表1,β-actin 作为内参基因。每个基因进行3 次平行实验,采用2-ΔΔCt相对定量法计算基因的相对表达量。

表1 qRT-PCR 引物序列Tab. 1 Primer sequences used in qRT-PCR
1.8 数据分析
所有实验数据均以平均值±标准差(mean±SD)表示。使用SPSS 22.0 软件进行数据的统计学分析,并使用单因素方差分析(One-Way ANOVA)中的Duncan 氏进行多重比较,P<0.05 表示差异有统计学意义。
2 结果
2.1 长期饥饿下贻贝的存活率
与对照组相比,饥饿组贻贝的存活率随饥饿时间的延长呈不断下降趋势。在实验的0~8 d,两组贻贝存活率均为100%,饥饿组从实验第9 天开始出现死亡,且实验第9~35 天饥饿组贻贝存活率显著下降(P<0.05),最低值达到87%。在实验第36~90 天,饥饿组贻贝存活率变化不显著(P>0.05),第60 天时饥饿组存活率达到最低86%,随后保持不变直至实验结束。对照组存活率呈下降趋势,但差异不显著(P>0.05),在实验第60 天时,存活率达到最低值96%后保持稳定(图1)。

图1 不同饥饿时间下饥饿组和对照组厚壳贻贝的存活率Fig. 1 Survival rate of M. coruscus in starvation and control group at different starvation times
2.2 转录组数据的统计及参考基因组比对
通过对对照组和饥饿组的厚壳贻贝性腺组织进行转录组测序,分别平均获得46 519 559 和46 310 607 条原始数据。碱基质量及组成分析显示,GC 含量区间为36.49%~37.45%,各样品Q20碱基百分比均超过97.6%,Q30 碱基百分比超过92.76%。对原始数据过滤,即去除含有带接头的、低质量的序列,分别平均获得43 496 069 和46 310 607条的clean reads。将clean reads 与参考基因组进行精确比对,获取reads 在参考基因组上的定位信息后,对照组和饥饿组各获得平均为26 810 506 和31 596 762 的唯一比对上的数据(表2)。所有样本转录组测序的原始数据已上传至NCBI 的SRA 数据库(https: //submit.ncbi.nlm.nih.gov/subs/sra),登录号为PRJNA930147。

表2 转录组数据统计情况Tab. 2 Data statistics of transcriptome
2.3 差异表达基因分析
通 过 DESeq2 软 件 在P<0.05 且|log2(Fold Change)|≥1、Padj≤0.05 条件下进行基因的差异表达分析[16],饥饿组和对照组相比较,共有4 130 个显著差异表达基因,其中显著上调的基因有2 082 个,显著下调的基因有2 048 个(图2)。显著上调表达的基因主要有C 型凝集素、过氧化物酶、热休克蛋白70、磷酸烯醇式丙酮酸羧激酶以及wnt等。显著下调的基因主要包括微小染色体维持蛋白(mcm)家族、DNA 连接酶、6-磷酸葡萄糖酶、mtorc1 以及复制蛋白A 等。
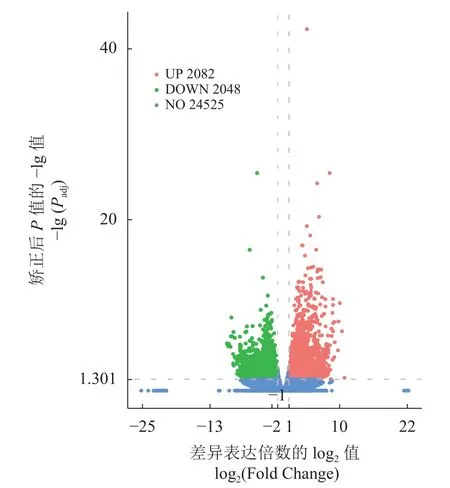
图2 饥饿组和对照组差异表达基因火山图火山图中的点代表有显著表达差异的基因,红色点代表上调差异表达基因,绿色点代表下调差异表达基因,蓝色点代表非差异表达基因。Fig. 2 Volcano plot of differentially expressed genes between starvation and control groups Each dot in the volcano plot represents a gene, where red dots represent up-regulated differentially expressed genes, green dots represent downregulated differentially expressed genes, and blue dots represent non-differentially expressed genes.
2.4 差异表达基因GO 功能注释和富集分析
GO 功能注释结果表明,共有13 383 个基因注释到847 个GO term 中,其中包括:生物过程481 个、细胞组分98 个、分子功能268 个。通过差异基因的富集分析,差异基因最显著富集的GO 功能主要包括蛋白质复合物、细胞器部分、细胞内细胞器部分、非膜性细胞器、细胞内膜结合细胞器、酸酐水解酶活性、水解酶活性、脱氧核糖核酸代谢过程、细胞器组织、应激反应等(图3)。

图3 厚壳贻贝性腺组织差异表达基因的GO 功能富集分析结果1. 水解酶活性,2. DNA 催化作用,3.酸 酐水解 酶活性,4.ATP 依赖性微管运动活动,5.微管,6. 微管动力活性,7. 运动活性,8.肿瘤坏死因子受体,9. 肿瘤坏死因子受体结合,10. 细胞因子受体结合,11. 细胞内细胞器部分,12. 蛋白质复合物,13. 细胞器部分,14. 蛋白酶体核心复合物,15. 细胞内膜结合细胞器,16. 非膜性细胞器,17. 染色体部分,18. 内肽酶复合物,19. 蛋白酶体复合物,20. 染色体,21. 脱氧核糖核酸代谢过程,22. 应激反应,23. 微管过程,24. 细胞器组织,25. 微管运动,26. 细胞或亚细胞成分的运动,27. 染色体组织,28. 免疫反应,29. DNA 复制,30. 免疫系统过程。Fig. 3 GO functional enrichment statistics chart of differentially expressed genes of gonad tissue 1. hydrolase activity, 2. catalytic activity, acting on DNA, 3. hydrolase activity, acting on acid anhydrides, 4. ATP-dependent microtubule motor activity, 5. microtubule, 6. microtubule motor activity, 7. motor activity,8. tumor necrosis factor receptor, 9. tumor necrosis factor receptor binding, 10. cytokine receptor binding, 11. intracellular organelle part, 12.protein-containing complex, 13. organelle part, 14. proteasome core complex, 15. intracellular non-membrane-bounded organelle, 16. nonmembrane-bounded organelle, 17. chromosomal part, 18. endopeptidase complex, 19. proteasome complex, 20. chromosome, 21. DNA metabolic process, 22. response to stress, 23. microtubule-based process, 24.organelle organization, 25. microtubule-based movement, 26. movement of cell or subcellular component, 27. chromosome organization, 28.immune response, 29. DNA replication, 30. immune system process.
2.5 差异表达基因的KEGG 功能注释和富集分析
KEGG 代谢通路注释分析显示,共有8 119个差异表达基因被分类到128 条KEGG 代谢通路中。本实验选取了前20 个最显著富集代谢通路,依次是泛素介导性蛋白酶解、吞噬体、DNA 复制、剪接体、氨基糖和核苷酸糖代谢、Fanconi 贫血通路、蛋白酶体、核苷酸切除修复、真核生物的核糖体生物合成、错配修复、氨基酸的生物合成、同源重组、甘氨酸和丝氨酸以及苏氨酸代谢、基底切除修复、半乳糖代谢、AGE-RAGE 信号通路、甘油脂代谢、丙氨酸、天冬氨酸和谷氨酸代谢、叶酸合成和叶酸-碳库代谢通路(图4)。在饥饿胁迫下,DNA 复制、错配修复、碱基切除修复等与细胞分裂有关的代谢通路显著富集,相关基因的表达量显著下降。

图4 厚壳贻贝性腺组织差异表达基因的KEGG 功能富集分析结果1. 剪接体,2. 叶酸合成,3. 氨基糖和核苷酸糖代谢,4. 甘油脂代谢,5. 丙氨酸、天冬氨酸和谷氨酸代谢,6. AGE-RAGE 信号通路,7. 甘氨酸、丝氨酸和苏氨酸代谢,8. 氨基酸的生物合成,9. 吞噬体,10. 同源重组,11. 真核生物核糖体合成,12. 半乳糖代谢,13.泛素介导蛋白水解,14. 碱基切除修复,15. 叶酸-碳库代谢通路,16. 核酸切除修复,17. 范可尼贫血通路,18. 错配修复,19. 蛋白酶体,20. DNA 复制。Fig. 4 KEGG functional annotation statistics of differentially expressed genes of gonad tissue 1. spliceosome, 2. folate biosynthesis, 3. amino sugar and nucleotide sugar metabolism, 4. glycolipid metabolism, 5. alanine, aspartate and glutamate metabolism, 6. AGE-RAGE signaling pathway, 7. glycine,serine and threonine metabolism, 8. biosynthesis of amino acids, 9. phagosome, 10. homologous recombination, 11. ribosome biogenesis in eukaryotes, 12. galactose metabolism, 13. ubiquitin mediated proteolysis,14. base excision repair, 15. one carbon pool by folate, 16. nucleotide excision repair, 17. Fanconi anemia pathway, 18. mismatch repair, 19.proteasome, 20. DNA replication.
2.6 荧光定量PCR 验证
从显著差异表达基因中随机挑选14 个进行qRT-PCR 验证(图5)。结果显示,被测基因的表达变化与转录组测序结果一致,表明转录组数据分析是真实可信的。

图5 差异表达基因的qRT-PCR 及转录组的比较分析Fig. 5 Comparison of DEGs by qRT-PCR and transcriptome analysis
2.7 贻贝性腺组织中能量代谢相关酶活性
饥饿胁迫处理对厚壳贻贝性腺组织中HK、PFK、SDH、MDH 和PEPCK 的活性变化见图6。结果显示,与对照组相比,饥饿胁迫90 d 后,厚壳贻贝性腺组织中HK、PFK、MDH 和SDH 活性均显著降低(P<0.05),而7PEPCK 活性显著升高(P<0.05)。

图6 饥饿胁迫下贻贝性腺组织中能量代谢相关酶活性变化PEPCK.磷酸烯醇式丙酮酸羧激酶,HK.己糖激酶,PFK.磷酸果糖激酶,MDH.苹果酸脱氢酶,SDH.琥珀酸脱氢酶,下同 “*” 代表显著差异,P<0.05; “**” 代表极显著差异,P<0.01。Fig. 6 Changes of enzyme activities related to energy metabolism in gonad tissue under starvation PEPCK. phosphoenolpyruvate carboxykinase, HK. hexokinase, PFK.phosphofructokinase, MDH. malate dehydrogenase, SDH. succinate dehydrogenase, the same below. "*" represents significant difference,P<0.05; "**" represents very significant difference, P<0.01.
3 讨论
厚壳贻贝因其营养价值高、养殖周期短、抗病能力强而成为我国沿海地区重要的经济贝类之一,特别是在浙江省嵊泗县,贻贝养殖已成为该地区经济发展的支柱产业[1]。由于贻贝自稚贝包苗开始就全部依靠海域的天然饵料摄食生长,因此,海域饵料的分布不均和寒冷季节的饵料不足严重影响贻贝的生长和育肥[17]。而目前关于饥饿胁迫对贻贝影响的研究较少,对贻贝的饥饿耐受力以及适应机制并不清楚。贻贝体内的性腺是最大的可食用部分,也是育肥和繁殖后代最为关键的组织,饥饿胁迫下性腺的生理变化和分子调控直接影响到贻贝的品质。目前已有研究表明,在短期饥饿状态下贻贝通过激活自噬途径以及能量代谢方面的调控来调节代谢水平和能量分配[4],以适应食物短缺对机体造成的威胁。本项目以厚壳贻贝为研究对象,探究其长期饥饿下的存活状况并进行性腺组织的转录组测序分析,结合性腺中能量代谢相关酶活性的变化,全面解析厚壳贻贝在长期饥饿下的生存策略以及能量调控和代谢响应机制。
本研究的结果显示,厚壳贻贝在90 d 的饥饿胁迫下存活率为86%,相较于鱼类和其他软体动物类而言,贻贝具有较强的饥饿耐受性。如管角螺(Hemi fusus tuba)在饥饿40 d 后存活率为100%[18],但随着饥饿时间的延长,其存活率是否下降并没有可参考数据。仿刺参(Apostichopus japonicus)在饥饿40 天后存活率下降至32%[19]。马氏珠母贝(Pinctada martensi)在 饥 饿 12 d 后 存 活 率 为89.9%[20]。雪蟹(Chinopecetes opilio)在饥饿119 d时存活率仅为40%[21]。由此可见,贻贝在软体动物中对长期饥饿具有一定的耐受力,这种饥饿耐受的背后必然存在一定的生理响应和分子调控机制。
为深入解析贻贝长期饥饿下的生理变化和调控机制,对饥饿90 d 的厚壳贻贝性腺组织进行了转录组测序和分析,结果显示与细胞增殖有关的DNA 复制通路显著富集,该通路上的相关基因表达量均显著下降(图7)。DNA 复制是细胞有丝分裂过程中的重要环节,有丝分裂细胞周期是一个受多种因素调控的复杂过程,其中细胞周期蛋白是控制细胞周期的重要因素,可被细胞周期蛋白依赖的激酶激活[22]。如Wee1 作为一种核激酶,可以通过抑制细胞周期蛋白CDK 的活性来阻止细胞进行有丝分裂,CDK 则在细胞分裂过程中维持正常的有丝分裂,发挥着关键作用[23]。长期饥饿下性腺的转录组数据表明,细胞周期蛋白依赖性蛋白1 (CDK1)和细胞周期蛋白依赖性蛋白2(CDK2)的表达均显著下调,由此说明贻贝通过调节细胞周期蛋白依赖性蛋白表达来调控细胞周期,减少细胞分裂过程以维持饥饿过程。

图7 饥饿胁迫下葡萄糖代谢和DNA 复制相关差异表达基因网络图红色字体为显著上调表达基因,绿色字体为显著下调基因。Fig. 7 Network diagram of differentially expressed genes related to glucose metabolism and DNA replication under starvation stress Significantly up-regulated genes are marked in red, significantly down-regulated genes are in green.
此外,E2F3 在细胞周期调控中也到起关键作用[24],CDC6 和MCM 蛋白等大量编码DNA 复制活性基因的转录受细胞生长调节,并依赖于E2F3[25],CDC6 是DNA 复制的重要调节因子,在细胞周期进程从S 期到M 期的转变中发挥重要作用[26]。MCM 是一种由6 个亚基组成的DNA 解旋酶,是基因组DNA 复制所必需的[27]。CDC20 对于细胞分裂的调节也是必不可少的,此外它另一个重要的功能是促进染色单体的分离[28]。本研究显示,在饥饿胁迫下贻贝性腺中e2f3、cdc20 和mcm5 基因的表达量均显著下调,转录组数据进一步显示,长期饥饿下贻贝性腺中的mcm2、mcm3、mcm4、mcm5、mcm6 和mcm7 的表达量均显著下调,而复制蛋白A (RPA)在饥饿时也显著下调,它是一种普遍存在的ssDNA 结合蛋白,在许多DNA 加工途径中起作用,以维持基因组完整性[29]。
有丝分裂是细胞周期的一部分,其中复制的姐妹染色单体被分成两条染色体,本研究中与有丝分裂相关的基因(ncapd2、ncapg、smc2 和smc4)表达也均显著下调,而NCAPD2、NCAPG、SMC2 和SMC4 参与染色体凝聚[26-27,30]。已有的研究显示,细胞增殖可以使水生生物因饥饿而无法生存[31]。而在饥饿胁迫下贻贝下调的基因多与细胞周期、DNA 复制、有丝分裂等相关,KEGG 代谢通路富集结果进一步显示细胞周期显著富集。由此可推断,当贻贝遭受长期饥饿胁迫时,其细胞生长几乎处于停滞状态,细胞内的DNA 复制均处于较低的水平,以尽可能地维持细胞稳态,从而减少能量消耗,最大程度延长生命和代谢。
此外,在贻贝转录组的差异表达基因中也筛选到多个与糖酵解和糖异生等能量代谢相关的差异表达基因。糖酵解和糖异生是碳水化合物合成和分解的重要代谢途径[32],在饥饿状态下,水生动物一般会通过抑制糖酵解和磷酸戊糖相关酶和激活糖异生相关酶活性来调节糖代谢从而维持血糖平衡[33]。葡萄糖是许多组织所必需的能量来源,为了在长期饥饿中满足代谢要求并保持稳定的葡萄糖水平,水生动物会激活糖原分解或从头合成葡萄糖的过程(糖异生)[34]。因此,促进非碳水化合物前体从头合成葡萄糖的糖异生作用对水生动物的葡萄糖动态平衡至关重要[35]。饥饿胁迫会引起大多数水生动物的糖异生的增加和与糖异生相关的基因的mRNA 水平的上调。在本研究中,饥饿3 个月的贻贝葡萄糖异生酶PEPCK 的表达显著上调,PEPCK 能催化草酰乙酸脱羧转变为磷酸烯醇式丙酮酸,是糖异生途径的关键限速酶。控制PEPCK 活性对维持正常血糖水平有着重要意义,PEPCK 的上调意味着饥饿胁迫下贻贝的糖异生速率增加,通过糖异生作用的促进来维持饥饿期间的葡萄糖动态平衡,类似的结果在异育银鲫[11]和中华蛸(Octopus sinensis)[36]中也有报道。此外,饥饿胁迫也会抑制葡萄糖利用率。糖酵解途径的主要限速酶是PFK 和HK,其中PFK 是衡量碳水化合物利用的重要代谢酶[37],HK 催化葡萄糖生成6-磷酸葡萄糖[38]。本研究中,与对照组相比,饥饿胁迫组的PFK 和HK 活性均显著下调,说明贻贝在饥饿状态下通过抑制糖酵解来调节葡萄糖代谢。生物在遭受饥饿胁迫时,需要降低能量的消耗来维持基本生存,糖酵解限速酶的下调意味着与葡萄糖转化相关的信号通路受到抑制。如虹鳟的葡萄糖激酶编码基因表达水平和酶活性在禁食14 d 后下降[9],与本研究的结果一致。另外,SDH 和MDH 是三羧酸循环途径中调控脱氢反应的关键酶[39],可在一定程度上反映水生动物的有氧代谢水平[40],本研究表明,贻贝在饥饿后性腺组织SDH 活性和MDH 活性显著降低,说明在饥饿状态下尽可能减少葡萄糖的消耗以维持血糖浓度,这也是贻贝面对饥饿胁迫时的能量代谢生理响应对策[41]。由此可见,贻贝在饥饿状态下通过调节能量代谢有关的酶活性来维持血糖水平从而维持基本生存。
综上所述,贻贝对饥饿胁迫的响应是受一系列的生理活动的调节和能量代谢的调控而实现的,与细胞周期相关的代谢通路DNA 复制、能量代谢相关的基因表达以及相关酶活性的改变构成了贻贝长期饥饿下的复杂的代谢调控网络(图7),而该代谢网络图上的相关基因功能的验证还有待进一步研究。
4 结论
本实验探究了长期饥饿下贻贝的存活率和性腺的转录组变化以及能量代谢相关酶活性的变化,全面解析贻贝对长期饥饿的生理响应策略和分子调控机制。研究结果显示,厚壳贻贝在饥饿90 d时的存活率为86%,相较于鱼类和其他软体动物而言,贻贝具有较强的饥饿耐受性。通过比较饥饿组和对照组的转录组,发现贻贝通过调控能量代谢相关基因以及DNA 复制相关通路来应对长期饥饿造成的损伤。转录组鉴定到一些参与贻贝能量代谢相关酶的基因(hk、pfk、mdh、sdh、pepck和pc等),表明贻贝在长期饥饿胁迫下通过调控相关的能量代谢酶来满足代谢要求,并保持稳定的葡萄糖水平,从而维持自身生存。此外在饥饿胁迫下,DNA 复制、错配修复、碱基切除修复等与细胞分裂有关的代谢通路显著富集,相关基因的表达量显著下降,从而减少不必要的能量消耗,最大程度地延长生命和代谢。本研究为深入解析贻贝饥饿胁迫下的生理生化响应和分子机制提供重要的理论依据,同时为揭示其适应饥饿胁迫的能量利用和分配策略提供有效的途径。
(作者声明本文无实际或潜在的利益冲突)

