芸薹根肿菌遗传多样性分析
左丹丹 尚静 黄云

摘要 [目的]研究来自全国范围内芸薹根肿菌的遗传多样性,并探索ISSR分子标记与Williams生理小种鉴定结果之间的关系。[方法]对100条ISSR引物进行筛选,将筛选出的引物对48株根肿菌DNA进行扩增,根据扩增的多态性条带建立根肿菌聚类分析图,对根肿菌进行遗传多样性分析,并与Williams生理小种鉴定结果进行对比。[结果]100条ISSR引物中筛选出3条引物能扩增48株根肿菌DNA,获得清晰条带。ISSR聚类分析结果可将11号生理小种与其他生理小种区分开来,但不能以Williams生理小种鉴定结果标准将根肿菌完全区分开来,并且根肿菌的地理来源与其遗传距离并无相关性。[结论]该研究为利用分子手段鉴别根肿菌生理小种的研究奠定了基础。
关键词 芸薹根肿菌;ISSR;分子标记;遗传多样性
中图分类号 S432.4 文献标识码 A 文章编号 0517-6611(2024)09-0079-05
doi:10.3969/j.issn.0517-6611.2024.09.017
开放科学(资源服务)标识码(OSID):
Evaluation of Genetic Diversity of Plasmodiophora brassicae
ZUO Dan-dan1, SHANG Jing2, HUANG Yun2
(1.Meishan Pharmaceutical College, Meishan, Sichuan 620200;2.Sichuan Agricultural University, Chengdu, Sichuan 611130)
Abstract [Objective]To study the genetic diversity of Plasmodiophora brassicae and to explore the relationship between ISSR markers of Plasmodiophora brassicae and Williamsrace identification results. [Method]100 ISSR primers were screened, and the selected primers were used to amplify the DNA of 48 P.brassicae from all over the country. Based on the amplified polymorphic bands, a cluster analysis chart was established to analyze the genetic diversity of P.brassicae and compare the results with Williamsrace identification.[Result]Three primers were screened out of 100 ISSR primers to amplify the DNA of 48 Plasmodiophora brassicae to obtain clear bands. The results of ISSR clustering analysis could distinguish the Williamsrace No.11 from other races, but could not completely distinguish Plasmodiophora brassicae, and the geographical origin of P.brassicae had no correlation with its genetic distance.[Conclusion]This study lays a certain foundation for the molecular identification of Plasmodiophora brassicae.
Key words Plasmodiophora brassicae;ISSR;Molecular marker;Genetic diversity
作者簡介 左丹丹(1993—),女,贵州贵阳人,讲师,硕士,从事根肿病及中草药种植研究。*通信作者,教授,从事植物病理研究。
收稿日期 2023-07-07;修回日期 2023-07-28
根肿病是由芸薹根肿菌(Plasmodiophora brassicae Woronin)侵染引起的主要发生在十字花科作物上的一种世界性土传病害[1-2],随着全球范围内农产品生产与运输,根肿菌会随着商品的运输而传播。研究发现,根肿病发病时最高可导致减产50%以上,危害面积可达到实际生产面积的30%以上[3],对十字花科植物的产量和品质危害严重。根肿病的主要症状为植株根部膨大,秋天叶片变黄,春天营养输送受到阻碍,植株逐渐枯萎,这些症状都会导致作物的产量以及质量受到严重影响[4-5]。现已研究出划分根肿菌生理小种的几种鉴别系统,使用范围最广的是Williams鉴别系统和ECD鉴别系统。不论是Williams还是ECD鉴别方法,都耗时长、劳动力需要量大,结果易受环境影响,且由于根肿菌田间分离系的异质性的影响,生理小种鉴定结果的解释通常会受不均匀反应限制[6]。此外,致病基因型可能会被病根的非致病基因型所掩盖[7-8]。
前人已有研究表明,根肿菌田间分离系的遗传多样性能使病原菌克服植株抗性[9],这对根肿病抗性品种的研究是一个挑战[10]。近几年,分子标记已用于鉴别根肿菌田间分离系[11-16]。
简单重复序列间(ISSR)分析是常用的测定遗传多样性技术。优点是有很多可见位点,丰富的多态性,可靠的鉴别信息以及更低的技术要求及花费[17]。因此,ISSR已被广泛用于检测很多物种的遗传多样性及变异性[18-24]。
根肿菌现有的生理小种鉴别体系存在很多问题,如耗时长,劳动量需求大,结果不稳定,易受环境影响。近年来,利用分子标记方法区分根肿菌生理小种的研究越来越多,用分子方法建立起一套快速、科学的芸薹根肿菌生理小种鉴定体系已成为研究趋势。笔者通过ISSR分子标记方法,对来自我国不同地区的根肿菌进行遗传多样性分析,形成的UPMGA聚类分析图与生理小种鉴别结果进行对比,以期探索不同地理来源的根肿菌与其遗传背景的关系以及生理小种的分布与其遗传背景之间的关系,为利用分子手段建立生理小种体系的研究奠定基础。
1 材料与方法
1.1 菌株来源
菌根主要采自我国根肿病高发地区的9个省市。菌根材料清洗干净,室内晾干后储藏在-20 ℃冰箱备用。48个根肿菌材料采样地点见表1。菌株生理小种采用Williams鉴别体系进行鉴定。
1.2 菌悬液的制备
休眠孢子悬浮液的制备参照马淑青[25]的方法并进行修改:取保存于-20 ℃冰箱内的肿根,在25 ℃下腐熟5 d。将根瘤切成小块,用匀浆机加入无菌水打磨3 min左右,打磨后的液体通过8层纱布过滤,滤液移入50 mL干净离心管,静置10 min左右。将静置液体的上清液及上层的灰色沉淀移入另一支50 mL干净离心管中,加30 mL无菌水悬浮,5 000 r/min离心5 min。留沉淀,加入5 mL 50%蔗糖溶液,用涡旋振荡仪振荡,使其混合均匀,5 000 r/min离心10 min。上清液移入另一支50 mL干净离心管,加45 mL无菌水,5 000 r/min离心10 min,沉淀溶于45 mL无菌水中,5 000 r/min离心10 min,留沉淀(该步骤重复2~3次),沉淀溶于5 mL灭菌水中。4 ℃保存备用,24 h内提取DNA。
1.3 休眠孢子DNA提取及DNA质量检测
DNA的提取采用传统CATB方法并进行改良。配制1.0%琼脂糖凝胶,倒胶点样成像,将跑出清晰条带的DNA保存备用,不清晰、被降解的弃去。
1.4 ISSR-PCR扩增
ISSR引物来自加拿大Brithsh Colunbia大学公布的100条引物,由江苏金唯智生物技术合成公司合成。ISSR-PCR反应体系总体积15 μL,其中包含1 μL DNA模版(80 ng/μL),0.5 μL引物(10 μmol/L),7.5 μL 2×PCR Mastermix(每1 mL含40 U Taq酶)以及6 μL ddH2O。ISSR-PCR扩增反应程序包括94 ℃预变性持续5 min,30 s 94 ℃变性、45 s Tm ℃ 复性、2 min 72 ℃延伸、7 min 72 ℃后延伸共重复40次,最后于12 ℃ 保存。PCR反应体系先进行退火温度和引物的预筛选。条带结果用2%凝胶电泳进行检测。
1.5 数据处理
ISSR属于显性标记,同一引物扩增产物中电泳迁移率一致的条带被认为具有同源性,相同移位上有带的记为“1”,无带的记为“0”,弱带也记为“1”,形成只有“1”和“0”的二次元数据矩阵。用NTSYSpc-2.01软件进行数据处理,建立UPGMA树状图。
2 结果与分析
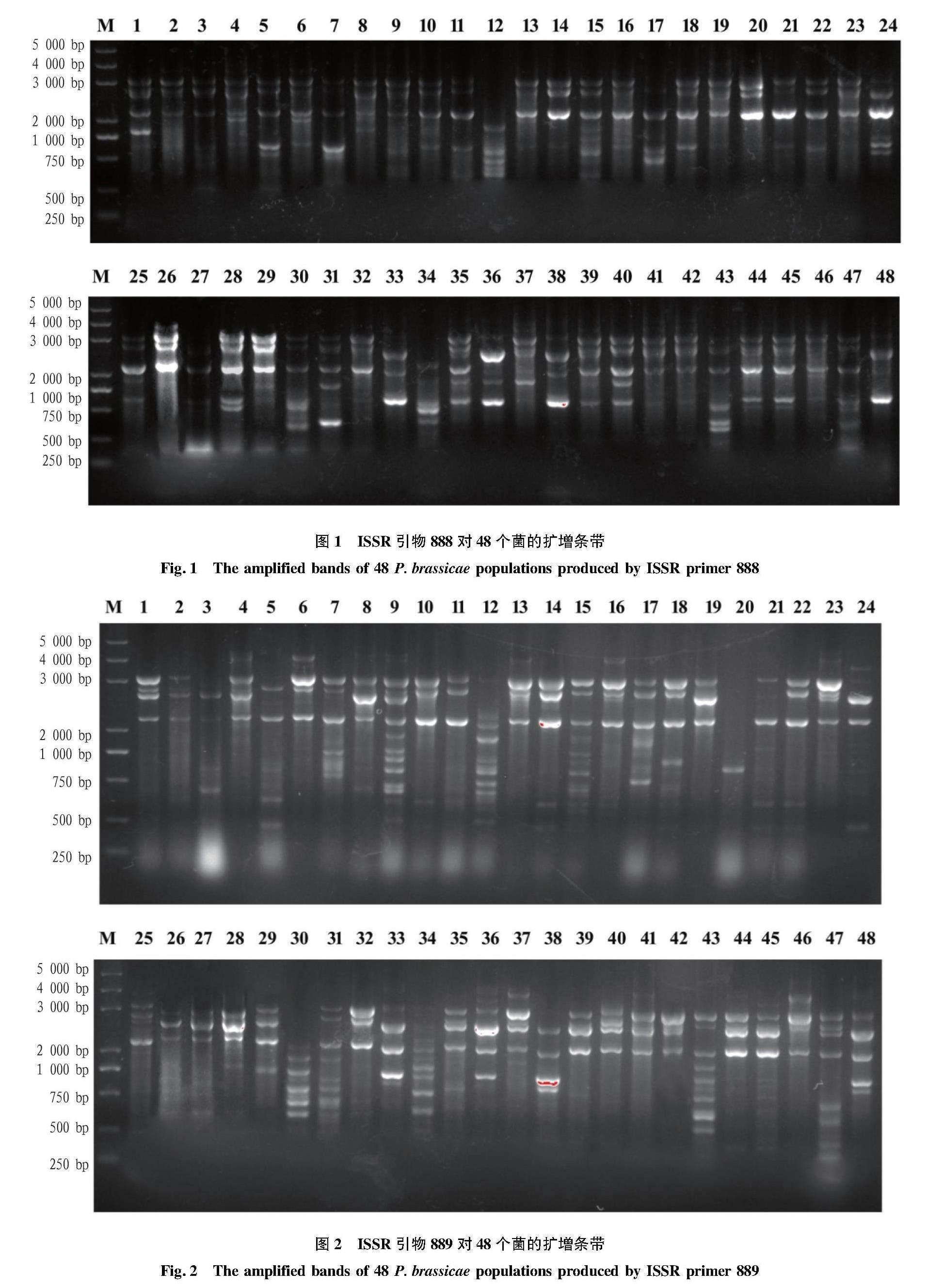
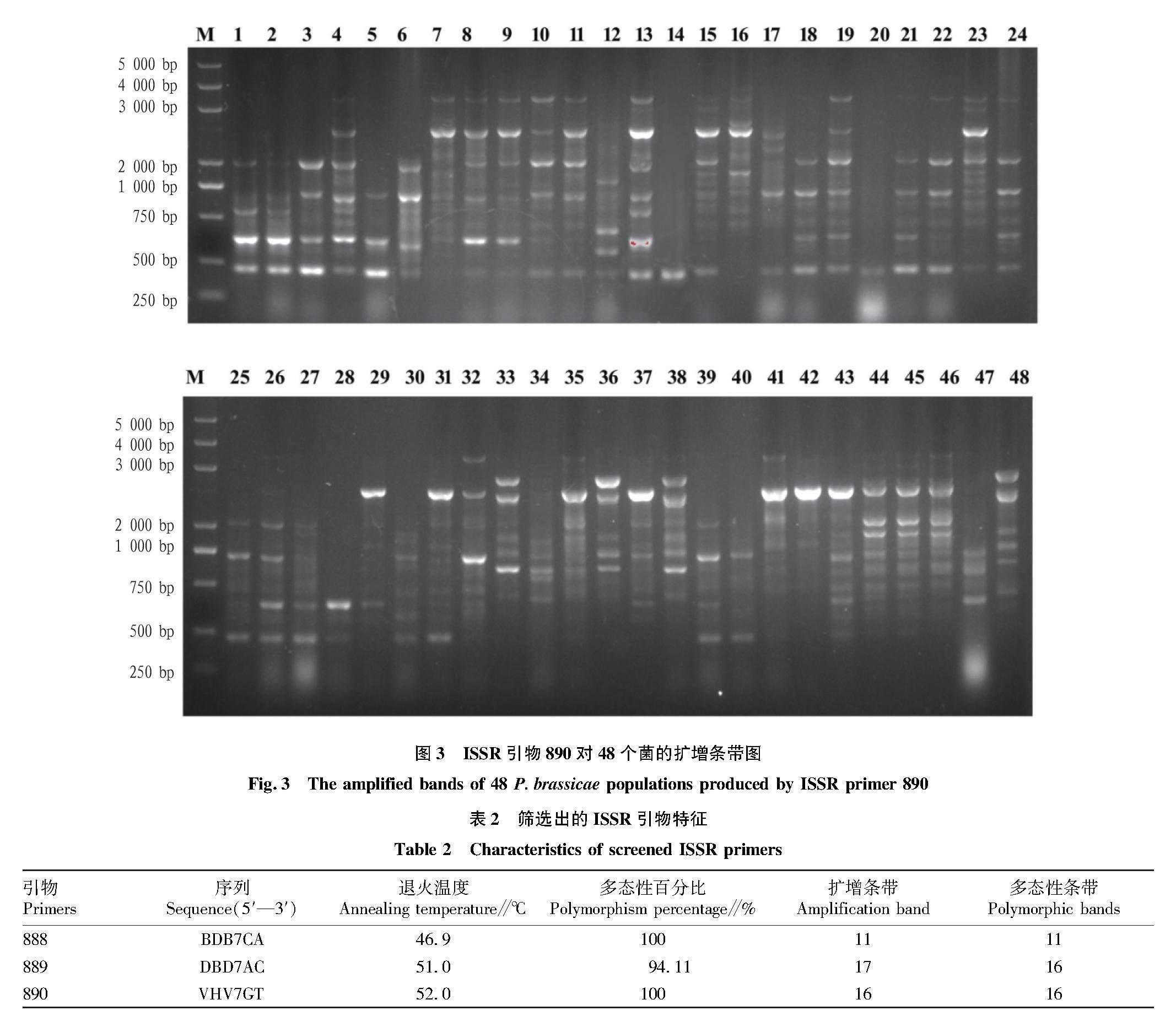
2.1 ISSR-PCR擴增结果
根据引物的扩增结果,从100条引物中筛选出3条扩增条带清晰、多态性明显、重复性好的引物,分别是888、889、890(图1~3)。将出现频率为5%~95%的条带视为多态性条带,3条ISSR引物对病原菌共扩增出44条带,其中多态性条带数为43,多态性比例为97.73%,平均每条引物的多态性条带数14.33(表2)。3条引物中引物889的多态性比例最小,为94.11%,另外2条的多态性比例均达到100%。引物扩增条带大小在250~4 000 bp,引物的扩增条带数在1~17。
2.2 根肿菌ISSR遗传多样性分析
结果表明,48株根肿菌菌株之间的遗传相似系数为0.58~1.00。聚类结果显示(图4),同是来自江西省南昌市南昌县的33号、34号菌株聚在两大不同类群上,33号归为类群Ⅰ,34号归为类群Ⅱ,2菌株遗传相似系数只有0.580 5,而来自四川省凉山州冕宁县的27号菌以及来自安徽省宣城市绩溪县的39号菌遗传相似系数为1.00,来自黑龙江省哈尔滨阿城区的44号菌以及来自四川省德阳市罗江区的46号菌遗传相似系数高达0.954 5,这说明菌株的地理来源与其遗传背景相关性不大。同为11号生理小种的30号以及34号菌归为类群Ⅱ,遗传相似系数为0.772 7,而遗传相似系数高达1.00的27号以及39号的菌株为4号生理小种,遗传相似系数为0.954 5的1号以及2号菌株,遗传相似系数为0.931 8的10号和11号菌株以及遗传相似系数为0.931 8的33号、38号菌株也都是4号生理小种。4号生理小种被聚类到多个支上,同为4号生理小种的31号菌和1号菌遗传相似系数为0.647 3,说明ISSR聚类分析结果可以将11号生理小种与其他生理小种区分开来,4号生理小种遍布遗传背景差异较大的菌至遗传背景差异较小的菌。同为7号生理小种的18号菌以及14号菌遗传相似系数为0.782 4,但与同为7号生理小种的46号菌株的遗传相似系数只有0.725 6,说明ISSR聚类分析结果并不能以Williams生理小种鉴定结果标准把不同根肿菌完全区分开来。
3 讨论
关于根肿菌遗传多样性,国外从1996年就开始进行研究。Mller等[26]用RAPD引物对根肿菌进行分子鉴定,结果
表明,引物OPA09对3个单孢系扩增出了相同的条带,引物
OPA07扩增条带的结果与ECD生理小种鉴定结果一致。Yano等[27]用32条RAPD引物对来自日本7个地区的16个
根
肿菌样品进行PCR扩增,UPGMA聚类结果显示,9号生理小种的2个菌单独聚在一类,与4号生理小种和1号生理小种的菌样区分开来,但不能把对白菜抗病品种有致病性的5个菌和其他11个菌区分开来。Manzanares-Dauleux等[15]用19个RAPD引物对来自7个不同田块的37个根肿菌单孢分离系进行分析,并用some鉴别体系对这37个单孢系进行生理小种的鉴别,结果表明,属于P1生理小种的菌都能在约1 200 bp处扩增出1条特异的共有的1个片段——OPLl4(1 200),对这个片段进行胶回收、克隆转化和测序,建立其SCAR标记,设计出了用于检测P1生理小种的特异性引物。吴健雄[28]用13条RAPD引物对来自湖北的24个根肿菌进行扩增,并将条带图进行UPGMA聚类,聚类结果发现,Williams生理小种鉴定结果与聚类结果不能一一对应,同一小种间也存在着遗传差异。柴阿丽等[29]利用肌动蛋白和多聚泛素蛋白这2个位点基因,设计出特异性引物对ActF1/ActR1 和UbiF1/UbiR1 对来自我国11个省份、12个寄主的43个根肿菌样进行PCR扩增。UPGMA聚类分析结果表明,根肿菌遗传变异与地理来源密切相关,与寄主无关。任静[30]对40 份来源于不同省份和地区的根肿菌进行了SSR 多态性分析发现,根肿菌生理小种的遗传分化与其地理来源有密切关系。许路阳[31]筛选出4 个SRAP引物组合,对72 个根肿菌样本进行遗传多样性分析,发现SRAP分子标记技术能将9 号生理小种分离开来。沈旭[32]采用RAPD 和SRAP 方法对48 个根肿菌单孢系进行了遗传多样性分析,发现RAPD 和SRAP 能在一定程度上对单孢系按照生理小种和地理位置进行分类,但并不能完全准确地区分生理小种。馬坡等[33]根据SSR引物聚类结果发现,同一地区的根肿菌大多聚类到一起,但同时存在变异。赵辉等[34]利用SSR技术对32 个主栽油菜品种上分离的 100株油菜根肿病菌的遗传分化进行研究发现,油菜根肿病菌的遗传分化与地理来源和寄主品种都有着一定相关性,但主要依赖地理来源间的差异,相近地理距离的根肿菌有着更近的亲缘关系。
笔者尝试用ISSR分子标记方法将11号生理小种与其他生理小种的菌区分开来,且遗传相似系数较高的菌为相同生理小种,这是用ISSR方法探索根肿菌与生理小种之间的关系,这对于用分子手段鉴别根肿菌生理小种的研究奠定了一定的基础。
参考文献
[1] DIXON G R.The occurrence and economic impact of Plasmodiophora brassicae and clubroot disease[J].Journal of plant growth regulation,2009,28(3):194-202.
[2] STRELKOV S E,DIXON G R.Clubroot(Plasmodiophora brassicae)on canola and other brassica species-disease development,epidemiology and management[J].Canadian journal of plant pathology,2014,36:1-4.
[3] 朱琢然.油菜根肿病的危害及综合防控技术[J].安徽农学通报,2013,19(16):84,99.
[4] JOHNSON T W,KARLING J S.The Plasmodiophorales[J].BioScience,1970,20(10):632.
[5] WALLENHAMMAR A C.Monitoring and control of Plasmodiophora brassicae in spring oilseed brassica crops[J].Acta horticulturae,2010(867):181-190.
[6] JONES D R,INGRAM D S,DIXON G R.Characterization of isolates derived from single resting spores of Plasmodiophora brassicae and studies of their interaction[J].Plant pathology,1982,31(3):239-246.
[7] VOORRIPS R E.Production,characterization and interaction of single-spore isolates of Plasmodiophora brassicae[J].European journal of plant pathology,1996,102(4):377-383.
[8] SOME A,MANZANARES M J,LAURENS F,et al.Variation for virulence on Brassica napus L.amongst Plasmodiophora brassicae collections from France and derived single-spore isolates[J].Plant pathology,1996,45(3):432-439.
[9] HIRAI M.Genetic analysis of clubroot resistance in Brassica crops[J].Breeding science,2006,56(3):223-229.
[10] DONALD E C,PORTER L J,FAGGIAN R,et al.An integrated approach to the control of clubroot in vegetable brassica crops[J].Acta horticulturae,2006(706):283-300.
[11] BUHARIWALLA H,GREAVES S,MAGRATH R,et al.Development of specific PCR primers for the amplification of polymorphic DNA from the obligate root pathogen Plasmodiophora brassicae[J].Physiological & molecular plant pathology,1995,47(2):83-94.
[12] ITO S,MAEHARA T,TANAKA S,et al.Cloning of a single-copy DNA sequence unique to Plasmodiophora brassicae[J].Physiological and molecular plant pathology,1997,50(5):289-300.
[13] ITO S,MAEHARA T,MARUNO E,et al.Development of a PCR-based assay for the detection of Plasmodiophora brassicae in soil[J].Journal of phytopathology,1999,147(2):83-88.
[14] FAGGIAN R,BULMAN S R,LAWRIE A C,et al.Specific polymerase chain reaction primers for the detection of Plasmodiophora brassicae in soil and water[J].Phytopathology,1999,89(5):392-397.
[15] MANZANARES-DAULEUX M J,DIVARET I,BARON F,et al.Assessment of biological and molecular variability between and within field isolates of Plasmodiophora brassicae[J].Plant pathology,2001,50(2):165-173.
[16] KLEWER,LUEREN,GRAF,et al.Restriction fragment length polymorphism markers to characterize Plasmodiophora brassicae single-spore isolates with different virulence patterns[J].Journal of phytopathology,2001,149(3/4):121-127.
[17] LABASTIDA E,COBIN D,HNAUT Y,et al.The use of ISSR markers for species determination and a genetic study of the invasive lionfish in Guanahacabibes,Cuba[J].Latin American journal of aquatic research,2015,43(5):1011-1018.
[18] HUANG Y,ZHANG C Q,LI D Z.Low genetic diversity and high genetic differentiation in the critically endangered Omphalogramma souliei(Primulaceae):Implications for its conservation[J].Journal of systematics and evolution,2009,47(2):103-109.
[19] ARRIGONI-BLANK M F,SANTOS M S,BLANK A F,et al.Analysis of genetic diversity of Laeliinae(Orchidaceae)in the State of Sergipe using ISSR markers[J].Genetics & molecular research,2016,15(2):1-9.
[20] SHARMA P,TIWARI S,TRIPATHI N,et al.Polymorphism analysis in advanced mutant population of oat(Avena sativa L.)using ISSR markers[J].Physiology and molecular biology of plants,2016,22(1):115-120.
[21] SAKI S,BAGHERI H,DELJOU A,et al.Evaluation of genetic diversity amongst Descurainia sophia L. genotypes by inter-simple sequence repeat(ISSR)marker[J].Physiology and molecular biology of plants,2016,22(1):97-105.
[22] GUO W L,GONG L,DING Z F,et al.Genomic instability in phenotypically normal regenerants of medicinal plant Codonopsis lanceolata Benth.et Hook.f.,as revealed by ISSR and RAPD markers[J].Plant cell reports,2006,25(9):896-906.
[23] SHARAFI A A,ABKENAR A A,SHARAFI A,et al.Genetic variation assessment of acid lime accessions collected from south of Iran using SSR and ISSR molecular markers[J].Physiology & molecular biology of plants,2016,22(1):87-95.
[24] XING P Y,LIU T,SONG Z Q,et al.Genetic diversity of Toona sinensis Roem in China revealed by ISSR and SRAP markers[J].Genetics & molecular research,2016,15(3):1-12.
[25] 馬淑青.油菜根肿病菌生物学特性、PCR检测及同工酶研究[D].雅安:四川农业大学,2006.
[26] MLLER M,HARLING R.Randomly amplified polymorphic DNA(RAPD)profiling of Plasmodiophora brassicae[J].Letters in applied microbiology,1996,22(1):70-75.
[27] YANO S,TANAKA S,ITO S I,et al.Variations of random amplified polymorphic DNA(RAPD)patterns among field populations of Plasmodiophora brassicae[J].Japanese journal of phytopathology,1997,63(3):179-182.
[28] 吴健雄.湖北省十字花科根肿病菌生理小种鉴定及遗传多样性分析[D].武汉:华中农业大学,2013.
[29] 柴阿丽,朱发娣,王惟萍,等.我国芸薹根肿菌遗传类群分化研究[J].华北农学报,2015,30(S1):309-315.
[30] 任静.芸薹根肿菌遗传多样性研究[D].沈阳:沈阳农业大学,2018.
[31] 许路阳.根肿菌的SRAP遗传多样性分析及大白菜抗根肿菌转录组分析[D].雅安:四川农业大学,2018.
[32] 沈旭.根肿病的再侵染及根肿菌单孢系生物学特性、遗传多样性、生理小种分子鉴定[D].雅安:四川农业大学,2019.
[33] 马坡,李晓楠,庞文星,等.芸薹根肿菌SSR标记的开发及遗传多样性研究[J].中国油料作物学报,2018,40(6):872-878.
[34] 赵辉,林宇丰,钟杰,等.湖南油菜根肿病菌遗传多样性分析[J/OL].分子植物育种,2022-05-30[2023-07-07].http://kns.cnki.net/kcms/detail/46.1068.s.20220527.1135.002.html.

