基于线粒体COX1和ND1基因顺序的黄河上游大鼻吻遗传多样性分析
杨立强,刘彦斌,苟金明,王吉祥,刘 凯,肖 伟,王永杰,杨瑞兰,柳 婷,刘 哲,连总强
(1.甘肃农业大学动物科学技术学院,兰州 730070;2.宁夏回族自治区水产研究所,银川 750001;3.宁夏渔业工程技术研究中心,银川 750001;4.喀什大学生命与地理科学学院,新疆喀什 844000;5.宁夏回族自治区水产技术推广站,银川 750001)
本研究利用线粒体细胞色素C氧化酶I(COX1)基因序列和线粒体NADH脱氢酶第一亚基(ND1)基因序列,初步研究了黄河上游3个不同地理群体大鼻吻的遗传多样性,探讨其群体关系及系统地位,为大鼻吻遗传资源保护和利用提供参考依据。
1 材料与方法
1.1 材料
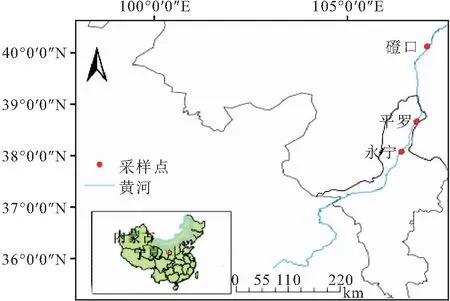
图1 大鼻吻采样水域Fig.1 Sampling location map of R. nasutus
1.2 基因组DNA提取
采用血液/细胞/组织DNA提取试剂盒(北京天根生化科技有限公司)提取大鼻吻样本基因组DNA。采用Nano-30核酸浓度测定仪(杭州奥盛仪器有限公司)检测浓度及OD值,采用1%琼脂糖凝胶电泳检测DNA的完整性后,于-20 ℃保存备用。
1.3 PCR扩增及测序
用于扩增目的片段COX1和ND1的引物序列分别为:COX1F:5′-TTTATCTTGTATTTGGTGC-3′和R:5′-CTTCAGAAGAAAGTGACAGAGTGGT-3′;ND1F:5′-ATACTGGGGTGGCAGAGCA-3′和R:5′-GGCGA AGGTTAGGGTGGTT-3′。大鼻吻线粒体基因COX1和ND1的PCR反应体系均为30 μL,其中,正反向引物各1 μL(10 pmol/μL),DNA样本 1 μL(50 ng),Premix 2×Taq(南京诺唯赞生物科技股份有限公司)15 μL,ddH2O 12 μL。PCR 反应程序为94 ℃预变性5 min;94 ℃变性30 min,两基因退火温度分别为COX1 55 ℃、ND1 51 ℃,退火30 s,72 ℃延伸1 min,35个循环;72 ℃终延伸10 min,产物4 ℃保存。PCR产物用2%琼脂糖凝胶电泳检测,将检测合格的样品送上海生工生物有限公司测序,测序引物与PCR扩增引物相同。
1.4 数据分析
利用DNASTAR Lasergene 11 Core Suite软件包中的SeqMan Pro软件对测序所得的原始序列峰图进行人工分析校对[9]。将合格的碱基序列采用Bioedit.exe软件进行多重对比,参照吻(GenBank登录号:NC 0244232.1)线粒体COX1和ND1基因序列,经人工校对、剪切后,保留同等长度的有效片段用于进一步分析[10]。通过MEGA 10.0统计COX1和ND1基因序列各碱基组成、保守位点、变异位点、简约信息位点及单突变位点,同时基于Kimura-2-parameter(双参数)模型计算两两群体之间的遗传距离(D),并用邻近法(Neighbor Joining,NJ)构建系统进化树[11]。采用DnaSP v6软件计算各群体单倍型数、变异位点数、单倍型多态(Hd)、核苷酸多态性(π)以及平均核苷酸差异(K)[12]等遗传多样性参数。Arlequin 3.0软件[13]进行群体间遗传分化指数检验和Tajima′sD中性检验及遗传变异分析。采用NETWORK 10.0软件来构建线粒体COX1和ND1基因序列的单倍型网络结构图[14]。
2 结果与分析
2.1 碱基组成及与序列变异
将分别测得的145条COX1、ND1序列进行比对,各群体碱基组成见表1。结果表明COX1序列长度为1 466 bp,变异位点11个,占全序列的0.75%,其中简约信息位点7个,单突变位点4个,分别占全序列的0.47%,0.27%。145条COX1序列中A、T、C、G碱基的平均含量分别为27.40%、29.85%、24.77%、17.98%;ND1序列的长度为975 bp,变异位点8个,占全序列的0.82%,其中简约信息位点7个,单突变位点1个,分别占全序列的0.72%,0.10%。145条ND1序列中A、T、C、G碱基的平均含量分别为29.22%、27.59%、27.90%、15.29%。由此可知,大鼻吻线粒体COX1、ND1基因在碱基组成上存在一定差异,但均具有明显的A/T碱基组成偏向性,这一特征与其他硬骨鱼类相类似[15]。
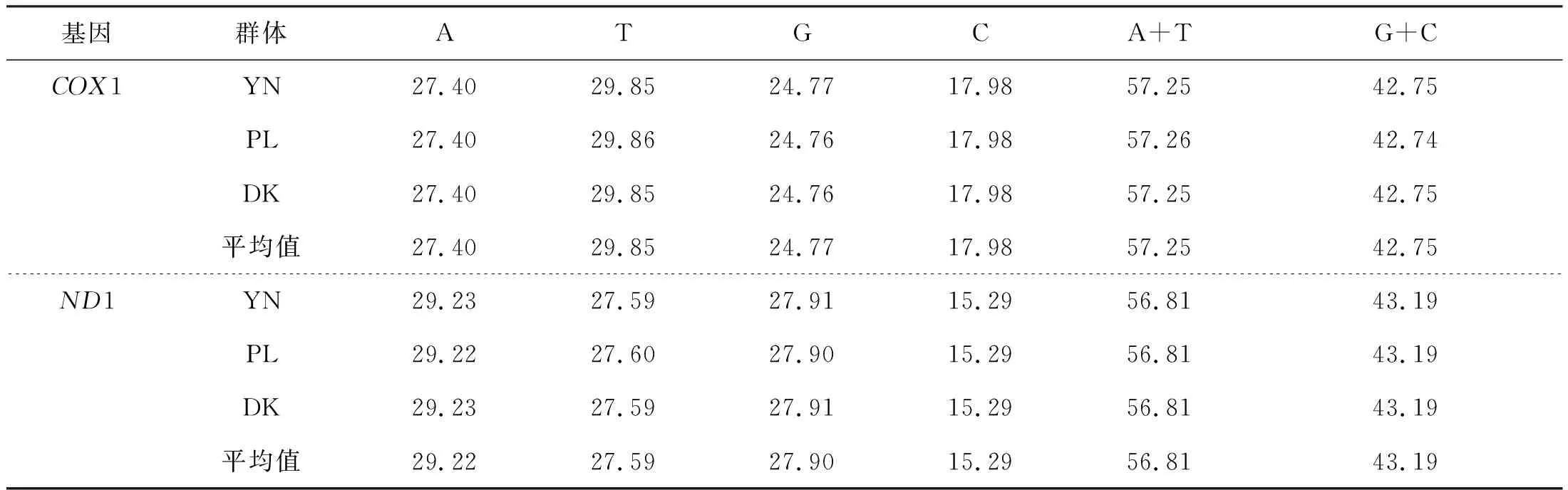
表1 基于线粒体DNA COX1和ND1序列的碱基组成Tab.1 Base competition based on mtDNA COX1 and ND1 sequences %
2.2 遗传多样性
在145条COX1基因序列中,共定义了20个单倍型,各群体单倍型多样度及核苷酸多样度见表2。由表2可知,大鼻吻YN群体单倍型多态性最高(Hd=0.764),PL群体单倍型多态性最低(Hd=0.709)。PL群体核苷酸多态性最高(π=0.001 32),YN群体核苷酸多态性最低(π=0.001 22);在145条ND1基因序列中,共定义了17个单倍型,各群体单倍型多样度及核苷酸多样度见表3。由表3可知,大鼻吻PL群体单倍型多态性最高(Hd=0.443),YN群体单倍型多态性最低(Hd=0.288)。PL群体核苷酸多态性最高(π=0.000 63),YN群体核苷酸多态性最低(π=0.000 31)。
表2 基于线粒体DNA COX1序列的3个大鼻吻群体的遗传信息Tab.2 Genetic information of three populations of R.nasutus based on mitochondrial DNA COX1 gene
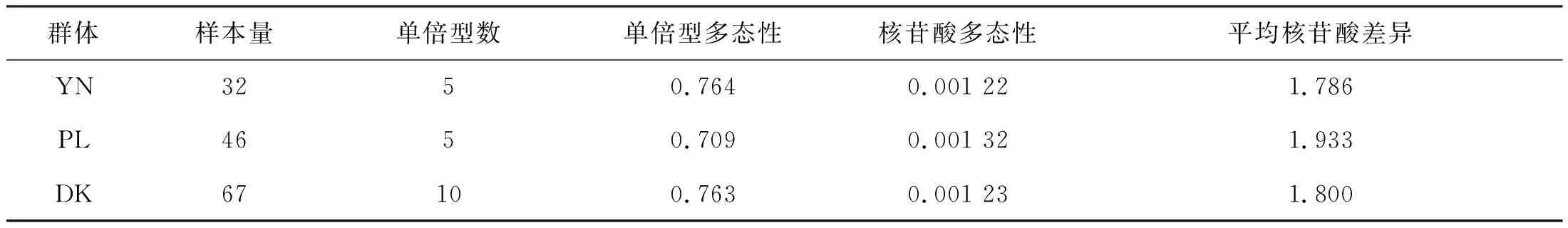
表2 基于线粒体DNA COX1序列的3个大鼻吻群体的遗传信息Tab.2 Genetic information of three populations of R.nasutus based on mitochondrial DNA COX1 gene
群体样本量单倍型数单倍型多态性核苷酸多态性平均核苷酸差异YN3250.7640.001 221.786PL4650.7090.001 321.933DK67100.7630.001 231.800
表3 基于线粒体DNA ND1序列的3个大鼻吻群体的遗传信息Tab.3 Genetic information of three populations of R.nasutus based on mitochondrial DNA ND1 gene
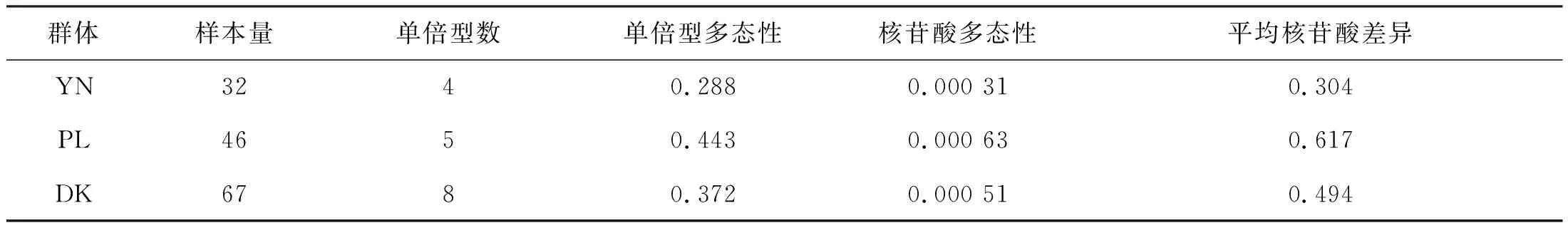
表3 基于线粒体DNA ND1序列的3个大鼻吻群体的遗传信息Tab.3 Genetic information of three populations of R.nasutus based on mitochondrial DNA ND1 gene
群体样本量单倍型数单倍型多态性核苷酸多态性平均核苷酸差异YN3240.2880.000 310.304PL4650.4430.000 630.617DK6780.3720.000 510.494
基于COX1基因序列共检测出11个单倍型,其中Hap1、Hap2、Hap3、Hap4为3个群体共享单倍型,占总数的36.36%;其余的均为单一单倍型,占总数的63.64%。Hap5是YN群体特有的单倍型;Hap6为YN群体和PL群体共享单倍型;Hap7、Hap8、Hap9、Hap10、Hap11为DK群体所独有;基于ND1基因序列共定义了9个单倍型,其中Hap1,Hap2为3个群体共享单倍型,占总数的22.22%;Hap3和Hap4为YN群体和PL群体共享单倍型,占总数的22.22%;Hap5为PL群体和DK群体共享单倍型,占总数的11.11%;其余的均为DK群体所特有的单一单倍型,占总数的44.44%。
2.3 遗传距离与遗传分化
基于COX1基因序列的Kimura双参数模型(Boot-strap=1 000)的遗传距离见表4。由此可知,大鼻吻3个群体组间遗传距离相同,组内遗传距离为0.001 22~0.001 32,组内遗传距离接近组间遗传距离;基于ND1基因序列的Kimura双参数模型(Boot-strap=1 000)遗传距离见表5。由此可知,大鼻吻3个群体组间遗传距离为0.000 41~0.000 558,群体间遗传距离接近。组内遗传距离为0.000 44~0.000 73,组内遗传距离接近组间遗传距离。
表4 基于COX1序列的大鼻吻群体间的遗传距离D和遗传分化指数FstTab.4 Genetic distance D and genetic differentiation index Fst among populations of R.nasutus based on COX1 sequence
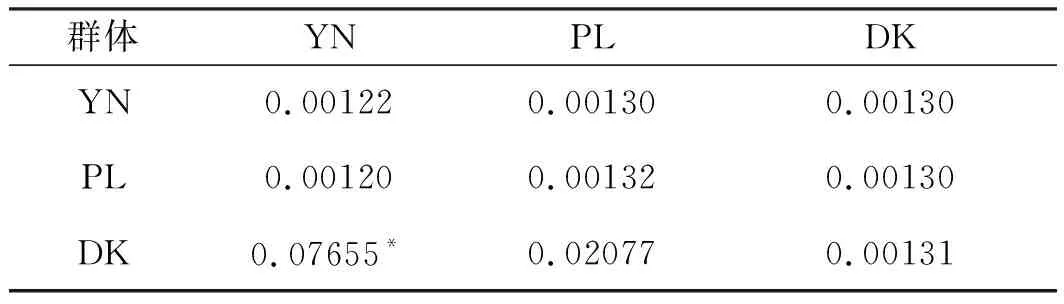
表4 基于COX1序列的大鼻吻群体间的遗传距离D和遗传分化指数FstTab.4 Genetic distance D and genetic differentiation index Fst among populations of R.nasutus based on COX1 sequence
群体YN PLDKYN0.001220.001300.00130PL0.001200.001320.00130DK0.07655*0.020770.00131
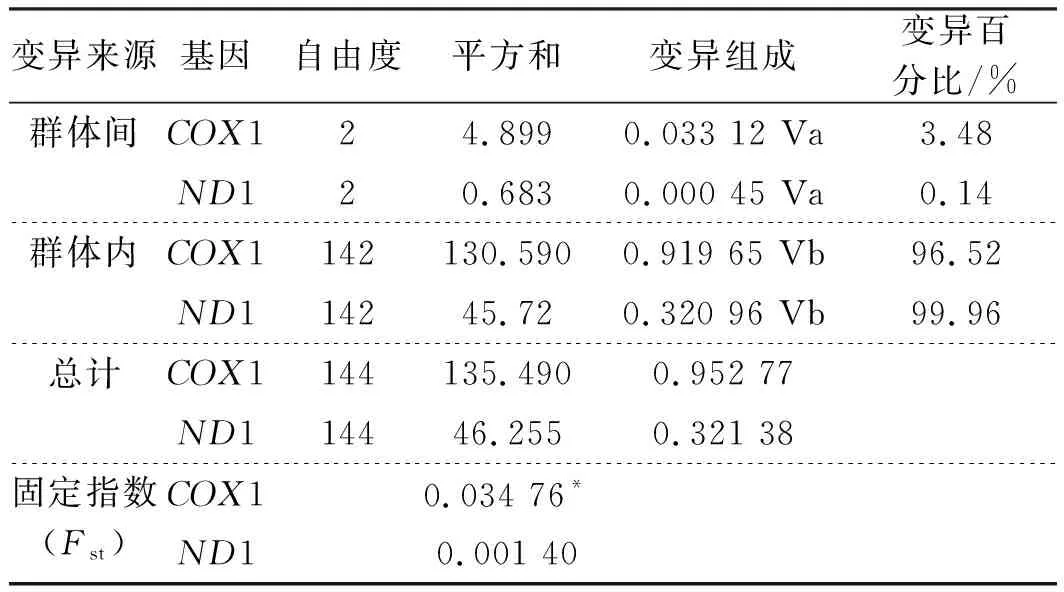
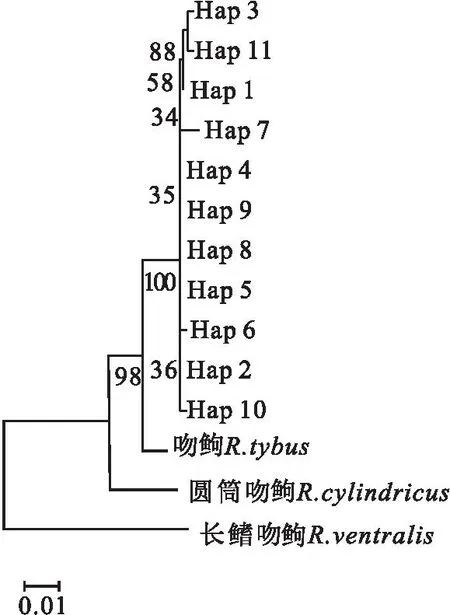
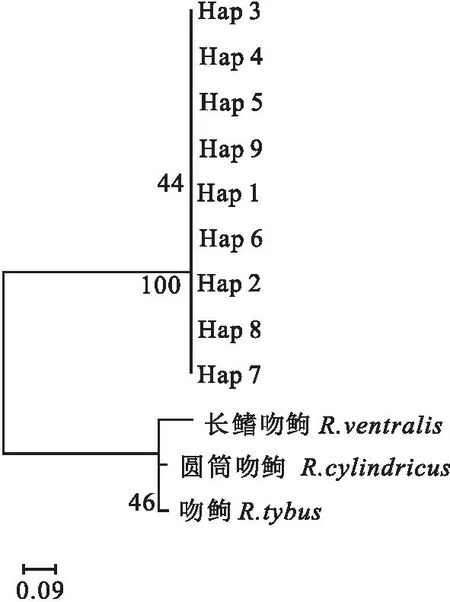
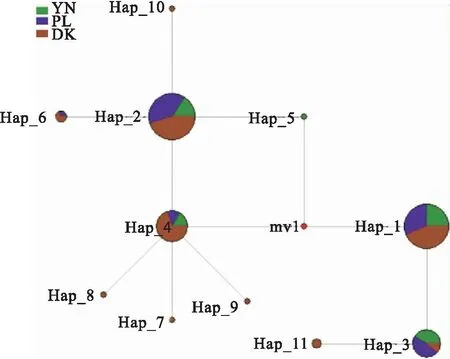
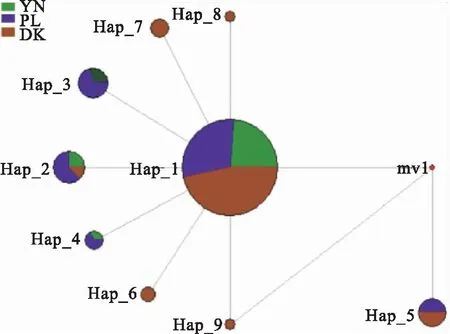
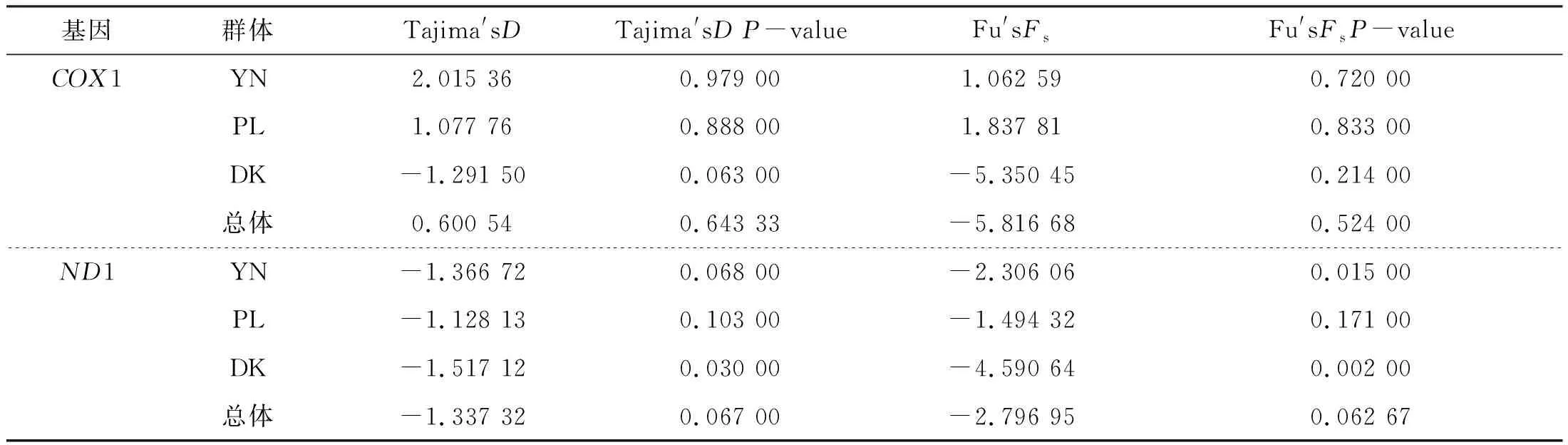
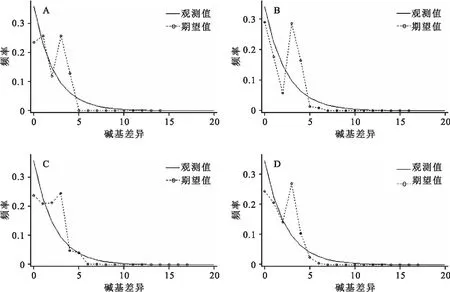
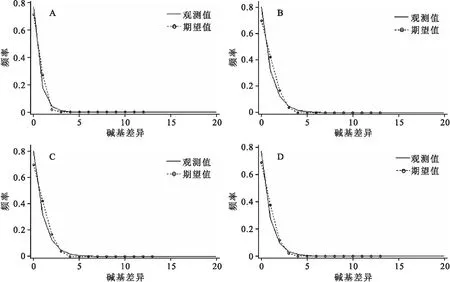
注:对角线为群体内遗传距离D;对角线左下方表示群体间分化指数Fst;对角线右上方表示群体间遗传距离D;*表示差异显著(0.01 表5 基于ND1序列的大鼻吻群体间的遗传距离D和遗传分化指数FstTab.5 Genetic distance D and genetic differentiation index Fst among populations of R.nasutus based on ND1 sequence 表5 基于ND1序列的大鼻吻群体间的遗传距离D和遗传分化指数FstTab.5 Genetic distance D and genetic differentiation index Fst among populations of R.nasutus based on ND1 sequence 群体YN PLDKYN0.000 440.000 470.000 41PL0.003 740.000 730.000 58DK0.003 590.008 350.000 67 基于COX1基因序列的大鼻吻群体间遗传分化指数见表4。结果显示,大鼻吻3个群体组间遗传分化指数为0.001 20~0.076 55,YN群体与DK群体间的遗传分化指数最大(0.076 55),YN群体与PL群体的遗传分化指数最小(0.001 20);基于ND1基因序列的大鼻吻群体间遗传分化指数见表5。结果显示,大鼻吻3个群体组间遗传分化指数为0.003 59~0.008 35,PL群体与DK群体间的遗传分化指数最大(0.008 35),YN群体与DK群体的遗传分化指数最小(0.003 59)。 基于COX1基因序列的分子方差(AMOVA)分析结果见表6,结果显示,大鼻吻3个群体的群体内遗传变异占96.52%,群体间的遗传变异占3.48%;基于ND1基因序列的分子方差(AMOVA)分析结果见表6,结果显示,3个群体的大鼻吻群体内遗传变异占99.96%,群体间的遗传变异占0.14%。 表6 大鼻吻群体间分子变异分析Tab.6 Analysis of molecular variance(AMOVA) among R.nasutus populations 表6 大鼻吻群体间分子变异分析Tab.6 Analysis of molecular variance(AMOVA) among R.nasutus populations 变异来源基因自由度平方和变异组成变异百分比/%群体间COX124.8990.033 12 Va3.48ND120.6830.000 45 Va0.14群体内COX1142130.5900.919 65 Vb96.52ND114245.720.320 96 Vb99.96总计COX1144135.4900.952 77ND114446.2550.321 38固定指数(Fst)COX10.034 76*ND10.001 40 注:*表示差异显著(0.01 基于线粒体COX1和ND1基因的单倍型序列,以吻(GenBank登录号:NC 0244232.1)、圆筒吻(GenBank登录号:KU 379652.1)、长鳍吻(GenBank登录号:KU 379653.1)为外群,用邻近法(Neighbor Joining,NJ)构建系统发育树(图2和图3)。结果显示,大鼻吻3个地理群体的单倍型均无明显的谱系分化,未发现构成一个单独的分支的群体。 图2 基于线粒体DNA COX1基因的单倍型NJ系统树Fig.2 Haplotype NJ system tree based on mitochondrial DNA COX1genes 图3 基于线粒体DNA ND1基因的单倍型NJ系统树Fig.3 Haplotype NJ system tree based on mitochondrial DNA ND1 genes 基于COX1基因序列的单倍型网络图见图4,结果显示,大鼻吻3个群体共鉴定出11个单倍型,11个单倍型无明显的支系划分,结果与NJ进化树一致。图中每个不同颜色代表不同的群体,每个圆圈代表一种单倍型,圆圈的面积大小表示其频率的高低。圆圈之间的连接线表示突发事件;单倍型网络图是以高频率优势单倍型H1、H2、H3、H4为中心,低频单倍型以网状围绕在其周围,表明大鼻吻3个群体遗传结构较为简单;基于ND1基因序列的单倍型网络图见图4,结果显示,大鼻吻3个群体共鉴定出9个单倍型,9个单倍型无明显的支系划分,结果与NJ进化树一致。单倍型网络图是以高频率优势单倍型H1为中心,低频单倍型以星状围绕在其周围,亦表明大鼻吻3个群体遗传结构较为简单。 图4 基于线粒体DNA COX1序列的大鼻吻单倍型网络图Fig.4 Network of R.nasutus based on concatenated DNA COX1 sequences haplotypes 图5 基于线粒体DNA ND1序列的大鼻吻单倍型网络图Fig.5 Network of R.nasutus based on concatenated DNA ND1 sequences haplotypes 基于COX1和ND1基因序列的中性检验结果显示见表7。鉴于大鼻吻3个地理群体间的遗传距离较小、遗传分化程度较弱,故分析其历史动态信息时将3个群体作为一个整体进行分析,COX1和ND1基因序列总体的中性检验结果表明Tajima′sD和Fu′sFs值均为负值,P值也均不显著,说明大鼻吻群体符合中性进化。基于COX1和ND1基因序列的3个大鼻吻群体增长均服从constant模型,核苷酸不配对分布曲线均呈现单峰形(图6和图7),表明大鼻吻近期可能经历了群体扩张过程。 表7 大鼻吻群体COX1和ND1基因的中性检验Tab.7 Neutrality test of COX1 and ND1 genes in R.nasutus populations 表7 大鼻吻群体COX1和ND1基因的中性检验Tab.7 Neutrality test of COX1 and ND1 genes in R.nasutus populations 基因群体Tajima'sDTajima'sD P-valueFu'sFsFu'sFsP-valueCOX1YN2.015 360.979 001.062 590.720 00PL1.077 760.888 001.837 810.833 00DK-1.291 500.063 00-5.350 450.214 00总体0.600 540.643 33-5.816 680.524 00ND1YN-1.366 720.068 00-2.306 060.015 00PL-1.128 130.103 00-1.494 320.171 00DK-1.517 120.030 00-4.590 640.002 00总体-1.337 320.067 00-2.796 950.062 67 图6 基于COX1 基因序列的大鼻吻群体错位分布图Fig.6 Dislocation distribution map of R.nasutus major population based on COX1 gene sequencesA表示YN群体;B表示PL群体;C表示DK群体;D表示总体 图7 基于ND1基因序列的大鼻吻群体错位分布图Fig.7 Dislocation distribution map of R.nasutus major population based on ND1 gene sequencesA表示YN群体;B表示PL群体;C表示DK群体;D表示总体 遗传多样性是物种进化和适应性的基础,遗传多样性水平可有效反映物种对环境适应能力的大小,遗传多样性水平越低表明该物种面临资源衰退甚至濒临灭绝,反之则意味着该物种对环境的适应能力越强[16-18]。单倍型多样性(Hd)和核苷酸多样性(π)作为衡量一个物种群体多样性的两个重要指标,通常被用来评价群体遗传多样性水平的高低[19,20]。由于核苷酸多样性(π)考虑了各种线粒体DNA单倍型在群体中所占的比例,因此更能准确反映群体的遗传多样性,π值越高则说明群体的遗传多样性较高[21]。根据GRANT等[22]制定的以单倍型多样性Hd=0.5和核苷酸多样性π=0.005为界的分类标准,本研究中,基于COX1基因的三个群体均呈现高单倍型多样度(Hd>0.5)低核苷酸多样度(π<0.005)的状态,其原因可能是一个较小的种群在快速进化过程中,种群数量增加的同时单倍型多样度也在提高,但无足够时间使得核苷酸发生变异[23,24]。基于ND1基因的3个群体均呈现低单倍型多样度(Hd>0.5)低核苷酸多样度(π<0.005)的状态,表明此小群体并未出现快速进化的过程,亦未发生明显的核苷酸变异。造成两种基因差异的原因可能是,由于基因序列差异,导致遗传多样性的大小存在一定的差异,这可能与线粒体DNA不同序列的基因长度不同以及保守性差别有关[25]。对比同属黄河上游分布的极边扁咽齿鱼[26](Platypharodonextremus)(Hd为0.993~1.000,π为0.0107~0.0158)和花斑裸鲤[27](Gymncypriseckloni)(Hd为0.75~0.89,π为0.06~0.11),大鼻吻遗传多样性表现出较低的水平。推测可能是黄河干流水利水电设施建设,洄游通道被阻断,加之历史上过度捕捞、水域污染以及周围环境的破碎化等因素,导致大鼻吻种质资源数量大幅度下降[28],出现瓶颈效应,从而使得群体遗传多样性水平下降。因此,为了有效恢复其种群大小及遗传多样性水平,必须加强对现有野生资源的科学管理和保护。 群体间的遗传距离(D)和遗传分化指数(Fst)通常用来衡量两个群体间的遗传分化程度,其值越大,表明群体分化程度越高[33]。根据NEI[30]制定的以遗传距离0.05、0.02为界的分类标准,本研究中,基于3个不同地理群体的线粒体COX1和ND1基因序列分析,群体间的遗传距离均在0~0.5之间,符合同一物种不同群体间遗传距离变动规律,且3个群体之间的遗传距离均处于同一水平,说明各群体之间在基因交流上没有受到影响。根据BALLOUX等[31]制定的以遗传分化指数0.05、0.15和0.25为界的分类标准,本研究基于3个地理群体的线粒体COX1基因序列分析,YN和DK两群体之间遗传分化指数(0.076 55)处于中等遗传分化水平,其余群体间的遗传分化指数均在0~0.05之间,处于低等分化水平。而ND1基因序列中群体间的遗传分化指数均在0~0.05之间,表明群体遗传分化水平较低。通过线粒体不同基因序列所得出3个群体的遗传距离和遗传分化指数结果不同,这可能与COX1和ND1基因的进化速率的大小有关。同时,大鼻吻群体间及群体内的AMOVA分析结果也表明,大鼻吻的遗传变异主要来自群体内,只有少量来自群体间。基于COX1基因和ND1序列构建的NJ树中,3个群体的单倍型相互混杂,未发生明显谱系分化,这些结果均表明3个群体没有显著的遗传分化,遗传分化较小的特点。推测出现这种现象与环境的胁迫对群体的遗传结构会产生较大影响[32],以及繁殖习性有关:本研究中3个群体分布于黄河上游河段,河流处于贯通状态,其气候、温度和盐度等环境因素都大致相同,严格意义上3个群体并未形成地理隔离;而大鼻吻作为一种典型的半洄游性鱼类,在每年繁殖期,其性成熟个体溯河洄游至黄河上游的急流浅滩处产卵,受精卵孵化过程贯穿于中游至上游河段,促进了种群的扩散,使得大鼻吻整体上呈现出遗传分化弱的现象。这与亚科鱼类的蛇[33](Saurogobiodabryi)、圆筒吻[34]和长鳍吻[35]等的报道结果相似。鉴于3个群体之间及群体内部的遗传分化水平较低且遗传距离相近,因此,建议将大鼻吻作为一个整体进行就地保护。 综上所述,本研究基于COX1和ND1基因序列对中国黄河上游YN、PL和DK 3个不同地理群体的大鼻吻进行了遗传多样性分析。结果表明,大鼻吻3个群体的亲缘关系较近,无明显谱系分化,结构比较单一,未发现构成一个单独分支的群体;从COX1基因序列来看,大鼻吻3个群体遗传多样性处于较高水平。从ND1基因序列来看,大鼻吻3个群体遗传多样性处于较低水平;大鼻吻3个群体的遗传变异主要来自于群体内部;中性检验结果表明大鼻吻进化历程符合中性进化假设,且存在群体扩张可能。因此,建议将大鼻吻3个群体作为一个整体加强对大鼻吻群体的保护,设立有效的保护措施,加大宣传保护力度,主要加强就地保护,改善其栖息环境,逐步完善对大鼻吻群体的保护措施,使得其种质资源得到有效的恢复。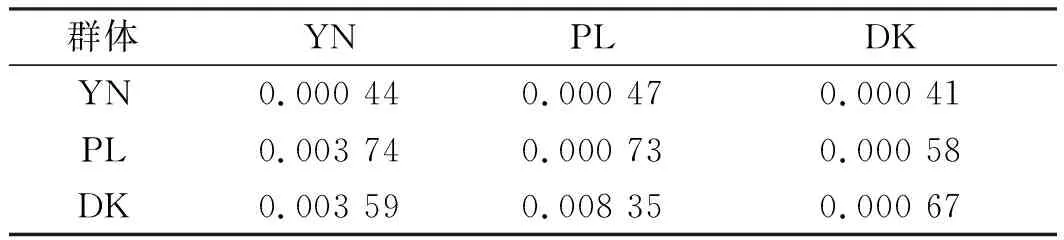
2.4 系统发育关系
2.5 群体历史动态分析
3 讨论
3.1 遗传多样性
3.2 群体遗传分化
3.3 保护建议

