苹果响应盐胁迫相关miRNA的深度测序数据集
摘要:土壤盐渍化已成为限制农作物产量与质量的重要因素之一,影响着全世界范围内的农作物产量。苹果作为一种重要的水果作物,盐胁迫的危害及耐盐砧木的缺乏已成为威胁现代苹果产业健康可持续发展的重要问题。因此,研究苹果对盐胁迫的反应和适应具有重要的现实意义和紧迫性。本研究分别以0%、0.2%和0.6% NaCl处理0、4、8和12天后的珠美海棠实生苗为试验材料,从叶片及根系中提取总RNA,构建RNA-seq文库,共鉴定获得575个miRNA,包括315个已知mdm-miRNA和260个新miRNA。此外,Differential Expression(DE) miRNA对盐胁迫反应具有组织特异性表达模式。同时提供珠美海棠miRNA深度测序的数据集,结合生物信息学方法对原始數据质量控制,获得高质量序列并利用BLAST软件将预测靶基因序列与NR、Swiss-Prot、Pfam等数据库比对,获得响应盐胁迫相关基因的注释信息。GO分析表明与盐胁迫有关miRNA及靶基因主要参与生物过程中对刺激的反应。本研究为珠美海棠的利用提供理论数据基础,并为耐盐砧木的选育提供有力支持。
关键词:苹果;盐胁迫;miRNA;深度测序;生物信息学
1 引言
盐胁迫是制约全世界农业生产的主要环境因素之一[1]。据估计目前全球约有20%的灌溉土壤正在遭受盐胁迫的危害,预计到2050年全世界将会有超过50%的可耕地土壤出现土壤盐渍化问题[2]。植物受到盐胁迫时会影响体内Na+、Cl- 等含量的积累,从而破坏细胞内的离子平衡,进而影响植物细胞膜上相关酶的活性,破坏膜结构。此外盐胁迫会造成光合作用速率下降,增加呼吸消耗,加速植物衰老,并且积累大量有毒代谢物质,对植物造成不利影响[3-4]。然而植物为适应高盐环境已进化出应对盐胁迫的策略,包括积累渗透调节物质以适应渗透损伤[5-6];激活活性氧清除系统保护植物免受盐胁迫引发的氧化损伤[7-8];调控盐响应基因的表达以保护细胞机制免受胁迫[9]等方式来提高植物耐盐性。因此,揭示更多耐盐植物分子机制对未来农业生产具有重要意义。
MicroRNA (miRNA)是一类广泛存在于动物、植物中的非编码小RNA(sncRNA),长度为20-24个核苷酸(nt),被认为是一种重要的基因表达调控因子[10]。miRNA最初发现于秀丽小杆线虫[11],随着对miRNA的深入研究,越来越多的植物、动物和病毒中都发现了miRNA的存在。截至目前已从82种植物中共鉴定出10 405个成熟miRNA,归属于上百个miRNA家族[12]。miRNA与反向互补序列结合形成RNA诱导的沉默复合物,从而通过转录后水平切割或抑制翻译来负调控其靶基因[13]。此外,miRNA参与了植物种子萌发和幼苗的生长发育、植物形态结构调控和产量形成,以及对许多生物和非生物胁迫的响应[14-16]。已有大量研究证明miRNA在植物响应盐胁迫中发挥着关键作用。例如,miR169c的靶基因可以控制玉米叶片气孔的开启和关闭,从而减少水分流失抵抗盐胁迫[17]。棉花中miR414c过表达可负调控铁超氧化物歧化酶基因,增强植物对盐胁迫的耐受性[18]。此外,越来越多的耐盐miRNA-mRNA模块也得到了功能验证。miR156/SPL模块通过上调苹果的MdWRKY100来增强其耐盐性[19]。在水稻中miR528-AO (l-抗坏血酸氧化酶)模块可以调节抗坏血酸和脱落酸的代谢以及清除ROS,从而增强耐盐能力[20]。这些研究表明了解miRNA介导的植物耐盐生理调控机制,可以为揭示植物耐盐胁迫复杂的分子和遗传机制提供必要的基础。
苹果是全世界重要的栽培果树之一,亦是乡村振兴和脱贫攻坚的优势树种。然而一些苹果优势产区由于土壤盐渍化严重,造成树体营养失衡、生理病害加重、果实产量及品质下降等问题,土壤盐渍化已经成为威胁果树产业健康可持续发展的重要问题[21]。苹果耐盐性与其品种和砧木类型密切相关,选择耐盐性强的苹果砧木类型可以有效提高苹果耐盐性[22]。因此对耐盐砧木的挖掘、筛选及利用将成为现代苹果产业和“四荒”用地上新果园发展的重要发展趋势。
我国拥有着丰富的苹果种质资源。据报道,珠美海棠可耐0.6%以上盐含量,具有耐盐、耐碱、适应性强等优良特性。miRNA深度测序作为高通量测序技术的重要应用,通过运用生物信息学技术对数据分析可从测序得到原始数据中筛选出相关盐胁迫响应基因,极大提高了植物与耐盐相关基因种类鉴定的准确性。因此本研究以珠美海棠实生苗为材料,通过miRNA测序获得样品的miRNA序列数据,并通过生物信息学注释了匹配到与盐胁迫响应相关的靶基因,为珠美海棠的砧木利用提供理论数据基础,并为解决耐盐砧木的利用提供有力支持。
2 数据采集和处理方法
2.1 植物材料和盐的处理
试验材料来自中国农业科学院果树研究所国家苹果种质资源圃(辽宁兴城),挑选健康的珠美海棠种子于4℃层积60 d。将层积的种子播种在营养钵中,随后移栽在花盆中,每盆各一株。挑选150株生长一致、高度约30 cm的一年生苗,分别进行0、0.2%和0.6% 的NaCl胁迫处理,每50株为一个处理组。随后在盐胁迫第0、4、8和12 d时选取整齐一致的植株用超纯水洗净,分别采集叶片和根系液氮速冻,每个处理重复3次。取样完成后,收集的组织样本在液氮速冻后放入干冰中寄送到测序公司。
2.2 sRNA测序
每个样本中的RNA提取参照植物RNA分离试剂盒的说明进行分离(Aidlab EASYspin, Beijing, China),随后用1.0 %琼脂糖凝胶电泳检测RNA降解和污染,RNA浓度和纯度采用Nucleic Proteometer Thermo NANODrop 2000(Thermo Fisher Scientific, Wilmington, DE, USA)进行检测,使用RNA Nano 6000检测试剂盒评估RNA的完整性(Agilent Technologies, Santa Clara, CA, USA)。只有260/280比值为1.8-1.9,260/230比值为2.0-2.5、RNA完整性(RNA integrity number)大于6.0的RNA样本可以被认为是合格样品。
获得高质量RNA后,使用Ribo-ZeroTM试剂盒(Epicentre, Madison, WI, USA)去除样本中rRNA以富集原核mRNA。随后,使用片段缓冲液随机中断富集的mRNA片段,用六碱基随机引物和逆转录酶合成第一链cDNA,用DNA聚合酶I、RNase H、dNTP和buffer solution合成第二链cDNA,使用AMPure XP beads纯化cDNA片段。在PCR扩增前将纯化的双链cDNA片段末端修复、添加碱基并与NEBNext适配器连接cDNA 15 min。PCR产物用AMPure XP系统(Beckman Coulter, Beverly, MA, USA)纯化,并由Agilent 2100 Bioanalyzer系统进行文库质量评估。测序采用Illumina HiSeq高通量测序平台,经过碱基识别和分析后,从测序数据中获得FASTQ格式的原始数据。
2.3 生物信息学分析
为了进行后续分析,采用‘Golden Delicious GDDH13_V1.1作为参考基因组进行序列比对。如图1所示,Illumina测序平台的工作流程如下:首先经过碱基识别,即Base Calling,原有的图像数据文件会被转换为原始测序序列,这也就是所谓的Raw Data或者Raw Reads。
这些收集到的原始序列可能会包含有接头序列或者质量较低的序列。为了保证分析数据的准确性,需要对这些初始数据进行质量检查,然后再生成高质量的序列,即Clean Reads。随后用短序列比对工具Bowtie将Clean Reads比对到Silva数据库、GtRNAdb数据库、Rfam数据库以及Repbase数据库。此流程有助于过滤掉核糖体RNA(rRNA)、转运RNA(tRNA)、核内小RNA(snRNA)、核仁小RNA(snoRNA)等非编码RNA以及重复序列。在与参考基因组比对完成后,将比对结果与miRBase(v22)数据库内已有的miRNA成熟序列及其上下游序列进行比对。此阶段最多允许一个错配,被满足条件的reads将被确认为已知miRNA。对于没有能够匹配到已知miRNA序列的数据,使用miRDeep2软件工具来进行处理。该软件通过分析reads在基因组上的位置信息,能够推断出潜在的前体序列。然后根据reads在前体序列中的分布和前体的结构能量信息,利用贝叶斯模型进行评分,以预测新的miRNA。随后,对已知的miRNA和新预测的miRNA进行结构和碱基编辑等相关分析,将获得的靶基因与数据库中的数据进行比对和分类注释。最终基于这些注释,确定与响应盐胁迫相关的miRNA。
3 数据样本描述
3.1 sRNA深度测序数据
从珠美海棠对照组0 NaCl(0 d)和NaCl(0.2%和0.6%)胁迫条件处理4 d、8 d、12 d的叶片(L)及根系(R)部位中提取总RNA,构建了包括3个生物学重复共42个样品的RNA-seq文库,并利用Illumina Hiseq X-ten平台进行高通量测序。对所获得的原始数据进一步过滤,最终得到用于后续分析的高质量小RNA reads。通过质量控制,每个样品的Clean Data均大于9.31 M。随后将每个样本中获取的Clean Reads与‘Golden Delicious GDDH13_V1.1参考基因组进行比对,Q30和GC含量分别为99.06%-99.35%、41.02%-49.37%(表1)。
3.2 miRNA分析
miRNA的起始转录位置往往在基因间隔区、内含子与编码序列的反向互补序列上。这些miRNA的前体都体现出所具有的特殊的发夹结构,而其成熟体则是要依赖于Dicer或DCL酶的剪切才能实现。基于miRBase(v22)数据库和miRDeep2软件包,我们对42个样品进行了深入分析,共鉴定出575个miRNA。其中,已知的miRNA有315个,而新预测的miRNA则有260个。此外,miRDeep2软件还预测了新miRNA的候选前体结构及其序列深度信息,并生成相应的pdf文件(图2)。
注:图A为miRDeep2软件对miRNA前体的打分和比对到成熟序列、环状结构、star序列上的reads数及其前体的二级结构预测图:红色为成熟序列,黄色为环状结构,紫色为star序列;图B显示比对到本条前体上的reads分布;圖C包含了成熟序列、环状结构、star序列的位置,紫色为miRDeep2软件预测的star序列,亮蓝色为测序reads支持的star序列。
3.3 盐胁迫下miRNA的差异表达分析
为了在珠美海棠中发现响应盐胁迫的miRNA,分析了在0.2%和0.6% NaCl处理下叶片和根系中不同处理时间(4 d、8 d、12 d)下盐响应差异miRNA的相对表达水平。研究发现在0.2% NaCl处理4 d、8 d、12 d后的叶片中分别有10、126和15个DE miRNA特异性表达,3个DE miRNA在盐处理4 d、8 d、12 d后的叶片中共同表达。在0.6% NaCl处理4 d、8 d、12 d后的叶片中分别有29、29和30个DE miRNA特异性表达,9个DE miRNA共同表达(图3a)。此外,在0.2% NaCl处理4 d、8 d、12 d后的根中分别有38、34和27个DE miRNA特异性表达,10个DE miRNA共同表达。在0.6% NaCl处理4 d、8 d、12 d后的根中分别有55、39和9个DE miRNA特异性表达,22个DE miRNA共同表达(图3b)。总之,这些miRNA对盐胁迫反应具有组织特异性表达模式。
3.4 miRNA靶基因预测及注释
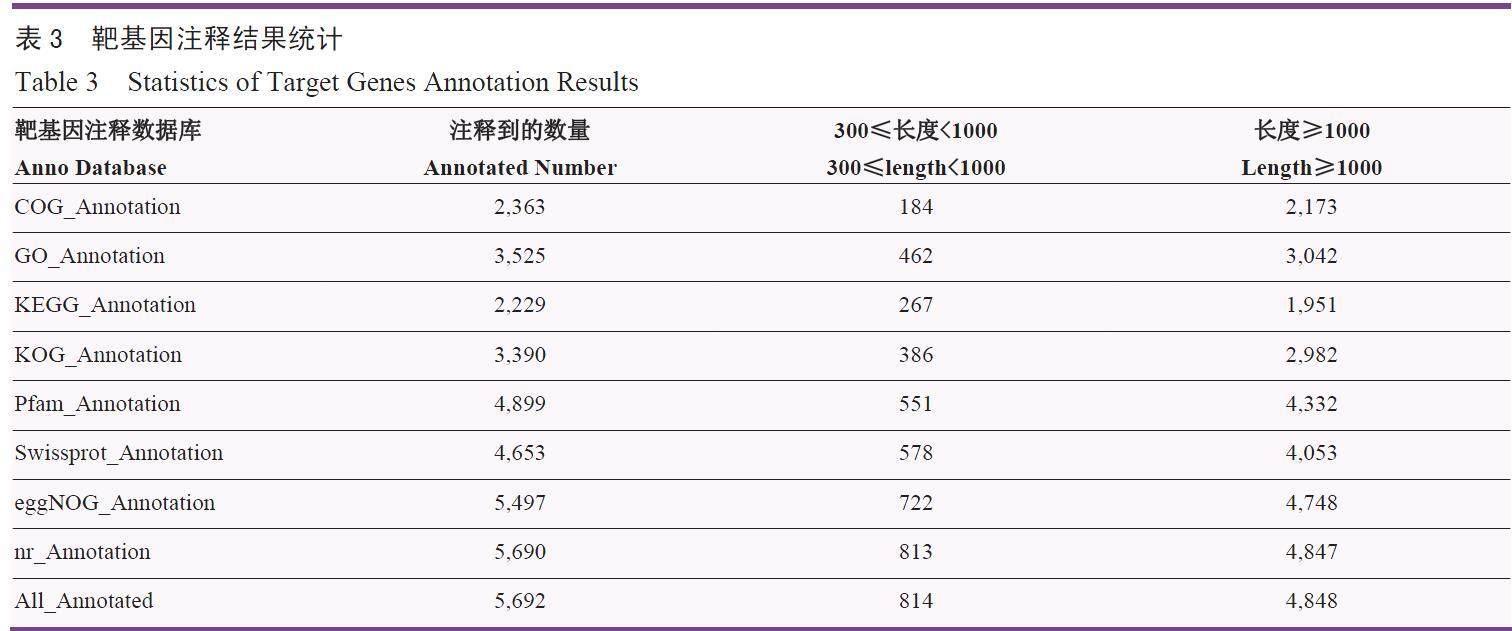
通过已知的miRNA与新预测出的miRNA,再结合对应物种的基因序列信息,运用TargetFinder软件预测可能的靶基因,预测结果如表2所示。以BLAST软件为工具,针对预测出的靶基因序列和NR、Swiss-Prot、Pfam等数据库进行比对,进一步了解这些靶基因,以此获取靶基因的注释信息。在5 812个预测出的靶基因中,共有5 692个获得了注释信息。全部miRNA靶基因注释数量和不同样品miRNA靶基因注释数量见表3。
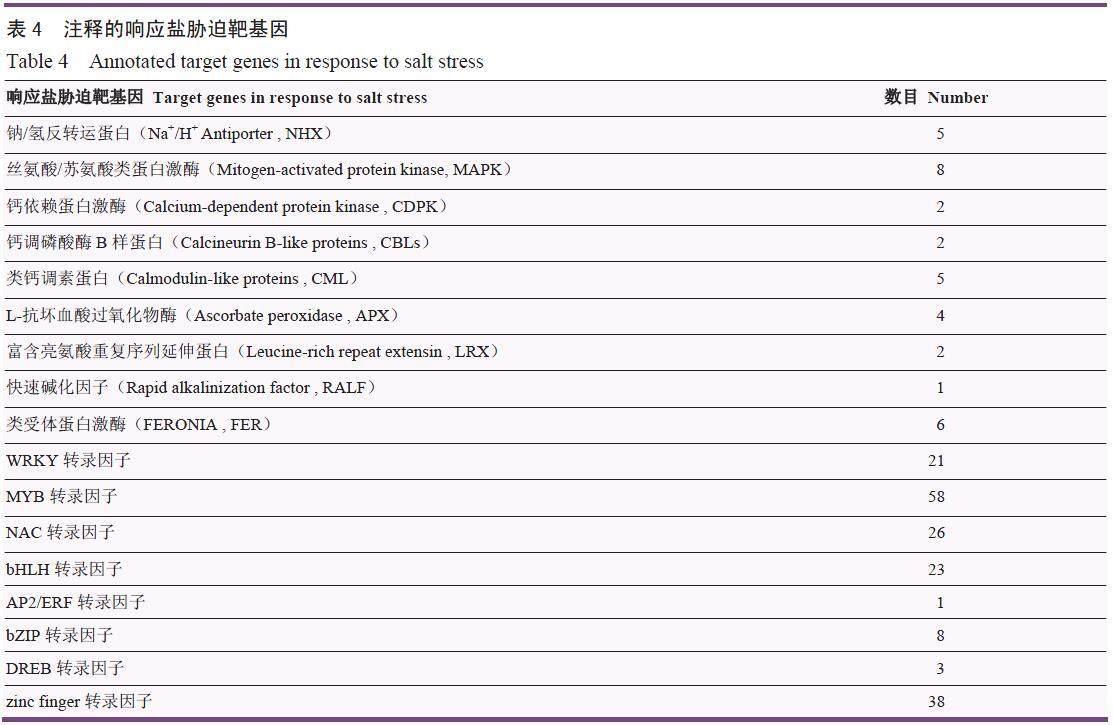
3.5 注释到的响应盐胁迫靶基因数据及GO分析
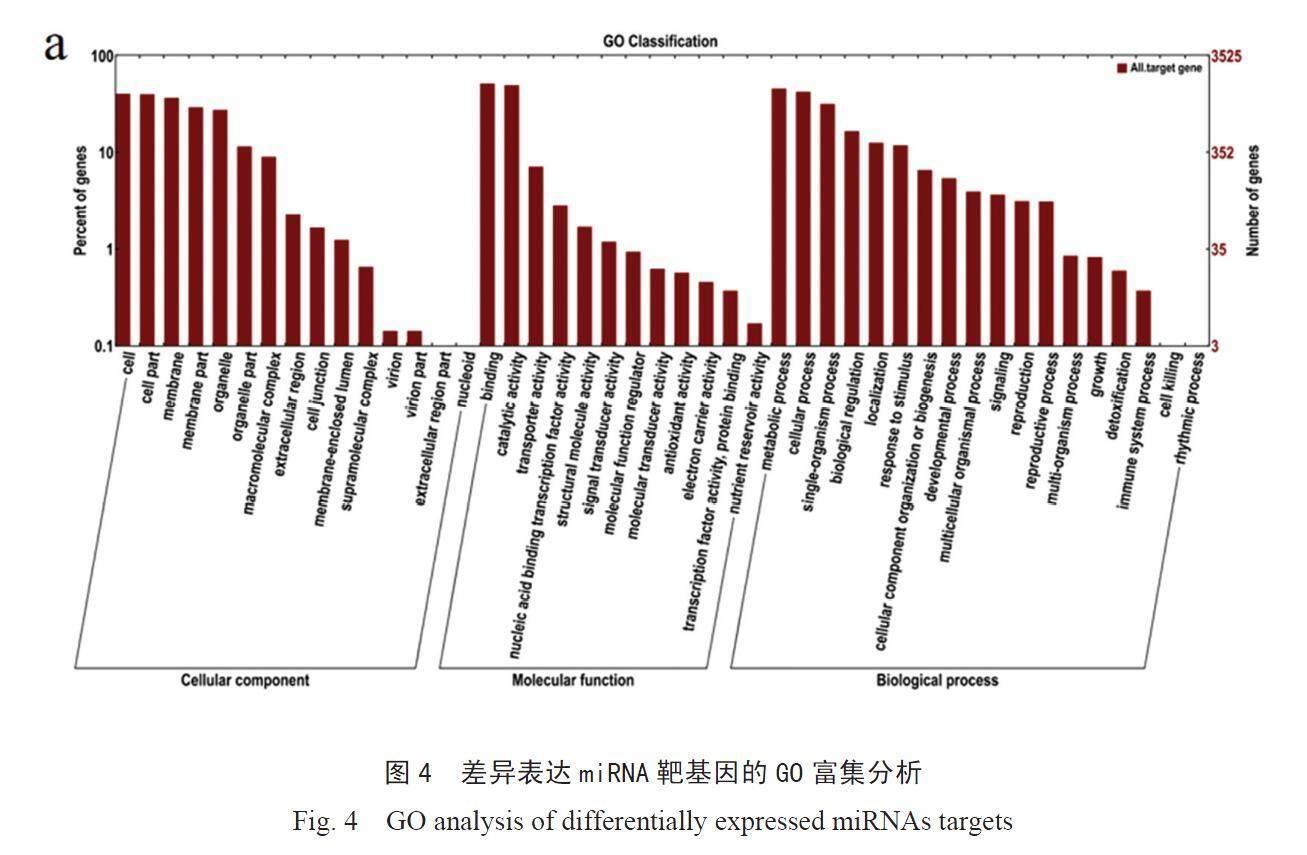
注释到响应盐胁迫相关的靶基因数据以.xlsx文件格式保存。其中,注释到盐胁迫的靶基因见表4。基于细胞成分、分子功能和生物过程对预测的靶基因进行GO富集分析。GO分析显示,差异表达的靶基因被注释为44个GO术语。在细胞成分类别中,主要富集的术语是“Cell”(GO:0005623)、“Cell part”(GO:0044464)、“membrane”(GO:0016020)和“organelle”(GO:0043226)。就其分子功能而言,主要富集的术语是“binding”(GO:00055488)、“catalytic activity”(GO:0003824)、“transporter activity”(GO:0005215)和“nucleic acid binding transcription factor activity”(GO:0003676)。涉及生物过程的主要术语是“metabolic process”(GO:0008152)、“cellular process”(GO:0009987)、“single-organism process”(GO:0044699)、“biological regulation”(GO:0065007)、“developmental process”(GO:0032502)和“response to stimulus”(GO:0050896)(图4)。
4 数据质量控制与评估
为了保证数据的质量和可靠性,试验样品寄送到北京百迈客生物科技有限公司后,由专业人员进行总RNA的提取以及miRNA的深度测序,并使用百迈客云平台进行生物信息学分析。同时确保所提取的RNA满足构建miRNA文库的要求。miRNA测序后的原始序列首先通过内部perl脚本进行处理,隨后通过去除接头;去除短于18或长于30个核苷酸的序列;去除对于每个样本中质量值低的序列;去除未知碱基N含量大于等于10%的Reads以获得Clean Reads。共得到648.15 M Clean Reads,各样品不少于9.31 M Clean Reads。同时计算清理数据的Q30、GC含量和序列重复水平。以上数据的整理过程均由专业人员进行核查。
将rRNA、tRNA、snRNA、snoRNA等ncRNA以及重复序列过滤后,选用非冗余蛋白序列数据库(NR)、同源蛋白家族数据库(Pfam)、人工注释的非冗余蛋白序列数据库(Swiss-Prot)等数据库对靶基因进行注释,并根据不同数据库的注释最终确定与响应盐胁迫相关的靶基因。响应盐胁迫靶基因筛选按照如下标准筛选:当只在其中一种数据库中注释到与耐盐相关的基因,则保留该注释的基因;当在两种或以上数据库中均注释到响应盐胁迫相关的基因,则保留该注释的基因。
5 数据价值与使用建议
该数据集通过miRNA深度测序获得样品的序列数据,并利用生物信息学技术进行联合分析,注释到珠美海棠与响应盐胁迫相关的靶基因,可为珠美海棠的砧木利用提供理论数据基础。同时为解决耐盐砧木的利用提供科学依据和数据支撑。
6 数据可用性
中国科技资源标识码(CSTR): 17058.11.sciencedb. agriculture.00097;
数据对象标识码(DOI): 10.57760/sciencedb. agriculture.00097。
数据服务系统网址:https://www.scidb.cn/s/iqmQfi。
作者分工与贡献
刘昭,数据整理汇总和论文撰写。
高源,数据质量控制与方法设计。
王昆,数据质量控制与方法设计。
孙思邈,数据收集与可视化。
代瑛姿,前期数据处理和分析。
路翔,样品收集及质量控制。
田雯,数据汇总处理。
王大江,论文构架及质量控制。
冯建荣,论文构架及质量控制。
伦理声明
本研究未涉及伦理。
利益冲突声明
作者声明,全部作者均无会影响研究公正性的财务利益冲突或个人利益冲突。
参考文献
[1] Zhu J K. Abiotic stress signaling and responses in plants[J]. Cell, 2016, 167: 313-324. https://doi.org/10.1016/j.cell.2016.08.029.
[2] Syed A, Sarwar G, Shah S H, et al. Soil salinity research in 21st century in Pakistan: its impact on availability of plant nutrients, growth and yield of crops[J]. Communications in Soil Science and Plant Analysis, 2021, 52 (3): 183-200. https://doi.org/10.1080/ 00103624.2020.1854294.
[3] Evans M J, Choi W G, Gilroy S, et al.A ROS-assisted calcium wave dependent on the AtR BODH NADPH oxidase and TPC1 cation channel propagates the systemic response to salt stress[J]. Plant Physiology, 2016, 171(3): 1771-1784. https://doi.org/10.1104/pp.16. 00215.
[4] Li J J, Ma J J, Guo H L, et al. Growth and physiological responses of two phenotypically distinct accessions of centipedegrass (Eremochloa ophiuroides (Munro) Hack.) to salt stress[J]. Plant Physiology and Biochemistry, 2018, 126: 1-10. https://doi.org/10.1016/j.plaphy.2018. 02.018.
[5] Niazian M, Sadat-Noori S A, Tohidfar M, et al. Agrobacterium- mediated genetic transformation of ajowan (Trachyspermum ammi (L.) Sprague): an important industrial medicinal plant[J]. Industrial Crops and Products, 2019, 132: 29-40. https://doi.org/10.1016/j.indcrop.2019. 02.005.
[6] Li H Y, Tang X Q, Yang X Y, et al. Comprehensive transcriptome and metabolome profiling reveal metabolic mechanisms of Nitraria sibirica Pall. to salt stress[J]. Scientific Reports, 2021, 11(1): 12878. https://doi.org/10.1038/s41598-021-92317-6.
[7] Aghaei K, Ehsanpour A A, Komatsu S. Potato responds to salt stress by increased activity of antioxidant enzymes[J]. Journal of Integrative Plant Biology, 2009, 51: 1095-1103. https://doi.org/10.1111/j.1744- 7909.2009.00886.x.
[8] Gill S S, Tuteja N. Reactive oxygen species and antioxidant machinery in abiotic stress tolerance in crop plants[J]. Plant Physiology and Biochemistry, 2010, 48(12): 909-930. https://doi.org/ 10.1016/j.plaphy.2010.08.016.
[9] Tang J, Liu Q Q, Yuan H Y, et al. Molecular analysis of a novel alkaline metal salt (NaCl)-responsive WRKY transcription factor gene IlWRKY1 from the halophyte Iris lactea var. Chinensis[J].International Biodeterioration and Biodegradation, 2018, 127: 139-145. https://doi. org/10.1016/j.ibiod.2017.11.021.
[10] Sun G L. MicroRNAs and their diverse functions in plants[J]. Plant Molecular Biology, 2012, 80: 17–36. https://doi.org/10.1007/s11103- 011-9817-6.
[11] Lee R C, Feinbaum R L, Ambros V. The C-Elegans heterochronic gene lin-4 encodes small RNAs with antisense complementarity to lin-14[J]. Cell, 1993, 75: 843-854. https://doi.org/10.1016/0092-8674 (93)90529-Y.
[12] Parmar S, Gharat S A, Tagirasa R, et al. Identification and expression analysis of miRNAs and elucidation of their role in salt tolerance in rice varieties susceptible and tolerant to salinity[J]. PLoS One, 2020, 15: e0230958. https://doi.org/10.1371/journal.pone.0230958.
[13] Tyagi S, Sharma S, Ganie S A, et al. Plant microRNAs: biogenesis, gene silencing, web-based analysis tools and their use as molecular markers[J]. 3 Biotech, 2019, 9: 413. https://doi.org/10.1007/s13205- 019-1942-y.
[14] Chandran V, Wang H, Gao F, et al. MiR396-OsGRFs module balances growth and rice blast disease-resistance[J]. Frontiers in Plant Science, 2019, 9:1999. https://doi.org/10.3389/fpls.2018.01999.
[15] Meng Y, Mao J P, Tahir M M, et al. Mdm-miR160 participates in auxin-induced adventitious root formation of apple rootstock[J]. Scientia Horticulturae, 2020, 270: 109442. https://doi.org/10.1016/j. scienta.2020.109442.
[16] Wang Y M, Liu W W, Wang X W, et al. MiR156 regulates anthocyanin biosynthesis through SPL targets and other microRNAs in poplar[J]. Horticulture Research, 2020, 7: 118. https://doi.org/10. 1038/s41438-020-00341-w.
[17] Luan M D, Xu M Y, Lu Y M, et al. Expression of zma-miR169 miRNAs and their target ZmNF-YA genes in response to abiotic stress in maize leaves[J]. Gene, 2015, 55: 178-85. https://doi.org/10.1016/j. gene.2014.11.001.
[18] Wang W, Liu D, Chen D D, et al. MicroRNA414c affects salt tolerance of cotton by regulating reactive oxygen species metabolism under salinity stress[J]. RNA Biology, 2019,16: 362-375. https:// doi.org/10.1080/15476286.2019.1574163.
[19] Ma Y, Xue H, Zhang F, et al. The miR156/SPL module regulates apple salt stress tolerance by activating MdWRKY100 expression[J]. Plant Biotechnology Journal, 2021, 19: 311-323. https://doi.org/10.1111/ pbi.13464.
[20] Wang M, Guo W, Li J, et al. The miR528-AO module confers enhanced salt tolerance in rice by modulating the ascorbic acid and abscisic acid metabolism and ROS scavenging[J]. Journal of Agricultural and Food Chemistry, 2021, 69(31): 8634-8648. https:// doi.org/10.1021/acs.jafc.1c01096.
[21] 王繼勋,梅闯,闫鹏,等.新疆苹果产业现状、存在问题及对策建议[J].农村科技,2022(3):65-70.
[22] 张志晓,曾丽蓉,赵嘉菱,等.五种苹果砧木的生长及生理特性对盐胁迫的响应[J].北方园艺,2017(3):19-25.
引用格式:刘昭,高源,王昆,孙思邈,代瑛姿,路翔,田雯,王大江,冯建荣.苹果响应盐胁迫相关miRNA的深度测序数据集[J].农业大数据学报,2024,6(1): 14-23. DOI: 10.19788/j.issn.2096-6369.100007.
CITATION: LIU Zhao, GAO Yuan, WANG Kun, SUN SiMiao, DAI YingZi, LU Xiang, TIAN Wen, WANG DaJiang, FENG JianRong. Deep Sequencing Dataset of miRNAs in Response to Salt Stress in Apples[J]. Journal of Agricultural Big Data, 2024,6(1):14-23. DOI: 10.19788/j.issn.2096-6369.100007.
Deep Sequencing Dataset of miRNAs in Response to Salt Stress in Apples
LIU Zhao1,2, GAO Yuan2, WANG Kun2, SUN SiMiao2, DAI YingZi1, LU Xiang1,2, TIAN Wen1,2, WANG DaJiang2,*, FENG JianRong1,*
1 College of Agriculture, Shihezi University, Xinjiang Production and Construction Corps Key Laboratory of Special Fruits and Vegetables Cultivation Physiology and Germplasm Resources Utilization, Shihezi 832000, Xinjiang , China; 2 Research Institute of Pomology , Chinese Academy of Agricultural Sciences/Key Laboratory of Horticultural Crops Germplasm Resources Utilization, Ministry of Agriculture and Rural Affairs, Xingcheng 125100, Liaoning, China
Abstract: Soil salinization has become an important factor limiting crop yield and quality, affecting crop yields worldwide. As an important fruit crop, the harm of salt stress and the lack of salt tolerant rootstocks have become important issues threatening the healthy and sustainable development of the modern apple industry. Therefore, studying the response and adaptation of apple trees to salt stress is of great practical significance and urgency. In this study, total RNA was extracted from the leaves and roots of Malus zumi (Mats.) Rehd. seedlings were treated with 0, 0.2% and 0.6% NaCl after 0, 4, 8 and 12 days, and RNA-seq libraries were constructed. A total of 575 miRNAs were identified, including 315 known mdm-miRNAs and 260 new miRNAs. In addition, differentially expressed (DE) miRNAs exhibited tissue-specific expression patterns in response to salt stress. At the same time, a data set for deep sequencing of miRNA in Malus zumi (Mats.) Rehd was provided, and bioinformatics methods were used to control the quality of the original data. High quality sequences were obtained, and BLAST software was used to compare the predicted target gene sequences with NR, Swiss Prot, Pfam and other databases to obtain annotation information of salt tolerance related genes. GO analysis shows that miRNAs and target genes related to salt stress are mainly involved in the response to stimuli in biological processes. This will provide theoretical data basis for the utilization of Malus zumi (Mats.) Rehd and favorable support for the breeding of salt tolerant rootstocks.
Keywords: apple; salt stress; miRNA; deep sequencing; bioinformatics

