9种狼尾草属植物叶绿体基因组特征分析*
胥富强,王海洋,师春娟,辛培尧,段利武,王齐
(1.甘肃省小陇山林业调查规划院,甘肃 天水 741020;2.云南林业职业技术学院,云南 昆明 650000;3.西南林业大学 林学院,云南 昆明 650224)
狼尾草属(Pennisetum)为禾本科(Poaceae)1 a生或多年生的草本植物,约有140个种,是热带地区的饲料作物和主要粮食作物[1-2]。该属植物的中心分布区域为非洲,在热带和亚热带地区居多,少数种可达温寒地带。我国境内被记载的狼尾草属植物主要分布在河北、江西、海南、重庆、四川、云南、甘肃、青海等地[1]。1991年,同文轩[3]发现了宝鸡狼尾草(P.baojienseTong,sp.nov.),为狼尾草的新种;2004年,吴玉虎[4]发现了青海白草(P.centrasiaticumTzve1.var.Qinghaiensis Wu.),为青海狼尾草的一个新变种。至今我国有狼尾草属植物共计12个种,3个变种,其中包括引进的4个种[5]。
狼尾草作为我国的重要牧草资源,狼尾草属植物在分子生物学方面的研究较多[6-7]。叶健军等[8]用引物组合法对133份狼尾草属植物样本进行基因分型,将所得条带的聚类分析结果分为五大、五小规模集群,研究发现就算排除了不良种质管理做法、高层次的遗传一致性、非最佳引物等因素,DNA扩增片段长度多态性技术的数据也不能清晰划分狼尾草属植物样本的界限。刘伟民等[9]和姚运法等[10]通过基于RAPD及SRAP标记的UPGMA聚类分析可以把彼此间亲缘关系较近的‘矮象草’‘桂牧1号’‘象草’‘王草’‘N51’等区分开来,证明了在种间鉴别及遗传关系分析方面RAPD及SRAP分子标记具有很好的应用价值。目前对狼尾草属植物分类,基因组研究方面非常有限,国内狼尾草属植物的种质资源多而杂,其中含有各个地域野生种、引种栽培的变种以及培育的新种,再加上各地的互相引种,导致了名称和分类归属的混乱,而且有多种狼尾草属植物都有相同或相近的别称,如中型狼尾草(P.longissimusS.L.Chen et Y.X.Jin var.intermediumS.L.Chen et Y.X.Jin)与长序狼尾草(P.longissimusS.L.Chen et Y.X.Jin)均有别称“白草”。还有拉丁名为“P.alopecuroides(L.)Spreng.”,有与该属名相同的中文命名“狼尾草”[11]。虽然现今已有分子标记技术、形态学、细胞学等方面的研究,但是在分类归属上问题仍然比较多。
叶绿体是植物光合作用和固碳的主要场所。高等植物叶绿体基因组是一个双链环状DNA分子,大小在72~217 kb之间,包含约130个基因。叶绿体基因组具有典型的四分体结构,在大多数植物中包括一个大的单拷贝区(LSC)、一个小的单拷贝区(SSC)和一对反向重复序列(IR)[12-14]。与核基因组相比,叶绿体基因组的独特之处在于其母系遗传、体积小、结构简单和序列保守[15-16]。因此,叶绿体基因组序列被广泛用于系统发育和适应性进化研究。
本研究以9种狼尾草属植物共10个样本为研究对象,利用浅层测序技术获得叶绿体全基因组序列,对其进行组装和注释,通过比较基因组学进行研究分析,阐明狼尾草属植物叶绿体基因组的结构和组成特点,丰富狼尾草属叶绿体基因组数据库,为后续研究该属物种提供更多序列信息,同时也为狼尾草属植物种质资源的评价、开发、利用以及重要经济性状的遗传改良提供理论依据。
1 材料与方法
1.1 材料
分析材料来自云南林业职业技术学院引种栽培的9种狼尾草属植物共10个样本(表1),所有样本皆经云南林业职业技术学院组织的专家鉴定。

表1 实验材料信息及标本号
1.2 方法
1.2.1 DNA的提取与测序
将狼尾草属植物10个样本新鲜叶片送至中国科学院昆明植物研究所进行DNA提取及测序。新鲜植物叶片采用改良的CTAB法[17]提取总DNA,在Ilumina二代基因组分析平台上对总DNA进行双向末端测序,每个样品添加标签混合建库,文段的片段大小为500 bp,双向150 bp或250 bp测序,每个样本确保获取不低于5G的测序数据。
1.2.2 叶绿体基因组的组装和注释
使用GetOrganelle软件[18]组装叶绿体基因组,对于很难用软件组装的有杂质或者低质量的序列,采用人工组装。具体方法为:以狼尾草属中的象草(P.purpureus,GenBank:MF594682)基因组序列为参考序列,结合使用BioEdit v7.2.5和Geneious R8.1.3软件,把Contig组装成完整的叶绿体基因组。获取Contig有两种方式:(1)通过GetOrganelle程序运行后输出的“scaffolds.fasta”文件中即含Contig;(2)运行NGSQC Tool kit v.2.3.3程序,过滤测序获取到的DNA序列,即得到高质量的片段(read),用CLCGenomics Workbench v6.5软件来组装read得到长度更长的片段(contig)[19]。最后对狼尾草属叶绿体基因组进行注释,注释程序选择在线程序GeSeq(https://chlorobox.mpimp-golm.mpg.de/geseq.html)[20],绘制狼尾草属叶绿体基因组物理图选择在线程序OGDRAW (https://chlorobox.mpimp-golm.mpg.de/OGDraw.html)。
1.2.3 狼尾草属植物叶绿体基因组特征分析及突变检测
对狼尾草属10个叶绿体基因组、大单拷贝区(LSC区)、小单拷贝区(SSC区)和反向重复去(IR区)的大小及GC含量进行统计,然后对10个狼尾草属植物的叶绿体基因组的基因数量和种类进行统计,其中包括编码蛋白质、tRNA和rRNA。
MISA是一个用perl语言写的从fasta序列中鉴定简单重复序列(Simple sequence repeats,SSRs)的程序[21],利用MISA软件检测10个狼尾草属植物叶绿体基因组简单序列重复数量。搜索的标准是:重复单元1~5 bp,单核苷酸重复序列最小重复数为10,二核苷酸重复序列的最小重复数为5,三、四、五核苷酸重复序列的最小重复数均为4,六核苷酸重复序列的最小重复数均为3,两个重复序列间隔最小值为100 bp。然后用REPuter在线程序(https://bibiserv.cebitec.uni-bielefeld.de/reputer)检测叶绿体基因组非简单序列重复中的reverse repeats(反向重复)、forward repeats(正向重复)、complement repeats(互补重复)和palindromic repeats(回文重复),按照如下参数设置:(1)sequence identity:90%;(2)minimum repeat size:30 bp;(3)Hamming distance:3。然后用DNAMAN在线程序(https://tandem.bu.edu/trf/home)检测串联重复(tandem repeats),按照如下参数设置:Mismatch:7;Indel:7;maximum period size:500;Match:2;minimum alignment score:80。
1.2.4 序列差异分析
对狼尾草属10个叶绿体基区组的4个区段进行比较,通过对比IR区边界区域的基因分布分析不同狼尾草属叶绿体基因组结构差异。采用DnaSP v6软件对狼尾草属10个叶绿体基因组进行滑动窗口分析,评估叶绿体基因组之间的核苷酸变异性(Pi)[22],步长设置为200 bp,窗口长度设置为600 bp。
1.3 系统发育关系重建
利用本研究10条狼尾草属植物叶绿体基因组序列,加上从NCBI数据库下载获得的21条狼尾草属植物完整叶绿体基因组序列,将狗尾草属的2个物种和钝叶草属(StenotaphrumTrin.)的1个物种做为外类群,共计34条叶绿体基因组序列进行系统发育关系重建。首先将叶绿体基因组序列用在线MAFFT Version 7 软件[23]进行比对,然后进行手工调整和矫正,得到可靠的矩阵用于系统发育分析。分别使用最大似然法(Maximum likelihood,ML)和贝叶斯推理法(Bayesian inference,BI)来构建系统发育树。构建BI树先使用jModeTest2.1.10选择的最合适的DNA替换模型进行系统发育重建[24]。采用MrBayes 3.1.2软件构建BI树[25]。马尔科夫链蒙特卡洛迭代运算1×108代,每1 000代抽样一次,当Average sandard deviation of split frequencies (P)小于0.01时,用于构建系统发育树。使用IQ-TREE 1.6.7进行ML分析[26],使用UFBoot2[27]和Colla-psing近零分支选项进行1 000个引导重复,ML支持率(bootstrap support values)大于70%时认为支持率较好。用Figtree v1 1.4.3软件进行结果对比,查看和美化。
2 结果与分析
2.1 狼尾草属植物叶绿体基因组基本信息
10个狼尾草属植物叶绿体基因组均为典型的四分体结构(图1),其大小在137 929~138 554 bp之间,最大差距为625 bp。叶绿体基因组最小的是非洲狼尾草,大小为137 929 bp,最大的是羽绒狼尾草,大小为138 554 bp。LSC区的大小为80 850~81 421 bp,最大差距为571 bp,最小的是东非狼尾草,长度为80 850 bp,最大的是羽绒狼尾草,长度为81 421 bp;IR区的大小为22 288~22 382 bp,最大差距为94 bp,最小的是长序狼尾草,长度为22 288 bp,最大的是羽绒狼尾草,长度为22 382 bp;SSC区的大小为12 189~12 437 bp,最大差距为248 bp,最小的是非洲狼尾草,长度为12 189 bp,最大的是绒毛狼尾草,长度为12 437 bp。GC含量范围为38.6%~38.7%,其中LSC区GC含量范围为36.4%~36.6%,IR区GC含量范围为44%~44.1%,SSC区GC含量范围为33%~33.2%(表2)。
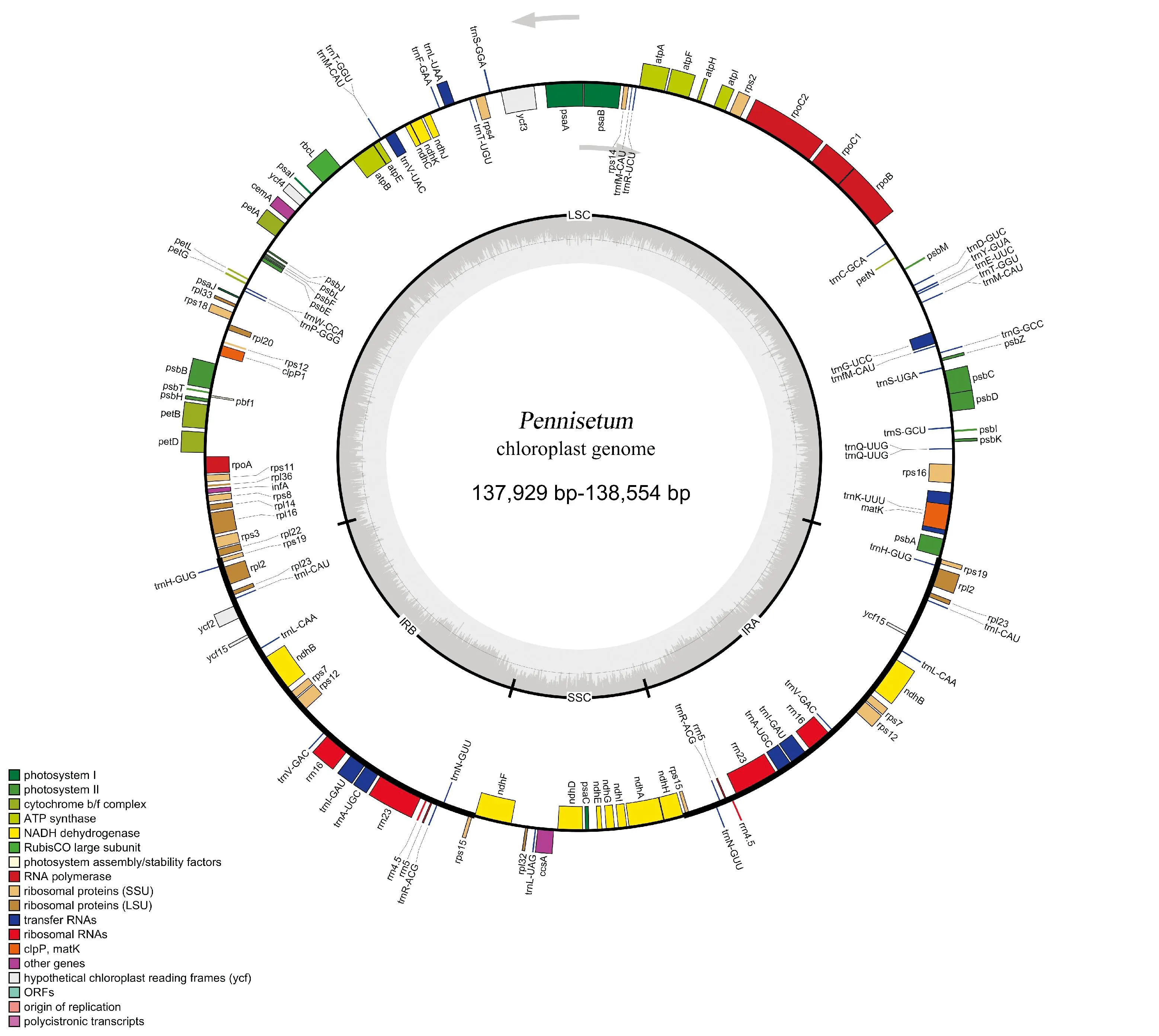
图1 狼尾草属植物叶绿体基因组物理图谱
共注释了113个基因,包括31个tRNA 基因,78个编码蛋白基因和4个rRNA 基因。17个基因在IR区重复,为互相拷贝,包括6个蛋白编码基因、4个rRNA 基因和7个tRNA基因。根据功能对基因进行分类,和光合作用有关的基因有43个(photosystemI、photosystemI、cytochrome b/f complex、ATP synthase、NADH dehydrogenase、Rubis CO large subunit),和转录、翻译相关的基因有25个(Ribosomal proteins、DNA dependent RNA polymerase),RNA基因35个(Ribosomal RNA genes、Transfer RNA genes),其他的一些基因和未知功能的基因(Conserved reading frames)一共10个(Maturase、Envelop membrane protein、ATP dependent protease)(表3)。

表3 10个狼尾草属植物样本叶绿体基因组基因分布
2.2 10个狼尾草属植物样本叶绿体基因组的突变类型
2.2.1 叶绿体基因组的IR区可视化
分析发现,10个狼尾草属植物叶绿体基因组的结构、基因顺序无明显差异,在IR/SC交界区表现出微小差异。叶绿体基因组IR 区大小为22 288 bp~22 382 bp,IRb与LSC区边界基因为rps19,与SSC区边界基因为ndhF;IRa与LSC区边界基因为rps19,与SSC区边界基因为ndhH。东非狼尾草 的rps19基因距IRb/LSC边界42 bp,其余物种的rps19基因距离IRb/LSC边界41 bp。所有物种的ndhF基因跨越IRb区的长度为29 bp,长序狼尾草 的ndhF基因跨越SSC区的长度为2 194 bp,其余物种的ndhF基因跨越SSC区的长度为2 188 bp。10个样本IRa与SSC区边界的ndhH基因长度都为1 181 bp。东非狼尾草的rps19基因距IRa与LSC区边界43 bp,其余样本的rps19基因距IRa与LSC区边界42 bp(图2)。

图2 10个狼尾草属植物叶绿体基因组LSC,SSC及IR边界比较
2.2.2 叶绿体基因组的重复序列检测
检测了10个狼尾草属植物叶绿体基因组的简单重复序列数量。共检测到389个SSRs位点。检测到的位点包括单核苷酸重复,双核苷酸重复及复合型核苷酸重复共5种,其中单核苷酸重复数量最多,为314个,其次是双核苷酸重复50个,三核苷酸重复11个,四、六核苷酸重复均为7个,未检测出五核苷酸重复(图3)。
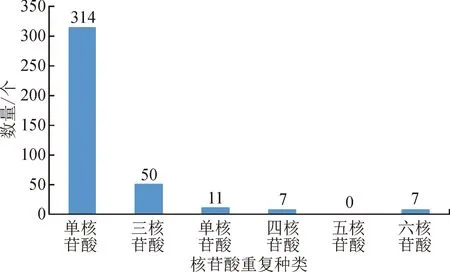
图3 10个狼尾草属植物叶绿体基因组中检测到的SSR数量及类型
分别检测10个狼尾草属植物叶绿体基因组的forward repeats(正向重复)、reverse repeats(反向重复)、palindromic repeats(回文重复)、complement repeats(互补重复)和tandem repeats(串联重复)的数量(图4)。结果显示正向重复的数量为268个,反向重复的数量为15个,回文重复的数量为182个,串联重复的数量为328个,未检测到互补重复。

图4 10个狼尾草属植物叶绿体基因组非简单序列重复类型和数量
2.2.3 mVISTA序列差异分析
以长序狼尾草叶绿体基因组序列作为参考的序列同源性比较表明,供试的狼尾草属植物叶绿体基因组结构基本一致,种间序列整体较为保守,尤其是CDS基因区。在整体变异上,基因非编码的基因间区和部分内含子区变异率要高于CDS基因区,LSC区和SSC区的变异率高于rRNA基因所在的IR区(图5)。
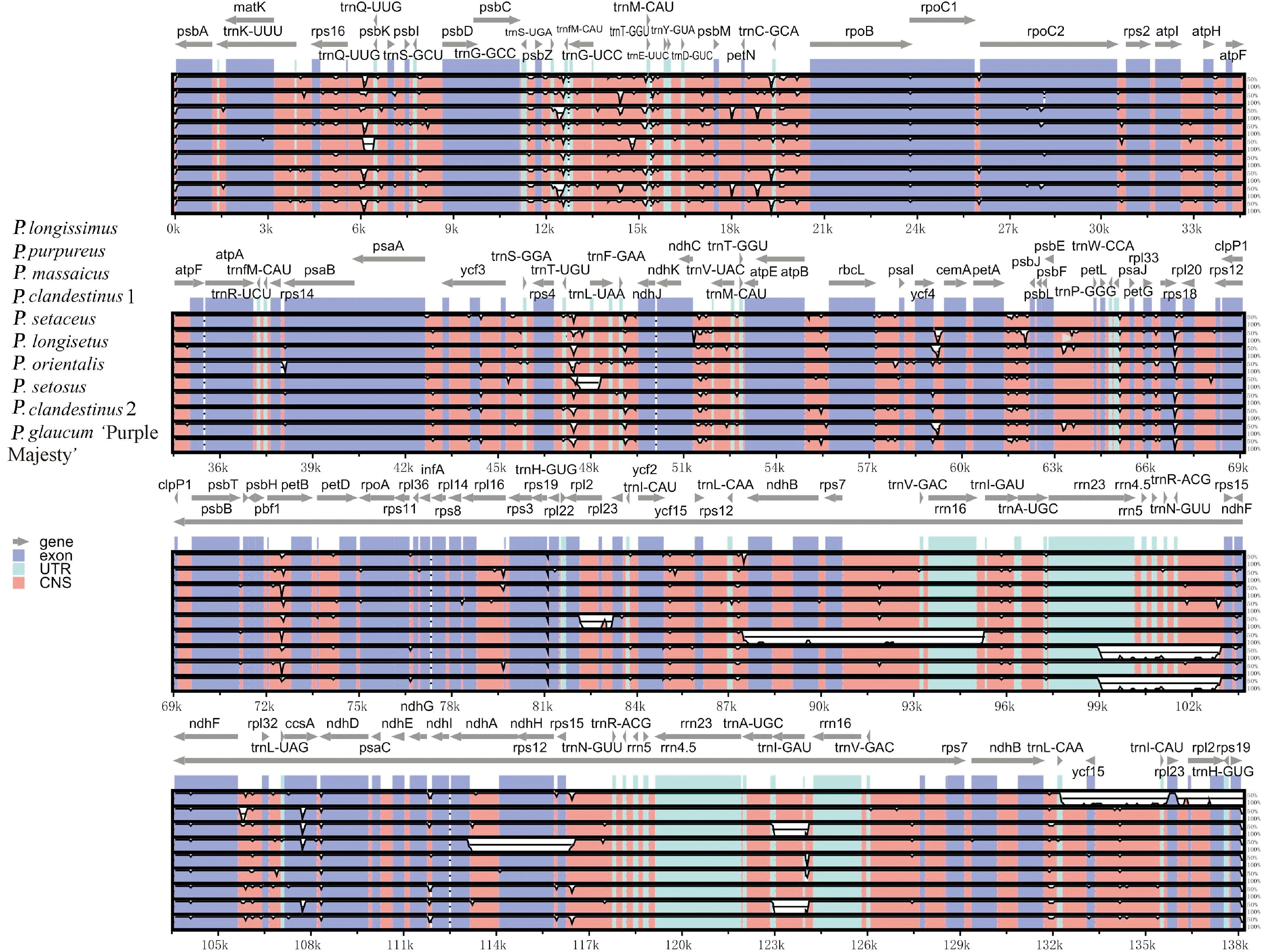
图5 10个狼尾草属植物叶绿体基因组序列对比图
2.2.4 突变热点筛选
利用DnaSP软件计算10个狼尾草属植物叶绿体基因组在600 bp范围内的核苷酸变异度 (Pi)。结果表明10个叶绿体基因组核苷酸变异度为0~0.022 07,平均值为0.005 27。有10个存在明显序列变异的高变区域(Pi>0.015),分别为trnK(UUU)-rps16,psbZ-trnM-CAU,trnS-CGA-trnT-GGU,rpl2-trnE-UUC,trnD-GUC-psbN,petN-rpoB,trnL-UAA-ndhJ,petA-psbJ,psbE-petL,ndhF-ccsA,其中9个位于LSC区,1个位于SSC区,IR区未发现变异较高的序列片段(图6A)。核苷酸变异度为0~0.021 87,平均值为0.005 19。有9个存在明显序列变异的高变区域(Pi>0.015),分别为rps16,rps16-psbK,trnS-CGA-trnT-GGU,petN-rpoB,rps4-trnL-UAA,trnL-UAA-ndhJ,petA-psbJ,psbE-petL,ndhF-ccsA。
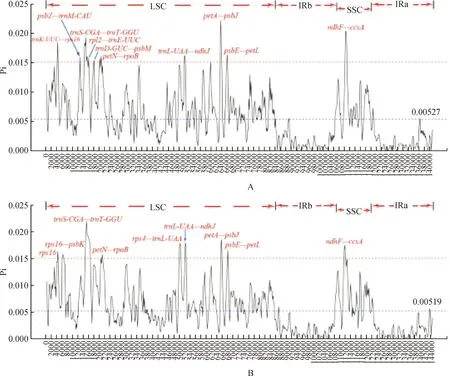
图6 狼尾草属植物10个叶绿体基因组核苷酸变异度(Pi)的比较(A),狼尾草属植物27个叶绿体基因组核苷酸变异度(Pi)的比较(B)
2.3 基于叶绿体基因组的狼尾草属植物系统发育分析
基于叶绿体基因组序列构建的狼尾草属植物BI和ML发育树具有较好的支持率,发育树结果一致(图7)。两个发育树均形成Clade Ⅰ和Clade Ⅱ两个大的分支,其中Clade Ⅰ分支(BS=100%,PP=1)包括5个物种:羽绒狼尾草(P.setaceus)、非洲狼尾草(P.massaicus)、白穗狼尾草(P.alopecuroides)、P.compressus、东非狼尾草(P.clandestinus),其中东非狼尾草的4个样本聚在一个亚分支(BS=100%,PP=1),与非洲狼尾草、P.alopecuroides、P.compressus形成姐妹关系(BS=100%,PP = 0.99)。
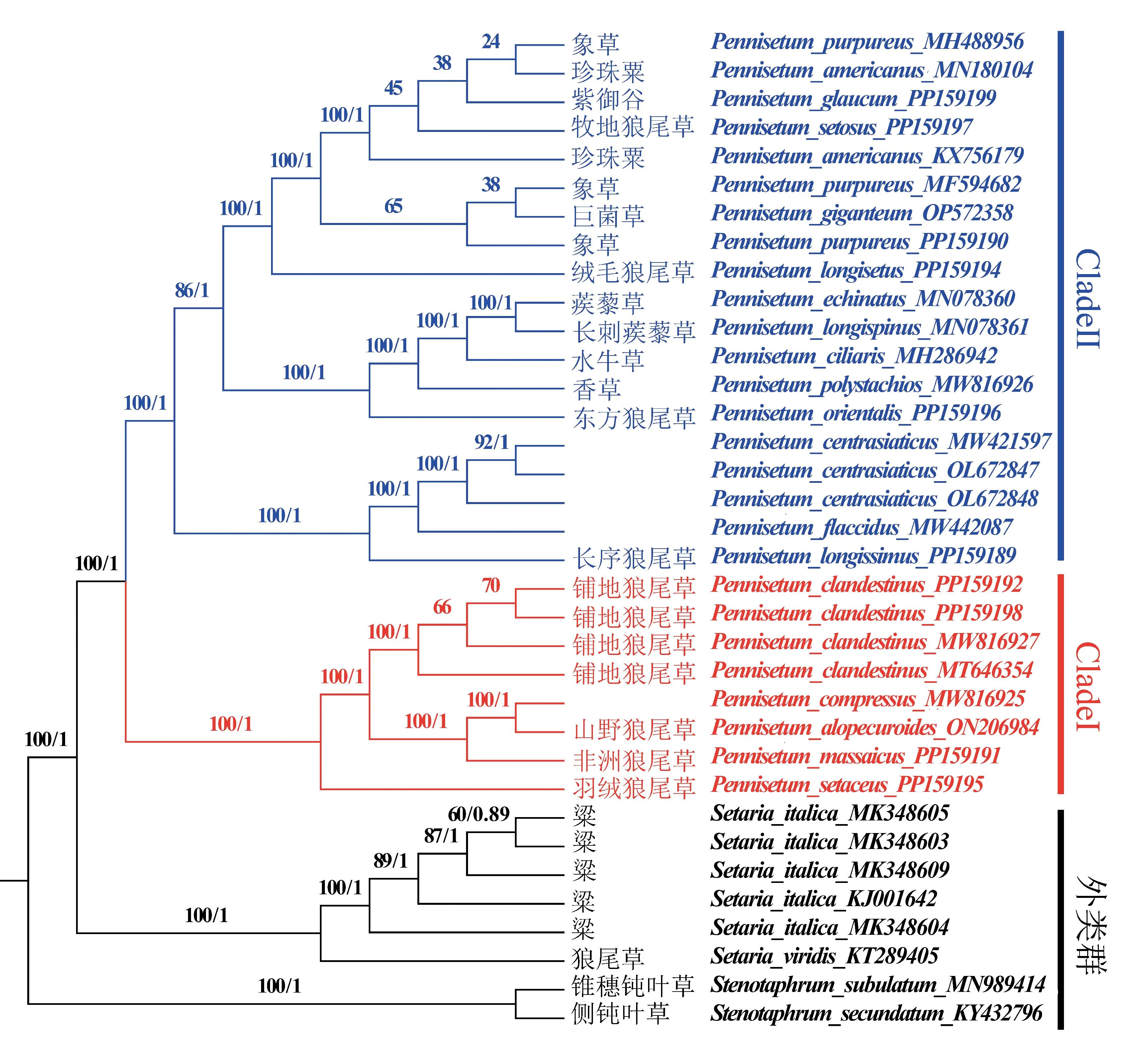
图7 基于叶绿体基因组数据构建狼尾草属植物ML和BI系统发育树
CladeⅡ分支(BS=100%,PP=1)又可分为三个亚支,第一个亚支(BS=100%,PP=1),包括长序狼尾草(P.longissimus)、P.flaccidus、P.centrasiaticus,其中,P.centrasiaticus的3个样本聚在一起,P.flaccidus和长序狼尾草与P.centrasiaticus形成姐妹关系(BS=100%,PP=1);第二个亚分支(BS=100%,PP=1),包括东方狼尾草、P.polystachios、P.ciliaris、P.longispinus、P.echinatus,且与第一个亚支形成姐妹关系(BS=55%,PP=1);第三个亚支(BS=100%,PP=1)包括绒毛狼尾草、象草(P.purpureus)、P.giganteum、紫御谷(P.glaucum‘Purple Majesty’)、牧地狼尾草(P.setosus)、P.americanus,其中象草的4个样本并未聚在一起,象草(MH488956)这个样本嵌套在另一小支中。
3 讨论与结论
3.1 讨论
高等植物的叶绿体基因组的序列长度一般在120~160 kb之间,如冉然[28]对5种蒿属(ArtemisiaLinn.)植物研究发现其叶绿体基因组序列长度在151 076~151 318 bp之间;岳杰[29]对4种人参属(PanaxLinn.)植物研究发现其叶绿体基因组序列长度在155 993~156 359 bp之间;顾丽[30]对13种天胡荽属(HydrocotyleL.)植物研究发现其叶绿体基因组大小在152 659~153 669 bp之间。本研究新测序的10个狼尾草属植物叶绿体基因组大小在137 929~138 554 bp之间,最大差距为625 bp,最小的是非洲狼尾草,为137 929 bp,最大的是羽绒狼尾草,为138 554 bp。狼尾草属植物叶绿体基因组均为典型四分体结构,完全符合高等植物叶绿体基因组特征。一般来说每个物种都有一个相对独立且固定的GC含量,如石蒜科(Amaryllidaceae)[31]、蔷薇科(Rosaceae)[32]等部分植物均存在此特征,本研究中10个狼尾草属植物的叶绿体基因组GC含量范围为38.6%~38.7%,且IR区GC含量(44%~44.1%)高于LSC区(36.4%~36.6%)和SSC区(33%~33.2%),推测IR区rRNA基因的存在可能是导致IR区GC含量较高的主要原因[33]。
完整的叶绿体基因组是用于系统发育分析的有价值的遗传标记来源,因为其基因组结构相对保守[34-35]。而IR区是植物叶绿体基因组中最保守的区域,IR区的长度、结构及与SC区的边界都表现出较高的保守性[36-37],IR区的扩张和收缩作为引起植物叶绿体基因组长短变化的重要因素,大多数物种的IR区的扩张与收缩体现在IR/SC边界在几个固定基因内的少量偏移[38],这种偏移可能导致部分基因成为假基因。本研究中10个狼尾草属植物基因组的IR边界区域比较保守,几乎没有差异。叶绿体基因组均有113个基因,包括78个编码蛋白基因,31个tRNA基因和4个rRNA基因,叶绿体基因组的大小、基因顺序和组成与已报道的狼尾草属植物基因组高度相似[2]。
在基因组中,并不是所有的基因突变事件都是随机的,而是聚集性的[39-40],并且这些突变位点在基因组中创造了高度可变的区域[41]。在已完成测序的狼尾草属植物中,广泛用作植物DNA条形码的matK、rbcL和psbA-trnH基因在叶绿体基因组中的遗传变异低于预期,但却发现9个其他高变区,并且有两个高变区(psbE-petL与petA-psbJ),这与Xu等[2]研究结果一致。基因组中存在的高变区可用做DNA条形码鉴别物种[42-43],也可作为重建狼尾草属系统发育关系的潜在标记,因此,这些高度分化的区域能为狼尾草属植物鉴定、系统发育、分子标记开发提供丰富的信息。
本研究基于叶绿体全基因组构建的贝叶斯树和最大似然树,具有较高的支持率,ML树和BI树均支持分成2个大的分支,其中1个分支分为3个亚枝,在基于叶绿体全基因组构建的发育树中,东非狼尾草与P.alopecuroides(NC—064146)和P.alopecuroides(ON 206984)聚在一起,其亲缘关系较近,这与Xu等[2]的研究结果相同,另外Liu等[44]研究中发现,P.americanus(KX756179)、P.americanus(MN180104)、象草、绒毛狼尾草在系统发育树中聚在一起,这也与本研究中系统发育分析结果相吻合,值得一提的是,所有发育树中象草 的4个样本并未聚在一起,象草(MH488956)样本嵌套在其他亚支中,推测该种可能为杂交种或地域原因导致的遗传差异。
3.2 结论
本研究测序的10个狼尾草属植物叶绿体基因组均为典型的四分体结构,序列长度在137 929~138 554 bp之间,其结构无明显差异,GC含量在38.6%~38.7%之间,编码113个基因。重复序列检测分析表明:在简单序列重复检测中单核苷酸重复数量最多,而在非简单序列重复检测中串联重复所占比例最高。结构突变以及序列差异的分析显示:IR区边界无明显伸缩或扩张现象,LSC区和SSC区的变异率较高。系统发育关系分析显示:10 份狼尾草属植物样本间界限清晰,发育树形成两个大的分支,其中第一个分支又可分为3个亚支,且第一亚枝与第二亚枝为姐妹群。对目前已发表叶绿体基因组的狼尾草属植物的系统发育分析揭示了物种之间的亲缘关系,为狼尾草属植物开发利用以及遗传改良提供理论基础。

