利用酵母双杂交系统筛选玉米ZmPRR73的互作蛋白
王盼盼 王雷立 张严玲 刘青青 董柯清 李安然 王翠玲
摘 要 关键词 玉米;酵母双杂交; ZmPRR73;生物钟;互作蛋白
生物钟(Circadian clock)是生命体对地球一年四季的季节变化和一天内24 h昼夜节律的长期适应而演化产生的一种复杂的内在计时器。生物钟可以帮助生命体感知并响应周围环境的周期性变化,从而调控生物体的新陈代谢、生长发育和增加对逆境胁迫环境的适应性[1-4]。PRRs(pseudo-response regulators)家族蛋白基因是生物钟中央振荡器的重要组成部分,与两个编码MYB蛋白的基因LHY(late elongated hypocoty1)和CCA1(circadian clock associated 1)一起组成转录翻译反馈环,三者在转录和翻译水平的昼夜周期性变化维持了生物节律钟的昼夜节律[5-7]。
目前关于PRRs蛋白的研究主要集中LHY、CCA1 和PRRs的转录反馈抑制调控环上,着重于上游基因结合在下游基因的启动子上抑制其表达,通过ChIP-Seq技术和转录组分析,科学家对拟南芥PRR9、PRR7、PRR5和TOC1转录因子的结合区域进行研究,结果表明PRRs家族转录因子直接调控植物的生长、光信号传导、非生物胁迫和光周期调控开花途径[8-11]。研究显示LHY、CCA1 和PRRs的相互抑制调控不是上游基因直接结合在下游基因的启动子上,而是与其他基因互作,形成复合体后结合在后者的启动子上抑制其表达,例如,TOC1 与TCP21(TEOSINTE BRANCHED1-CYCLOIDEA-PCF21)互作来抑制CCA1的表达[12]。PRR9、PRR7和PRR5蛋白能通过其EAR(ERF-associated amphiphilic repression)结构域与TOPLESS和HDA6(histone deacetylase 6)形成一个复合体,结合到CCA1和LHY的启动子上从而抑制CCA1和LHY的转录,调节生物钟周期[13]。因此筛选生物钟基因PRRs的互作蛋白,对研究这些基因的反馈调控机制具有非常重要的意义。
与拟南芥和水稻中各存在5个PRR基因家族成员不同的是,玉米中共鉴定到9个ZmPRR基因( ZmPRR1-1、 ZmPRR1-2、 ZmPRR37-1、 ZmPRR37-2、 ZmPRR73、 ZmPRR59-1、 ZmPRR59-2、 ZmPRR95-1和 ZmPRR95-2),除 ZmPRR73外,這些基因中分别有两个与拟南芥AtPRR或水稻OsPRR基因家族成员高度同源[14-15]。河南科技大学玉米分子育种课题组前期对调控玉米开花时间和光周期敏感性的主效QTL qDPS9进行了精细定位,并克隆了候选基因 ZmPRR73,生物信息学分析表明 ZmPRR73基因编码的蛋白属于伪应答调节蛋白PRRs家族成员,与拟南芥 AtPRR7基因同源性最高[16]。研究表明 PRR7及其同源基因在拟南芥、水稻、高粱、小麦、大麦等植物的开花时间的光周期调控中发挥重要作用[17-19]。近期研究表明PRRs家族不但在生物钟节律中起关键作用,在植物应对寒冷、干旱、氧化应激、病原体攻击等生物钟基因在胁迫响应调节中也起着非常重要的作用[20-23]。 ZmPRR73基因作为玉米生物钟中央振荡器的重要组成部分,在玉米开花时间调控中的分子功能和参与玉米逆境胁迫响应的分子机制尚未见 报道。
酵母双杂交技术是一种检测细胞内蛋白间互作的技术,不仅可以检测到蛋白之间稳定的相互作用,也能够敏感地检测到蛋白质之间微弱或短暂的相互作用,成为目前高通量筛选互作蛋白的最常用的方法。许多研究者构建酵母双杂交cDNA文库,利用酵母双杂交技术筛选到很多非常重要的与诱饵蛋白相互作用的蛋白[24-26]。本研究旨在从长日照处理的热带玉米自交系的cDNA文库中初步筛选与ZmPRR73互作的蛋白,进一步揭示 ZmPRR73基因在玉米光周期反应和环境胁迫应答中的调控机制及生物学通路。
1 材料与方法
1.1 试验材料
长日照诱导热带玉米自交系CML288的酵母双杂交cDNA文库和携带目的基因 ZmPRR73的PMD19T-ZmPRR73质粒由河南科技大学玉米分子育种课题组构建并保存。质粒提取试剂盒和酵母质粒提取试剂盒购自OMEGA公司。酵母菌株Y2H Gold、酵母双杂交载体pGBKT7-53、pGBKT7-Lam、pGADT7-T、pGBKT7等购自上海欧易公司。Infusion连接酶和限制性内切酶购自TaKaRa公司。酵母缺陷型培养基和X-α-Gal等购自北京酷来搏科技有限公司。
1.2 诱饵载体pGBKT7-ZmPRR73的构建
根据 ZmPRR73基因和pGBKT7载体序列设计带酶切位点的特异性引物( ZmPRR73-in-F:CATGGAGGCCGAATTCATGGGCAGTGCT-TGCCAAGCTGGC; ZmPRR73-in-R:GCAGGTCGACGGATCCTCATCTTTCTGAACCTT-GCGCTGT)。以PMD19T-ZmPRR73质粒为模板使用高保真酶进行PCR 扩增,回收、纯化并鉴定目的片段;同时使用限制性内切酶EcoRⅠ和BamHⅠ对pGBKT7载体进行双酶切,胶回收并纯化线性化后的载体;然后按照Infusion试剂盒的方法,利用同源重组技术将 ZmPRR73基因CDS序列插入到pGBKT7载体的酶切位点EcoRⅠ和BamHⅠ之间,构建诱饵载体pGBKT7-ZmPRR73。
1.3 诱饵载体pGBKT7-ZmPRR73毒性及自激活检测
把pGBKT7-53+pGADT7-T作为阳性对照,pGBKT7-Lam+pGADT7-T作为阴性对照, pGBKT7-ZmPRR73+pGADT7作为自激活检测组,将对照组与自激活检测组载体分别转化酵母Y2HGold感受态细胞。分别挑取阳性单克隆接种到SD/-Leu/-Trp/X-α-Gal和SD/-Leu/-Trp/-His/-Ade/ X-α-Gal /AbA营养缺陷型培养基上,30 ℃恒温培养3~5 d,观察酵母菌落生长情况。
1.4 酵母双杂交文库筛选及阳性克隆的鉴定
制备新鲜的酵母感受态细胞,将诱饵质粒pGBKT7-ZmPRR73与pGADT7-cDNA文库质粒共转化Y2H Gold酵母感受态细胞。将转化产物均匀涂布于SD/-Leu/-Trp/-His/ X-α-Gal固体培养基上进行初筛,挑选蓝色阳性单克隆菌,接种到SD/-Leu/-Trp/-His/-Ade/X-α-Gal/AbA培养基上进行高严谨度筛选。30 ℃恒温培养3~5 d,菌落生长至约2~3 mm,挑选蓝色阳性克隆单菌落接种于SD/-Leu/-Trp液体培养基中, 30 ℃培养16~18 h。菌液PCR 验证,对阳性菌液提取酵母质粒,用热激法转入大肠杆菌DH5α感受态中,送阳性菌液至上海生工测序。
1.5 互作蛋白的点对点验证
将构建好的pGBKT7-ZmPRR73质粒与pGADT7-候选互作蛋白质粒,共转于Y2H Gold酵母感受态细胞,涂布于SD/-Leu/-Trp/-His平板中,培养3~5 d。挑取单菌落在YPDA 液体培养基中30 ℃恒温培养至菌液OD600约为0.8时,将菌液分别稀释10倍、100倍、1 000倍、 10 000倍,各取5 μL点样在SD/-Leu/-Trp培养基和SD/-Leu/-Trp/-His/-Ade/X-α-gal/AbA培养基上,30 ℃倒置培养3~5 d,观察共转酵母质粒的生长情况。以pGBKT7-53+pGADT7-T为阳性对照, pGBKT7-lam+pGADT7 和pGBKT7-ZmPRR73+pGADT7为阴性对照,平行重复3次。
2 结果与分析
2.1 诱饵载体pGBKT7-ZmPRR73的构建
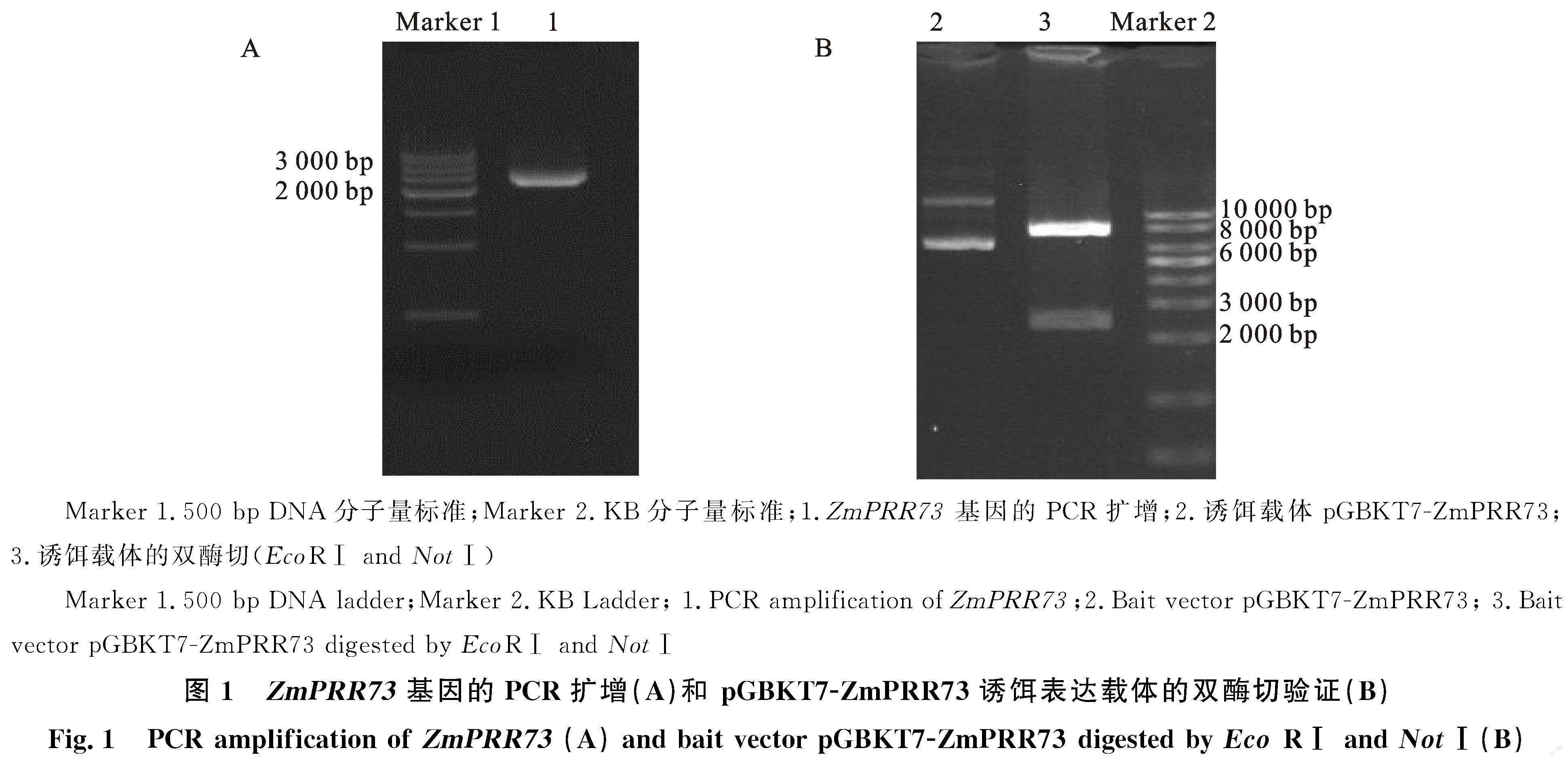
前期从热带玉米自交系CML288中克隆到 ZmPRR73基因的CDS序列,片段大小为2 298 bp(图1-A),以PMD19T-ZmPRR73 质粒为模板,用引物ZmPRR73-in-F / ZmPRR73-in-R 使用高保真酶进行PCR 扩增目的片段并验证,片段大小和测序结果与目的基因完全一致(图1-A)。在此基础上,利用同源重组的方法将目的片段 ZmPRR73与线性化的pGBKT7载体进行连接。连接产物转化到大肠杆菌DH5α感受态中,挑取单克隆菌落做菌液PCR,测序结果显示 ZmPRR73已插入到序列的正确位置,重组载体读码框正确,无移码突变,说明目的基因与载体融合,pGBKT7-ZmPRR73诱饵载体构建成功(图1-B)。
2.2 诱饵载体毒性及自激活分析
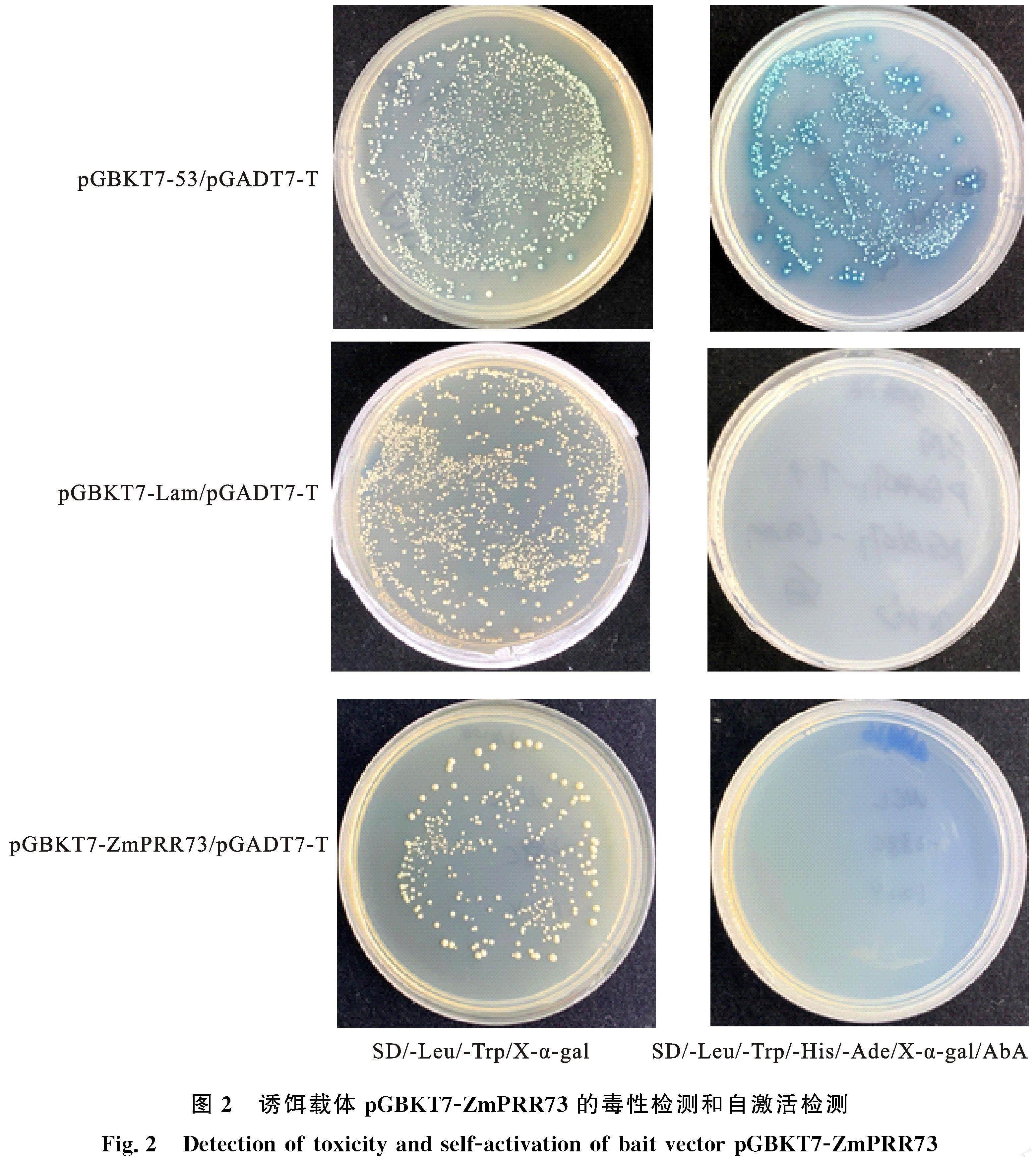
将阳性对照pGBKT7-53/pGADT7-T、阴性对照pGBKT7-Lam/pGADT7-T和pGBKT7-ZmPRR73/pGADT7分别转化Y2H Gold酵母感受态细胞,分别涂布在SD/-Leu/-Trp/X-α-Gal和SD/-Leu/-Trp/-His/-Ade/X-α-Gal/AbA培养基上培养,在30 ℃条件下培养3~5 d。结果显示阳性对照pGBKT7-53/pGADT7-T在SD/-Leu/-Trp/X-α-Gal培养基上有克隆生长且变蓝,阴性对照pGBKT7-Lam/pGADT7-T和诱饵载体pGBKT7-ZmPRR73/pGADT7在SD/-Leu/-Trp/X-α-Gal培养基上有白色克隆生长,但不变蓝,说明pGBKT7-ZmPRR73质粒成功转入酵母菌株中,且诱饵载体pGBKT7-ZmPRR73对宿主酵母菌无毒性(图2)。
阳性对照pGBKT7-53/pGADT7-T在SD/-Leu/-Trp/-His/-Ade/X-α-gal/AbA 培养基上蓝色克隆生长,阴性对照pGBKT7-Lam/pGADT7-T和诱饵载体pGBKT7-ZmPRR73/pGADT7在SD/-Leu/-Trp/-His/-Ade/X-α-gal/AbA培养基上无克隆生长,说明pGBKT7-ZmPRR73不存在自激活现象(图2)。综上所述,诱饵载体pGBKT7-ZmPRR73在和空AD质粒pGADT7共转时,在Y2H Gold酵母菌株中没有自激活活性,可用于后续筛库实验。
2.3 互作蛋白的筛选与分离
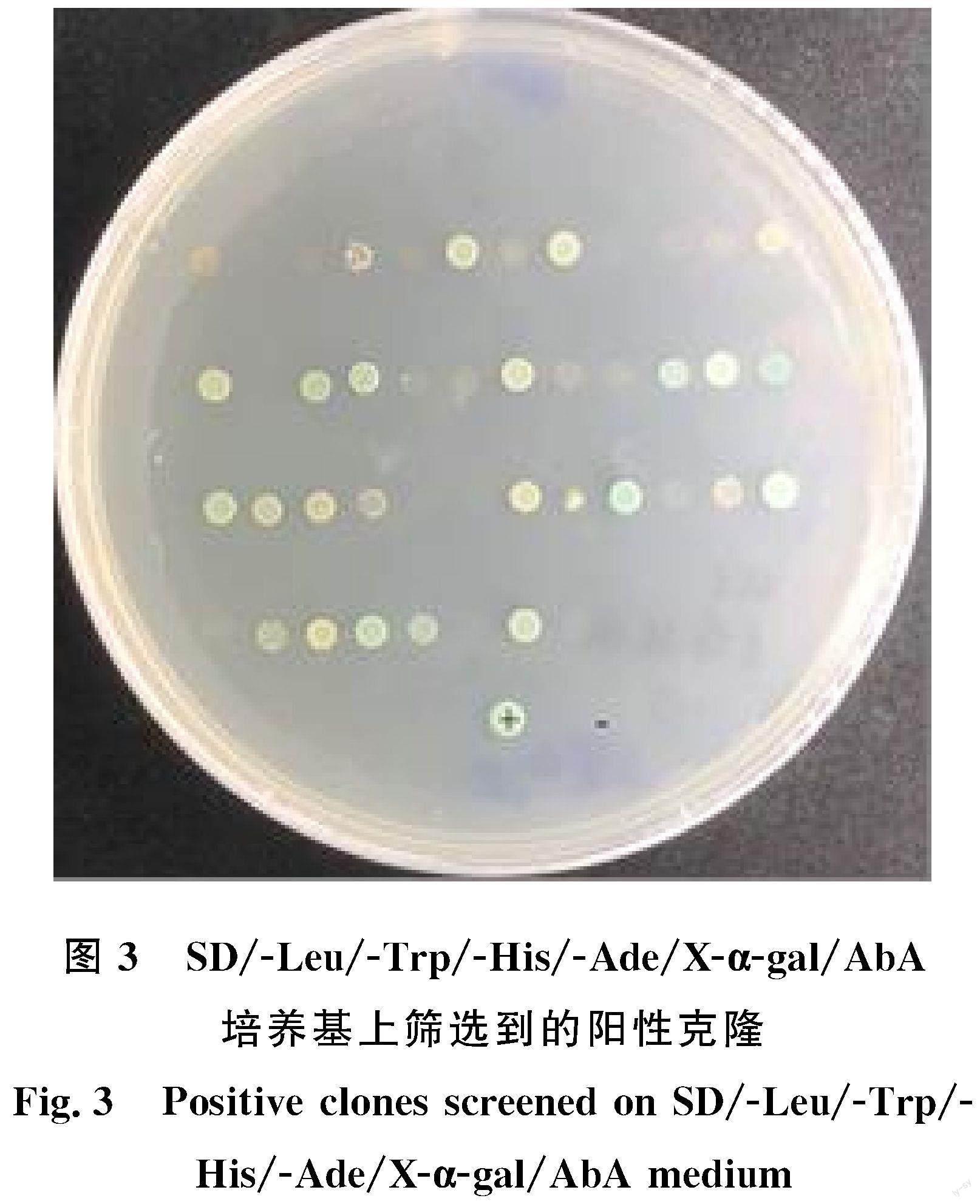
以pGBKT7-ZmPRR73为诱饵蛋白,与热带玉米自交系CML288的叶片和根组织等量混合构建的cDNA文库共同转化酵母感受态细胞Y2H Gold,涂布到三缺培養基SD/-Leu/-Trp/-His/X-α-Gal上进行初筛,挑取直径大于2 mm且生长健康的蓝色克隆,接种到四缺培养基SD/-Leu/-Trp/-His/-Ade/X-α-Gal/AbA上进行高严谨度筛选。结果显示,在SD/-Leu/-Trp/-His/X-α-Gal缺陷培养基筛选平板上共有44个克隆生长变蓝,再次转到四缺培养基SD/-Leu/-Trp/-His/-Ade/X-α-Gal/AbA筛选平板上复筛后,共获得18个候选阳性克隆(图3)。对阳性克隆菌落提取酵母质粒,并转化入大肠杆菌DH5α感受态细胞,再次培养后送大肠杆菌阳性菌液至上海生工测序,测序结果显示除去重复序列后最终确定有12个互作阳性克隆。
2.4 互作蛋白的测序及比对分析
测序结果显示除去重复序列后最终确定有12个互作阳性克隆。生物信息学分析表明这些基因功能涉及植物的转录调控、离子跨膜转运的调节、信号转导、电子传递链等多个方面(表1)。值得注意的是,第2号蛋白编码S-腺苷甲硫氨酸合成酶,具有甲硫氨酸腺苷转移酶活性,可能参与S-腺苷甲硫氨酸生物合成过程[27-28]。第3号蛋白编码拟南芥铁氧还蛋白Ferredoxin5(Fd5)的同源基因,具有电子转移活性和金属离子结合功能,可能参与电子传递链,对植物的生长发育至关重要[29]。第11号蛋白编码拟南芥AP2/EREBP类转录因子EREB17的同源基因,可能参与下游基因的转录调控,在植物的生长发育的信号转导中发挥重要的作用[30]。互作蛋白中还有4个未知蛋白,其可能的功能仍需进一步探究。
2.5 互作蛋白的点对点验证
为进一步验证ZmPRR73与筛选得到的蛋白之间存在的互作关系,从中选取1号蛋白Zm00001eb210500和2号蛋白Zm00001eb354640的cDNA 插入序列转化含有pGBKT7 或pGBKT7-ZmPRR73转化子的酵母感受态中,进行点对点验证(图4)。阴性对照pGBKT7-Lam+pGADT7-T、阳性对照pGBKT7-53+pGADT7-T、空载对照pGBKT7-ZmPRR73+pGADT7以及pGBKT7-ZmPRR73+pGADT7-Zm00001eb210500和pGBKT7-ZmPRR73+pGADT7-Zm00001eb354640均能在SD/-Leu/-Trp培养基上正常生长。在SD/-Leu/-Trp/-His/-Ade/X-α-Gal/AbA培养基上,只有阳性对照pGBKT7-53+pGADT7-T和pGBKT7-ZmPRR73+pGADT7-Zm00001eb210500以及pGBKT7-ZmPRR73+pGADT7-Zm00001- eb354640能正常生长且显现蓝色,而阴性对照pGBKT7-Lam+pGADT7-T和空载对照pGBKT7-ZmPRR73+pGADT7均未见菌斑,表明Zm00001eb210500蛋白和Zm00001eb354640蛋白与ZmPRR73在酵母体内均能够发生相互作用,排除了假阳性的干扰。
3 讨 论
挖掘 ZmPRR73基因的互作蛋白是解析其功能及分子通路的关键。目前有关植物蛋白质互作的研究方法有很多,包括酵母双杂交系统(Yeast two-hybrid system,Y2H)、免疫共沉淀(Co-Immunoprecipitation,Co-IP)、双分子荧光互补技术(Bimolecular Fluorescence Complementation,BiFC)、pull-down技术、荧光共振能量转移技术(Fluorescence Resonance Energy Transfer,FRET)、蛋白质芯片技术(Protein chip)、亲和纯化-质谱技术(AP-MS)和邻近标记技术(Proximity labeling)等[31]。其中酵母双杂交技术是一种简单易行、高通量、高灵敏度的检测细胞内蛋白质相互作用的技术,不论是对蛋白质之间稳定的相互作用还是微弱而短暂的相互作用均能够敏感地检测到。通过构建不同处理条件下的酵母双杂交文库,将目的基因作为诱饵,利用酵母双杂交技术筛选与诱饵相互作用的蛋白,找到基因之间的联系,构建蛋白质互作网络,建立蛋白连锁图,已经成为解析重要基因的分子功能及信号传导、代谢途径、分子通路的最常用的方法。本研究利用酵母双杂交技术挖掘出了 ZmPRR73基因的一些重要的互作蛋白,为进一步解析 ZmPRR73基因的功能及作用机制和调控网络奠定了基础。鉴于酵母双杂交系统可能存在假阳性问题,需要继续用双分子荧光互补试验对互作蛋白进行逐一验证,并通过互作蛋白之间的相互作用研究 ZmPRR73基因的调控机制和生物学通路。
本研究中筛选到12个与ZmPRR73相互作用的候选蛋白,包括外包膜孔蛋白24A、S-腺苷甲硫蛋氨酸合成酶1、铁氧还蛋白5、富半胱氨酸/组氨酸C1结构域家族蛋白、PTR 家族蛋白、核糖体蛋白、LBD转录因子、BTB/POZ结构域蛋白、DUF3143家族蛋白、AP2/EREBP类转录因子17、DUF1997家族蛋白等。其中筛选到的2号基因( Zm00001eb354640)编码S-腺苷甲硫氨酸合成酶( ZmSAMS1),是植物代谢过程中的一个关键酶,它催化ATP和L-甲硫氨酸反应生成S-腺苷甲硫氨酸,在干旱、高温、低温、盐和ABA等逆境胁迫环境下调节相关基因的表达以适应环境的变化,对植物的适应性具有重要意义[27-28]。本研究筛选鉴定到ZmSAMS1蛋白與生物钟中央振荡器核心元件ZmPRR73蛋白存在直接互作,表明S-腺苷甲硫氨酸合成酶不但参与了干旱、高温、低温、盐和ABA等逆境胁迫响应,而且还与玉米的光周期反应有关。生物信息学分析显示玉米S-腺苷甲硫氨酸合成酶基因 ZmSAMS1的启动子区域包含包括G-Box、AAGAA-motif、ATCC-motif、Box 4,Box-W1、GATA-motif、 I-box,chs-CMA2bGA-motif、GAG-motif、circadian等重要的光应答元件,表明ZmSAMS基因的表达可能受光周期反应的调控[28]。具体生物钟基因 ZmPRR73如何影响S-腺苷甲硫氨酸的合成,或者S-腺苷甲硫氨酸合成酶 ZmSAMS1如何参与玉米的生物钟反应,深入研究二者相互作用的分子机制和生物学功能具有非常重要的意义。
筛选到的3号蛋白与拟南芥铁氧还蛋白Ferredoxin5(Fd5)同源,有研究报道在植物光合过程中,光合型铁氧还蛋白作为唯一的可溶性电子受体,可将来自光系统I的电子传输到下游各种代谢过程,将光能转化为化学能,是光合电子向叶绿素代谢、碳氮同化、光敏色素合成、脂肪酸合成等生物学途径分配的中枢元件,对植物的生长发育至关重要。水稻铁氧还蛋白I突变后会造成水稻光合电子传递受阻,光合碳同化过程被破坏,从而引起突变体内源光合产物合成不足,导致突变体快速褪绿并致死的表型[29]。筛选到的5号蛋白(Zm00001eb251540)编码植物硝酸盐转运蛋白NRT1/PTR FAMILY 8.3,植物硝酸盐转运蛋白(Nitrate transporter,NRT)是植物氮素营养系统的关键生物大分子之一,在吸收和感知外界硝酸盐及信号调控方面有着重要的作用。NRT1/PTR FAMILY 8.3是二肽转运体,在氮循环中很活跃的,可以影响信号传导过程和次生代谢产物的生产过程,干旱促进NRT1/PTR FAMILY 8.3基因的表达,随着干旱程度的增加,其表达量显著上调[32]。有文献表明硝酸盐转运蛋白的表达活性受昼夜节律调节[33-34],但是其相关分子机制迄今未见报道,本研究鉴定到生物钟元件ZmPRR73蛋白与玉米硝酸盐转运蛋白Zm00001eb251540存在互作,进一步深入研究二者的互作机制,将为探索硝酸转运蛋白的昼夜节律表达的分子机理奠定基础。11号蛋白编码AP2/EREBP转录因子家族蛋白EREB17,研究表明AP2-EREBP家族蛋白家族是植物特有的一类转录因子,广泛参与了植物的细胞分化、激素调控、病源反应以及生物和非生物胁迫应答反应等各个生命过程,在植物的生长发育以及逆境胁迫应答等信号转导中发挥重要的作用[30,35]。以上这些互作蛋白的发现,表明 ZmPRR73基因除了维持生物钟节律外,还广泛参与玉米生长发育、逆境胁迫响应、氮素营养调控、光合电子传递等重要的生理生化过程。
因此在本研究的基礎上,进一步开展玉米ZmPRR73与候选互作蛋白EREB17、SAMS1、Protein NRT1/ PTR FAMILY 8.3和FDX5的互作关系研究,对深入了解玉米 ZmPRR73基因的功能及其参与逆境胁迫响应机制有重要意义。
参考文献 Reference:
[1] 谢启光,徐小冬.植物生物钟与关键农艺性状调控[J].生命科学,2015,27(11):1336-1344.
XIE Q G,XU X D.Circadian clock and the control of key agronomic traits in higher plants[J].Chinese Bulletin of Life Sciences,2015,27(11):1336-1344.
[2] 王 晗.生物钟生物学及其研究进展[J].生命科学,2015,27(11):1313-1319.
WANG H.Circadian biology and its recent progresses[J].Chinese Bulletin of Life Sciences,2015,27(11):1313-1319.
[3] YAN J,KIM Y J,SOMERS D E.Post-Translational Mechanisms of Plant Circadian Regulation [J].Genes,2021, 12(3):325.
[4] 谢启光,徐小冬.作物生物钟的研究进展与前瞻[J].植物生理学报,2022,58(1):39-51.
XIE Q G,XU X D.Progress and prospects of circadian clock in agriculture[J].Plant Physiology Journal,2022,58(1):39-51
[5] SHIM J S,KUBOTA A,IMAIZUMI T.Circadian clock and photoperiodic flowering in Arabidopsis:Constans is a hub for signal integration[J].Plant Physiology,2017,173(1):5-15.
[6] NOHALES M A,KAY S A.Molecular mechanisms at the core of the plant circadian oscillator[J].Nature Structural & Molecular Biology,2016,23(12):1061-1069.
[7] 魏 华,王 岩,刘宝辉,等.植物生物钟及其调控生长发育的研究进展[J].植物学报,2018,53(4):456-467.
WEI H,WANG Y,LIU B H, et al.Deciphering the underlying mechanism of the plant circadian system and its regulation on plant growth and development[J].Chinese Bulletin Botany,2018,53(4):456-467
[8] HUANG W,PREZ-GARCA P,POKHILKO A,et al.Mapping the core of the Arabidopsis circadian clock defines the network structure of the oscillator[J].Science,2012,336(6077):75-79.
[9] NAKAMICHI N,KIBA T,KAMIOKA M,et al.Transcriptional repressor PRR5 directly regulates clock-output pathways [J].Proceedings of the National Academy of Sciences of the United States of America,2012,109(42):17123-17128.
[10] LIU T,CARLSSON J,TAKEUCHI T,et al.Direct regulation of abiotic responses by the Arabidopsis circadian clock component PRR7[J].The Plant Journal,2013, 76(1):101-114.
[11] CERVELA-CARDONA L,YOSHIDA T,ZHANG Y,et al.Circadian control of metabolism by the clock component TOC1 [J]. Frontiers in Plant Science,2021,12:683516.
[12] NAKAMICHI N.The transcriptional network in the Arabidopsis circadian clock system[J]. Genes(Basel),2020,11(11):1284.
[13] WANG L,KIM J,SOMERS D E.Transcriptional corepressor TOPLESS complexes with pseudoresponse regulator proteins and histone deacetylases to regulate circadian transcription [J].Proceedings of the National Academy of Sciences of the United States of America,2013, 110(2):761-766.
[14] WANG C L,WANG L L,LIU Q Q, et al.Genome-wide identification and characterization of PRR gene family and their diurnal rhythmic expression profile in maize[J].International Journal of Genomics,2022,2022:6941607.
[15] LAI X,BENDIX C,YAN L,et al.Interspecific analysis of diurnal gene regulation in panicoid grasses identifies known and novel regulatory motifs[J].BMC Genomics,2020,21:428.
[16] 趙淑靓.玉米伪应答调节基因 ZmPRR73的克隆与表达分析[D].河南洛阳:河南科技大学,2018.
ZHAO SH J.Gene cloning and expression analysis of ZmPRR73 in Zea mays[D].Luoyang Henan:Henan University of Science and Technology,2018.
[17] WANG L,SUN S,WU T,et al.Natural variation and CRISPR/Cas9-mediated mutation in GmPRR37 affect photoperiodic flowering and contribute to regional adaptation of soybean[J].Plant Biotechnology Journal,2020, 18(9):1869-1881.
[18] 梁力文.水稻CCT家族基因OsPRR73和OsCO3调控抽穗期的分子机制研究[D].武汉:华中农业大学,2020.
LIANG L W.Study on the molecular mechanisms of rice CCT family genesOsPRR73 and OsCO3 in regulating heading date[D].Wuhan:Huazhong Agricultural University,2020.
[19] ZHANG W,ZHAO G,GAO L,et al.Functional studies of heading date-related gene TaPRR73,a paralog of Ppd1 in common wheat[J].Frontiers in Plant Science,2016,7:772.
[20] YANG M,HAN X,YANG J,et al.The Arabidopsis circadian clock protein PRR5 interacts with and stimulates ABI5 to modulate abscisic acid signaling during seed germination[J].The Plant Cell,2021,33(9):3022-3041.
[21] ZHANG Y,PFEIFFER A,TEPPERMAN J M,et al.Central clock components modulate plant shade avoidance by directly repressing transcriptional activation activity of PIF proteins[J].Proceedings of the National Academy of Sciences of the United States of America,2020,117(6):3261-3269.
[22] LI N,ZHANG Y,HE Y, et al.Pseudo response regulators regulate photoperiodic hypocotyl growth by repressing PIF4/5 transcription [J].Plant Physiology,2020,183(2):686-699.
[23] 李剑峰,李 婷,贾小平.PRRs家族功能基因的研究进展[J].植物遗传资源学报,2019,20(6):1399-1407.
LI J F,LI T,JIA X P.Advances on unlocking the functional basis of PRRs family genes[J].Journal of Plant Genetic Resources,2019,20(6):1399-1407.
[24] 郑 凯,曲延英,倪志勇,等.海岛棉纤维均一化酵母双杂交文库的构建与GbTCP5互作蛋白的筛选[J].核农学报,2019,33(10):1928-1939.
ZHENG K,QU Y Y,NI Z Y,et al.Construction of yeast two-hybrid normalized cDNA library and screening of interaction proteins of GbTCP5 in Gossypium barbadense fiber[J].Journal of Nuclear Agricultural Sciences,2019,33(10):1928-1939.
[25] 刘永惠,沈 一,沈 悦,等.利用酵母双杂交系统筛选花生AhMYB44互作蛋白[J].植物遗传资源学报,2020, 21(1):201-207.
LIU Y H,SHEN Y,SHEN Y, et al.Yeast two-hybrid screening of the proteins interacting with AhMYB44 from peanut[J].Journal of Plant Genetic Resources,2020, 21(1):201-207.
[26] 范延艮,王 域,刘富浩,等.茶树Cs HIPP26.1互作蛋白的筛选与验证[J].中国农业科学,2022,55(8):1630-1641.
FAN Y G,WANG Y,LIU F H, et al.Screening and verification of CsHIPP26.1 interaction protein in tea plant[J].Scientia Agricultura Sinica,2022,55(8):1630-1641.
[27] SUN F,MA J,WANG P, et al.Genome-wide identification of the SAMS gene family in upland cotton(Gossypium hirsutum L.) and expression analysis in drought stress treatments[J].Genes,2022,13(5):860.
[28] 余愛丽,赵晋锋,王高鸿,等.玉米SAMS家族基因鉴定及其在逆境胁迫下的表达分析[J].中国农学通报,2016, 32(8):30-36.
YU A L,ZHAO J F,WANG G H,et al.Identification of SAMS genes from maize and its expression under adversity stresses[J].Chinese Agricultural Science Bulletin,2016,32(8):30-36.
[29] HE L,LI M,QIU Z, et al.Primary leaf-type ferredoxin 1 participates in photosynthetic electron transport and carbon assimilation in rice [J].The Plant Journal,2020, 104(1):44-58.
[30] YU F,LIANG K,FANG T,et al.A group VII ethylene response factor gene, ZmEREB180,coordinates waterlogging tolerance in maize seedlings[J]. Plant Biotechnology Journal,2019,17(12):2286-2298.
[31] QIN W,CHO K F,CAVANAGH P E,et al.Deciphering molecular interactions by proximity labeling [J].Nature Methods,2021,18(2):133-143.
[32] CORRATG-FAILLIE C,LACOMBE B.Substrate(un)specificity of Arabidopsis NRT1/PTR FAMILY(NPF) proteins[J].Journal of Experimental Botany,2017, 68(12):3107-3113.
[33] 贾宏昉,张洪映,刘维智,等.高等植物硝酸盐转运蛋白的功能及其调控机制[J].生物技术通报,2014(6):14-21.
JIA H F,ZHANG H Y,LIU W ZH,et al.Function and regulation mechanisms of nitrate transporters in higher plants[J]. Biotechnology Bulletin,2014(6):14-21.
[34] 徐海荣,谷俊涛,路文静,等.水稻硝酸盐转运蛋白基因 OsTNrt2.1的编码蛋白特征和表达[J].作物学报,2007,33(5):723-730.
XU H R,GU J T,LU W J,et al.Characterization and expression of nitrate transporter gene OsTNrt2.1 in rice(Oryza sativa L.) [J].Acta Agronomica Sinica,2007, 33(5):723-730.
[35] JISHA V,DAMPANABOINA L,VADASSERY J,et al.Overexpression of an AP2/ERF type transcription factor OsEREBP1 confers biotic and abiotic stress tolerance in rice [J].PloS One,2015,10(6):e0127831.
Screening of Proteins Interacting with ZmPRR73 in Maize Using Yeast Two-hybrid System
Abstract To elucidate its biological function,the bait vector PGBKT7-ZMPRR73 was constructed,and the proteins interacting with ZmPRR73 was screened from the yeast cDNA library of a tropical maize inbred line induced by long-day photoperiod treatment,using yeast two-hybrid technique.The results showed that the bait vector pGBKT7-ZmPRR73 exhibited no toxicity to yeast strains and did not activate the reporter gene on its own.A total of 12 candidate proteins that interacted with ZmPRR73 were identified.Bioinformatics analysis showed that these candidate interacting proteins were involved in various pathways,such as plant transcription regulation,ion transmembrane transport regulation,signal transduction and electron transport chain.This suggests that ZmPRR73 may participate in multiple signal transduction and metabolic pathways by interacting with the identified interaction proteins.The results enhance our understanding of the signal transduction and the regulatory pathways associated with ZmPRR73 and provide molecular evidence for further investigations into its molecular function and regulatory mechanism as a core component of circadian clock in maize.
Key words Maize; Yeast two-hybrid system; ZmPRR73; Circadian; Interacting protein

