CPEB4的表达与肿瘤临床病理特征和预后关系的生物信息学分析*
曹浩然,梁懿婷,黄金晨,张希成,施嘉怡,黄奕,何金波,李曼,余滋中,李国义
[1.湖北医药学院,湖北 十堰 442000;2.十堰市太和医院(湖北医药学院附属医院) 麻醉科,湖北 十堰 442000;3.十堰市太和医院(湖北医药学院附属医院) 耳鼻咽喉科,湖北 十堰 442000]
COVID-19 的出现导致全球范围内持续性肿瘤患者照护减少从而不可避免的出现患者死亡率增高。但是,我们可以采取更具创新性的解决办法来加强公共卫生和预防措施,辅助治疗的手段可以从传统的方法迈向基因靶向治疗与预测的阶段,因此对目标肿瘤诊断标志物的筛选与分析相关肿瘤患者的预后情况有着重要的研究目的。
CPEB4 是CPEB 家族的成员之一,定位于人染色体5q21,相对分子质量为80021,长度为7 769 bp,编码一段含729 个氨基酸残基的多肽[1]。有研究证明CPEB4通过正向调节t-纤溶酶原激活剂/PLAT的翻译,可作为促进肿瘤生长和进展的癌基因发挥作用[2]。CPEB4既可激活蛋白质翻译也可以抑制一些靶基因的翻译,CPEB4 蛋白具有促进mRNA 的翻译,影响细胞周期蛋白、细胞分裂、肿瘤细胞迁移和侵袭能力[3-4]。CPEB4结合的mRNA主要包括Ras信号通路中的相关分子、染色质重塑相关蛋白、细胞周期蛋白、凋亡相关分子、压力和炎症相关因子、代谢酶及侵袭转移相关基因等[5]。在如今越来越多的研究证明CPEB4与肿瘤的发生有着显著的相关性,CPEB4的过表达存在于许多种类的肿瘤中,例如胶质母细胞瘤(GBM)、胰腺癌(PAAD)、皮肤黑色素瘤(SKCM)、结直肠癌(CRC)、肾透明细胞癌(KIRC)、子宫内膜癌(UCEC)等均存在过表达现象[6]。研究发现GBM 中CPEB4 表达与晚期和较短的总生存期相关且CPEB4表达上调与世界健康组织(WHO)分级、卡氏功能状态评分标准(KPS)显著相关,CPEB4 过表达促进了GBM 的进展,其高表达可促进GBM 细胞的侵袭和转移,而低表达可抑制细胞的转移,故CPEB4 可能是一种更具侵袭性的GBM 表型的生物标志物[7-9]。研究发现胃腺癌(STAD)中CPEB4 低表达患者预后差于高表达患者,CPEB4还可以通过激活zeb1 介导的EMT 促进STAD 细胞的生长、侵袭和转移,从而可能为癌症患者的临床新靶点的开发提供依据[10-11]。万仁辉等[12]研究发现CPEB4 蛋白在多数CRC组织中呈阳性表达,其阳性表达可能与CRC的发生发展有一定相关性且阳性表达的CRC 患者预后较差。CPEB4在多种肿瘤中异常高表达,作为致癌因子在肿瘤生长、侵袭及血管生成中发挥作用,然而在肝癌和非小细胞肺癌中则相反[13-15]。因此需要整合33种肿瘤的突变数据和临床数据等,对其一一进行精细分析并观察CPEB4 蛋白在哪些肿瘤中有相关性亦或起正负反馈的作用。
笔者基于各领域肿瘤研究的基础上,探究CPEB4 在各个肿瘤中的临床病理特征与临床预后价值,使用Meta分析共纳入5类癌症(1种GBM、1种STAD、2 种非小细胞肺癌(NSCLC)、2 种CRC、1 种KIRC癌)分别对其临床数据进行整合,根据差异性摘选出有意义的结果并判断结局指标是否具有参考价值;在泛癌差异分析中,拓展出更多的肿瘤中CPEB4 表达含量与其之间的深层关系并判断在哪些肿瘤中与正常组织表达具有显著差异,在哪些癌症患者中具有较高的预后价值并探究是否可发展为临床肿瘤病理分级诊断指标。
1 CPEB4 关于临床肿瘤患者Meta 分析
1.1 Meta 分析资料与方法
1.1.1 文献检索途径 综合检索在PubMed、知网、万方、维普等数据库中的文献,选择”CPEB4”AND “cancer”OR “tumor”OR“neoplasm”OR“phyma”遵循《系统综述和荟萃分析优先报告条目》(PRISMA)[2]系统回顾,并手动查阅了相关的参考文献以便精确化纳入研究。
1.1.2 纳入与排除标准 纳入CPEB4 与癌症相关的研究,根据文章是否有足够的临床患者病理情况的数据并遵循随机对照试验作为标准,导入文献管理器筛选并删除各数据库的重复与缺失临床相关数据的文献。
1.1.3 数据提取与质量评价 由两人对16 篇文章提取出文章第一作者、国籍、发表年限、肿瘤类型、患者性别、患者数量、肿瘤分化程度、肿瘤情况、淋巴结转移程度以及具体的TNM 分期进行筛选统计,如果出现分歧由第三方仲裁决定。为实现Meta 生存分析,对生存资料提取,获取95%CI 的上下限和LogHR 值,计算出SE 标准值汇入表中。使用Engauge Digitizer10.8 软件汇总相关肿瘤患者的生存(KM)曲线,直观检测出CPEB4 是否为一个良好的预后因素。采用Cochrane 评估工具从以下六个维度评估每个RCT的偏倚风险:随机序列的产生、分布隐藏、盲性、结果数据不完整、选择性报告和其他偏倚[16]。
1.1.4 文献筛选流程及纳入结果 在数据库中搜索出194 篇与CPEB4 和肿瘤相关的文献,筛重后共排除132 篇重复文献,对摘要、文章内容、临床数据进行审查后通过定性定量合成最后获得16 篇文章纳入Meta分析(图1),包括1 949例肿瘤患者进行荟萃分析,研究的癌症类型包括:KIRC、NSCLC、肺鳞状细胞癌(LUSC)、GBM、CRC、结肠腺癌(COAD)、STAD。
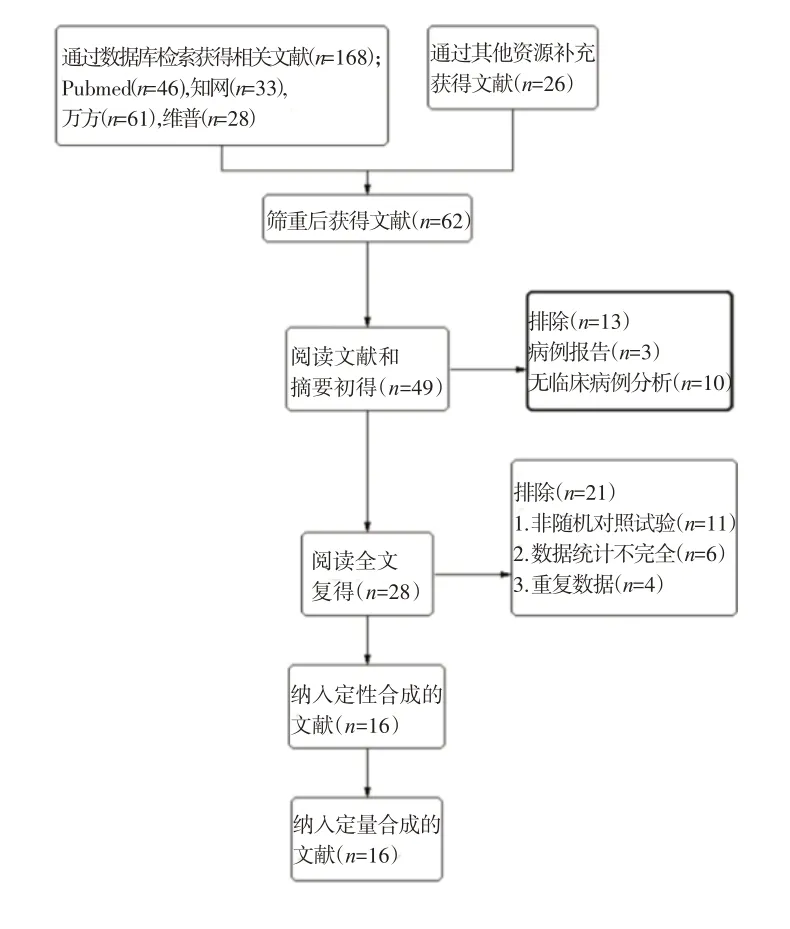
图1 文献纳入流程图
1.2 Meta 分析研究结果
1.2.1 统计学分析 采用干预性系统评价中二分类变量对患者的肿瘤分化程度(图2A)、淋巴结转移情况(图2B)、肿瘤直径(图2C)、TNM 分期(图2D)与CPEB4 表达的关系进行定量合成,四组分析中控制组与对照组差异有统计学意义(P<0.05),均以(比值比)作为效应量。用Review Manager 5.4 对数据进行荟萃分析,结果以森林图进行展现,使用I2统计量和卡方检验[5]评估异质性作为异质性评价标准,采用固定效应模型对单项研究合并效应量及其相应可信区间,若存在明显异质性(I2>50%且P<0.05),则采用随机效应模型。结果以P-value<0.05 为差异有统计学意义。为了评判是否具有发表偏倚,对纳入10 篇文献以上的研究[6]采用Begg's 和Egger's 检验对漏斗图的对称性进行定量分析。
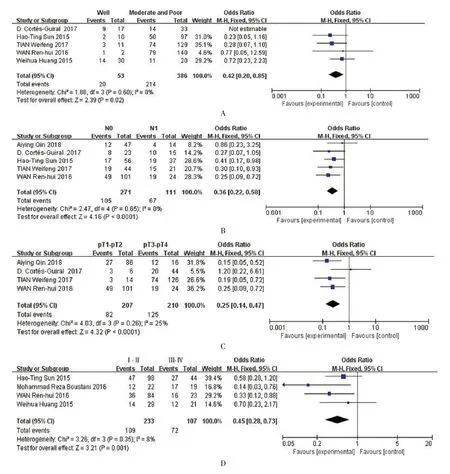
图2 CPEB4 与肿瘤分化程度、淋巴结转移、肿瘤大小、TMN 分期的Meta 分析
1.2.2 CPEB4 表达与预后的关系 结果显示CPEB4 在肿瘤高分化和中分化、同侧淋巴结转移、肿瘤最大径>5 并侵袭任一器官、TNM 分期为Ⅲ或Ⅳ分期表达含量较高,且结果属于低异质性(I2<30%)具有较强可靠性。研究结果均为总体生存期(OS),利用Engauge Digitizer10.8 软件从图形中提取出七篇原始数据并绘制出KM 总生存曲线,以风险比值()作为时间事件结局指标,可观测120 月内CPEB4 高表达患者较低表达患者生存人数较少(图3),CPEB4 为不良预后因素,但相关的研究临床指标较少、文献数量有限,结果还有待验证。

图3 CPEB4 表达与患者总生存的Meta 分析(HR)与生存曲线(OS)
1.3 肿瘤的单个差异分析验证
提取癌症基因组图谱(TCGA)数据库中的KIRC、CRC、GBM、STAD、NSCLC的临床样本,观察CPEB4 表达含量在正常组样本和肿瘤组样本中的差异情况,验证Meta分析的结果,5类数据皆显示在正常样本中基因表达含量呈上调趋势(含量高)且差异有统计学意义(P<0.05)(图4)。
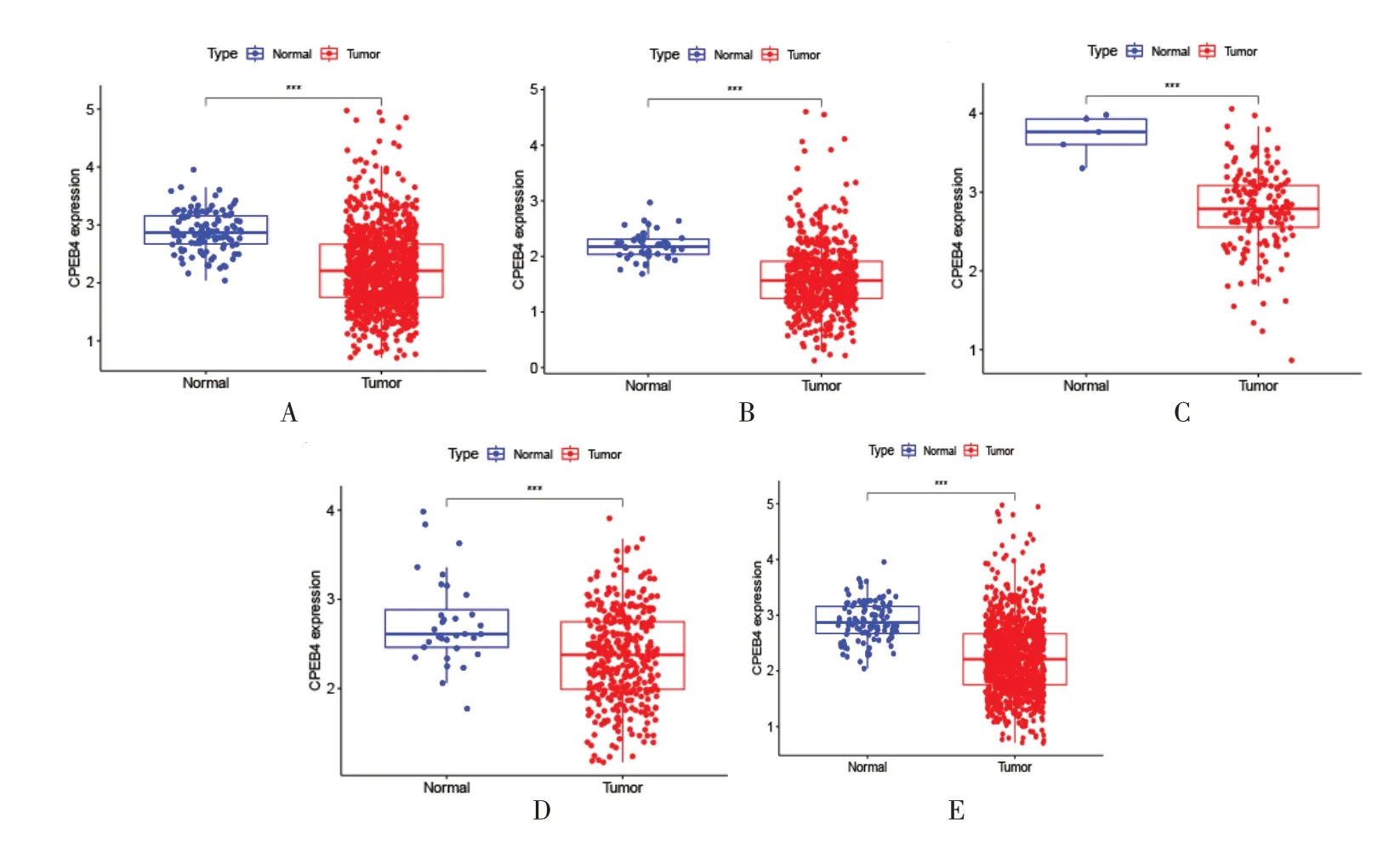
图4 CPEB4 在肿瘤组与相应癌旁组中差异对比情况
1.4 小结
综上所述,荟萃分析结果显示CPEB4 呈高表达是不利于晚期的KIRC、CRC、GBM、STAD、NSCLC 患者预后的因素(=3.35;95%CI:2.69~4.18;P<0.0001);提取出文章中五个肿瘤信息,进行单个肿瘤差异分析(图4),结果显示在所有正常样本中CPEB4 都呈高表达而肿瘤样本中表达含量较少,但是对CPEB4于患者不同临床分级和与免疫相关的表达尚且未知。为更深入的研究,我们从TCGA 数据库中提取33 种癌症的样本对CPEB4 进行泛癌分析,对CPEB4 在不同肿瘤中的微观分析与相关性比较。
2 TCGA 数据对CPEB4 泛癌分析
2.1 材料与方法
2.1.1 对数据库信息进行提取与整理 以33 种类型癌症为实验对象,提取TCGA 数据库与UCSC Xena 数据库中临床生存数据、肿瘤样本中转录组基因表达数据和突变的数据,筛选掉不符合规定和缺失的数据,汇总出33 种肿瘤样本与正常样本的对照数据,接着从各肿瘤表达中将目标基因表达含量提取出,为后期分析做准备。
2.2 泛癌分析研究结果
2.2.1 CPEB4 泛癌差异分析 通过提取的基因表达数据对33 个肿瘤样本与正常样本中CPEB4 表达量绘制出差异图(图5)。其中膀胱移行细胞癌(BLCA)、乳腺浸润性癌(BRCA)、胆管癌(CHOL)、COAD、食管癌(ESCA)、GBM、头颈部鳞状细胞癌(HNSC)、KIRC、肾乳头状细胞癌(KIRP)、肝细胞癌(LIHC)、LUAD、LUSC、前列腺腺癌(PRAD)、直肠腺癌(READ)、STAD、甲状腺癌(THCA)、UCEC组织中CPEB4 呈下调趋势,表达低于正常组织;此外与正常组织相比,肾嫌色细胞癌(KICH)中CPEB4 在肿瘤组织中呈上调趋势,表达含量较高。以上结果表明,CPEB4在肿瘤组织与正常组织中表达含量差异有统计学意义(P<0.05)。初步证实CPEB4 既可成为部分肿瘤的抑癌因子也可成为部分肿瘤的癌因子。
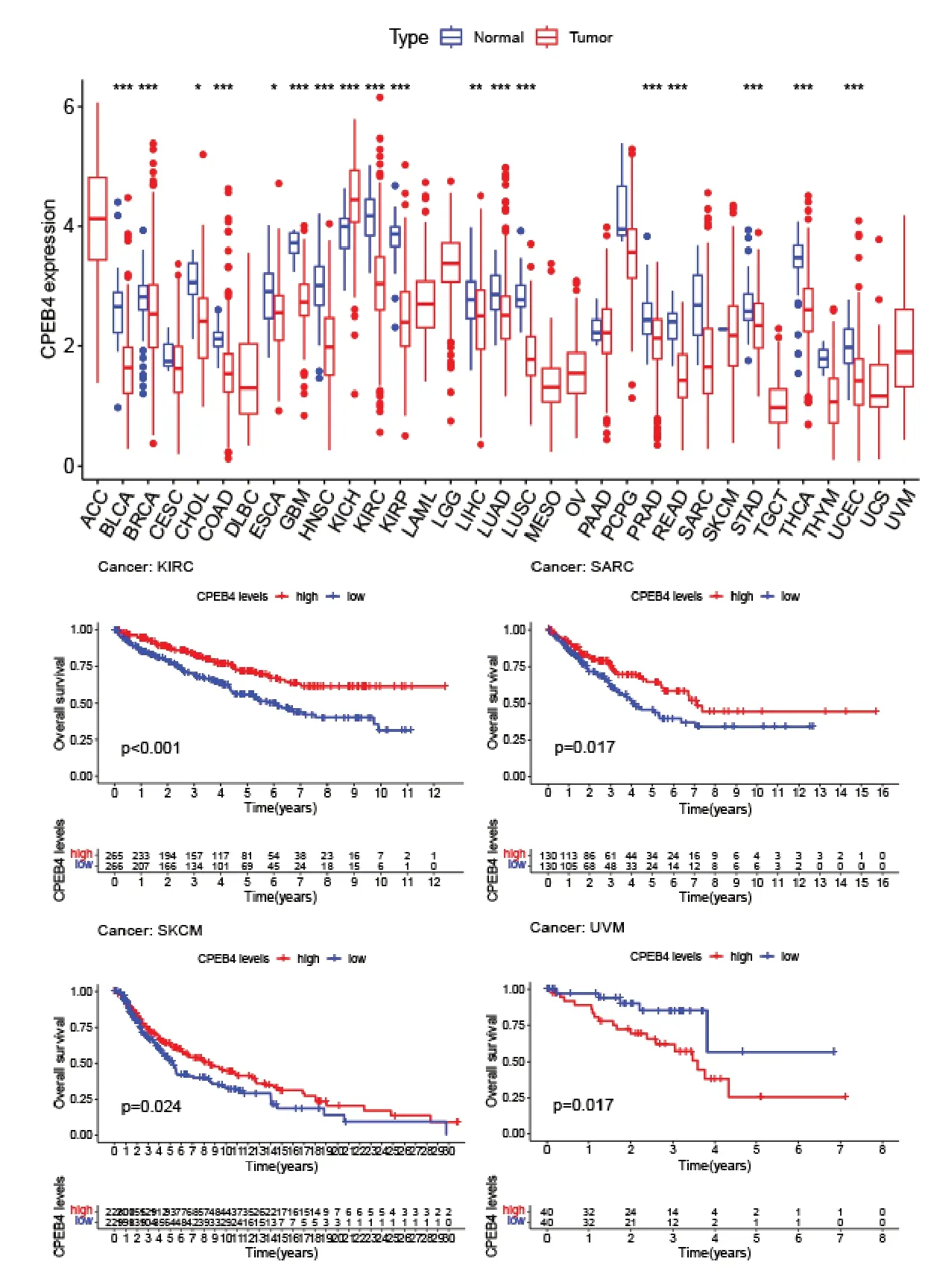
图5 CPEB4 在泛癌中表达状况与生存曲线
2.2.2 泛癌中CPEB4 表达与生存分析 对33 类癌症临床生存数据进行筛选,剔除掉生存数据信息缺失的患者,以中位基因表达为界限将患者划分为高表达、低表达两组,根据已有的随访总生存数据进行生存分析,筛选出高低表达差异有统计学意义(P<0.05)的癌症患者数据。如图5,在KIRC、肉瘤(SARC)、皮肤黑色素瘤(SKCM)中CPEB4 低表达的患者较低生存率较短;而葡萄膜黑素瘤(UVM)中CPEB4 高表达患者有着较低的生存率。
为确定数据的准确性,在Kaplan-Meier Plotter数据库中,对泛癌进行生存数据绘图。挑选出CPEB4高低表达差异无统计学意义(P>0.05)的癌症患者的生存曲线(图6)。BLCA、LUSC、ESCA、THCA中CPEB4 高表达不利于患者生存;卵巢癌(OV)、KIRC中CPEB4低表达不利于患者生存。
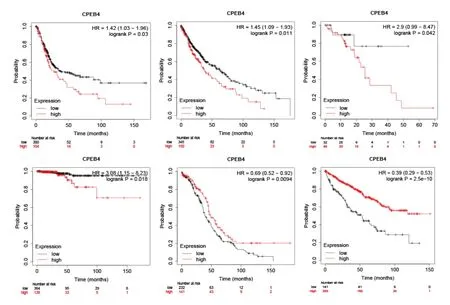
图6 Kaplan-Meier Plotter 数据库中生存曲线
为进一步验证基因表达与患者生存相关的准确性,用Cox 分析观测CPEB4 与患者生存状态、较低生存率的相关性,森林图对值可视化。CPEB4 在UVM、LUSC 中为高风险基因(>1);SKCM、SARC、KIRC、肾上腺皮质癌(ACC)为低风险基因(<1)。综上所述,在UVM、BLCA、LUSC、ESCA、THCA的患者中CPEB4高表达为一不利预后因素;CPEB4高表达于OV、KIRC、SARC、SKCM患者为一良好的预后因素(图7)。

图7 Cox 分析(OS)
2.2.3 泛癌中CPEB4 表达与临床相关性分析 从TCGA 数据库提取纳入研究的临床数据,包括患者的年龄、性别、临床分期、肿瘤大小、淋巴管浸润程度,进行临床相关性分析,设定P<0.05 为有意义的观测数据。可发现女性的CPEB4 表达量在HNSC、胸腺瘤(THYM)显著比男性高(图8A);BLCA、BRCA、HNSC、KIRC、THCA 中的StageⅠ患者的基因表达量大多高于其他分期且差异有统计学意义(P<0.05),CPEB4 大量存在于早期患者肿瘤细胞中(图8B)。
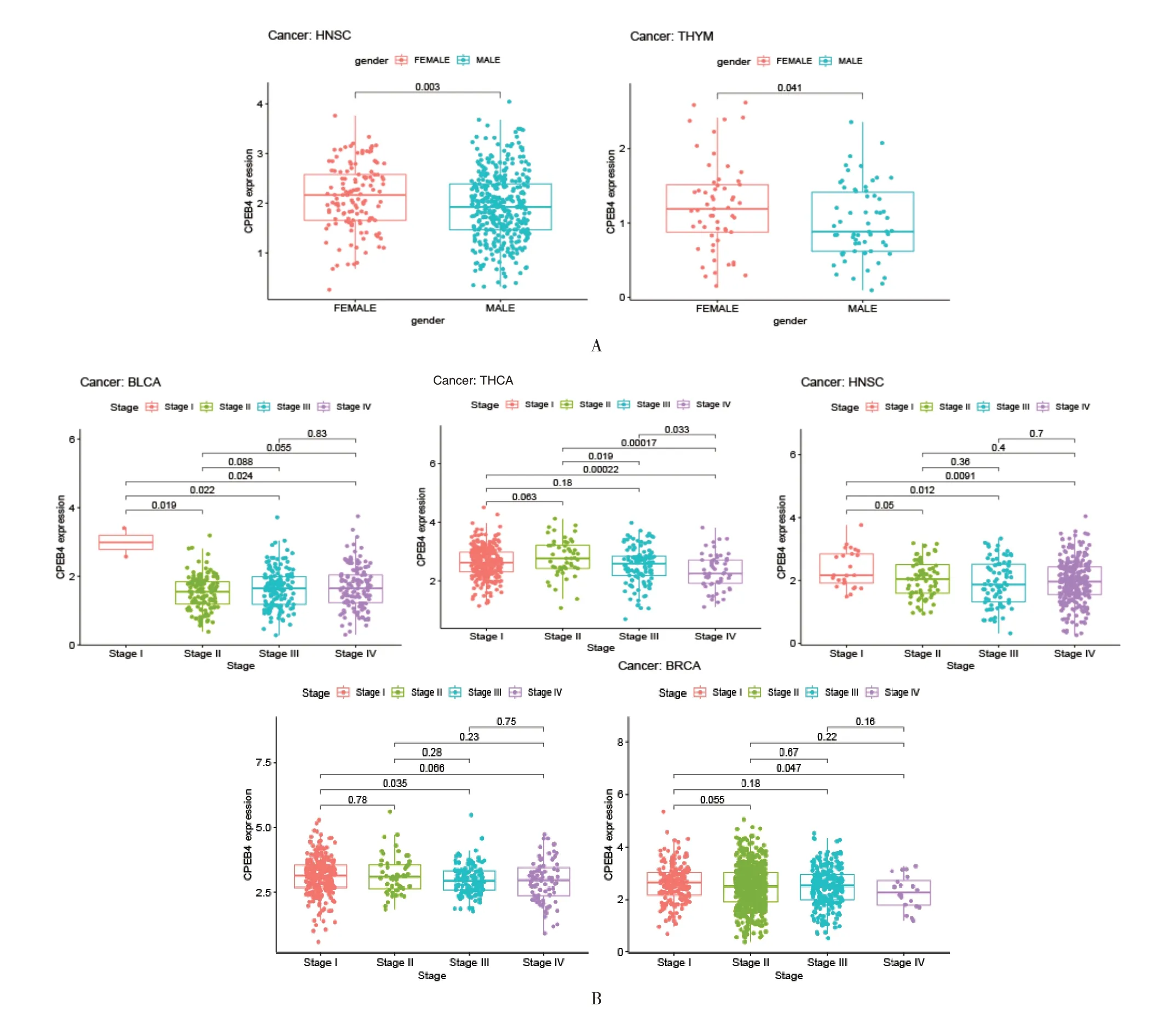
图8 CPEB4 表达与临床相关性分析
2.2.4 泛癌中CPEB4 表达与肿瘤突变负荷(TMB)的关系 TMB定义为肿瘤基因组编码区体细胞突变的总数,从几个到成千上万[17]。近年来发现TMB 可用于预测免疫治疗疗效的独立生物标记物,在免疫治疗中的预测能力不仅限于NSCLC、SKCM 等“热点肿瘤”,还能作为其它癌症的生物标志物[18]。本研究旨在泛癌中分析CPEB4 表达与肿瘤突变的关系,结果以雷达图显示(图9A)。其中以BLCA、BRCA、HNSC、LUAD、LUSC、胰腺癌(PAAD)、STAD、THYM 肿瘤中碱基数显著突变,仅THYM 中基因表达与肿瘤突变负荷呈正相关,对免疫检查点抑制的应答反应较好,可更好的预测免疫治疗的疗效。
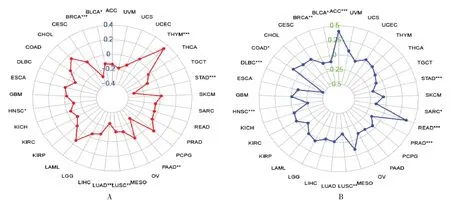
图9 肿瘤突变负荷与肿瘤MSI 微卫星不稳定分析
2.2.5 泛癌中CPEB4 表达与微卫星不稳定性(MSI)相关性分析 MSI 在大多实体瘤中为一个重要的分子生物标志物,有研究表明MSI 肿瘤微环境中淋巴细胞的浸润增多,MSI 肿瘤患者能够获得更好的临床缓解,延长较低生存率[19-20]。MSI肿瘤因存在高度突变的潜质,突变基因编码翻译的蛋白可以成为有潜力的肿瘤抗原而被免疫系统识别并清除[21],故推测MSI 可能与肿瘤微环境中免疫细胞相关并可预测免疫治疗疗效。如图9B,在ACC、COAD、READ 中CPEB4 表达与MSI 呈正相关,说明在这些实体瘤中CPEB4 表达量越高肿瘤基因越有利于突变成肿瘤抗原基因;在BLCA、BRCA、DLBC、HNSC、LUSC、PRAD、SARC、STAD 中CPEB4 呈高表达不利于肿瘤的抗原突变。
2.2.6 泛癌中CPEB4 表达与肿瘤微环境的相关性分析 肿瘤微环境在肿瘤细胞生长中具有促进的作用但肿瘤微环境中也存在免疫细胞影响着癌细胞的增殖与扩散[22]。肿瘤细胞可以调控免疫细胞的代谢,进一步影响免疫细胞发育、分化与功能发挥,使其往促肿瘤型转化,极大限制了抗肿瘤免疫活性。因此对肿瘤微环境的探究显得十分重要。通过分析免疫细胞和基质细胞的含量可预测非肿瘤细胞的浸润。验证肿瘤微卫星不稳定是否易被免疫系统识别,对22 种免疫细胞与基因表达关系进行分析,筛选出与基因表达相关的免疫细胞(图10)。

图10 免疫细胞相关性分析
因此验证出CPEB4 在肿瘤中与免疫细胞相关性,BRCA、COAD、LIHC、LUAD、STAD、THCA、THYM、GBM等在CPEB4高表达时易被免疫系统识别,证明CPEB4在上述肿瘤中对肿瘤生长扩散有抑制作用;UVM、SKCM、TGCT、PRAD、ACC、BLCA、CHOL、CESC、LUSC、KIRC、HNSC 等在CPEB4 低表达时易被免疫细胞识别并抑制肿瘤生长扩散。
2.2.7 泛癌中CPEB4 与免疫和肿瘤基因的共表达分析 为更确切的说明CPEB4 在肿瘤中的表达量与免疫相关,筛选出47 个免疫相关基因,对其共表达分析(如图11)。在UVM、THYM、UCEC、TGCT、SKCM、READ、PRAD、LUSC、LUAD、LAML、DLBC、COAD 中CPEB4 与免疫基因表达量呈正向调控;THCA、STAD、THCA、STAD、SARC、PCPG、PAAD、LIHC、LGG、KIRC、KICH、HNSC、GBM、BRCA、BLCA中CPEB4与免疫基因呈负向调控关系,不利于机体对肿瘤的免疫。进一步证明CPEB4 参与肿瘤免疫系统的调控。
2.2.8 GSEA 富集分析结果 运用R 语言包提取GSEA 数据库中对于GO 富集的数据,筛选出GO 富集功能相近的前5 个通路,对差异有统计学意义(P<0.05)的通路进行可视化且校正FDP(P<0.25),因此CPEB4 通过五个通路功能调节每个肿瘤的发生。在STAD、OV、SKCM、LUAD、COAD、BRCA中与CPEB4 表达正相关的基因较活跃,通路峰值向上,说明基因表达越高功能越活越;GBM、LGG、ACC 中与基因表达负相关的差异基因富集在通路上,故CPEB4高表达时功能通路越沉默(图12)。
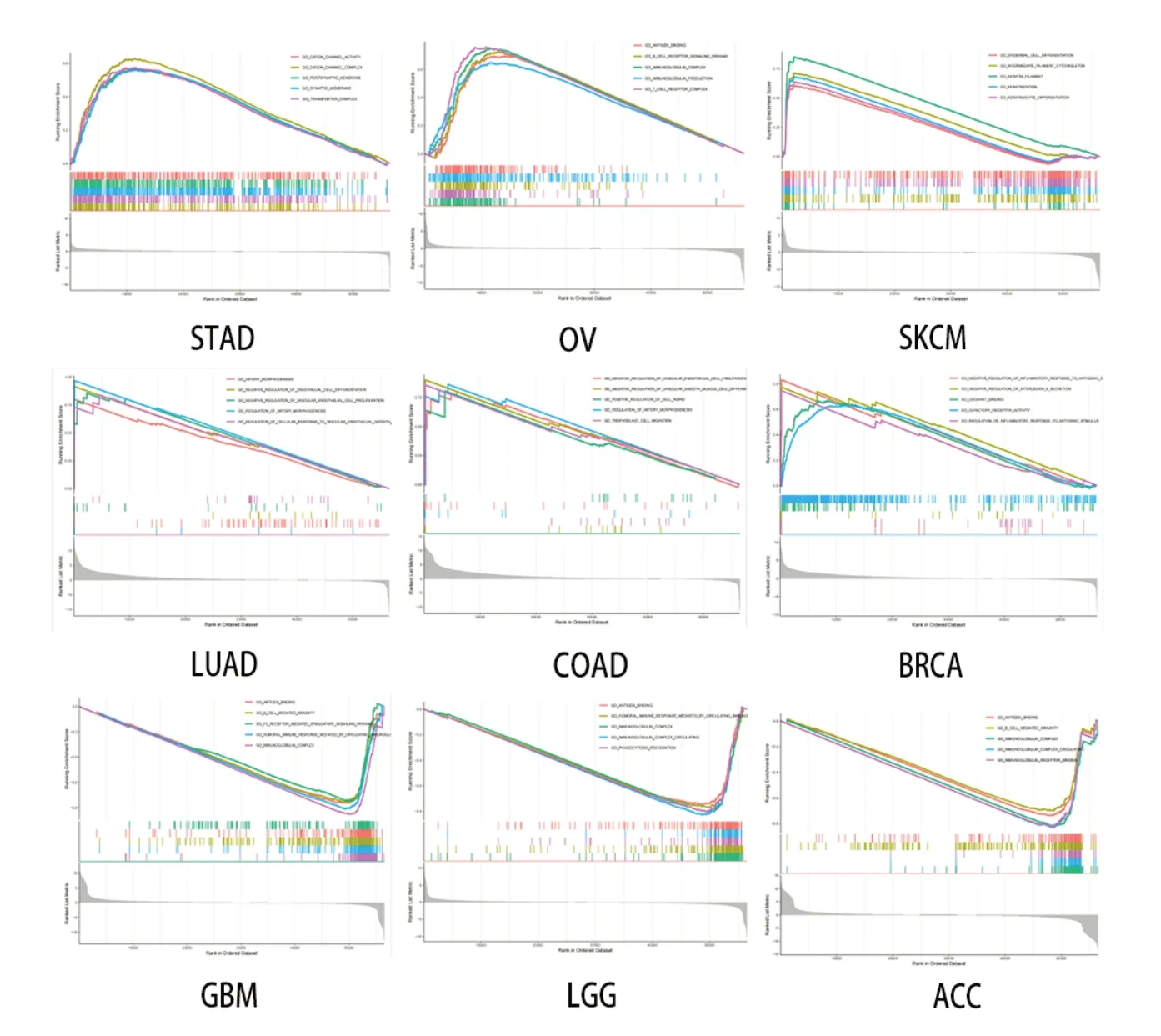
图12 CPEB4 泛癌GSEA 富集通路
3 CPEB4 蛋白表达于正常细胞的位置
通过HPA 数据库中特异性抗体与细胞中CPEB4 蛋白的免疫荧光染色,检测到CPEB4 蛋白主要定位于核质体、高尔基体,其次定位于囊泡中(图13),故猜测CPEB4 在正常组织的细胞中可能参与细胞基因表达、蛋白质的加工运输,调控细胞内蛋白正常表达。

图13 对抗体、细胞核、微管、内质网进行免疫荧光染色并观察
4 结论
通过以上对CPEB4相关临床、基因突变等数据的整理与Meta 泛癌联合分析。Meta 分析中发现CPEB4上调表达在KIRC、NSCLC、GBM、CRC、STAD的患者中,并且成为一个不利的预后因素。由于CPEB4在癌症中扮演矛盾的角色,例如胰腺导管腺癌(PDAC)和GBM 中的致癌启动子,但HCC 中的肿瘤抑制因子[23]。为验证CPEB4 表达在早期样本上调,但在晚期癌症中降低的结论,笔者采用单个肿瘤差异分析强化荟萃分析的结果,都显示在早期样本CPEB4 蛋白含量高于晚期肿瘤样本。为了更进一步探究CPEB4在肿瘤中的临床价值,笔者实施泛癌肿瘤突变负荷与免疫相关的分析并与免疫相关基因共表达分析,结果统计仅在ACC、READ、THYM、BRCA、COAD、LIHC、LUAD、STAD、THCA、GBM、UVM、UCEC、睾丸生殖细胞肿瘤(TGCT)、SKCM、PRAD、LUSC、急性髓性白血病(AML)、弥漫性细胞淋巴瘤(DLBCL)中CPEB4 蛋白高表达有助于增高免疫细胞的数量(尤在THYM、COAD、LUAD、READ 中最为显著)。在肿瘤分级系统中显示CPEB4低表达会伴随着肿瘤分级越高,意味着患者的危险因素随之上升。综上所述CPEB4 可作为上述肿瘤患者良好的预后因素。

