基于EST-SSR标记的沙棘品种鉴定及指纹图谱构建
赵雨欣,张哲文,考惠霞,孙永江,辛智鸣,赵 喆,董树斌①,程 瑾② (.北京林业大学生物科学与技术学院/ 林木育种与生态修复国家工程中心/ 花卉种质创新与分子育种北京市重点实验室,北京 0008;.北京林业大学林学院/ 森林培育与保护教育部重点实验室,北京 0008;.中国林业科学研究院沙漠林业实验中心,内蒙古 磴口 0500)
我国沙棘(Hippophaerhamnoides)天然资源和人工种植面积均居世界首位,截至2018年我国沙棘种植总面积约占全球沙棘种植总面积的93%,截至2022年我国83%的人工沙棘林分布于内蒙古、山西、甘肃、青海和陕西5个省(区)[1-2]。沙棘具有适应性强、耐寒、耐旱、耐风蚀、抗盐碱、防病虫害和根系较发达等特性,常被视为水土保持、气候调节和沙漠治理的重要先锋树种,在我国三北地区得到大范围推广和种植[3]。此外,沙棘为药食同源植物,其各个器官,尤其是果实、根、茎、叶中都富含营养物质和生理活性物质等[4],如维生素、类胡萝卜素、有机酸、不饱和脂肪酸和一些必需氨基酸等,在饮食品、医药和保健品等领域得到广泛利用,具有良好的经济价值和生态效益[5]。
近几年,随着我国对沙棘价值的持续关注,杂交育种、分子育种等技术在沙棘品种选育中得到了广泛应用,再加上国外优良沙棘种质资源的不断引进,出现了一大批亲缘关系较近,生物学特性、生态适应性、品质性状良莠不齐的品系,给沙棘的良种鉴定和审定,种苗的保存、生产和管理,沙棘产业的发展和水土保持重要品种的选择带来了困扰。鉴于上述情况,传统的形态学、细胞学鉴定方法已不能很好满足对沙棘品种进行快速准确鉴定和研究的需求,孙燕琳等[6]通过筛选葡萄的SSR引物对沙棘遗传多样性进行分析,赵春娥等[7]利用相关序列扩增多态性(sequence-related amplified polymorphism,SRAP)进行沙棘聚合酶链式反应(PCR)程序的优化和引物筛选,但这两种标记都存在引物挑选和组合的局限性;邵珊珊[8]利用随机扩增多态性(random amplified polymorphic,RAPD)对沙棘品种进行鉴定,但RAPD由于没有DNA探针等原因造成结果稳定性差,可重复率较低。
简单重复序列(SSR)又称微卫星序列标记(MS)或短串联重复标记(STR),是以PCR为基础的DNA分子标记技术[9]。SSR具有高度重复性、丰富多态性、共显性和高度可靠性等优点[10],被广泛应用于梨[11]、杨树[12]和建兰[13]等植物品种鉴定、遗传多样性研究及核心种质评价等相关领域。SSR可分为两类:分布在整个基因组序列中的基因组SSR(gSSR)和嵌入转录序列中的表达序列标签SSR(EST-SSR)[14]。EST-SSR具有易操作、易重复、准确度较高和变异较丰富的优点,具有很好的种间传递性,且出现无效等位基因的概率小[15-16]。不仅如此,由于EST-SSR是在基因组的编码区域中被识别的,与gSSR相比,EST-SSR的使用成本相对较低,已广泛应用于很多无参考基因组的物种[17]。目前,EST-SSR多用于植物遗传多样性分析、遗传图谱构建和品种鉴定等研究工作。
“实优1号”是我国在20世纪80年代后期从芬兰引进的沙棘中选育的新品种,生长速度快,产量高,沙棘油含量高,有较好的抗寒、抗旱、耐盐碱及抗干缩病能力[18-20],在我国多个省份得到广泛栽培。笔者研究对沙棘品种“实优1号”的转录组数据进行分析,挖掘出大量EST-SSR序列,使用微卫星识别软件(microsatellite identification tool,MISA)识别SSR序列并根据其序列特征进行引物设计,结合TP-M13-SSR毛细管电泳技术筛选出一套SSR多态性较高的引物,对收集的沙棘材料进行亲缘关系分析和指纹图谱构建,为沙棘优良品种选育提供参考。
1 材料与方法
1.1 材料的采集与处理
供试的42份沙棘材料由国家林业和草原局国有林场和种苗管理司、中国林业科学研究院沙漠林业实验中心、山西省林业和草原科学研究院、山西省桑干河杨树丰产林实验局等多家单位提供(表1[18-31])。试验材料为沙棘的无病虫害健康叶片、枝条等,以幼嫩叶片为优(指位于沙棘植株枝条上部,刚发育形成、尚未完全开展或刚开展的叶片),每份材料至少随机抽取3个单株,采样后置于装有变色硅胶的自封袋中备用(变色硅胶量没过样品即可)。

表1 42份沙棘品种信息[18-31]Table 1 Information of 42 Hippophae rhamnoides varieties
1.2 DNA提取
利用自动磨样机(MiniBeadbeater-96)结合天根生物公司DP320-02试剂盒提取DNA,通过琼脂糖凝胶电泳和分光光度计(NanoDrop 2000,Thermo Scientific)检测DNA质量、浓度和纯度,样品D260/D280值以1.7~1.9之间为宜,于-20 ℃条件下储存。
1.3 EST序列来源
采集“实优1号”新鲜的幼嫩叶片用于RNA提取和cDNA文库构建。经文库质检后使用Illumina NovaSeq 6000(Illumina,USA)S4套组组件,利用Illumina Hiseq 2000平台(Illumina,USA)进行高通量测序,将所得序列利用Trinity[32]拼接成独立基因集,即一个由EST序列拼接成的转录组。
1.4 SSR位点识别及引物合成
利用MISA(http:∥pgrc.ipk-gatersleben.de/misa/misa.html,默认参数)查找沙棘EST-SSR位点,利用Primer 3(version 2.3.4)设计引物。对原有上游引物5′端进行M13(5′-TGTAAAACGACGGCCAGT-3′)修饰,然后合成3′端具有荧光的M13接头〔6-羧基荧光素(6-carboxy-fluorescein,FAM)、六氯-6-甲基荧光素(6-hexachloro-fluorescein,HEX)、甲基-X-罗丹明(6-carboxy-x-rhodamine,ROX)、6-NED炔烃(6-NED alkyn,NED)〕,通过序列互补进行标记,检测PCR扩增产物。
1.5 PCR扩增及SSR基因分型检测
25 μL的PCR反应体系:2×TaqPCR预混试剂Ⅱ 17.5 μL,10 μmol·L-1正、反向引物各1.0 μL,10 μmol·L-1M13荧光标记引物0.5 μL,基因组DNA 0.8 μL,ddH2O 4.2 μL。PCR反应程序:94 ℃预变性5 min;94 ℃变性30 s,60 ℃退火30 s(每个循环降低1 ℃),72 ℃延伸30 s,共6个循环;94 ℃变性30 s,54 ℃退火30 s,72 ℃延伸30 s,共30个循环;72 ℃延伸10 min,12 ℃保存。采用ABI-3730XL基因分析仪(Applied Biosystems,Foster City,CA)对扩增PCR产物进行毛细管电泳,检测荧光信号和位点出峰情况。
1.6 数据统计及分析
利用28对引物对42份沙棘材料进行多态性分析,用GeneMarker(version 2.2.0)(SoftGenetics,USA)读取毛细管电泳产物大小,根据产物大小对数据进行判读和整理,记录结果时,纯合位点的等位变异数据记录为X/X,其中X为该位点等位变异的大小;杂合位点的等位变异数据记录为X/Y,其中X、Y分别为该位点上两个不同的等位变异,小片段数据在前,大片段数据在后,采用Excel对相关结果进行统计;利用Convert(version 1.31)进行格式转换,再利用Popgene(version 1.32)和Cervus(version 3.0.7)计算每对引物的遗传多样性参数,比较引物多态性;利用OriginPro 2010(version 9.8.0.200)进行Pearson相关分析;利用NTSYSpc(version 2.10e)分析遗传相似性系数,绘制UPGMA聚类树状图[33],并基于NTSYSpc(version 2.10e)软件中的MxComp功能分析相似性系数间的相关性,评价系统树状图的质量,分析品种间的亲缘关系;最后优选引物组合鉴定所有品种,将所选引物的多态性位点数(n)代入公式(P=1/2n)计算相同指纹图谱出现的概率(P)[34]。
2 结果与分析
2.1 沙棘转录组的产出及质量检测
转录组测序结果显示,“实优1号”获得的原始序列数(raw reads)为22 456 907条,对原始数据进行过滤、质控后获得高质量干净读长(clean reads)为21 738 111条,占原始序列的96.80%;碱基质量值Q20为97.46%,碱基质量值Q30为92.91%;序列拼接后GC含量为41.87%。以上数据表明测序数据可靠,品质较好,可进行下一步分析。
2.2 沙棘转录组SSR位点信息及组成
利用MISA识别“实优1号”转录本的unigene序列,检测到总序列数为25 286条,总序列长度为33 604 162 bp;对SSR位点进行搜索发现含SSR的序列为5 205条,SSR位点总数为6 196个,其中复合型(C型)SSR位点仅为447个,占位点总数的7.21%;完全重复型(P型)SSR位点为5 749(92.79%)个。
沙棘转录组中SSR位点信息见表2。

表2 沙棘转录组中SSR位点信息Table 2 Information of SSR loci in transcriptome of Hippophae rhamnoides
表2显示,在沙棘P型SSR位点中,单核苷酸重复型、二核苷酸重复型和三核苷酸重复型为主要重复类型,其中单核苷酸重复型基序为3 019个,在总位点数中占比最高,为48.72%。对P型SSR位点进行搜索共发现182种基序,(A/T)n占绝对优势,共3 003个,占此重复基序的99.47%,占总位点数的48.47%;二核苷酸重复型中(AC/GT)n最少,仅占总位点数的0.44%;三核苷酸和四核苷酸重复型中,(GAA/TTC)n和(AAAG/CTTT)n出现频率较高;五核苷酸和六核苷酸重复型中,主要基序分别为(AAAAG/CTTTT)n和(TCTGCT/AGCAGA)n。
2.3 沙棘转录组SSR位点基序重复次数
沙棘转录组SSR位点中,不同基序类型SSR重复次数情况(图1)显示,单、二和三核苷酸基序重复次数集中在10~15、6~10和5~7次;四、五和六核苷酸基序重复次数集中在5~6次。其中,当重复次数为10时,SSR位点数为1 726个,占总SSR位点数比例最高(27.86%);5次和6次重复次数的SSR位点数分别为630(10.17%)和637个(10.28%);当重复次数为20时,SSR位点数占比最少,仅21个(0.34%)。上述结果也表明,SSR位点中不同重复类型的频率,随重复次数的增加而减小。
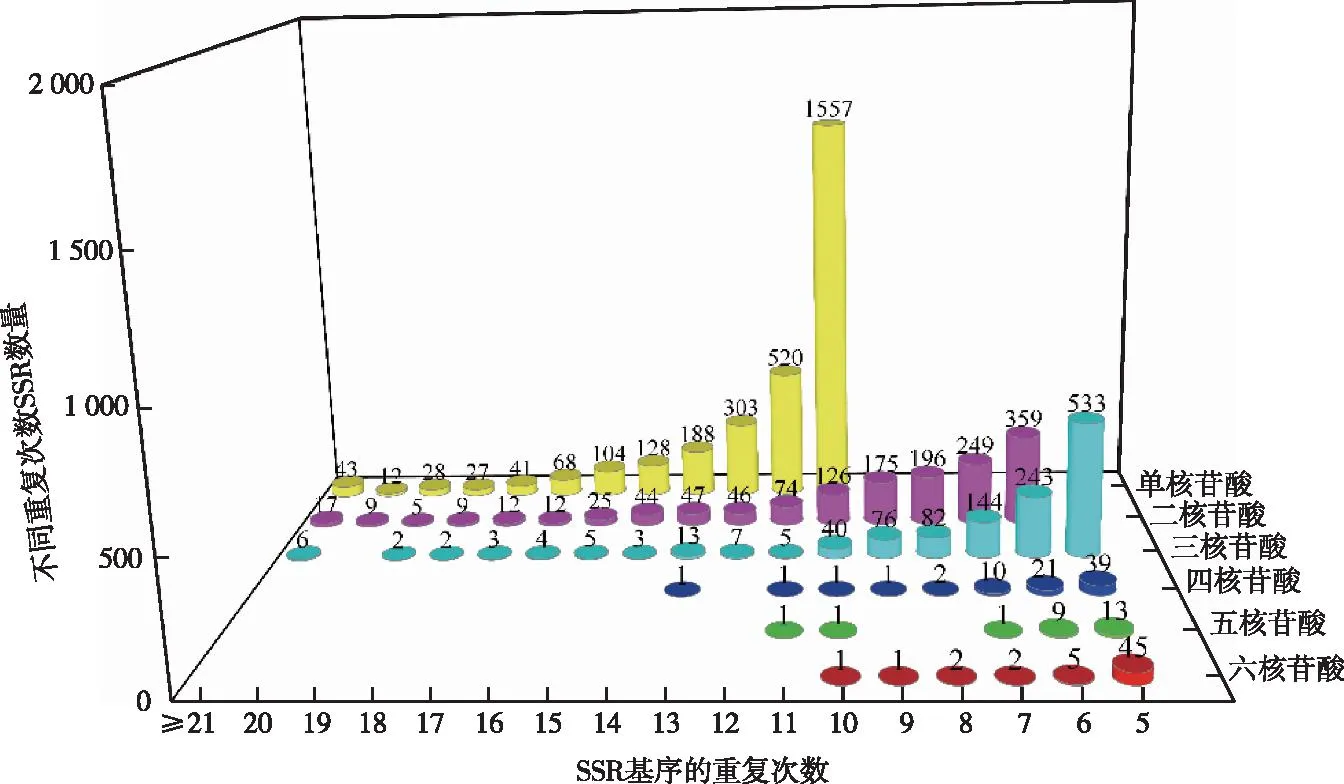
图1 转录组中SSR位点的基序重复次数分布Fig.1 Distribution of repeat numbers of SSR motifs in transcriptome
2.4 沙棘转录组SSR序列长度及变异
沙棘转录组SSR序列长度为10~264 bp,平均长度为19.05 bp。SSR序列中10 bp的序列数量最多,占总位点数的25.13%(图2)。
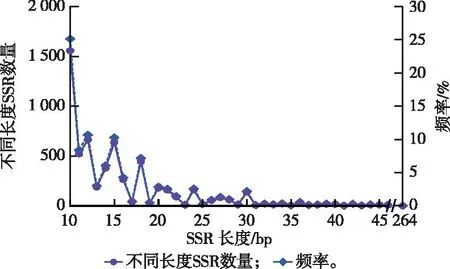
图2 沙棘转录组不同SSR序列长度的出现频率Fig.2 Frequency of SSR with different lengths in transcriptome of Hippophae rhamnoides
P型SSR序列中,基序长度变异情况非常丰富,同时随着不同重复型SSR长度的增加其序列出现频率也逐渐降低(图3)。单核苷酸重复的长度变化最大,为81 bp,平均长度为11.56 bp(表2);(C/G)n与(A/T)n平均长度分别为12.50和11.55 bp;二核苷酸重复的各基元类型中,(AC/GT)n平均长度为14.22 bp(最短),(AG/CT)n平均长度为18.25 bp(最长)。Pearson相关分析表明重复单元长度与SSR平均长度呈正相关(P<0.005)。

图3 不同重复单元SSR序列长度的变异情况Fig.3 Variation in sequence length of different types of SSR
2.5 沙棘SSR引物扩增产物的多态性及遗传多样性参数
根据转录组数据分析获得沙棘SSR位点信息,利用6 196个SSR位点设计出4 681对SSR引物,设计成功率为75.55%。为了验证SSR的有效性,随机选取不同类型SSR位点进行验证,从扩增成功的引物中选取28对多态性好的引物。利用这28对引物在42份沙棘品种中检测出193个等位基因位点,每个位点的等位基因数(Na)为3.000~13.000,均值为6.964。这些沙棘品种的有效等位基因数(Ne)为1.849~6.380,均值为3.495;多态性信息含量(PIC)为0.398~0.824,均值为0.623;Shannon信息指数(I)最大值为2.071,最小值为0.779,均值为1.384;观测杂合度(Ho)和期望杂合度(He)分别为0.310~0.857和0.459~0.843,均值分别为0.617和0.671(表3)。
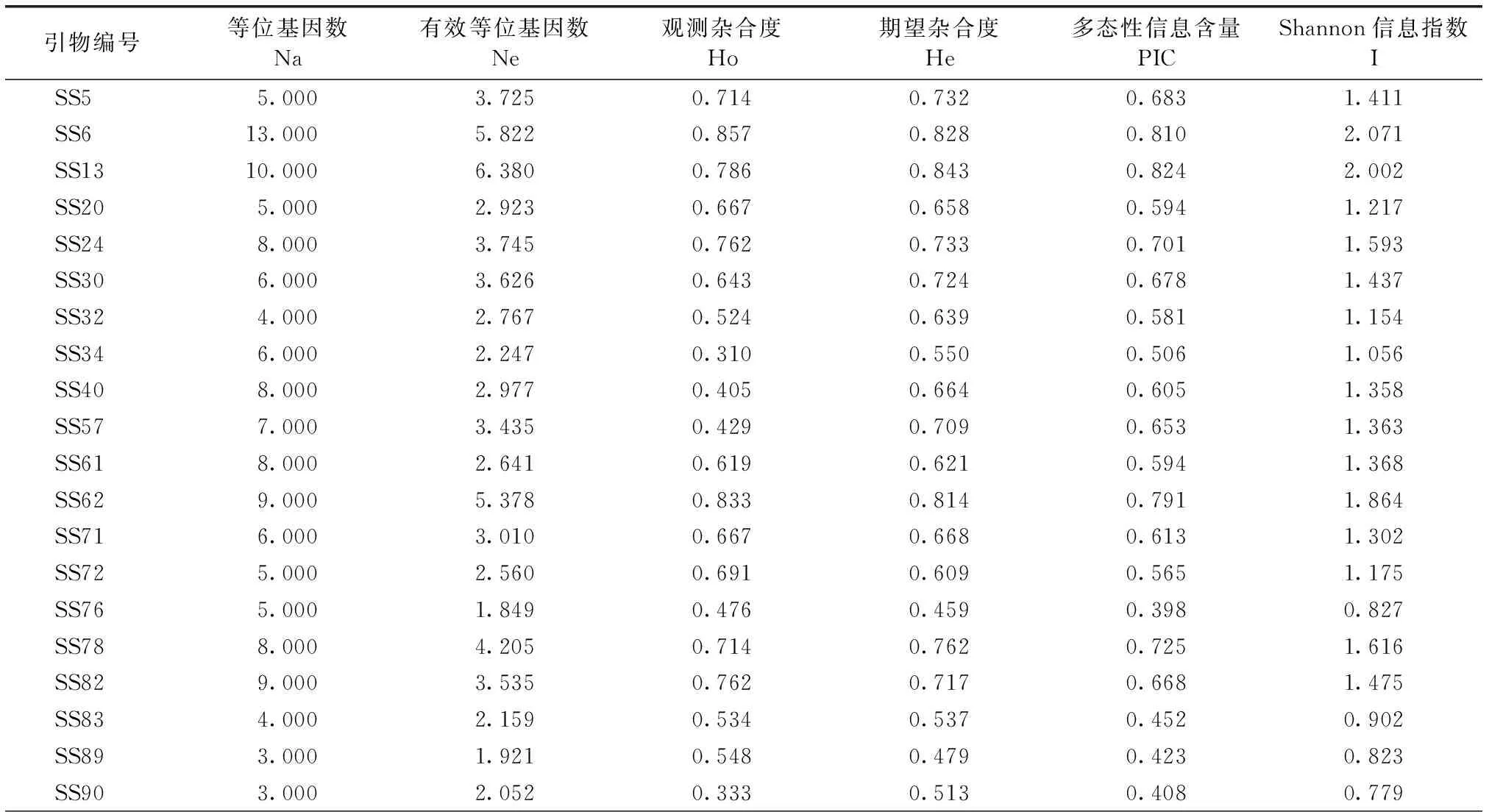
表3 28对SSR引物在42份沙棘品种中的遗传信息Table 3 Genetic information of 28 pairs of SSR primers in 42 varieties of Hippophae rhamnoides
2.6 沙棘品种间亲缘关系
遗传相似性系数可用于评估遗传相似程度,遗传相似性系数越大,表明两者间的相似程度越高,亲缘关系越近。在42份沙棘品种及近缘种中,遗传相似性系数最小的为“深秋红”和“无刺雄”(0.601),说明两者相似程度低,亲缘关系远。遗传相似性系数最大的为“状元黄”和“楚伊”(0.990)、“丰产”和“特丰1号”(0.990)以及“阿列依”和“乌兰沙林”(0.990),说明两者间相似程度高,亲缘关系近。UPGMA聚类结果显示,当遗传相似性系数为0.694时,42份材料被分为2组,当遗传相似性系数约为0.740 2时,所有品种被分为3组(图4)。基于MxComp功能分析相似性系数间的相关系数为0.782,数值接近于1,表明聚类分析结果较好。

图4 42份沙棘品种的聚类分析Fig.4 Dendrogram of 42 Hippophae rhamnoides varieties
2.7 沙棘品种指纹图谱
根据28对引物的扩增结果,综合分析PIC值、扩增片段大小和等位基因数等指标,选取SS6、SS13、SS30、SS40、SS62和SS189这6对引物构建沙棘DNA指纹图谱(表4),表4显示,“乌兰沙林”经引物SS6扩增后有两个不同的等位变异,分别为123和125 bp;经引物SS13扩增后有两个不同的等位变异,分别为272和287 bp;经引物SS30扩增后有一个等位变异,为257 bp;经引物SS40扩增后有一个等位变异,为312 bp;经引物SS62扩增后有两个不同的等位变异,分别为286和292 bp;经引物SS189扩增后有两个不同的等位变异,分别为264和273 bp,表4中其他数据同上述意义。这些引物在全部品种中检测到的多态性位点共57个,即两个品种之间相同指纹图谱出现的概率为1/257,表明出现相同指纹图谱的概率极低。因此,选用这6对引物构建沙棘DNA指纹图谱,可以实现沙棘品种的快速准确鉴定。

表4 42份沙棘品种指纹图谱Table 4 Fingerprint of 42 Hippophae rhamnoides varieties
3 讨论
笔者对“实优1号”转录组数据进行分析,得到的干净读长占原始序列的96.80%;Q30值高达92.91%;转录本unigene序列为25 286条;SSR位点总数为6 196个。不同植物中主要的SSR重复类型不同,但大部分为二、三核苷酸重复,如榆树[35]、大麻[36]。沙棘P型SSR位点重复类型中,优势基序分别为(A/T)n、(AT/TA)n和(GAA/TTC)n。影响SSR中短重复单元含量的因素有物种进化时间、物种变异频率等[37],沙棘转录组中SSR重复类型较丰富且短重复单元含量多,这与WANG等[17]的研究结果一致,除了单核苷酸重复外,二核苷酸重复是沙棘EST-SSR中最常见的位点,其次是三核苷酸重复,这也与李珊珊等[38]根据蒙古沙棘转录组开发EST-SSR的研究结果相似。从沙棘SSR重复基序类型来看,AT含量越高的基序出现的频率也就越高,而且SSR重复基序的长度越长,SSR发生的频率越低,这也与戴亚平[39]的研究结论一致。沙棘基因组的编码区域SSR位点丰富多样,为SSR引物开发及后续研究提供可能。
笔者研究中沙棘材料主要为国家林业和草原局林木品种审定委员会及地方林业和草原部门审定的林木良种,生态适应性较强,是适用于干旱半干旱广大区域种植的主栽品种,基于这些研究材料的SSR引物的筛选和亲缘关系分析具有重要参考价值。利用28对多态性引物对42个品种进行分析,Na、Ne、Ho、He、PIC和I等遗传多样性参数均明显高于李贺等[40]利用基于RNA-seq数据的17对引物对沙棘不同品种进行SSR的试验结果。笔者研究中PIC范围为0.398~0.824,均值为0.623;而李贺等[40]的试验结果中PIC为0.150 7~0.588 6,均值为0.271 0,该PIC较低的原因可能是供试沙棘材料有多个杂交品种/品系,亲本亲缘关系较近导致多样性降低。与上述沙棘遗传多样性研究相比,笔者研究中沙棘品种来源丰富度高,包括引种、选育和杂交等不同类型,且包括多种不同杂交组合(表1),因此,这些品种的遗传多样性更高。PIC可用来衡量微卫星DNA的变异程度:0表示无多态性;PIC值越趋近于1,表示多态性越高。BOTSTEIN等[41]首次提出:当PIC>0.5时,引物具有高度多态性;当0.5 我国沙棘良种的选育首先集中于对我国天然沙棘的研究和筛选,如“无刺雄”“森淼”;随后开始从国外引进优良品种,如俄罗斯大果沙棘“阿列依”“向阳”“丰产”“楚伊”“浑金”“橙色”和“阜欧”等,从芬兰引进“实优1号”“海滨沙棘”等,从蒙古引进“乌兰格木”等。在品种引进的基础上,开始选育或杂交育种工作,如“乌兰沙林”“草新2号”是由“乌兰格木”实生子代选育的,“棕丘”“辽阜1号”“白丘”是由“楚伊”实生子代选育的。杂交组合后代优势明显,如“中棘1号”“中棘2号”“中棘3号”“中棘4号”“中棘25号”“红棘1号”“红棘2号”都是以蒙古沙棘亚种优良品种“乌兰格木”为母本,以中国沙棘优良无性系“丰宁”为父本杂交的[42]。UPGMA聚类中,当遗传相似性系数约为0.740 2时沙棘品种分为3支,其中“乌兰格木”“丰宁”和中棘系列分别聚类为一支,且当遗传相似性系数为0.694时“乌兰格木”与中棘系列聚为一支,这一结果与RUAN[43]和李贺等[40]对蒙古沙棘、中国沙棘杂交品种/品系的研究结果一致。 该研究分析了沙棘转录组数据,挖掘出SSR位点6 196个,筛选出28对多态性引物,选用SS6、SS13、SS30、SS40、SS62和SS189这6对引物,构建了沙棘指纹图谱,对42份沙棘品种进行快速准确鉴定,相关研究结果能够为沙棘的种质鉴定、分类、保护和利用等提供支撑。4 结论

