基于三维密集连接卷积网络鉴别高级别胶质瘤与单发脑转移瘤
张斌,黄陈翠,薛彩强,李昇霖,周俊林*
1.兰州大学第二医院放射科,兰州大学第二临床医学院,甘肃省医学影像重点实验室,医学影像人工智能甘肃省国际科技合作基地,甘肃 兰州 730030;2.北京深睿博联科技有限责任公司,北京 100080;
脑胶质瘤是成人中枢神经系统最常见的原发肿瘤。2016年WHO中枢神经系统肿瘤分类将脑胶质瘤分为WHO 1~4级,其中WHO 3级和4级胶质瘤统称为高级别胶质瘤(high-grade gliomas,HGGs),占原发性恶性脑肿瘤的60%以上[1-3]。脑转移瘤(brain metastases,BMs)是颅内最常见的恶性肿瘤,25%~50%的癌症患者最终会发展为脑转移[4-5]。与BMs通常采用姑息性治疗方案不同,HGGs标准治疗方式是最大范围手术安全切除同步放化疗[6-7],然而临床术前区分单发BMs与HGGs较为困难,通过影像学检查准确鉴别两者有助于制订临床决策,但其影像学征象有较多重叠。人工智能的深度学习(deep learning,DL)预测模型能够为脑肿瘤分类鉴别提供更好的临床适应性及灵活性[8],但该方法在BMs与HGGs鉴别诊断方面的研究较少。本研究拟利用三维密集连接卷积网络(three-dimensional densely connected convolutional networks,3D-DenseNet)分别构建独立的基于T2WI(T2-net)、T1WI增强(T1C-net)及T2WI和T1WI增强(TS-net)融合的预测模型,以期实现对两种肿瘤的精准鉴别诊断,为临床的治疗决策提供可靠的依据。
1 资料与方法
1.1 研究对象 回顾性分析兰州大学第二医院2016年6月—2021年6月经手术病理证实的颅内单发BMs 111例,其中男65例,女46例,年龄19~75岁,平均(54.63±9.98)岁;原发病灶来源于肺癌55例、胃肠道肿瘤17例、泌尿生殖系统肿瘤9例、乳腺癌4例、恶性黑色素瘤1例、骨肿瘤1例、原发灶未确定24例。HGGs 230例,其中男134例,女96例,年龄18~77岁,平均(52.66±11.23)岁;WHO 3级胶质瘤31例,4级199例。纳入标准:①根据2016年WHO中枢神经系统肿瘤分类[3],经术后病理学证实HGGs(WHO 3、4级)和BMs;②均为颅内孤立性病灶;③术前均进行MRI检查,检查序列包括T2WI、T1WI增强(T1C)序列。排除标准:①MRI扫描前有明确颅脑手术史、放化疗史;②图像存在明显伪影;③病灶弥漫,无明确颅内肿块;④颅内肿块直径<1 cm或多发病灶;⑤年龄<18岁。本研究经本院医学伦理委员会批准(2020A-070),豁免受试者知情同意。
1.2 仪器扫描参数 患者取仰卧位,使用西门子Verio 3.0T超导MRI进行头部平扫和增强扫描。扫描参数:T1WI(GRE序列),TR 550 ms、TE 11 ms、层厚5 mm、层间距1.5 mm、视野260 mm×260 mm、矩阵256×256;T2WI(TSE序列),TR 2 200 ms、TE 96 ms、层厚5mm、层间距1.5 mm、视野260 mm×260 mm、矩阵256×256。增强扫描在轴位、矢状位及冠状位上行T1WI扫描,对比剂为Gd-DTPA[(Bayer Schering Pharma AG)/kg],静脉滴注速度0.1 mmol/kg,流速3.0 ml/s,相应的扫描参数与平扫相同。
1.3 图像处理与模型的构建
1.3.1 图像预处理与分割 由于HGGs与BMs的瘤-脑界面模糊,瘤周水肿边界通常不清晰,本研究在参考T1C和T2WI图像的同时,在T1C和T2WI图像上手工标注肿瘤区域。所有图像均从PACS系统以DICOM格式输出,将获得的T2WI、T1C序列图像通过Python 3.6.11进行N4偏置场校准及配准。后续分析仅在感兴趣区体积(volume of interest,VOI)中进行,而非在MRI图像的所有层面中进行,以更好地聚焦于特征提取、减少计算负担。
肿瘤区域VOI由1名高年资神经影像诊断医师划定,使用开源的ITK-SNAP 3.8.0软件(http://www. itksnap.org)的动态轮廓分割模式在T2WI和T1C轴位图像上进行手工标注,将T2WI图像上的异常信号及高信号(包括瘤周水肿)认定为肿瘤区域,T1C图像上的异常强化信号及低信号(包括瘤周水肿)认定为肿瘤区域,避开脑脊液。在3D-DenseNet模型输入图像VOI的勾画中,将能够完全包含各层面肿瘤区域的最小三维矩形框定义为动态轮廓分割的VOI,利用三维分割病灶体积得到VOI[9](图1)。另一名高年资放射科医师检查所勾画的VOI,存在分歧时讨论达成一致。最后,将三维标注文件以nii.格式保存,以进行后续处理。
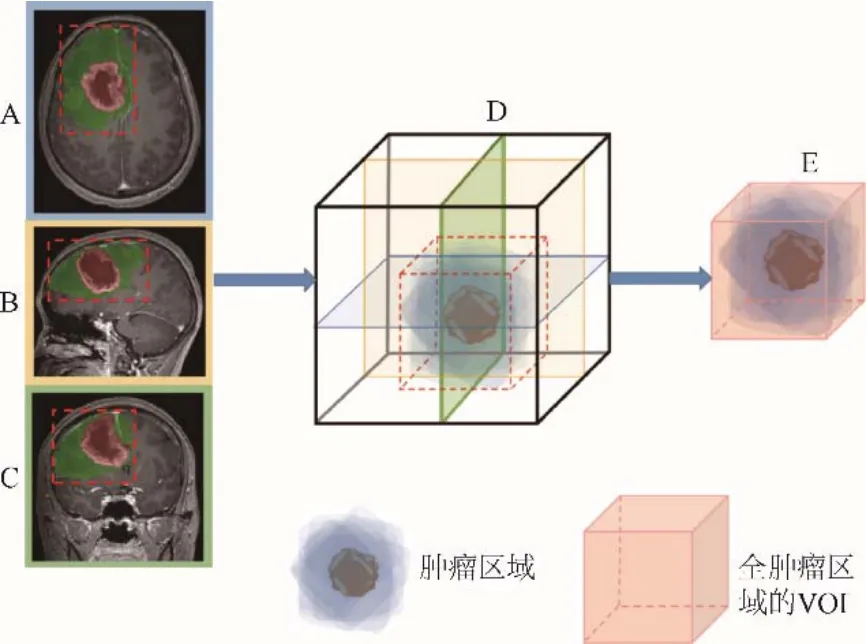
图1 3D模型的VOI分割方法。A~C:分别在三维重建图中横断位、矢状位和冠状位选取包含肿瘤区域的最小矩形区域(红色虚线框),其中红色为肿瘤瘤体,绿色为瘤周水肿区;D:能够完全包含各个方位肿瘤区域的最小三维矩形框;E:三维分割病灶体积得到全肿瘤区域的VOI
1.3.2 DenseNet 本研究选择DenseNet-169进行研究,它由4个密集块(Dense Block)构成,2个相邻模块间的层称为过渡层,通过卷积层和池化层改变特征映射的大小。图2为DenseNet的架构示意图。
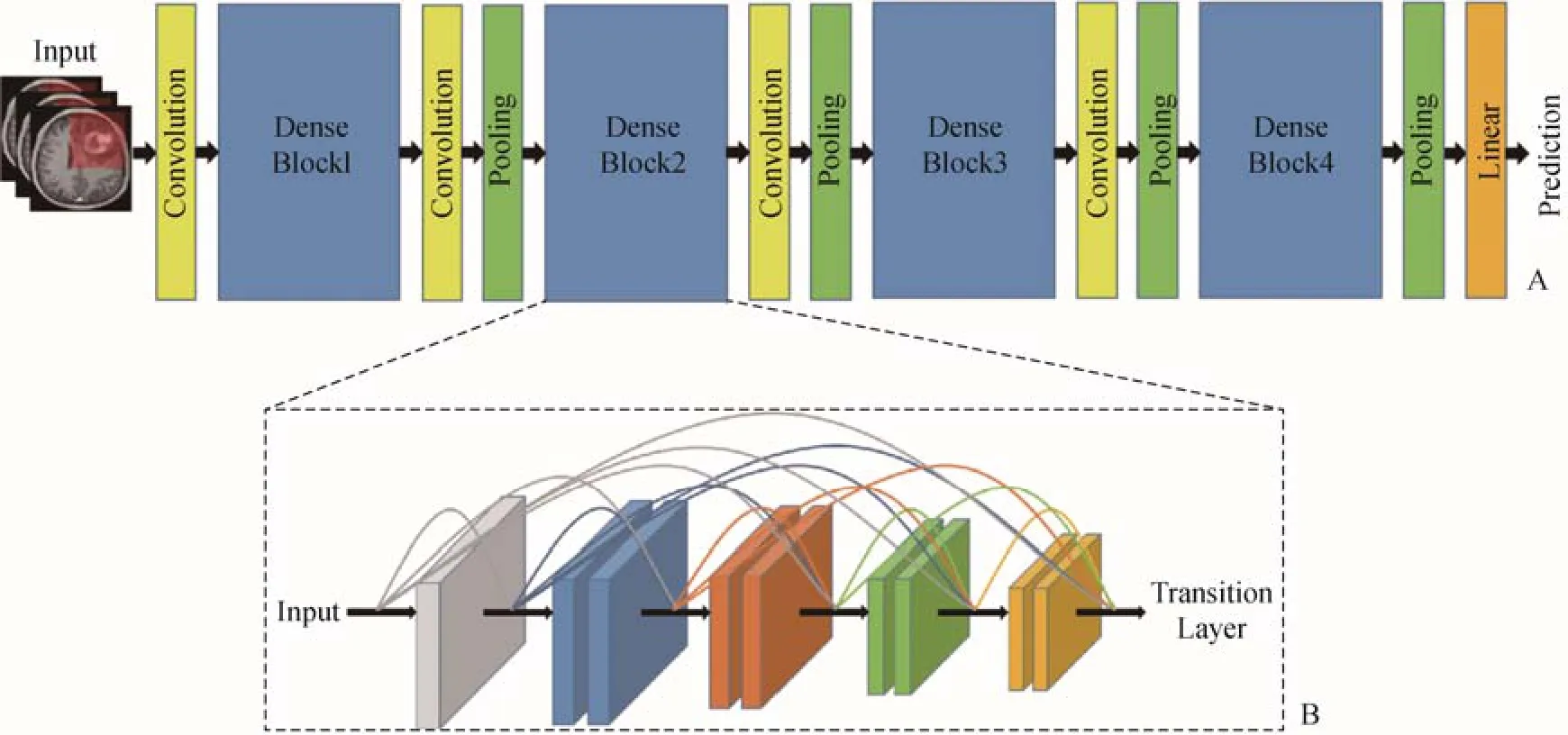
图2 密集连接卷积网络示意图。A.密集连接卷积网络构架的4个密集块;B.密集块结构示意图,每一层均将前面所有的特征图作为输入
1.3.3 卷积神经网络模型的训练及验证 将VOI的大小统一调整为64×64×64。将HGGs和单发BMs的标签分别设置为1和0,数据集按照7∶3随机分为训练集254例和验证集87例,利用训练集训练分类器,然后利用验证集对模型进行验证。对训练集数据采用多种数据增强方法,包括镜像、缩放、旋转、重采样、亮度、对比度及裁剪,尽可能减弱过拟合。
在训练过程中,根据最小的验证损失进行提前停止,得到最优模型。在训练时,卷积神经网络模型参数的更新梯度由损失函数Log损失计算,用于衡量预测值与标签值的差异,Log损失计算见公式(1)。其中,m为输入样本量,y为给定输入标签值。
参数优化算法采用Adam算法对学习率进行约束,学习速率设置为0.001。学习率调节方法采用StepLR等间隔调整学习率,调整倍数为0.1倍,调整间隔为1,间隔单位为遍历(epoch),每10个轮回epoch调整1次学习率,学习率调整为上一次的2倍。由于本研究是医学图像的二分类问题,在ImageNet自然图像分类预训练模型的基础上进行训练。模型迭代的轮数设置为100次,按批次放入模型训练的图像数量Batch_size设置为12[10]。DL模型输出为每个输入图像的分类概率,然后将患者所有VOI的结果结合,将概率最高的结果分配给该患者,从而给出每个患者的诊断,最终基于3D-DenseNet分别构建预测模型T2-net、T1C-net和TSnet。
1.4 统计学分析 DL模型统计学分析主要使用Python 3.6.11进行,卷积神经网络模型的构建使用PyTorch软件包,通过sklearn软件包输出受试者工作特征(ROC)曲线评价DL模型的鉴别效能。采用Delong检验比较不同序列及不同卷积神经网络所建模型的预测性能[10],P<0.05为差异有统计学意义。
2 结果
2.1 3D-DenseNet模型的预测性能及验证 3D-DenseNet模型在T1C及T2序列模型的训练集中HGGs 175例,BMs 79例;验证集中HGGs 55例,BMs 32例。
T1C-net在训练集和验证集的AUC分别为0.852(95%CI0.802~0.901)和0.853(95%CI0.773~0.934);T2-net在训练集和验证集的AUC分别为0.802(95%CI0.743~0.862)和0.721(95%CI0.607~0.835);TS-net在训练集和验证集的AUC分别为0.856(95%CI0.822~0.890)和0.745(95%CI0.667~0.822),见表1。
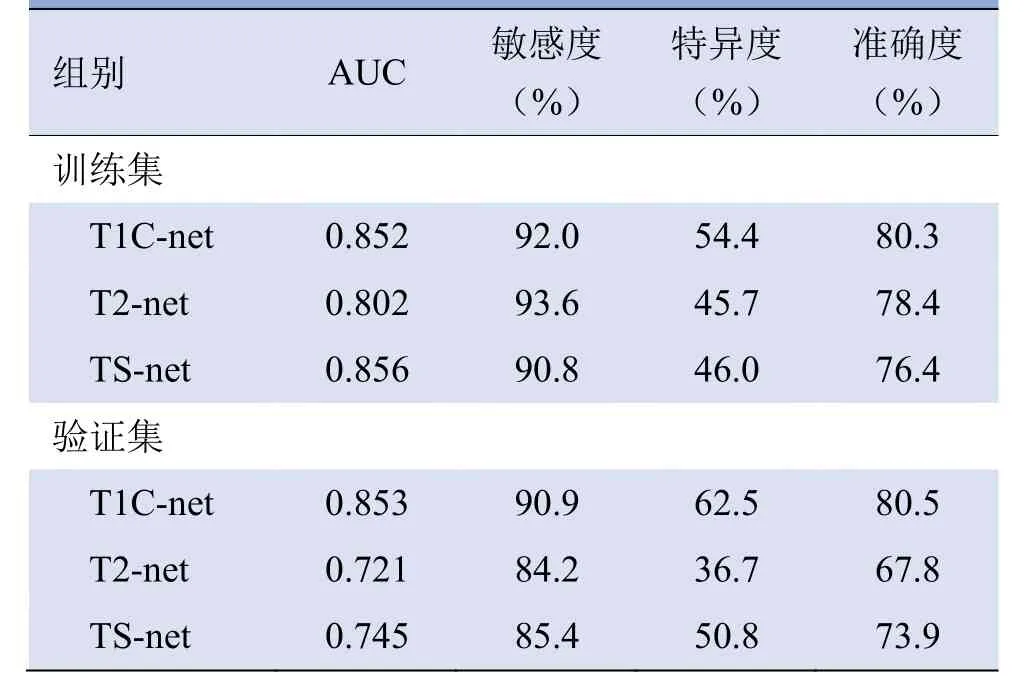
表1 3D-DenseNet模型鉴别HGGs和BMs的效能
2.2 T1C-net、T2-net和TS-net模型性能比较 T1Cnet验证集的AUC及准确度均高于T2-net和TS-net,TSnet验证集的AUC及准确度均高于T2-net(表1)。训练集中,T1C-net与T2-net、TS-net与T2-net模型的AUC差异有统计学意义(P<0.05),而T1C-net与TS-net模型的AUC差异无统计学意义(P>0.05);验证集中,T1Cnet与T2-net模型的AUC差异有统计学意义(P<0.05),而TS-net与T2-net、T1C-net与TS-net模型的AUC差异无统计学意义(P>0.05),见图3。

图3 不同序列各模型的Delong检验比较。A.训练集;B.验证集
综合比较发现,基于3D-DenseNet的T1C-net模型的性能最优(表1)。
3 讨论
BMs和HGGs在常规MRI上的影像学特征相似,即使已知原发恶性肿瘤病史,临床及影像医师仍需要进行活检或在可能的情况下进行病灶切除通过病理组织学确诊;然而部分BMs患者在行任何手术或药物治疗前,需要进行复杂的全身分期以确定原发癌的位置和评估其他远处转移[11]。由于两者在手术计划、治疗途径和预后方面存在较大差异,术前准确区分HGGs和BMs十分必要。
DL仅需要简单的注释,所提取的特征具有比影像组学特征更高的识别能力和稳定性,显示出其在医学图像分类中的优势[12]。因此,本研究以DL方法为基础,将MRI的T2WI及T1C图像作为输入图像鉴别两种肿瘤,在更大的患者队列中研究HGGs和BMs的差异,减少既往研究由于数据较少造成的偏倚;同时,DL方法具有更好的灵活性,能够进一步挖掘数据中更深层次的信息;此外,DL方法避免了复杂的感兴趣区勾画过程,本研究将能覆盖所有层面肿瘤区域的最小矩形框作为VOI,VOI内包括肿瘤实质及瘤周水肿区,跟既往的组学勾画相比,对肿瘤的边界并不需要细致的分割,使不同操作者间的可重复性较好。相较于依赖精确肿瘤边界注释的传统影像组学方法,该方法临床通用性更强,能更好地显示肿瘤本身以及瘤周微环境的特征,同时更为聚焦图像的特征提取[13]。
本研究选用的DenseNet通过特征重用来开发网络的潜力,产生易于训练和具有高参数效率的压缩模型,较既往的卷积神经网络在大多数技术上获得了显著改进[14],仅需较少计算实现更高性能,可以更有效地利用参数,使DenseNet过拟合的现象较为少见[15-16]。Remedios等[17]比较5个卷积神经网络(ResNet-50、DenseNet-121、EfficientNet-B0、PhiNet和Inception)在CT血管造影上检测大血管闭塞的性能,结果显示DenseNet-121在准确度、敏感度、特异度、召回率和F1评分方面的平均表现最好,其认为DenseNet大量使用密集连接,使来自较早层次的特征映射可以在网络中更深入地使用,同时有助于梯度流和正则化。本研究使用的三维网络结构优势在于密集的连接将信息从所有前面的层传送到下面的层,能够更准确地保存信息的完整性[18]。三维模型对病灶信息的掌握更加完整,且在扫描过程中不同序列的可变性较低,不同序列的数据匹配度更好,在一定程度上避免了二维数据的不连续性,使模型的适用范围更广[14,19-20]。本研究对3D勾画得到的VOI进行训练得到的模型具有良好的性能,训练集和验证集AUC分别为T1C-net:0.852、0.853;T2-net:0.802、0.721;TS-net:0.856、0.745。此外,临床统计显示,当BMs为单发灶且缺乏原发肿瘤病史时,与HGGs影像征象的相似性会导致40%以上的病例出现误诊[21],本研究T1C-net模型验证集准确度高达80.5%,远高于该水平。
T2WI和T1C序列是临床广泛应用于脑肿瘤诊断的MRI常规序列。T2WI序列图像质量好,伪影较少[22];而T1C序列病灶轮廓更加清晰,通过注入增强对比剂的方式,可以反映更多的肿瘤异质性[23]。本研究选择T2WI和T1C序列通过卷积神经网络分别建立T2-net、T1C-net和TS-net模型鉴别HGGs与BMs,T1C-net表现出最高的诊断效能,AUC为0.852,与T2-net模型有显著差异,并且在验证集中评估模型性能时同样具有很好的稳定性。血管增生和肿瘤血管生成是恶性脑肿瘤生物学行为中最重要的因素之一,T1C序列可以显示肿瘤血供的丰富程度,同时对肿瘤内血管细节的展示更为清晰[23-24]。此外,T2WI序列上肿瘤坏死囊变区域和周围水肿高信号区域间常有不规则等低信号边界,肿瘤中心高信号区域被低信号的边缘包围,在T1WI增强序列上通常为不规则环形强化区域,与T2WI相比,该环状强化区域在T1WI增强序列上对比度更明显[25]。因此,区分HGGs与BMs的关键在于检测瘤周区域及病灶边缘的变化。相较于T2WI,T1WI增强序列可以使病灶轮廓更为清晰,同时显示出更多病灶的异质性及病灶周围的信息,对肿瘤血管的显示较T2WI更加全面,使病灶边缘与瘤周水肿的对比显示更加强烈,这也解释了本研究所建立的DL模型中,T1C-net模型的诊断效能更高。
本研究仍存在一些局限性。由于本研究为单中心回顾性研究,未来仍需纳入多中心数据作为外部验证集,以提高DL模型的临床泛用性。此外,在创建卷积神经网络模型时特征提取的过程隐藏在黑盒中,未来需要一种新的算法澄清DL分析的内部决策过程。
总之,基于MRI常规序列的3D-DenseNet模型鉴别诊断HGGs和单发BMs具有良好的效能,在鉴别两者时T1C-net序列所建模型性能更好,DL模型可成为鉴别HGGs和BMs并指导临床制定精准化治疗方案的潜在工具。

