基于宏基因组测序技术的非靶向筛查方法检测肉类食品中的动物源性成分
丁清龙 杨丹婷 谢爱华 韦云 陈秀芬 周露


基金项目:国家市场监督管理总局科技计划项目(2019MK057);广东省市场监督管理局科技计划项目(2021ZS03)。
作者简介:丁清龙(1990—),男,河北沧州人,硕士,工程师。研究方向:食品安全检测。
通信作者:周露(1982—),女,河南洛阳人,博士,正高级工程师。研究方向:食品安全检测。E-mail:zhoulu1982@sohu.com。
摘 要:目的:建立肉类食品中动物源性成分非靶向筛查方法。方法:基于宏基因组测序技术建立肉类食品中动物源性成分非靶向筛查方法,将该方法用于模拟样品和实际样品检测,并用现有标准检测方法对实际样品检测结果进行确认。结果:模拟样品检测结果与模拟情况一致;在实际样品中检出与样品名称不一致或样品标签未标识的动物源性成分,且非靶向筛查方法检测结果与现有标准方法检测结果一致。结论:该方法可以快速锁定样品中未知的动物源性成分,检测结果准确可靠,可为政府打击肉类食品掺假提供更为有力的技术支撑。
关键词:宏基因组测序;动物源性成分;非靶向;掺假
Determination of Animal-Derived Ingredients in Meat Products by Non-Targeted Screening Method Based on Metagenomic Sequencing Technology
DING Qinglong, YANG Danting, XIE Aihua, WEI Yun, CHEN Xiufen, ZHOU Lu*
(Guangdong Institute of Food Inspection, Guangzhou 510435, China)
Abstract: Objective: To establish the non-targeted screening method for animal-derived ingredients in meat products. Method: The non-targeted screening method for animal-derived ingredients in meat products was established based on metagenomic sequencing technology. The method was applied to the determination of simulated samples and actual samples, and the results of actual samples were confirmed by existing standard determination methods. Result: The determination results of simulated samples were consistent with design of simulated samples. The animal-derived ingredients inconsistent with sample name or label identification were detected in actual samples, and the results of non-targeted screening method and existing standard methods were consistent. Conclusion: This method could quickly test the unknown animal-derived ingredients of samples, and the determination results were accurate and reliable. It could provide more powerful technical support to combat meat adulteration for the government.
Keywords: metagenomic sequencing technology; animal-derived ingredients; non-targeted; adulteration
肉類食品是人们餐桌必备的主要食品之一,常见的高经济价值的肉类食品有牛肉、羊肉及其制品等。但受经济利益驱动,肉类食品中掺杂使假、以次充好事件频繁发生。例如,欧洲市场的“马肉风波”,以鸭肉代替牛羊肉的“假羊肉串”“假牛肉干”“假肥牛片”等[1]。这种掺假行为不仅侵害了消费者的权益,扰乱市场,更降低了消费者对相关部门肉类食品监管的信任度。
我国现行有效的肉类食品动物源性成分检测标准仅能进行单一成分的定向检测。但随着监管力度的加大,肉类食品掺假物种的种类向多元化方向发展,样品掺假的物种及物种数目存在很大的不确定性。单一成分的定性检测极易造成漏检导致假阴性,给肉类食品市场监管带来巨大挑战。因此,急需建立肉类食品中动物源性成分非靶向筛查方法。
目前,可能实现动物源性成分非靶向筛查的技术分为两类:一类是基于质谱、光谱等产生的“指纹”图谱进行鉴别[2-3];一类是基于分子生物学的方法。“指纹”图谱方法对基质要求高,仅适用于未加工的肉类,同时该方法需要复杂的数学计算。在分子生物学方法中,多重PCR法和微流控芯片法可以实现对样品多个靶标的同时检测,但若样品中含有实验用引物探针检测靶标之外的成分,仍会存在漏检的情况[4-7]。将DNA条形码技术与Sanger测序技术相结合,挑取多个细菌克隆的方式具有很强的随机性,该方法漏检的可能性仍然很大[8]。而基于二代测序的宏基因组技术在鉴定土壤、粪便等含有混合微生物的样品中菌种方面已得到广泛应用,该技术可以实现对扩增产物中多种扩增条带的同时检测,为基于宏基因组测序技术建立肉类食品中动物源性成分非靶向筛查方法奠定基础[9]。
本研究基于宏基因组测序技术建立肉类食品中动物源性成分非靶向筛查方法,并将该方法用于模拟样品和实际样品检测,以期为相关部门打击肉类食品掺假提供可靠的技术支撑。
1 材料与方法
1.1 材料、试剂与仪器
1.1.1 实验样品
实验中用到的猪、水牛、黄牛、山羊、绵羊、鸡、鸭、鹅、马和驴等肉样于农贸市场购买;牦牛、鼠、雀、鸵鸟、貂、骆驼、乳鸽、猫、雉鸡、鸽子和貉肉源从中国检验检疫科学研究院获得;香辣味牛肉粒、猪血豆腐、牛排等样品于超市购买;餐馆牛肉和羊肉串在餐饮店获得。
1.1.2 仪器与试剂
T100 PCR仪(美国Bio-Rad公司),Biospec-nano核酸蛋白测定仪(日本岛津公司),1300系列A2级别生物安全柜(美国Thermofisher公司),Sorvall? ST 8离心机(美国Thermofisher公司),Sub-cell GT核酸电泳仪(美国Bio-Rad公司),PowerPac Basic电泳仪电泳(美国Bio-Rad公司),GelDoc XR+凝胶成像系统(美国Bio-Rad公司),QuantStudio 6 Flex荧光定量PCR仪(美国Thermofisher公司),Qubit 4.0荧光计(美国Thermofisher公司),MiSeq高通量测序仪(美国illumina公司)。
DNA提取试剂,DNeasy mericon Food Kit(QIAGEN,货号69514);DNA浓度检测试剂盒,QubitTM 1×dsDNA HS Assay Kit(Invitrogen,货号Q33231);PCR扩增试剂,PrimeSTAR? HS DNA Polymerase(Takara,货号R010A);基因文库构建试剂,NexteraR XT Index Kit 24 Indexes-96 samples(illumina,货号15055293);高通量测序试剂,PhiX Control Kit v3(illumina,货号FC-110-3001)和MiSeq Reagent Micro Kit v2(300-cycles)(illumina,货号FC-103-1002);荧光PCR试剂,Takara Premix Ex Taq (Probe qPCR)(Takara,货号RR390A)。
用于宏基因组测序的扩增子上游引物COI-F序列(5-3)为TCGTCGGCAGCGTCAGATGTGTATAAGAGACAGCTCAACYAATCAYAAAGATATYGGCAC,下游引物COF-R序列(5-3)为GTCTCGTGGGCTCGGAGATGTGTATAAGAGACAGGGYATNACTATRAAGAAAATTATTAC。该对引物由Illumina宏基因组测序引物和推荐性国家标准《动物制品中动物源性检测基因条码技术 Sanger测序法》(GB/T 35918—2018)中的微型基因条码引物合并而成。相关引物、探针合成及Sanger测序服务均委托生工生物(上海)股份有限公司完成。
1.2 实验方法
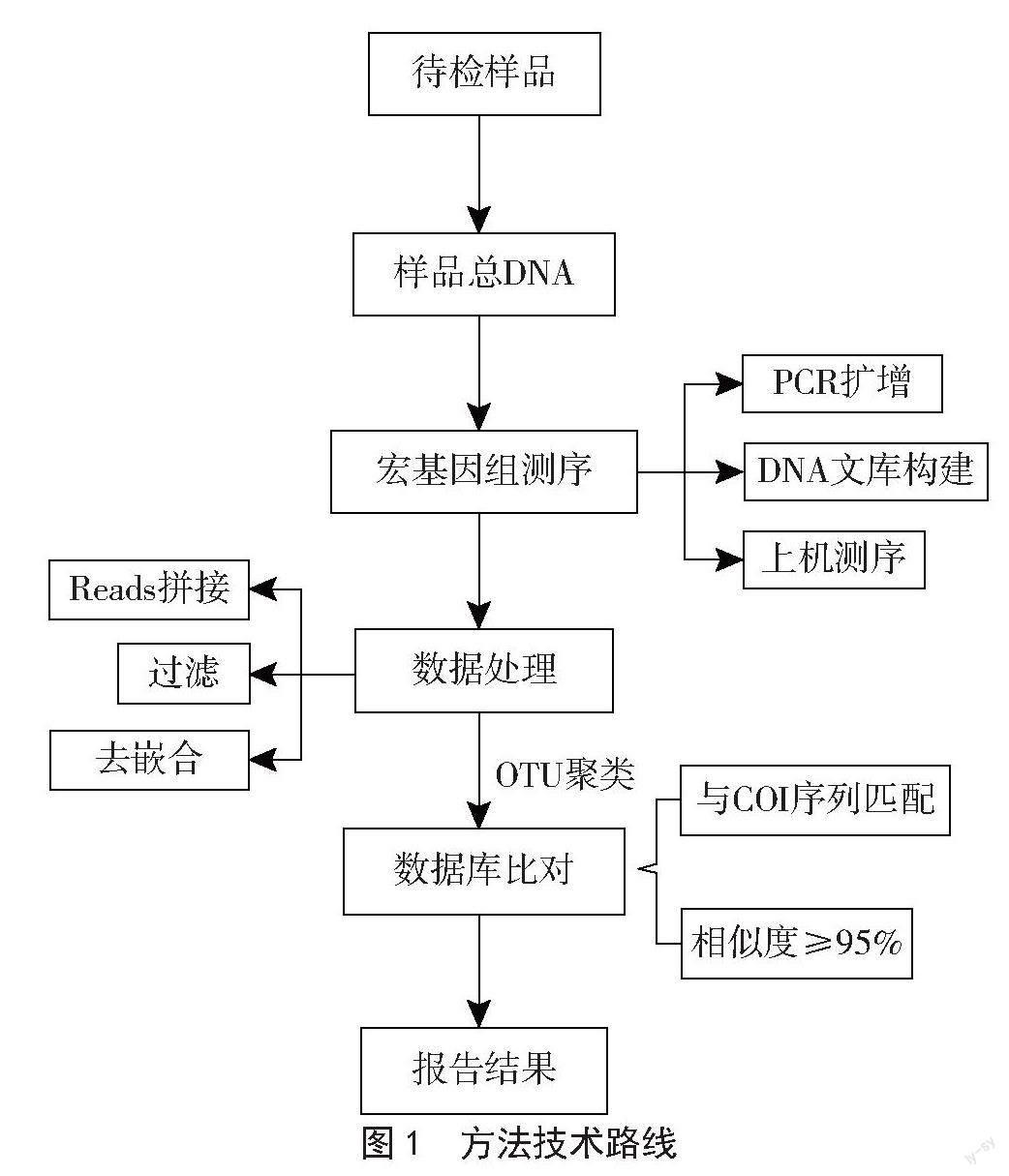
1.2.1 动物源性成分非靶向筛查方法的技术路线
动物线粒体DNA上编码细胞色素酶亚基I(COI)基因序列相对保守,不同种动物的COI基因序列存在差异。本方法使用的扩增子引物为通用引物,可以扩增出不同种动物的COI基因序列,技术路线如图1所示。使用该扩增子引物对样品中的所有动物源性成分进行扩增,利用宏基因组测序技术对扩增产物中不同种动物的COI基因序列进行分析检测,再将获得的序列在基因数据库(美国国家生物信息中心)中比对,从而实现样品中动物源性成分的非靶向检测。
1.2.2 样品DNA提取
按照商业化DNA提取试剂盒说明书的要求提取DNA。提取过程中设置提取对照,以灭菌去离子水代替样品进行DNA提取。
1.2.3 DNA浓度测定
取3.5 μL已提取的DNA样品,采用核酸蛋白分析仪测定DNA浓度。
1.2.4 PCR扩增及扩增产物电泳检测
采用25 μL PCR反应体系,其中10×PCR反应Buffer 2.5 μL、dNTP 2 μL、上下游引物各1 μL(引物浓度为10 μmol·L-1)及DNA模板25~100 ng,补充无菌双蒸水至总体积为25 μL。
PCR反应程序:94 ℃预变性5 min;94 ℃变性30 s,51 ℃退火30 s,72 ℃延伸30 s,40个循环;
72 ℃,10 min。
将5 μL扩增产物与适量上样缓冲液混合,用2%的琼脂糖胶进行电泳检测。
1.2.5 基因文库构建
采用COI-F和COI-R为引物,以样品DNA为模板,进行第1轮PCR。25 μL反应体系:模板
5 μL,5×Buffer 5 μL,dNTP 2 μL,上游引物1 μL,下游引物1 μL,DNA聚合酶(高保真)0.2 μL,超纯水10.8 μL。反应程序:95 ℃,3 min;95 ℃ 30 s,55 ℃ 30 s,72 ℃ 30 s,30个循环;72 ℃,5 min;4 ℃保存。采用磁珠法對第1轮扩增产物进行纯化,以灭菌超纯水重悬纯化后的DNA。
再以纯化后的DNA为模板,以Nextera○R XT Index Kit 24 Indexes-96 samples中的Index primer为引物,进行第2轮PCR(接头Index PCR)。25 μL反应体系:模板2 μL,5×Buffer 5 μL,dNTP 2 μL,Index 1引物2.5 μL,Index 2引物2.5 μL,DNA聚合酶(高保真)0.2 μL,超纯水12.8 μL。反应程序:95 ℃,3 min;95 ℃ 30 s,55 ℃ 30 s,72 ℃ 30 s,7个循环;72 ℃,5 min;4 ℃保存。采用磁珠法对第2轮扩增产物进行纯化,以灭菌超纯水重悬纯化后的DNA,即为构建好的基因文库。
1.2.6 宏基因组测序
采用Qubit 4.0荧光计对构建好的样本基因文库进行定量,并将其稀释至4 nmol·L-1。从每个稀释好的文库中取出5 μL DNA,混匀成为最终上机文库。取5 μL混合好的文库和5 μL 0.2 mol·L-1的NaOH,二者混合进行文库变性后,再向其中加入990 μL预冷的HT1溶液,得到20 pmol·L-1的变性文库。取
20 pmol·L-1变性文库210 μL,加入390 μL预冷的HT1溶液,得到终浓度为7 pmol·L-1的样本变性文库。制备终浓度为7 pmol·L-1的测序标准品PhiX的过程与上述操作一致。
将样本变性文库中掺入30%的PhiX,即420 μL终浓度为7 pmol·L-1的样本变性文库与180 μL终浓度为7 pmol·L-1的PhiX标准品变性文库混合,得到最终上机样本。将上机样本加入测序试剂盒的样本孔中,采用illumina Miseq测序仪上机测序。
1.2.7 测序数据分析
将下机数据中的FASTQ文件导入软件中,使用CLC Genomics Workbench 21.0.5软件Toolbox中的Trim工具包对数据进行剪切。然后使用Toolbox中的OTU clustering工具包,对剪切后的数据进行拼接和OTU聚类,最后将OTU聚类得到的序列在美国国家生物信息中心数据库(https://blast.ncbi.nlm.nih.gov/Blast)中进行比对。将比对结果为Cytochrome oxidase I基因且序列相似度≥95%的序列作为结果序列。
1.2.8 测序结果确认
采用现有动物源性成分检测标准方法对测序结果进行确认,其中牛源性成分和羊源性成分检测采用《常见畜禽动物源性成分检测方法 实时荧光PCR法》(GB/T 38164—2019);猪源性成分检测采用《食品及饲料中常见畜类品种的鉴定方法 第8部分:猪成分检测 实时荧光PCR法》(SN/T 3730.8—2013);马源性成分检测采用《食品及饲料中常见畜类品种的鉴定方法 第5部分:马成分检测 实时荧光PCR法》(SN/T 3730.5—2013),鸡源性成分检测采用《动物源性产品中鸡源性成分PCR检测方法》(SN/T 2978—2011)。
2 结果与分析
2.1 扩增子引物通用性评价
提取猪、水牛、黄牛、山羊、绵羊、牦牛、鸡、鸭、鹅、马、驴、鼠、雀、鸵鸟、貂、骆驼、乳鸽、猫、雉鸡、鸽子和貉等21个物种的DNA,分别以其为模板采用1.1.2中所述引物进行PCR扩增。将扩增产物进行电泳,电泳结果见图2。由图2可以看出,21种动物DNA的扩增产物均为单一的条带,条带长度为227 bp,包括COI序列192 bp和M13测序引物35 bp。
将扩增产物进行Sanger测序,利用NCBI数据库对测序结果进行比对,结果见表1。由表1可以看出,本方法所用引物(1.1.2中所述)对于测试的21种动物DNA均可以扩增出相应物种的COI基因序列,且序列的相似度均在95%以上,说明该引物通用性良好。
1—猪DNA;2—水牛DNA;3—黄牛DNA;4—山羊DNA;5—绵羊DNA;6—牦牛DNA;7—鸡DNA;8—鸭DNA;9—鹅DNA;10—马DNA;11—驴DNA;12—鼠DNA;13—雀DNA;14—鸵鸟DNA;15—貂DNA;16—骆驼DNA;17—乳鸽DNA;18—猫DNA;19—雉鸡DNA;20—鸽子DNA;21—貉DNA。
图2 引物通用性评价电泳图
2.2 样品宏基因组测序及结果确认
采用本研究建立的动物源性成分非靶向检测方法对在市场上获得的7份实际样品和3份实验室模拟样品进行检测,10份样品的样品信息见表2。
提取10份样品的DNA(DNA浓度见表2),按照1.1.2中所述引物进行PCR获得扩增产物。采用Miseq高通量测序仪对扩增产物进行宏基因组测序,将测序得到的数据进行拼接、过滤、去嵌合和OTU聚类获得最终的结果序列。将测序结果序列在NCBI数据中进行比对,结果见表3。由表3可知,模拟样品8检出猪源性成分和黄牛源性成分,模拟样品9检出黄牛源性成分、鸡源性成分、鸭源性成分,模拟样品10检出绵羊源性成分、鸡源性成分、猪源性成分,检测结果与实际掺入物种一致;而实际样品1、样品4、样品5和样品6除检出与其样品名称相符的牛源性成分外,还检出了猪源性成分、马源性成分、鸡源性成分等与样品名称不符或标签未标识的成分;实际样品2和样品3除检出与其样品名称相符的猪源性成分外,还检出了鸡源性成分。
为了验证本研究建立方法检测结果的准确性,采用现有标准方法对实际样品(1~7)的宏基因组测序结果(表3)进行确认,结果见表4。从表4中可以看出,采用宏基因组对7份实际样品进行非靶向检测的结果与采用现有标准方法的检测结果一致。综合模拟样品检测结果,说明本研究建立的食品中动物源性成分非靶向检测方法的检测结果准确可靠。
3 结论
肉类食品是人们餐桌必备的主要食品之一,但受经济利益驱动,肉类食品中掺杂使假、以次充好事件频繁发生。针对目前肉类食品动物源性成分检测标准仅能进行单一成分的定向检测而极易造成漏检的问题,本研究建立了一种食品中动物源性成分非靶向筛查方法,可以实现样品中常见动物源性成分的非定向同时检测。应用该方法对模拟样品和实际样品进行检测,模拟样品检测结果与模拟情况一致,且该方法对实际样品检测结果与现有标准方法检测结果一致,说明该方法准确可靠。该方法可以快速锁定样品中未知的动物源性成分,可为政府打击肉类食品掺假提供有力的技术支撑。
参考文献
[1]唐穗平,张燕,黄景辉.广东省牛羊肉及其制品中掺杂掺假情况的调查分析[J].食品安全质量检测学报,2016,7(5):1882-1886.
[2]白京,李家鹏,邹昊,等.近红外特征光谱定量检测羊肉卷中猪肉掺假比例[J].食品科学,2019,40(2):287-292.
[3]李莹莹,张颖颖,丁小军,等.液相色谱-串联质谱法对羊肉中鸭肉掺假的鉴别[J].食品科学,2016,37(6):
204-209.
[4]陈冉越,周庆,王昱,等.6种肉类成分多重PCR鉴别方法的建立及应用[J].食品安全质量检测学报,2019,10(19):6661-6666.
[5]薛超波,王萍亞,李素芳,等.一对同时鉴定8种动物源性成分的通用引物的制备及应用[J].现代食品科技,2017,33(6):271-275.
[6]步迅,张全芳,刘艳艳,等.快速鉴定食品或饲料中9种动物源性成分的引物、探针组合物、试剂盒及其检测方法和应用:201510695558.0[P].2023-11-09.
[7]周新丽,申炳阳,高丽娟,等.用于五种动物源性成分快速检测的离心式微流控芯片系统研制[J].食品与发酵工业,2020,46(3):229-234.
[8]钟文涛,王芳妹,李白玉,等.DNA条形码在肉制品掺伪中非定向筛查技术的研究[J].食品安全质量检测学报,2017,8(5):1547-1551.
[9]宋宇,王鹏,韦月平.基于宏基因组测序的稻蟹共作稻田根际土壤微生物群落功能分析[J].西北农业学报,2023,32(9):1466-1475.

