克什米尔熊蜂转录组分析及SSR位点开发








摘要:作为众多农作物和野生植物的重要传粉者,熊蜂在维持农业和生态系统平衡中发挥十分重要的作用。本研究以克什米尔熊蜂(Bombus kashmirensis)为研究对象,使用Illumina Novaseq平台进行转录组测序,并基于转录组数据进行微卫星位点的开发。结果显示,克什米尔熊蜂转录组测序共获得95 639条测序良好的unigene,GO注释和KEGG通路富集主要集中在分子功能、细胞进程和碳水化合物代谢上。基于转录组数据共获得27 414个微卫星位点,位点发生频率17.81%。简单重复序列主要重复类型为单碱基,其中A/T为优势基元,占总微卫星数量的55.89%,且微卫星主要重复次数范围在11~15次(48.61%)。用10头不同克什米尔熊蜂样品对30个微卫星位点进行验证,有17对引物可稳定扩增。本研究为克什米尔熊蜂的适应与进化等种群遗传学研究提供了重要参考。
关键词:克什米尔熊蜂;转录组测序;微卫星分析
中图分类号:Q969.557.7""" 文献标识码:A"""" 文章编号:1007-0435(2024)06-1742-10
Transcriptome Analysis and SSR Locus Development in Bombus kashmirensis
SUN Guo, YAN Jing-yan, LIANG Cheng-bo, MA Xiao-xuan, LIU Dao-xin*
(School of Agriculture and Animal Husbandry, Qinghai University, Xining, Qinghai Province 810016, China)
Abstract:As the essential pollinators of many crops and wild plants,bumblebees play an important role in maintaining the balance of agriculture and ecosystems. In this study,the Bombus kashmirensis was used for transcriptome sequencing by using Illumina Novaseq platform and microsatellite locus were development based on the transcriptome data. The results indicated that a total of 95 639 well-sequenced unigenes were obtained from the transcriptome sequencing of Bombus kashmirensis,with GO annotations and KEGG pathway enrichment focusing on molecular functions,cellular processes and carbohydrate metabolism. A total of 27 414 microsatellite loci were obtained based on transcriptome data,with a locus frequency of 17.81%. The major repeat type of microsatellites was Mono-nucleotide repeats,of which A/T was the dominant motif,accounting for 55.89% of the total microsatellites number,and the major microsatellites base repeats were in the range of 11~15 repeats (48.61%). Thirty microsatellite loci were validated using samples from 10 different Bombus kashmirensis,of which 17 primer pairs were stably amplified. This study provides an important reference for population genetics studies such as adaptation and evolution of Bombus kashmirensis.
Key words:Bombus kashmirensis;Transcriptome sequencing;Microsatellite analysis
熊蜂(bombus spp.)隶属于节肢动物门(Arthropoda)昆虫纲(Insecta)膜翅目(Hymenoptera)蜜蜂科(Apidae)熊蜂属(Bombus),是一类社会性传粉昆虫[1]。相比于蜜蜂,熊蜂体型较大,浑身长满绒毛,耐寒性强,能够分布在寒冷的区域为一些濒危植物进行传粉[2]。此外,由于具有耐湿性强、耐低光照以及声震传粉等特性,熊蜂成为了温室作物的理想授粉昆虫,尤其适宜为设施作物及深花冠植物授粉,能够显著提高农林果蔬产量及品质[3-4]。研究显示,熊蜂授粉可使大棚试验农作物坐果率提高30%以上,单株产量提高17%以上[5]。目前全球已知熊蜂种类260余种,我国有120多种,约占全球熊蜂资源的50%[6-7]。然而,气候变暖和频繁的极端天气导致全球熊蜂资源正在减少,包括我国熊蜂资源丰富的一些地区的熊蜂数量也在减少[8-9]。有研究表明,气候变暖引起的植物单一化已使某些熊蜂的饮食宽度发生改变,导致它们与植物互作关系中传粉功能的失配,已威胁到生态系统的平衡[10]。另外,农药的广泛使用更是直接威胁到各类传粉昆虫的生存,不仅引起熊蜂访花行为的异常,而且极大地造成其传粉能力的下降[11]。
为了维持熊蜂种群数量及多样性,除了要采取各种措施保护熊蜂赖以生存的自然环境外,更多科学家使用分子手段为熊蜂的保护提供支持[12]。例如,Wang等[13]和Williams等[14]利用多种分子标记重建全球15个亚属120多种熊蜂的系统发育关系,为熊蜂的分类提供了参考依据;Lin等[15]和Sadd等[16]通过分析重要熊蜂物种的基因组序列,探讨熊蜂基因组进化速率与海拔的关系以及熊蜂基因组进化过程中一些关键基因潜在的调控过程,为熊蜂的适应与进化奠定了遗传学基础。简单重复序列(Single sequence reperts,SSR)又称微卫星序列(microsatellite),是研究动植物基因遗传多样性的一种重要分子标记[17]。作为一种多态性高、稳定性好、共显性表达且易检测的分子遗传标记[18],微卫星被广泛应用于遗传多样性分析[19]、基因定位[20]和亲缘关系鉴定[21]等众多领域。目前熊蜂有关微卫星的研究主要集中在群体遗传方面,如Takeuchi等[22]利用微卫星标记研究北海道小峰熊蜂(Bombus hypocrita sapporoensis)的遗传结构;Cejas等[23]利用微卫星标记结合线粒体基因标记研究伊比利亚半岛地熊蜂(Bombus terrestris)的时空格局等;而不同熊蜂转录组分析以及基于转录组数据开发SSR位点的报道很少。
本研究对克什米尔熊蜂(Bombus kashmirensis)进行了转录组测序,分析了其基因表达特征,并基于转录组数据分析了SSR位点的分布特征,同时设计引物对部分SSR位点进行了有效性验证。研究结果可以为克什米尔熊蜂的适应与进化等种群遗传学研究提供参考。
1 材料与方法
1.1 供试昆虫
本试验中克什米尔熊蜂样品于2021年8—9月采集于三江源地区(31°39′~36°12′ N,89°45′~102°23′ E),将熊蜂样品置于液氮速冻后,保存在-80℃条件下,以便于后续RNA提取。另取10头工蜂用于微卫星引物的验证,使用Ezup 柱式动物基因组 DNA 抽提试剂盒(上海生工生物工程有限公司)提取DNA,经过微量核酸分光光度计和1%的琼脂糖凝胶电泳检测,结果满足进一步实验要求,置于-20℃保存备用。
1.2 克什米尔熊蜂总RNA的提取、建库及数据评估
转录组测序委托上海美吉生物医药科技有限公司进行。取克什米尔熊蜂的头、胸部肌肉且用MJZol total RNA extraction kit试剂盒(上海美吉生物医药科技有限公司)提取总RNA,使用Nanodrop 2000对所提RNA的浓度和纯度进行检测并用琼脂糖凝胶电泳检测RNA完整性;采用Illumina TruseqTM RNA sample prep Kit方法[24]进行文库构建;使用Illumina平台完成转录组测序,并对获得的测序数据进行质量控制;获得 RNA-seq高质量测序数据后,使用Trinity软件对转录组数据从头组装,并对初始组装序列进行优化过滤(TransRate软件和CD-HIT软件)并再次评估(BUSCO软件)。
1.3 克什米尔熊蜂转录组unigenes的功能注释
将转录组测序获得的所有转录本与NR(https://www.ncbi.nlm.nih.gov/public/),Swiss-Prot(ftp://ftp.uniprot.org/pub/databases/uniprot/current_release/knowledgebase/complete/uniprot_sprot.fasta.gz),Pfam(http://pfam.xfam.org/),eggNOG(http://eggnogdb.embl.de/#/app/home),GO(http://www.geneontology.org/),KEGG(http://www.genome.jp/kegg/)等六大数据库进行比对,获得在各数据库的注释信息。
1.4 SSR的筛选及引物的设计
利用 MISA 软件(http://pgrc.ipk-gatresleben.de/misa/)搜寻克什米尔熊蜂转录组组装后 unigene 数据中潜在的SSR位点。软件中参数设置为单碱基、二碱基、三碱基、四碱基、五碱基、六碱基的最短重复次数分别为10,6,5,5,5,5。将结果文件导入Primer3软件,设计SSR位点的相关引物。
1.5 SSR引物的验证
为了较全面的验证引物的有效性,从批量设计的引物中随机选出30对引物(表1)。(挑选原则为两碱基重复的16个,三碱基重复的7个,四碱基重复的7个;位点片段长度在150 bp以下的位点10个、150~200 bp的位点10个、200~300 bp的位点10个;重复次数15~20次的5个、21~25次的5个、26~30次的5个、31~35次的5个、36次以上的5个;完美型微卫星)交由生工生物工程(上海)股份有限公司合成。PCR反应体系为 25 μL:克什米尔熊蜂DNA模板 0.5 μL,上下游引物(0.2~1.0 μmol·L-1)各加 0.5 μL,2×Taq Master Mix* 12.5 μL,ddH2O 11 μL。反应程序:94℃预变性 1 min 30 s;94℃变性 20 s,59~60℃退火 20 s,72℃延伸 20 s,总共 35 个循环;72℃延伸 5 min,4℃保温。扩增产物用1.2%琼脂糖凝胶电泳检测(110V,35 min)。
2 结果与分析
2.1 克什米尔熊蜂转录组测序、组装及注释
通过对克什米尔熊蜂样品转录组测序,获得119.45 Gb Clean Data,各样品Clean Data均达到6.04 Gb以上,Q30碱基质量比值在92.98%以上,GC含量在37.36%~47.39%之间,说明测序质量较好。经de novo组装后,通用单拷贝同源基因基准值(Benchmarking universal single-copy orthologs,BUSCO)中有94.5%的测试基因被覆盖,其中有78.9%的基因经过比对发现是单拷贝,有15.6%的基因经过比对发现包含多拷贝(测试基因覆盖数=比对后的单拷贝数+比对后的多拷贝数)[BUSCO值:C:94.5%(S:78.9%:D:15.6%),C=S+D],共获得unigene 95 639条,平均长度为981.88 bp,N50长度为1 741 bp,unigene长度在200~500 bp之间,占总unigene的50%,随着序列长度的增加,unigene总体呈逐步递减趋势(表2)。将克什米尔熊蜂转录组测序获得的所有转录本分别与NR,Swiss-Prot,Pfam,eggNOG,GO 和 KEGG数据库进行比对注释,结果表明,在148 656条Transcript中,共有64.62%的Transcript被注释到相应的数据库,组装评估后58.41%的基因被注释到公共数据库;同时有58.64%表达unigene 被注释到公共数据库中(表3)。转录组数据被存入NCBI SRA 数据库中,克什米尔熊蜂的登录号为SRR19913284 至SRR19913300。
2.2 unigenes的GO功能注释
GO数据库的28 443个unigenes的功能注释结果表明,克什米尔熊蜂转录组功能分为分子功能(molecular function)、细胞组分(cellular component)和生物学进程(biological process)三大类和54个功能小类。注释到的生物学进程有22个功能小类,注释到的细胞组分有15个功能小类,注释到的分子功能有17个功能小类。在生物学进程中注释到的细胞进程(cellular process)功能的基因最多(13 091,36.46%),在细胞组分中注释到的细胞部分(cell part)功能的基因最多(13 758,30.62%),在分子功能中注释到的结合(binding)功能的基因最多(16 316,44.13%)(图1)。
2.3 unigenes的KEGG通路富集
KEGG数据库的31 212条unigenes 代谢通路富集分析表明,克什米尔熊蜂转录组代谢通路分别富集到环境信息处理(Envieonmental information processing)、新陈代谢(Metabolism)、遗传信息处理(Genetic information processing)、细胞进程(Cellular processes)和有机系统(Organismal system)五大类别、33个小类别中。其中富集到新陈代谢的基因数最多(9 552),富集到环境信息处理中的基因最少(3 428)。在新陈代谢途径中富集到氨基酸代谢(Amino acid metabolism)的基因最多(2 921,30.58%),在有机系统途径中富集到内分泌系统(Endocrine system)的基因最多(1 709,20.87%),在细胞进程途径中富集到运输与分解代谢(Transport and catabolism)的基因最多(2 223,44.65%),在遗传信息处理途径中富集到信号转导(Signal transduction)的基因最多(3 107,90.64%)(图2)。富集到的所有基因分布在342条代谢通路中,其中富集最多的10条代谢通路分别是核糖体(Ribosome)、戊糖和葡萄糖醛酸的相互转化(Pentose and glucuronate interconversions)、内质网中的蛋白质加工(Protein processing in endoplasmic reticulum)、氧化磷酸化(Oxidative phosphorylation)、产热(Thermogenesis)、胞吞作用(Endocytosis)、剪接体(Spliceosome)、辅助因子的生物合成(Biosynthesis of cofactors)、泛素介导的蛋白水解(Ubiquitin mediated proteolysis)、AMPK 信号通路(AMPK signaling pathway)(表4)。
2.4 克什米尔熊蜂转录组中SSR分布特征
搜寻克什米尔熊蜂95 639条unigenes中的SSR位点,结果表明:共找到SSR点27 414个,分布在17 029个unigenes中,SSR位点发生的频率为17.81%(SSR位点发生频率=含有SSR位点的unigenes/全部序列数量),含有1个以上SSR位点的unigenes序列有4 109条,单碱基至六碱基的SSR位点依次减少,占比分别为56.26%,23.31%,17.08%,2.17%,0.61%,0.57%(表5)。
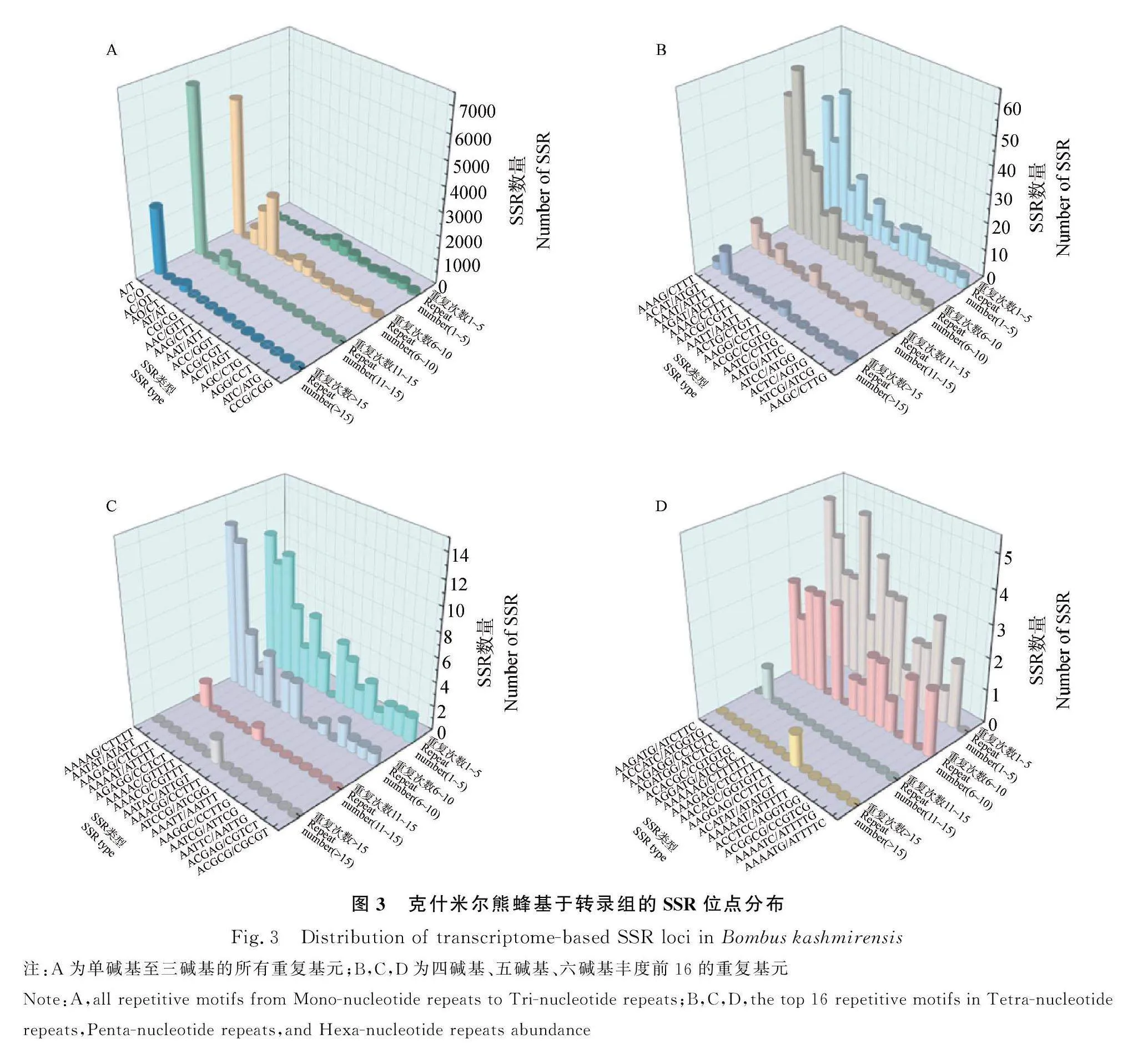
单碱基至六碱基的碱基重复类型分布结果表明:单碱基、二碱基和三碱基重复基元总共16种。单碱基中共有2种重复基元,(A/T)n重复11~15次的最多(6 836);二碱基中共有4种重复基元,(AT/AT)n重复6~10次的最多(2 477);单碱基(A/T)n,(C/G)n与二碱基(AC/GT)n,(AG/CT)n,(AT/AT)n,(CG/CG)n重复均在5次以上。三碱基中共有10种重复基元,(AAG/CTT)n重复1~5次的最多(498)(图3A)。四碱基重复基元共有29种,(ACAT/ATGT)n重复6~10次的最多(60)(图3B)。五碱基重复基元共有51种,(AAAAG/CTTTT)n重复6~10次的最多(13)(图3C),六碱基重复基元共有83种,(AAGATG/ATCTTC)n和(ACACGC/CGTGTG)n重复1~5次的最多,均为5次(图3D)。三碱基至六碱基基元类型的重复次数在1~5,6~10,11~15和大于15次区间内都有分布。
基元重复次数在11~15次的最多,为13 327(48.61%)(百分数=该重复基元次数的SSR位点数/SSR位点总数),基元重复次数在1~5次的最少,为2 777(10.13%)。单碱基的优势重复基元为A/T,共出现15 321次(55.89%)(百分数=该碱基所有重复次数的SSR位点数/SSR位点总数);二碱基的优势重复基元为AT/AT,共出现2 820次(10.29%);三碱基的优势重复基元为AAG/CTT,共出现1 033次(3.77%);四碱基的优势重复基元为AAAG/CTTT,共出现106次(0.61%);五碱基的重复基元为AAAAG/CTTTT,共出现24次(0.14%),六碱基由于各重复基元次数均未超过10次,优势重复基元不明显。
2.5 SSR分子标记开发
按照1.5中的挑选原则,从筛选出的微卫星引物中挑选30对引物进行验证,每对引物分别用10个不同地点的克什米尔熊蜂样品进行PCR扩增。结果显示:有17对引物可以在10个不同样品中稳定扩增(表1前17对SSR引物),SSR引物的有效扩增率为56.67%,部分验证结果如图4所示。同时部分引物呈现一定的多态性,初步表明上述SSR引物可靠性较好。(由于是用10个不同采样地的克什米尔熊蜂筛选SSR引物,部分引物出现1至2条非特异性或未扩增出来的条带时可将其视为偶然情况,仍可用于后续分析。)
3 讨论
3.1 克什米尔熊蜂转录组特点
转录组测序技术通过对不同对象、不同发育时期的不同组织中的转录组进行分析,构建基因调控网络,筛选出关键基因,用于相关的研究[25]。目前,克什米尔熊蜂转录组信息处于研究空白。使用不同样本材料测序,再将测序结果进行组装,可以保证数据尽量覆盖度广、信息全面、准确度高[26]。本研究基于此方法对克什米尔熊蜂进行转录组测序,95 639条unigene被注释到公共数据库,占unigene 总数的58.41%。所有试验样本Q30碱基质量比值均高于92.98%,N50长度为1 741 bp;组装后BUSCO值C:94.5%[S:78.9%;D:15.6%],说明转录组组装较完整;unigene平均长度为981.88 bp,长于中华蜜蜂(Apis cerana)(平均unigene 898 bp)[27]和泽兰实蝇(Procecidochares utilis)(平均unigene 457 bp)[28],但是短于椰心叶甲啮小蜂(Tetrastichus brontispae)(平均unigene 1 737.95 bp)[29]。有研究表明,序列长度是影响注释的重要因素[30],本次测序获得的N50、序列长度和碱基质量比值等均显示克什米尔熊蜂转录组的拼接注释效果良好。
本研究中,克什米尔熊蜂的GO功能注释主要集中在结合、催化活性、细胞部分、细胞进程和代谢进程等功能中,与夜蛾黑卵蜂[31]、绿眼赛茧蜂[32]和椰心叶甲啮小蜂[29]等膜翅目昆虫转录组功能注释结果相似。夜蛾黑卵蜂unigenes的GO功能注释主要参与结合(4 925条unigenes)、催化活性(3 542)、细胞进程(5 124)、细胞解剖实体(4 314)和代谢进程(4 479)等功能[31];绿眼赛茧蜂unigenes的GO功能注释主要参与细胞进程(5 230)、细胞部分(4 591)、代谢过程(4 068)、结合(6 652)和催化活性(5 302)等功能[32];椰心叶甲啮小蜂unigenes的GO功能注释主要参与细胞过程(1 527)、结合(1 733)和膜(1 324)等功能[29],这表明克什米尔熊蜂与这几种蜂类的基因功能类似。克什米尔熊蜂的KEGG通路富集主要在信号转导、碳水化合物代谢、氨基酸代谢、翻译、运输与分解代谢和内分泌系统等过程中,富集到的主要通路有核糖体、戊糖和葡萄糖醛酸的相互转化、内质网中的蛋白质加工、氧化磷酸化和产热等。其中富集最多的是与核糖体有关的基因,表明这些基因与克什米尔熊蜂蛋白的合成有关。
3.2 克什米尔熊蜂SSR位点发生频率及分布
本研究中,从克什米尔熊蜂的转录组中共识别SSR位点27 414个,以单碱基、二碱基和三碱基核苷酸重复为主。SSR位点的发生频率为17.81%,略高于意大利蜜蜂(Apis mellifera)(17.73%)[33],略低于中蜂(Apis cerana)(17.82%)[27],远高于黑翅土白蚁 (Odontotermes formosaanus) (9.98%)[34]和烟粉虱 (Bemisia tabaci) (5.07%)[35]等昆虫,表明其转录组中SSR相对比较丰富。不同昆虫SSR位点出现频率的差异与物种基因组特点有关,也与搜寻SSR位点时MISA软件的参数设置有关[36]。如搜寻意大利蜜蜂[33]和中蜂[27]的SSR时,参数设置为二碱基至六碱基的最小重复为6,5,5,5,5,本次搜寻SSR位点与它们的参数一致,说明研究结果可靠。
克什米尔熊蜂SSR单核苷酸重复中共有(A/T)n和(C/G)n两种,A/T为优势基元,这与梨小食心虫[37]和窄足真蚋[38]中优势碱基情况相同。且有研究表明,单碱基A/T既是单核苷酸重复中的优势碱基,也是所有重复类型的优势碱基[39]。克什米尔熊蜂SSR二碱基重复共有(AC/GT)n,(AG/CT)n,(AT/AT)n,(CG/CG)n四种,AT/AT为优势基元,与中华蜜蜂(Apis cerana cerana)[27]和意大利蜜蜂[33]相同,与青海草原毛虫(Gynaephora qinghaiensis)(AC/GT)[40]不同。克什米尔熊蜂三碱基重复的优势基元为AAG/CTT,相比于单碱基和二碱基的重复基元出现的次数,三碱基重复基元出现的次数更少,随着重复基元碱基数的增加,重复基元的类型越来越多,但是重复基元出现的次数越来越少。另外有研究表明,当SSR重复次数高于12次时,多态性位点的比例较大[41]。克什米尔熊蜂转录组SSR单碱基至六碱基的重复次数大多集中在11~15次(48.61%),说明其转录组SSR具有较高的多态性位点潜能。
4 结论
克什米尔熊蜂转录组的测序质量良好,组装注释完整,其中GO功能注释和KEGG通路富集主要集中于分子功能、细胞进程和碳水化合物代谢等一些途径中。基于转录组数据的SSR位点出现频率高、基元特征明显。本研究通过对克什米尔熊蜂转录组测序数据的分析和微卫星位点的筛选,丰富了克什米尔熊蜂的分子生物学研究,并设计开发出了17对能够稳定扩增的SSR引物,为进一步开展克什米尔熊蜂的遗传学研究奠定了基础。
参考文献
[1] 梁程博,王久利,孙国,等. 三江源地区熊蜂物种多样性研究[J]. 草地学报,2022,30(8):2126-2134
[2] 周峰,姚丽媛,石涵,等. 传粉熊蜂访花行为的研究进展[J]. 昆虫学报,2023,66(3):419-438
[3] 黄家兴,安建东. 中国熊蜂多样性、人工利用与保护策略[J]. 生物多样性,2018,26(5):486-497
[4] 黄家兴,安建东,吴杰,等. 熊蜂为温室茄属作物授粉的优越性[J]. 中国农学通报,2007(3):5-9
[5] 黄训兵,李辉,陈浩,等. 熊蜂授粉对北方冬季温室不同品种番茄品质的影响[J]. 北方园艺,2022(4):56-61
[6] CAMERON S A,SADD B M. Global trends in bumble bee health[J]. Annual Review of Entomology,2020,65:209-232
[7] SUN C,HUANG J,WANG Y,et al. Genus-wide characterization of bumblebee genomes provides insights into their evolution and variation in ecological and behavioral traits[J]. Molecular Biology and Evolution,2021,38(2):486-501
[8] KWONG W K,ENGEL P,KOCH H,et al. Genomics and host specialization of honey bee and bumble bee gut symbionts[J]. Proceedings of the National Academy of Sciences,2014,111(31):11509-11514
[9] SOROYE P,NEWBOLD T,KERR J. Climate change contributes to widespread declines among bumblebees across continents[J]. Science,2020,367(6478):685-688
[10]MILLER-STRUTTMANN N E,GEIB J C,FRANKLIN J D,et al. Functional mismatch in a bumble bee pollination mutualism under climate change[J]. Science,2015,349(6255):1541-1544
[11]SPRAYBERRY J D,RITTER K A,RIFFELL J A. The effect of olfactory exposure to non-insecticidal agrochemicals on bumblebee foraging behavior[J]. Plos One,2013,8(10):e76273
[12]孙国,梁程博,赵芳,等. 15种熊蜂全基因组SSR分布规律研究[J]. 南方农业学报,2023,54(11):3378-3387
[13]WANG L,LIU S,TANG Y,et al. Using the combined gene approach and multiple analytical methods to improve the phylogeny and classification of Bombus (Hymenoptera,Apidae) in China[J]. Zookeys,2020,1007:1
[14]WILLIAMS P H,BROWN M J,CAROLAN J C,et al. Unveiling cryptic species of the bumblebee subgenus Bombus s. str. worldwide with COI barcodes (Hymenoptera:Apidae)[J]. Systematics and Biodiversity,2012,10(1):21-56
[15]LIN G,HUANG Z,WANG L,et al. Evolutionary rates of bumblebee genomes are faster at lower elevations[J]. Molecular Biology and Evolution,2019,36(6):1215-1219
[16]SADD B M,BARRIBEAU S M,BLOCH G,et al. The genomes of two key bumblebee species with primitive eusocial organization[J]. Genome biology,2015,16(1):1-32
[17]毛轩睿,刘玉萍,苏旭,等. 沙鞭转录组简单重复序列(SSR)位点特征分析[J]. 草地学报,2022,30(8):1990-2001
[18]甘丽萍,田辉,唐恒,等. 6种鳞翅目昆虫全基因组SSR分布规律[J]. 基因组学与应用生物学,2021,40(3):1022-1030
[19]王日芳,李健,李吉涛,等. 脊尾白虾(Exopalaemon carinicauda)近交系3个家系33个微卫星座位的遗传分析[J]. 渔业科学进展,2017,38(4):78-86
[20]ZHOU L,LIU T,CHENG Y,et al. Molecular mapping of a stripe rust resistance gene in Chinese wheat landrace “Hejiangyizai” using SSR,RGAP,TRAP,and SRAP markers[J]. Crop Protection,2017,94:178-184
[21]李东宇,孔杰,孟宪红,等. 凡纳滨对虾(Litopenaeus vannamei)微卫星多重PCR体系的建立及其在家系亲权鉴定中的应用[J]. 渔业科学进展,2016,37(3):58-67
[22]TAKEUCHI T,TAKAHASHI M,NISHIMOTO M,et al. Genetic structure of the bumblebee Bombus hypocrita sapporoensis,a potential domestic pollinator for crops in Japan[J]. Journal of Apicultural Research,2018,57(2):203-212
[23]CEJAS D,DE LA RU′A P,ORNOSA C,et al. Spatial and temporal patterns of genetic diversity in Bombus terrestris populations of the Iberian Peninsula and their conservation implications[J]. Scientific Reports,2021,11(1):22471
[24]JIANG L,SCHLESINGER F,DAVIS C A,et al. Synthetic spike-in standards for RNA-seq experiments[J]. Genome research,2011,21(9):1543-1551
[25]郁伟杰,苗一凡,穆海婷,等. 转录组学解析紫花苜蓿雄性不育系花粉败育机制[J]. 草地学报,2023,31(6):1702-1713
[26]WANG Y,ZENG X,IYER N J,et al. Exploring the switchgrass transcriptome using second-generation sequencing technology[J]. Plos One,2012,7(3):e34225
[27]熊翠玲,张璐,付中民,等. 基于RNA-seq数据大规模开发中华蜜蜂幼虫的SSR分子标记[J]. 环境昆虫学报,2017,39(1):68-74
[28]高平,李丽芳,兰明先,等. 泽兰实蝇雌雄成虫转录组及差异分析[J]. 应用昆虫学报,2021,58(2):289-299
[29]刘华伟,李朝绪,李芬,等. 椰心叶甲啮小蜂转录组分析及基因功能注释[J]. 中国生物防治学报,2021,37(3):412-419
[30]ZHANG L,YANG X,QI X,et al. Characterizing the transcriptome and microsatellite markers for almond (Amygdalus communis L.) using the Illumina sequencing platform[J]. Hereditas,2018,155:1-9
[31]周婉,徐琢,杨文俊,等. 夜蛾黑卵蜂转录组及化学感受相关基因的分析[J]. 植物保护,2022,48(6):264-277
[32]王予彤,徐林波,段立清,等. 绿眼赛茧蜂转录组分析及嗅觉相关蛋白基因的鉴定[J]. 应用昆虫学报,2021,58(4):846-855
[33]郭睿,陈华枝,庄天艺,等. 利用转录组数据开发意大利蜜蜂的SSR分子标记[J]. 安徽农业大学学报,2018,45(3):404-408
[34]HUANG Q,SUN P,ZHOU X,et al. Characterization of head transcriptome and analysis of gene expression involved in caste differentiation and aggression in Odontotermes formosanus (Shiraki)[J]. Plos One,2012,7(11):e50383
[35]XIE W,MENG Q,WU Q,et al. Pyrosequencing the Bemisia tabaci transcriptome reveals a highly diverse bacterial community and a robust system for insecticide resistance[J]. Plos One,2012,7(4):e35181
[36]BISWAS M K,CHAI L,MAYER C,et al. Exploiting BAC-end sequences for the mining,characterization and utility of new short sequences repeat (SSR) markers in Citrus[J]. Molecular Biology Reports,2012,39:5373-5386
[37]冷春蒙,李引,胡迪,等. 梨小食心虫幼虫中肠转录组及SSR分子标记分析[J]. 昆虫学报,2018,61(11):1272-1283
[38]郭欢,王刚,张树田,等. 基于RNA-seq数据的窄足真蚋SSR分子标记开发[J]. 昆虫学报,2018,61(7):815-824
[39]王晖,王敬,高妍夏,等. 基于家蚕转录组测序的SSR序列分析[J]. 山东农业科学,2022,54(1):14-20
[40]南彦斌,许嘉诚,何啟玥,等. 青海草原毛虫转录组分析及SSR位点开发[J]. 草地学报,2023,31(9):2653-2662
[41]THAO D,YAMASHITA M,WATANABE A,et al. Development of tetranucleotide microsatellite markers in Pinus kesiya Royle ex Gordon[J]. Conservation Genetics Resources,2013,5:405-407
(责任编辑 闵芝智)

