基于形态性状和SSR 标记的克氏原螯虾养殖群体遗传多样性分析


关键词 克氏原螯虾; 形态性状; 微卫星; 遗传多样性
中图分类号 S917.4 文献标识码 A 文章编号 1000-2421(2024)03-0258-09
克氏原螯虾(Procambarus clarkii),俗称淡水小龙虾,因具有较高的商业价值和食用价值而深受养殖者及消费者的欢迎[1-2],现已被广泛养殖,是一种重要的水产养殖品种[3]。但由于克氏原螯虾属于外来物种,在我国的养殖时间较短,加之人们对其种质资源的保护和利用缺乏重视,这导致克氏原螯虾种质退化和病害肆虐等问题日益突出。为了克氏原螯虾种质资源的保护和良种培育,对克氏原螯虾群体的遗传多样性与遗传结构进行研究具有重要意义。
物种或种群的遗传多样性大小是其生存适应和发展进化的先决条件[4]。了解种群的遗传多样性和遗传结构有助于防止过度捕捞、保护种质资源并确保种群的可持续开发。形态学方法是进行种群遗传变异分析的最传统、最直接的方法,已被广泛应用于克氏原螯虾的种群变异及种群鉴定等研究中[5-7]。但由于形态性状受到遗传和外部环境的共同作用,会随着环境出现适应性进化[8],基于形态学的研究方法在一定情况下不能准确反映遗传变异信息[9],且易受生物生长阶段和数据处理方法的影响[10]。因此,形态学方法还需要与遗传分子标记方法相结合,才能对研究对象的遗传多样性进行客观的评估。微卫星分子标记因其具有多态性和共显性特征而在遗传分析中越来越受青睐,已被广泛应用于水产动物的遗传多样性分析、亲子鉴定、连锁图谱构建和QTL定位等领域[11-13]。因此,为进行更加客观地评估,本研究分别采用形态学和微卫星分子标记的分析方法对来自广西南宁(3 个群体)、湖北荆州(1 个群体)和江西湖口(1 个群体)共5 个克氏原螯虾群体的遗传多样性进行分析,旨在更好地了解广西南宁周边地区克氏原螯虾养殖群体的遗传多样性,为克氏原螯虾的综合养殖和科学育种提供参考资料。
1 材料与方法
1.1 样本采集
本试验采集的5 个克氏原螯虾群体,包括3 个广西南宁群体(埃及群体、良庆群体和上林群体)、1 个湖北荆州群体和1 个江西湖口群体。其中,埃及群体为2019 年从埃及引种抗逆性较强的克氏原螯虾品种亲本的F2代;良庆群体为2019 年从湖北荆州和江西鄱阳湖引种亲本自然交配产生的F3 代;上林群体为2018 年从湖北监利引种亲本的F4 代;荆州群体为2016 年采集自湖北荆州区长江中游南岸;湖口群体为2016 年采集自江西九江湖口县,为长江与鄱阳湖的交汇处。各群体具体采样信息见表1。对所有样本进行了形态参数的测量,并取腹部肌肉组织保存于无水乙醇中待用于提取DNA。
1.2 形态参数测量与数据处理
参照王克行[14]的测量方法,使用游标卡尺(精度为0.01 mm)测量每个个体的形态性状,包括全长(TL)、头胸甲长(CL)、头胸甲宽(CW)、腹节长(ASL)、腹节宽(ASW)(图1)。为了消除形态特征的个体大小依赖性,测量的5 项形态性状均标准化为以全长为基数的比值或2 个形态性状的比值。本研究共使用了6 项形态比例参数,分别为:CL/TL、CW/TL、ASL/TL、ASW/TL、CL/CW 和ASL/ASW。
1.3 DNA的提取和PCR扩增
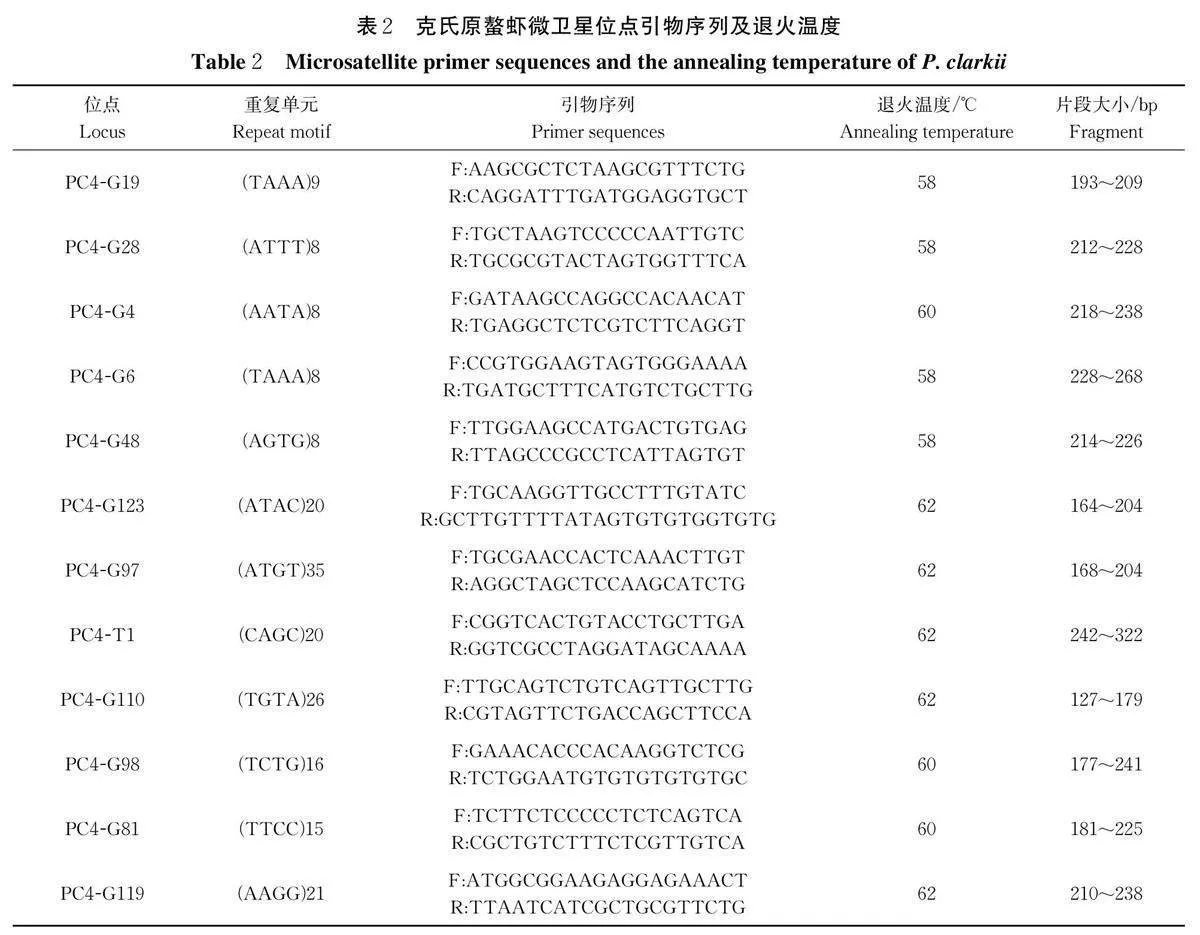
采用醋酸铵/异丙醇的方法[15]进行克氏原螯虾基因组DNA 的提取,检测浓度和质量后,稀释至100ng/μL 保存于‒20 ℃冰箱备用。从Wang 等[16]开发的克氏原螯虾微卫星标记中选择具有较高多态性的四碱基重复位点12 个,具体信息见表2。上述微卫星位点引物对的正向引物5'端分别用FAM、HEX、ROX进行荧光修饰并合成。12 对荧光引物在5 个克氏原螯虾群体上得到扩增并进行基因分型。PCR 扩增体系为10 μL,包括PCR Mix 5 μL,ddH2O 4 μL,正反引物(10 μmol/L)各0.25 μL 和DNA 模板(100 ng/μL)0.5 μL。PCR 扩增程序为:95 ℃预变性5 min;95 ℃变性30 s,最适退火温度下退火30 s,72 ℃延伸30 s,35 个循环;72 ℃延伸5 min,最后4 ℃保存。PCR 扩增产物由武汉擎科生物有限公司在ABI 3730XL 遗传分析仪上进行分离,利用Gene Marker v1.75 软件读取各个体在各微卫星位点的基因型。
1.4 数据分析
本研究中形态数据以平均值和标准差(SD)的形式呈现。采用SPSS 23.0 进行单因素方差分析,通过公式CV=(M1-M2)/(S1+S2)[17]计算差异系数(CV),式中M1和M2表示某项形态参数的平均值,S1和S2为标准差。若CV 值小于1.28,则表示2 个群体在这项形态参数上的差异未达到亚种水平。使用SPSS 23.0 软件对6 项经过标准化的形态参数采用逐步判别的方法进行判别分析,在计算每个群体的6 项形态比例参数的平均值后,采用欧式最短距离系统聚类法进行聚类分析。
在读取各群体在各微卫星位点的基因型并校准后,使用Cervus 3.0 软件计算每个位点的等位基因数(number of alleles,Na)、观测杂合度(observed heterozygosity,Ho)、期望杂合度(expected heterozygosity,He)和多态信息含量(polymorphism information content,PIC),使用GenAIEx v6.51 软件计算每个位点的有效等位基因数(effective number of alleles,Ne)、固定指数(fixation index,Fst)及Nei’s 遗传距离(geneticdistance,DA)。并基于Nei’s 遗传距离用Mega7.0 构建UPGMA 系统进化树。
2 结果与分析
2.1 形态学分析
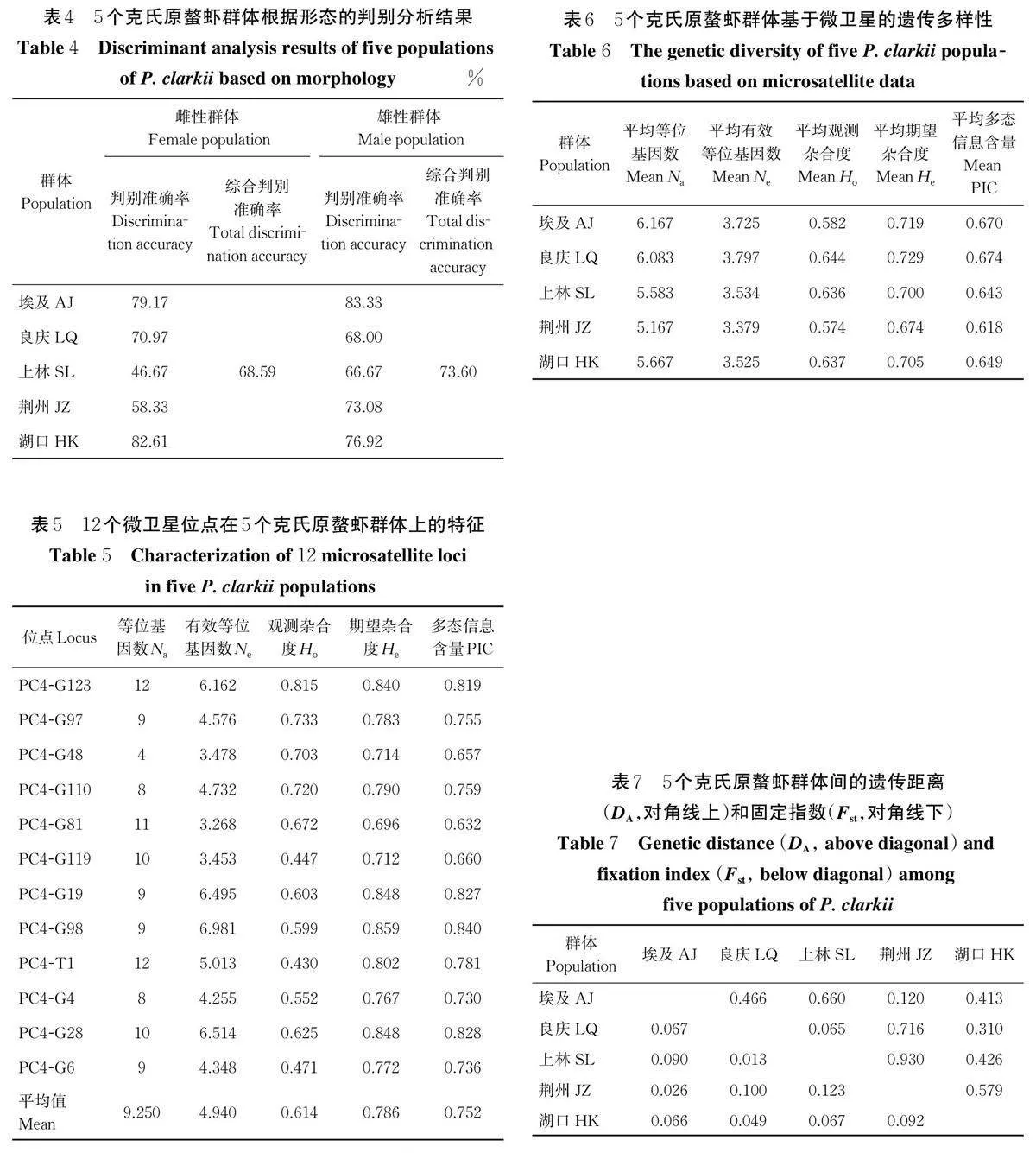
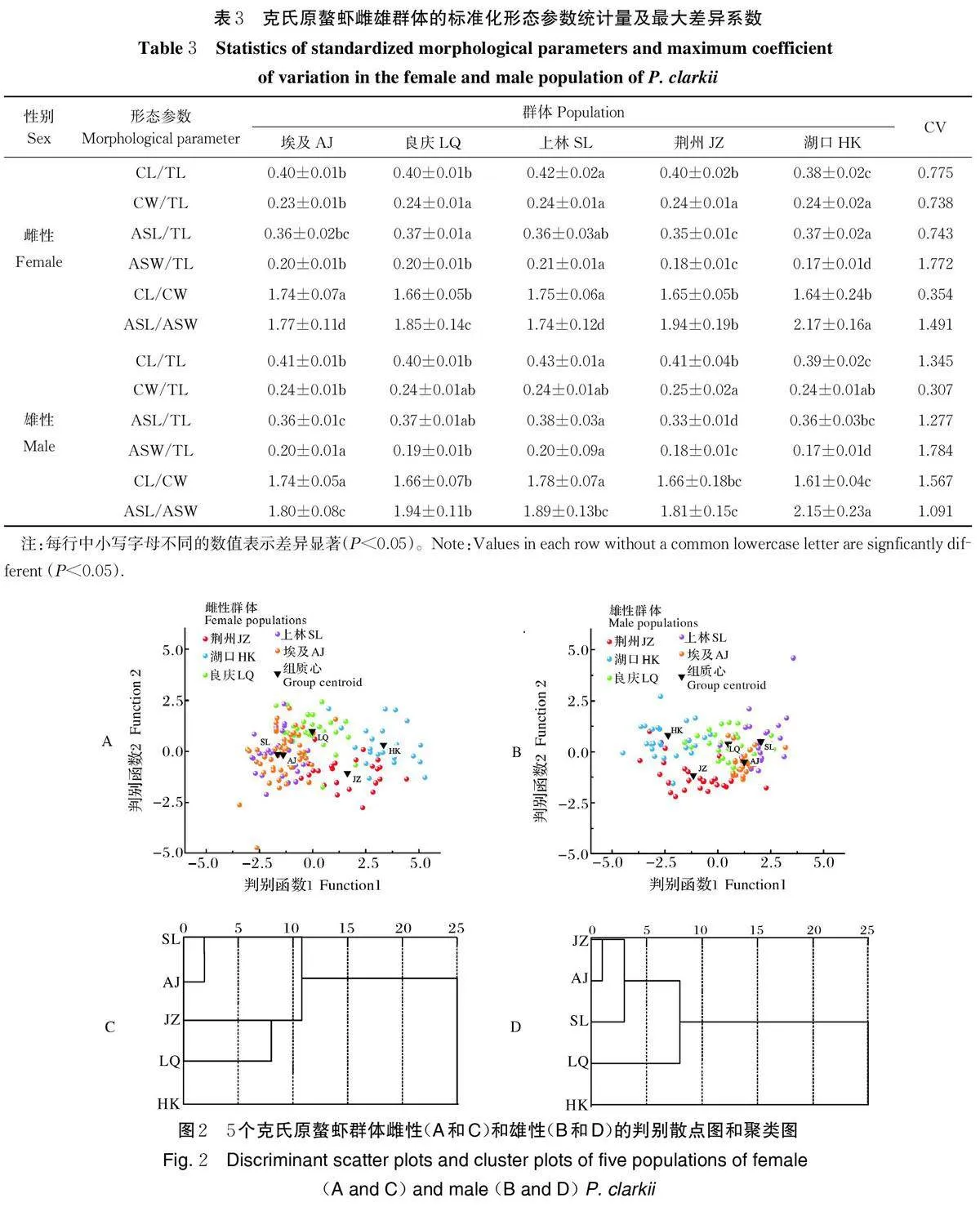
1)单因素方差分析。分别对克氏原螯虾雌雄群体的6 项形态比例参数进行单因素方差分析。结果表明,雌雄群体中,6 项形态比例参数在群体之间均存在显著性差异。对克氏原螯虾雌雄群体的形态比例参数进行差异分析,最大差异系数的结果显示,雌性群体中,在6 项形态比例参数中仅有2 项(ASW/TL 和ASL/ASW)的最大差异系数值大于临界值(1.28);而雄性群体中有3 项(CL/TL、ASW/TL 和CL/CW)的最大差异系数值大于临界值(表3)。群体之间的最大差异系数主要体现在上林群体和湖口群体之间,雄性群体间的形态差异大于雌性。
2) 判别分析。对克氏原螯虾雌雄群体6 项形态比例参数的判别F 检验结果表明,判别结果均较好(Plt;0.01)。利用前2 个判别函数绘制散点图可以更直观地表现出5 个克氏原螯虾群体的分离和重叠情况(图2 A、B)。雌雄群体的散点图显示了相似的结果:5 个克氏原螯虾雌性群体间存在不同程度的重叠,其中埃及群体和上林群体的重叠比例较大,相对难以区分,而良庆群体、荆州群体和湖口群体的重叠比例较小,相对容易区分。5 个克氏原螯虾雄性群体的散布趋势与雌性群体基于一致,但群体之间的重叠比例均较小。判别分析结果显示,雌性群体中,湖口群体的判别准确率最高为82.61%,而上林群体的判别准确率最低为46.67%,综合判别准确率为68.59%;雄性群体中,判别准确率最高的是埃及群体(83.33%),最低的仍是上林群体(66.67%),综合判别准确率为73.60%(表4)。
3)聚类分析。在形态比例参数平均值聚类分析中,雌性和雄性群体呈现出相似的分组(图2 C、D)。5 个群体被分为2 支,其中湖口群体单独聚为一支,其他4 个群体则聚为另一支。结果表明,埃及群体、上林群体和荆州群体的形态相似度较高,湖口群体与其他4 个群体的形态差异较大。
2.2 微卫星分析
1)群体遗传多样性分析。5个克氏原螯虾群体在12 个微卫星位点的等位基因数介于4~12 个(平均9.250 个),有效等位基因数介于3.268 ~ 6.981 个(平均4.940个),观测杂合度介于0.430~0.815,期望杂合度介于0.696~0.859,多态信息含量介于0.632~0.840(表5)。12 个微卫星位点均属于高度多态性位点(PICgt;0.5),可用于对克氏原螯虾遗传结构的分析。
5 个克氏原螯虾群体的遗传多样性参数如表6 所示。5 个群体的平均等位基因数在5.167~6.167,平均有效等位基因数在3.379~3.797,平均观测杂合度值在0.574~0.644,平均期望杂合度值在0.674~0.729,平均PIC 值在0.618~0.674。结果表明,5 个群体均具有较高的遗传多样性,具体表现为:良庆群体和埃及群体的遗传多样性最高,湖口群体和上林群体次之,荆州群体最低。
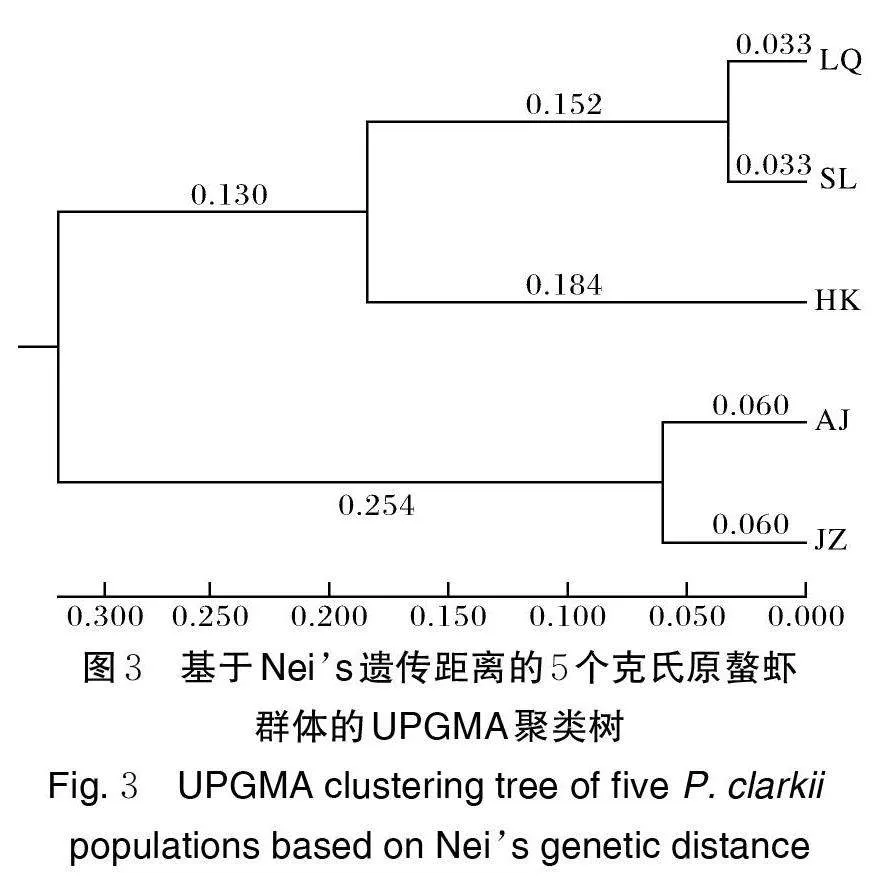
2)群体遗传分化分析。由表7 可见,5 个克氏原螯虾群体间发生了不同程度的分化。其中,上林群体和良庆群体间的固定指数(Fst)最小,其次是荆州和埃及群体(0.026)与良庆和湖口群体(0.049),均属于轻微分化水平(Fst lt;0.05),而其他群体间处于中度分化水平(0.05lt; Fst lt;0.15)。群体间遗传距离为0.065~0.930,结果显示,上林和良庆群体间的遗传距离最小(0.065),荆州和上林群体间的遗传距离最大(0.930)。
基于Nei’s 遗传距离,采用UPGMA 方法构建了5 个克氏原螯虾群体的聚类树。聚类结果显示,研究中涉及的5 个群体可分为两大分支(图3),埃及群体和荆州群聚类为一支,而良庆群体先与上林群体聚类为一支后再与湖口群体聚类为另一支。
3 讨论
形态学分析方法是研究物种遗传变异的有效方法之一[18]。在本研究中,单因素方差分析结果显示,6 项形态比例参数在群体之间均存在显著性差异,且最大形态差异主要体现在上林群体和湖口群体之间。判别分析结果显示,在281 尾克氏原螯虾样本中,有199 尾(70.82%)根据其外部形态被正确分类,分类正确率低于韩晓磊等[5]、张萌等[19]、徐滨等[6]的研究结果。这与本研究中使用的形态参数较少有很大关系。基于形态的聚类结果显示,埃及群体、上林群体和荆州群体的形态相似性最大,良庆群体与这3个群体聚为一支,而湖口群体单独聚为一支。聚类的结果与群体的地理分布的远近并无一致性,主要体现在荆州群体的异常分类。克氏原螯虾作为渔业产品,由于其经济的重要性被大量开发,并在世界范围内广泛养殖[20]。因此,其苗种或成虾可能通过贸易等其他非自然因素存在于地理位置较远的水域。Yue 等[21]的研究也表明,克氏原螯虾群体的遗传距离与其地理距离的远近并无显著相关性。遗传距离和固定指数反映了不同群体之间遗传关系的远近亲疏。不同群体间的固定指数越大,群体间的遗传分化水平就越高[10, 22],遗传距离越小,亲缘关系越近[23]。在本研究中,5 个克氏原螯虾群体的遗传距离趋势与遗传分化系数趋势一致。其中,埃及群体和荆州群体的遗传距离较小,亲缘关系较近,这与埃及群体与荆州群体的形态的相似性一致,具体原因有待进一步研究。良庆群体与上林群体的遗传距离最小,亲缘关系最近,表明两者的亲本在被引种前可能已产生了基因交流或两个群体在养殖过程中产生了基因交流。另外,良庆群体与其杂交亲本来源地的2 个早期野生群体的遗传距离较大,可能原因有:(1)因克氏原螯虾养殖热的兴起造成各地群体间的基因混杂,导致杂交亲本与早期野生群体间遗传差异较大;(2)杂交后的养殖群体在养殖过程中与其他群体产生了基因混杂。埃及群体作为一个从埃及引种的养殖群体,与上林群体和良庆群体的遗传距离较大,亲缘关系较远,但与生活在相近环境下的上林群体和良庆群体具有较高的形态相似度。这与甲壳动物较高的环境可塑性有关[24],此外,物种外部形态的变化也通常被解释为对栖息地环境的适应[25-26]。
物种的遗传多样性越高,对环境的适应能力越强,进化的潜力就越大。对物种遗传多样性的研究是育种研究的基础,明确其遗传多样性,获得理想的种质资源后才能更好地进行育种研究,提高育种效率[27-28]。本研究结果显示,12 个位点在5 个群体上均表现出高多态性(0.632lt;PIClt;0.840)[29],满足对克氏原螯虾遗传多样性分析的要求。杂合度表示位点上杂合子的频率,是对群体的遗传多样性评价的重要指标之一,杂合度越高,遗传变异程度越大。期望杂合度是指当种群处于平衡状态时的理论杂合子频率,与观测杂合度相比,能够更准确地反映群体的遗传多样性水平[10,30-31]。本研究中,12 个微卫星位点的期望杂合度平均值为0.786,表明群体杂合度水平较高,与以往的报道相比,高于江苏地区[32]、长江水系的一些群体[33-35]和广西地区的其他群体[36],与安徽地区[37-38]和原产地种群[39]相当。本研究中的5个克氏原螯虾群体均具有高水平的遗传多样性(0.674lt;Helt;0.840,0.618lt;PIClt;0.674),5 个群体的平均期望杂合度均远高于平均观测杂合度,说明在5 个克氏原螯虾群体中均存在杂合子丢失现象[40],这可能是由于群体的规模较小且出现了瓶颈效应,产生了遗传漂变等原因,导致了等位基因丢失[36]。
综上所述,本研究基于12 个微卫星位点对克氏原螯虾的养殖群体的遗传多样性进行评估,结果表明广西南宁周边的克氏原螯虾养殖群体具有较高的遗传多样性,从国外引种或国内不同地理来源群体的杂交可能是提高克氏原螯虾群体遗传多样性的重要途径,该研究为克氏原螯虾的综合养殖和科学育种提供了参考资料。
(责任编辑:边书京)

