一株反硝化惠氏副球菌的分离鉴定及脱氮特性研究


摘""要:"在城镇生活污水处理中,氮(N)的高效去除已成为一个重要的内容. 其中反硝化是脱氮的关键步骤. 研究筛选了一株惠氏副球菌(Paracoccus huijuniae),并通过生物强化的方法提高其反硝化的速率. 以普通反硝化污泥作为对照,将P."huijuniae加入到普通硝化污泥进行生物强化,在相同操作条件下,NO3--N的去除速率可以提高34%. 高通量测序结果表明:由于P."huijuniae的加入,具有反硝化功能的微生物菌属的丰度得到明显提高. 对P."huijuniae的全基因组测序可知,P."huijuniae包含有多个反硝化的关键基因,正是因为这些基因的加入,使反硝化速率明显加速.
关键词:"惠氏副球菌(Paracoccus huijuniae);"反硝化;"生物强化
中图分类号:"X 522 """文献标志码:"A """文章编号:"1000-5137(2024)04-0564-07
Isolation,"identification,"and the denitrification characteristics for a denitrifying strain:"Paracoccus huijuniae
HUANG Meng,"CHEN Songyun,"ZHANG Haiyun,"GAO Xiaobing,"ZHANG Yongming
(School of Environmental and Geographical Sciences,"Shanghai Normal University,"Shanghai 200234,"China)
Abstract:"Nitrogen (N)"removal become an important topic in municipal wastewater treatment,"in which denitrification seems more important step. A denitrifying strain,"Paracoccus huijuniae,"was isolated and was employed for accelerating denitrification by means of bioaugmentation. When"P."huijuniae"was added into common denitrifying sludge for denitrification,"NO3--N removal rate was increased by 34% compared with common denitrifying sludge. The results of high throughput sequencing indicated that abundance of genera with denitrification function increased obviously after P."huijuniae"was added into common denitrifying sludge. Meanwhile,"the whole genome sequencing indicated that P."huijuniae"contains multiple key genes for denitrification. This led a significant acceleration for denitrification rate.
Key words:"Paracoccus huijuniae;"denitrification;"bioaugmentation
0 "引言
总氮(TN)的去除在城市污水处理中变得越来越重要[1-2]. 不断有关于各种新型脱氮工艺的报道,其中短程硝化反硝化(PND)[3-4]和厌氧氨氧化(Anammox)[5-6]等是其中典型的代表. 但传统硝化、反硝化工艺仍然是当前城市污水处理的主流方法. 硝化是去除TN的第一步[7],而反硝化显得更为重要. 一般硝化反应只要确保足够的供氧量和适宜的泥龄,便能确保硝化反应顺利进行. 而反硝化反应则既要合适的缺氧条件,又要必要的有机碳源作为电子供体[8-9]. 因为碳源与污水处理成本密切相关,所以寻找低成本的碳源便成为控制污水处理成本的重要一环. 例如,有人使用具有高浓度生化需氧量(BOD)的工业废水[10-11]. 然而,提高反硝化的效率并不仅仅局限在利用经济的碳源,研究人员还在不断探索通过其他方法,例如生物强化的方法来提高反硝化的效率. TAN等[12]通过红球菌的生物强化使反硝化反应加速进行,同时还成功地将喹啉作为电子供体.
当采用生物强化来提高TN去除率时,有必要了解生物强化菌株所起的作用. 例如,KUYPERS等[13]通过研究发现,反硝化反应之所以得到加速,是因为生物强化菌株的丰度提高,其中的硝酸盐还原酶(NAR)和亚硝酸盐还原酶(NIR)起到了非常大的作用.
本研究的作者筛选得到了一株惠氏副球菌(Paracoccus huijuniae),并利用其来加速以丁二酸为电子供体的反硝化反应. 通过对该菌株的全基因组测序,发现该菌株中包含有多种NAR和NIR. 该项研究对采用生物强化加速反硝化反应具有重要的理论和现实意义.
1 "材料与方法
1.1 化学试剂和培养基的制备
本研究中所有化学药品均购置于上海泰坦科技股份有限公司. 试剂制备中的溶剂均使用电阻率为18.2 MΩ·cm的超纯水. 实验母液的配制:将30.35 g硝酸钠(NaNO3)、16.03 g丁二酸(C4H6O4)、28.21 g碳酸氢钠(NaHCO3)和12.32 g亚硝酸钠(NaNO2)溶解于250 mL的超纯水中.
低盐培养基(MSM)的制备:将4 g硫酸铵((NH4)2SO4)、3 g十二水合磷酸氢二钠(Na2HPO4"∙12H2O)、3 g磷酸二氢钾(KH2PO4)、0.196 g硫酸镁(MgSO4)、0.015 g氯化钙(CaCl2)和0.002 g七水合硫酸亚铁(FeSO4∙7H2O)加入到由2 L纯水组成的2 L容量瓶中.
不含铵的MSM的制备:在2 L的容量瓶中加入0.6 g Na2HPO4"∙12H2O,0.6 g KH2PO4,0.331 6 g六水合氯化镁(MgCl2"∙6H2O),0.015 2 g CaCl2和0.001 4 g四水合氯化亚铁(FeCl2∙4H2O),用纯水稀释到2 L.
Luria Bertani(LB)的制备:将溶解在纯水中的10 g胰蛋白酶、10 g氯化钠(NaCl)和5 g酵母提取物加入1 L容量瓶中,用纯水稀释到1 L.
固体培养基的制备:在基础MSM培养基中加入1.5~2.0 g的琼脂,形成固体培养基,用于分离纯反硝化细菌.
上述培养基均在121 ℃和0.10~0.15 MPa下灭菌20 min,所有溶液和培养基在使用前储存在4 ℃的冰箱中.
1.2 反硝化污泥的驯化
原始污泥取自上海长桥污水处理厂二沉池. 驯化前,对污泥清洗3次,以去除其中的杂质[14-15]. 将体积为400 mL的洗涤污泥加入1 L锥形烧瓶中,加入丁二酸作为反硝化活性污泥驯化的电子供体. 期间每天用新配制的液体替换反硝化液. 经过约30 d的驯化,得到成熟的反硝化污泥.
1.3 反硝化菌的筛选鉴定
取5 mL驯化的反硝化污泥的上清液在试管中稀释10倍,将200 μL的稀释溶液滴在培养皿中的无菌固体培养基上,并使用无菌玻璃棒将液滴均匀地涂布于固体培养基的表面. 随后,将该培养皿置于30 ℃的培养箱中,用于生长和菌落形成. 24 h后用无菌环挑选单个菌落,通过划线方法,经过10次重复,获得了一株纯菌,并将该纯菌接种到斜面上. 然后,将斜面送往上海派森诺生物科技股份有限公司进行菌种鉴定,所用引物分别是27F(5-AGAGTTGATCCTGTCTCCAG-3)和1492R(5-GGTTACCTTGTTACG ACTT-3). 用AxyPrep DNA凝胶回收试剂盒回收PCR产物,并用测序仪(ABI3730-XL)对纯化的PCR产物进行测序. 采用NCBI Blast程序将序列文件与NCBI 16S数据库中的数据进行比较,以获得基于最大序列相似性的物种识别. 最终分离到的细菌被鉴定为P."huijuniae,其最大序列相似性为99.34%,其基因登录号为NR_108224.1.
1.4 反硝化实验
反硝化实验分别用两种方法进行. 第一种是直接利用驯化的反硝化污泥进行,污泥的干重为1.0 g·L-1;第二种方法则是在反硝化污泥中加入P."huijuniae,污泥和P."huijuniae的干重分别是0.9 g·L-1和0.1 g·L-1,即所加P."huijuniae的干重占总重量的10%. 初始硝酸盐氮(NO3--N)的N的质量浓度为50 mg·L-1,以丁二酸作为电子供体进行反硝化,其加入量按照质量比为m(化学需氧量(COD))︰m(N)= 5︰1的比例加入. 即加入丁二酸之后,初始的COD为250 mg·L-1. 其中,P."huijuniae采用LB培养基进行扩增以用于反硝化反应.
将反硝化污泥或纯菌,以及硝酸盐、丁二酸等置于锥形瓶,并将其放入恒温摇动培养箱中,在30 ℃和150 r·min-1下进行反硝化实验. 所有的反硝化实验均是在密封(缺氧)条件下进行. 在实验之前,每个锥形瓶均用高纯氮气(N2)充气5 min以脱除瓶中的氧,使瓶中的溶解氧(DO)质量浓度小于0.5 mg·L-1. 实验过程中每间隔一定时间取样,分别测试NO3--N,NO2--N和总有机碳(TOC)浓度.
1.5 高通量测序
反硝化结束后,立即将2个样品污泥样品,即反硝化污泥加P."huijuniae(DS_P)和单独反硝化污泥(DS)送上海派森诺生物科技股份有限公司进行高通量测序. DNA提取和测序的步骤与ZHU等[15]描述的相同. 用NanoDrop 2000检测DNA浓度和纯度,用1%(质量分数)琼脂糖凝胶电泳检测DNA提取物的质量. 高通量测序所用上下游引物分别为338F (5’-AACTCCTACGGGAGGCAGCA-3’)和806R(5’- GACTACHVGGGTWTCTAAT-3’). PCR扩增区间为该细菌16S rRNA基因V3~V4区间.
1.6 对P. huijuniae全基因组测序
将P. huijuniae接种在含有LB培养基的锥形烧瓶中,并将烧瓶置于恒温培养箱中,在温度30 ℃和转速180 r·min-1条件下培养,直到P. huijuniae进入对数生长期(约36 h). 以10 000 r·min-1的转速离心8 min收集培养的菌株. 随后,用无菌水洗涤收集菌株3次以去除残留的底物. 洗涤后的样品被送往上海美吉生物医药科技有限公司(Majorbio)进行基因组学分析. 在此期间,根据土壤DNA试剂盒(Omega Bio-tek,Norcross,GA,U.S. )的说明提取菌株的总DNA. 使用PacBio Sequel IIe(PacBio,USA)和Illumina NovaSeq6000(Illumina,USA)测序仪的组合,采用原核生物的标准方案[16],对P. huijuniae进行完全测序. 将通过Illumina测序获得的配对末端原始读数映射到由PacBio测序读数组装的完整基因组序列,以确保准确性和完整性. Prodigal v2.63用于预测基因组中的编码序列(CDS),并使用序列比对工具(BLASTP、Diamond和HMMER等)对其进行注释.
1.7 分析方法
每个样品在测试前,均经过孔径为0.22 μm的膜过滤. NO2--N and NO3--N采用型号为Dionex ICS-5000的离子色谱测试. TOC的分析采用型号为SHIMADZU TOC-L CPN的TOC分析仪.
2 "结果与讨论
2.1 P."huijuniae对反硝化速率的影响
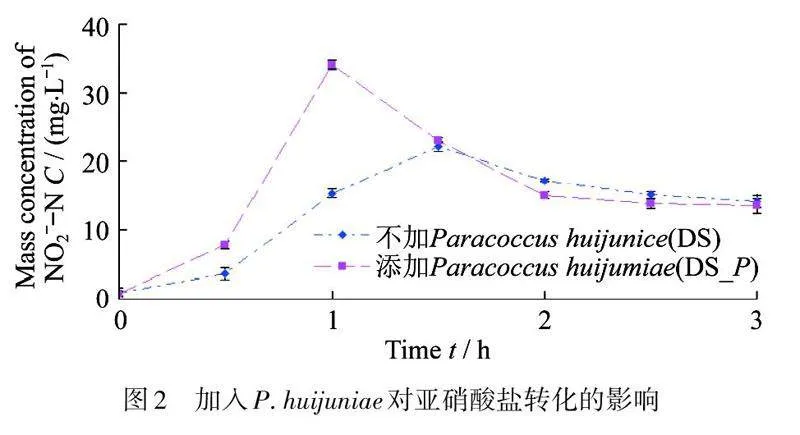
分别用驯化的反硝化污泥和反硝化污泥加P."huijuniae,NO3--N的去除情况如图1所示. 从图1中可以看出,NO3--N的去除速率符合零级反应动力学规律. 比较2种方法可以看出,加入了P."huijuniae后,NO3--N的去除速率提高了34%,这说明通过生物强化的方法,即加入P."huijuniae后,NO3--N的去除速率得到明显提高. 通常反硝化过程主要经过两步进行,即先转化为NO2--N,随后再转化为N2. 从图2中可以看出,加入了P."huijuniae后,NO2--N质量浓度先升高,随后降低. 但加入了P."huijuniae后的反硝化体系,在最初的2 h,NO2--N的质量浓度迅速升高,其生成速率比未加P."huijuniae的反硝化体系的生成速率快1.2倍. 根据推测,这一结果是由于具有反硝化功能菌属的含量增加的原因.
2.2 反硝化过程中电子供体的消耗
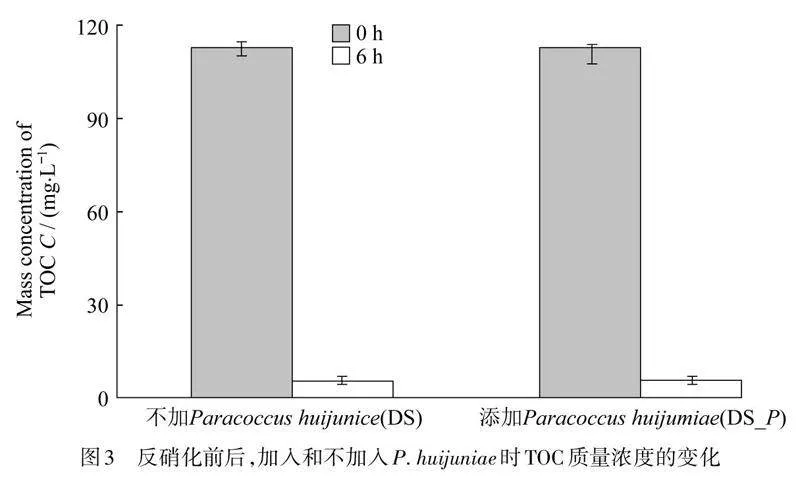
在2种反硝化体系中,反应前和反应后TOC的变化情况如图3所示. 根据图3所示,添加和未加P."huijuniae的反硝化系统,在反硝化过程中,TOC质量浓度的变化几乎一致. 这说明加入了P."huijuniae的反硝化DS_P体系中,电子利用的效率要高于未加入P."huijuniae的反硝化DS体系. 因此可以推断,在DS_P体系中具有反硝化功能的菌属其丰度更高.
2.3 加入P."huijuniae前后反硝化污泥系统中微生物群落结构分析
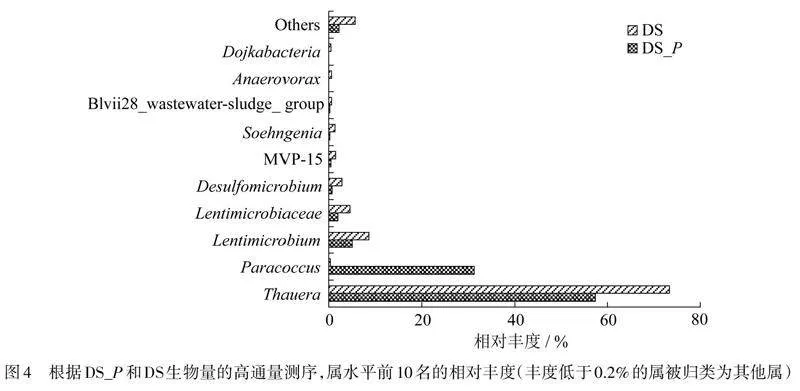
反硝化污泥在加入P."huijuniae前后微生物群落的变化情况如图4所示,是丰度在前10位的微生物丰度的汇总. 丰度小于0.2%的细菌汇总于其他属(Others)中. 其中相对丰度最高的是Thauera属. 加入P."huijuniae之前,Thauera的相对丰度为73%,而加入P."huijuniae之后,其相对丰度降至57%,其相对丰度下降值为16%. Thauera是典型的反硝化细菌,经常被选为反硝化的模式生物[17]. Thauera可以同时利用可溶性微生物产物(SMP)和细胞外聚合物(EPS)作为反硝化的电子供体[18].
当P."huijuniae加入到反硝化污泥系统后,DS_P中Paracoccus的丰度显著增加,净增加值高达26%. 有报道称,Paracoccus在PND过程中发挥着重要作用,其中Paracoccus可以使NO2--N积累率达到81.9%[19]. 其他微生物的丰度明显小于Thauera和Paracoccus这2种菌属. 因此,Thauera和Paracoccus这2种菌属之和在DS和DS_P中的丰度分别是74%和89%. 它们也主导了DS和DS_P系统的反硝化反应. 这说明,即使加入P."huijuniae之后,菌属Thauera的丰度有所下降,但具有反硝化功能的菌属总的丰度还是增加了许多. 这些微生物群落的变化说明正是由于P."huijuniae的加入,使NO3--N的去除速率明显提高. 此外,加入P."huijuniae的反硝化污泥中,"Lentimicrobiaceae"的丰度只有未加入了P."huijuniae的反硝化污泥的43%. 而Lentimicrobiaceae是降解硬脂酸并产甲烷的关键细菌[20],"即硬脂酸的降解导致TOC浓度的下降,但其主要产物是甲烷,但并没有释放出电子供体用于反硝化. 该实验结果也证明了前述的推测.
2.4 P. huijuniae"的基因分析
基因组测序的基因注释表明了NIR和NAR分别处于2个簇上. 结果如图5所示,NIR位于第一簇的767 000~768 000 bp之间,而NAR位于第二簇的1 168 000~1 177 000 bp之间. 第一簇包含有亚硝酸盐的还原酶(nirK);第二簇包含有多个NAR(narI,narJ,narH,anrG和narK). 比较而言,NIR只在第一簇上只有一种功能基因(nirK),而NAR则在第二簇上拥有5种硝酸盐还原功能基因. 这些功能基因能够将NO3--N转化为NO2--N[21]. 结合前述的实验结果(图1和图2),P. huijuniae之所以可以加速反硝化速率,是因为P. huijuniae中NAR在反硝化过程中起了重要的作用.
3 "结"论
从反硝化污泥中筛选的细菌P."huijuniae可以明显地加速普通的反硝化污泥脱氮速率. 相比未添加P."huijuniae反硝化污泥系统DS,添加P."huijuniae后,DS_P系统可以使NO3--N的去除速率提高34%. 这是因为DS_P系统中,具有反硝化功能的微生物菌属的丰度得到明显升高. 更为关键的是在P."huijuniae中,包含有硝酸盐还原功能基因和亚硝酸盐还原的功能基因,在P."huijuniae的作用下,可以有效地利用有机碳源所释放的电子,因而加速反硝化的反应速率.
参考文献:
[1] JIANG C,"XU S,"WANG R,"et al. Achieving efficient nitrogen removal from real sewage via nitrite pathway in a continuous nitrogen removal process by combining free nitrous acid sludge treatment and DO control [J]. Water Research,"2019,161:590-600.
[2] FU X,"HOU R,"YANG P,"et al. Application of external carbon source in heterotrophic denitrification of domestic sewage:"a review [J]. Science of the Total Environment,"2022,817:153061.
[3] JI Z,"CHEN Y. Using sludge fermentation liquid to improve wastewater short-cut nitrification-denitrification and denitrifying phosphorus removal via nitrite [J]. Environmental Science amp; Technology,"2010,44(23):8957-8963.
[4] KANG D,"YUAN Z,"LI G,"et al. Toward integrating EBPR and the short-cut nitrogen removal process in a one-stage system for treating high-strength wastewater [J]. Environmental Science amp; Technology,"2023,57(35):13247-13257.
[5] ADAMS M,"ISSAKA E,"CHEN C. Anammox-based technologies:"a review of recent advances,"mechanism,"and bottlenecks [J]. Journal of Environmental Science,"2025,148:151-173.
[6] LI W,"LI X,"ZHANG Q,"et al. Recent advances of partial anammox by controlling nitrite supply in mainstream wastewater treatment through step-feed mode [J]. Science of the Total Environment,"2024,912:168965.
[7] WU X,"YU Z,"YUAN S,"et al. An ecological explanation for carbon source-associated denitrification performance in wastewater treatment plants [J]. Water Research,"2023,247:120762.
[8] CAO Q,"LIU X,"RAN Y,"et al. Methane oxidation coupled to denitrification under microaerobic and hypoxic conditions in leach bed bioreactors [J]. Science of the Total Environment,"2019,649:1-11.
[9] TAN C,"ZENG Q,"ZHU G,"et al. Characteristics of denitrification in a vertical baffled bioreactor [J]. Environmental Research,"2021,197:111046.
[10] CHEN S,"ZHU X,"ZHU G,"et al. N-methyel pyrrolidone wastewater as electron donor for denitrification:"from bench to pilot scale [J]. Science of the Total Environment,"2024,912:169517.
[11] MA Y,"TAN C,"CHEN S,"et al,. Soybean-processing wastewater as an electron donor for denitrification [J]. International Biodeterioration amp; Biodegradation,"2024,188:105745.
[12] TAN C,"CHEN S,"ZHANG H. et al. The roles of Rhodococcus ruber"in denitrification with quinoline as the electron donor [J]. Science of the Total Environment,"2023,902:166128.
[13] KUYPERS M M M,"MARCHANT H K,"KARTAL B. The microbial nitrogen-cycling network [J]. Nature Reviews Microbiology,"2018,16(5):263-276.
[14] ZHU G,"XING F,"TAO J,"et al. Synergy of strains that accelerate biodegradation of pyridine and quinoline [J]. Journal of Environmental Management,"2021,285:112119.
[15] ZHU G,"ZHANG Y,"CHEN S. How bioaugmentation with Comamonas testosteroni"accelerates pyridine mono-oxygenation and mineralization [J]. Environmental Research,"2021,193:110553.
[16] WICK R R,nbsp;JUDD L M. GORRIE C L,"et al. Unicycler resolving bacterial genome assemblies from short and long sequencing reads [J]. Cold Spring Harbor Laboratory,"2016(6):1-20.
[17] WEI Q,"ZHANG J,"LUO F,"et al. Molecular mechanisms through which different carbon sources affect denitrification by Thauera linaloolentis:"electron generation,"transfer,"and competition [J]. Environmental Internation,"2022,170:107598.
[18] FAN J,"DU R,"LIV C L,"et al. Inducing high nitrite accumulation via modulating nitrate reduction power and carbon flux with Thauera"spp. Selection [J]. Bioresource Technology,"2022,354:127188.
[19] LUAN Y,"YIN Y,"GUO Z et al. Partial nitrification-denitrification and enrichment of paracoccus"induced by iron-chitosan beads addition in an intermittently-aerated activated sludge system [J]. Journal of Environmental Management,"2024,353:120189.
[20] AGHASA A,"CHOI S,"LEE J,"et al. Effect of initial bacterial diversity on anaerobic degradation of long-chain fatty acids [J]. Biomass and Bioenergy,"2022,162:106498.
[21] XIE C,"ZHANG Q,"LI X,"et al. Highly efficient transformation of slowly-biodegradable organic matter into endogenous polymers during hydrolytic fermentation for achieving effective nitrite production by endogenous partial denitrification [J]. Water Research,"2023,230:119537.
(责任编辑:郁慧)

