凡纳滨对虾(Litopenaeus vannamei)野生群体与养殖群体的线粒体遗传变异分析



摘 要:为研究凡纳滨对虾(Litopenaeus vannamei)的母系遗传背景以及遗传特性,运用线粒体基因( mtDNA) D-loop区分子标记技术,对凡纳滨对虾野生群体与养殖群体的遗传变异进行了比较分析。结果显示:野生群体与养殖群体的碱基组成一致,A+T的含量(81.52%)明显高于C+G的含量(18.48%);野生群体的单倍型多样性(Hd)为0.960±0.017,核苷酸多样性(π)为0.104±0.051,明显高于养殖群体的(Hd=0.680±0.075,π= 0.060±0.029)。经分子方差分析(AMOVA)发现,种群间的遗传变异率为13.09%,群体内的遗传变异率为86.91%,野生凡纳滨对虾群体与养殖群体的遗传分化存在显著差异(F st=0.131,P< 0.05)。本研究结果可为凡纳滨对虾的种质资源相关研究提供理论依据。
关键词:凡纳滨对虾;野生群体;养殖群体;mtDNA;遗传多样性
凡纳滨对虾(Litopenaeus vannamei)隶属于节肢动物门(Arthropoda)、甲壳动物亚门( Crustacea)、软甲纲(Malacostraca)、十足目(Decapoda)、对虾总科(Penaeoidea)、对虾科(Penaeidae)、滨对虾属(Litopenaeus),原产于南美洲太平洋沿岸水域,主要分布在秘鲁北部至墨西哥桑诺拉(Sonora)沿海一带,以厄瓜多尔沿岸分布最为集中。我国在1988年从美国引进凡纳滨对虾,发展至今已逾30年。该虾具有生长快、抗病性强等特点,因而获得大规模推广养殖,现已经成为当今世界三大养殖产量最高的品种之一 [1]。
相对于同工酶、RAPD等其他分子标记, DNA序列分析在用于群体遗传多样性分析时,其结果更直接、更准确和可靠 [2],而且线粒体基因(mtDNA)具有结构简单、进化速度快、呈母系遗传、种群间的遗产差异容易检出等特点 [3-6],其中关于12S rRNA、16S rRNA、COI,D-loop等基因片段的研究较多。水产种质资源发掘与利用教育部重点实验室于2016年运用mtDNA D-loop区分子标记对凡纳滨对虾养殖群体的遗传多样性进行了分析 [7],鉴于在全世界范围内凡纳滨对虾长期选育和养殖可能引起的种质退化问题,实验室在引进厄瓜多尔野生群体后,再次运用mtDNA D-loop区分子标记技术,对野生凡纳滨对虾以及人工养殖凡纳滨对虾群体进行了比较分析,以期揭示野生凡纳滨对虾与经长期人工选育和养殖的凡纳滨对虾群体之间在遗传多样性及遗传结构方面存在的差异,为凡纳滨对虾种质资源相关研究提供理论依据。
1 材料和方法
1.1 试验材料
野生凡纳滨对虾群体于2016年从厄瓜多尔引进后,暂养于上海市金山区,从中随机挑选51尾用于试验。养殖群体来自本实验室自2014到2017年保存的9个选育群体样品,从中随机选取51尾用于试验。全部试验样本取肌肉放在无水乙醇中于4 ℃下保存。采用海洋动物组织基因组DNA提取试剂盒[天根生化科技(上海)有限公司]提取基因组DNA。
1.2 引物序列与PCR扩增
以凡纳滨对虾mtDNA D-Loop区为标记,根据已公布的凡纳滨对虾mtDNA全序列设计引物,引物信息见表1。所有引物均由生工生物工程(上海)股份有限公司合成。
PCR反应体系为:总体积50 μL,其中2×Taq MasterMix混合液25 μL,上、下游引物(10 pmol/μL)各1 μL,DNA模板(100 pmol/μL)1 μL,ddH2O 22 μL。反应条件为:94 ℃预变性5 min;94 ℃变性1 min,58 ℃退火1 min,72 ℃延伸1 min,共35个循环;72 ℃延伸10 min;4 ℃保存。mtDNA扩增产物经1%的琼脂糖凝胶电泳,选择条带清晰的送至生工生物工程(上海)股份有限公司测序。
1.3 数据分析
将测序结果用BioEdit软件进行对比,去除多余序列,并以已发表的线粒体全基因序列( GenBank:EF584003)为对照,通过NCBI进行序列同源性比对,用软件DnaSP v5分析碱基组成、核苷酸保守位点、多态位点,以及x单倍型多样性(haplotype diversity,Hd)、核苷酸多样性(nucleotide diversity,π)和平均核苷酸差异数(average number of nucleotide differences,k),软件Arlequin v3.1基于Kimura 2P模型分析群体内与群体间分子方差(analysis of molecular variance,AMOVA),用MEGA 4.0软件建立单倍型进化树。
2 结果
2.1 扩增结果
凡纳滨对虾的mtDNA D-Loop区扩增产物如图1所示,目的条带清晰。试验中共获得102条野生对虾与养殖对虾的线粒体控制区序列,养殖群体的线粒体控制区序列大小在716~744 bp,野生群体的线粒体控制区序列大小在691~728 bp。
2.2 核苷酸变异与单倍型分布
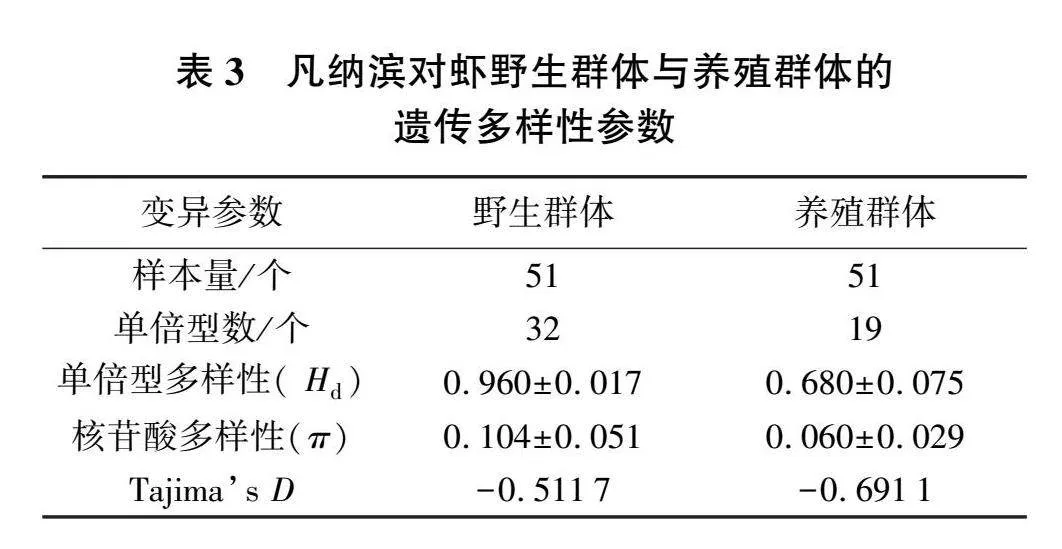
将凡纳滨对虾野生群体与养殖群体的扩增产物序列进行比较,结果见表2。在碱基组成上,mtDNA D-Loop区的碱基序列都富含A、T,碱基A、C、G、T的平均含量分别为34.80%、10.61%、7.87%、46.72%。其中A+T含量占总碱基的 81.52%,C+G含量占总碱基的18.48%。野生群体中,有效序列752 bp,其中含330个保守位点和422个多态位点。野生群体中简约信息位点197个,其中插入/缺失位点98个,发生转换241个,颠换175个,转换与颠换的比值(R=Si/Sv)为 1.38。在养殖群体中,有效序列754 bp,其中含485个保守位点和269个多态位点。养殖群体中简约信息位点104个,其中插入/缺失位点65个,发生转换119个,颠换116个,转换与颠换比值(R=Si/Sv)为1.03。
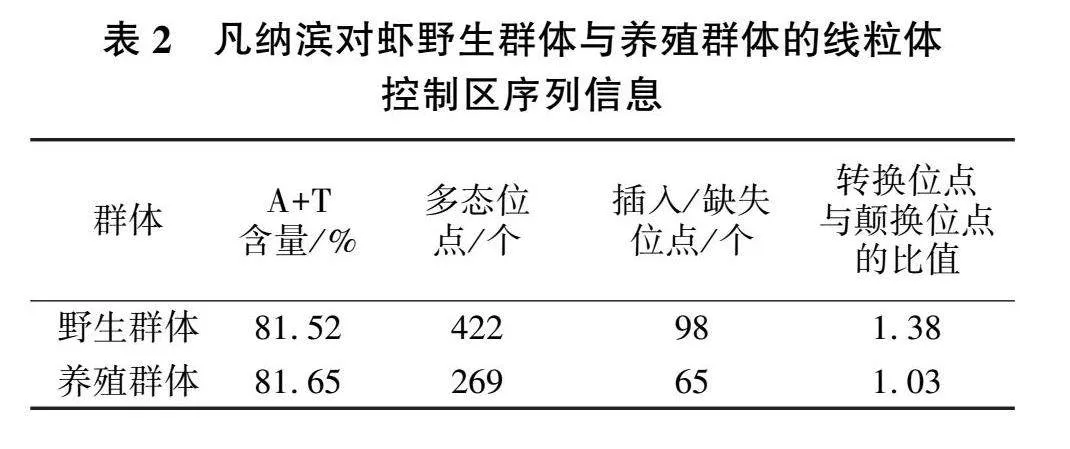
采用DnaSP v5软件对养殖群体和野生群体的线粒体控制区序列进行分析,在389个变异位点中发现了49个单倍型(见图2和表3)。野生群体有32个单倍型,其中30个为野生群体特有的单倍型;养殖群体有19个单倍型,其中17个为养殖群体特有的单倍型,2个为野生群体和养殖群体共享单倍型,两个群体合计共有49个单倍型。由图2可以看出,养殖群体的单倍型主要聚为3簇,而野生群体的单倍型基本呈均匀分布。
2.3 遗传多样性
遗传多样性分析结果见表3。野生群体的单倍型指数(Hd)为0.960±0.017,核苷酸多样性指数(π)为0.104±0.051;养殖群体的单倍型指数为0.680±0.075,核苷酸多样性指数为0.060± 0.029,说明野生群体的遗传多样性远高于养殖 群体。
2.4 遗传分化与遗传距离
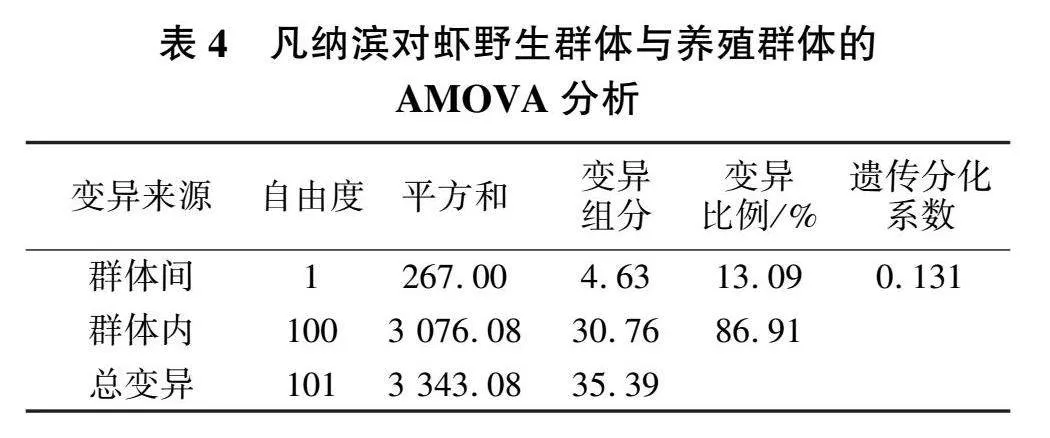
由表4的结果可知,群体间的遗传变异率为13.09%,群体内的变异为86.91%,可见凡纳滨对虾遗传变异的主要来源为种群内变异。遗传分化系数F st=0.131(P<0.05),说明野生群体与养殖群体已发生遗传分化。基因流(Nm=1.590)越小,表明遗传分化程度越高,两者共同阐明了两个群体之间较高的遗传分化水平。遗传距离结果显示,野生群体与养殖群体间的遗传距离为0.107,野生群体内的平均遗传距离为0.120,养殖群体内的平均遗传距离为0.068。
2.5 系统发育树
将野生群体和养殖群体的49种单倍型进行贝叶斯聚类分析,并用MEGA 5软件构建系统进化树,结果见图2。单倍型进化树结果显示: Hap_5、6、7、16、20、22、23、31、41、42、45是单独成群,其中Hap_5、6、7、16、20、22、23、31为野生群体所特有的单倍型,Hap_41、42、45为养殖群体所特有的单倍型。其他38个单倍型存在分支,养殖群体中的Hap_19、34、35、38、39、43、44、46、47、49亲缘关系较近,且与野生群体中的Hap_1、8、27聚为一支,Hap_33、36、37、40与野生群体中的Hap_3亲缘关系较近,养殖群体的Hap_48与野生群体的Hap_32聚为一支,亲缘关系较近。
3 讨论
目前凡纳滨对虾遗传育种的亲虾大多来自于引进群体,因此在凡纳滨对虾的育种计划中已有很多关于养殖群体的遗传背景分析。由于凡纳滨对虾原产地对种质资源的严格控制,mtDNA控制区序列进化较快 [8],是进行甲壳类种群内遗传多样性分析的理想分子标记。本研究是国内首次采用线粒体标记对凡纳滨对虾野生群体与养殖群体进行遗传比较分析,结果表明,野生凡纳滨对虾群体与养殖群体的遗传分化存在显著差异。
4种核苷酸(A、T、C、G)在线粒体基因组中的分布呈不均一性,是动物线粒体基因组的一个共性 [9]。本研究显示,凡纳滨对虾的野生群体与养殖群体在碱基组成上呈现一致现象,即组成中 A+T含量明显高于G+C含量,这与线粒体普遍存在的高AT碱基的规律相符 [10]。
从遗传学角度来说,物种遗传多样性水平的高低与其适应能力、生存能力和进化潜力密切相关 [11-14],群体遗传多样性每损失10%,会对其繁殖能力、存活率、生长等重要生理性状产生极大的负面影响 [15]。有研究表明,群体中线粒体DNA的核酸多样性(π)和单倍体多样性(Hd)是衡量群体多态程度和群体遗传分化的重要指标,π、Hd值越大,表示群体多态程度越高 [16],当核苷酸多样性指数在0.001 0~0.004 7时,其群体的遗传多样性较低 [17]。由表2可见,凡纳滨对虾野生群体的核酸多样性指数明显高于其他物种,如银鲳 [18](Pampus argenteus)、贻贝 [19](Mytilus coruscus)、许氏平鲉(Sebastes schlegeli) [20]、条石鲷 [21](Oplegnathus fasciatus)等,是上述物种野生群体核酸多样性的5~16倍,而团头鲂 [22](Megalobrama" amblycephala)野生群体的核酸多样性是本研究的3倍。以上结果说明,不同物种间核酸多样性丰富度不同也可能是采集样品地点不同导致的。本试验的野生群体捕捞自凡纳滨对虾的原产地厄瓜多尔,该群体可能保留着更丰富的核酸多样性;Sunden等 [23]研究发现,凡纳滨对虾野生群体的核酸多样性指数是其养殖群体的1.36~20.00倍,而本研究中,野生群体的核酸多样性(0.10)是养殖群体(0.06)的1.7倍;日本囊对虾 [24](Marsupenaeus japonicus)野生群体的核酸多样性是4个养殖群体的4~10倍,可能原因有物种本身核酸多样性丰富度的不同、亲本选择的数量以及对养殖环境的适应能力不同等;与刘红等 [7]研究的6个凡纳滨对虾养殖群体的遗传多样性相比,本研究结果与之相似。研究证实,养殖群体的核酸多样性低于野生群体,mtDNA遗传多样性在养殖群体中显著降低,这已成为养殖群体的遗传特征 [23]。人工养殖的亲本数量的选择、繁殖能力、存活率以及繁育过程中性状的选择等都会在很大程度上影响种群的遗传多样性。Verspoor [25]和Winans [26]认为,当群体有效亲本数量小于100时,会降低该群体的遗传多样性。
Li等 [27]认为,养殖群体繁殖亲本数太少易导致遗传漂变加剧,遗传漂变往往伴随着瓶颈效应,从遗传学的角度来讲,瓶颈效应的发生首先表现为稀有单倍型的消失,随着瓶颈效应的加剧就会表现出单倍型多样性的降低,继而会造成物种生长速度和抗病力的下降 [28]。本研究结果显示,野生群体的单倍型多样性(Hd=0.960)与贻贝 [19](Hd=0.718~0.923)以及团头鲂 [22](Hd=0.857~0.943)等野生物种相近,说明野生群体具有稳定的大群体;养殖群体的单倍型多样性(Hd= 0.680)低于刘红等 [7]研究的6个养殖群体的单倍型多样性(Hd=0.880~0.994),原因可能是该养殖群体的样品来源于这6个养殖群体的后代,与陈薇等 [29]研究的养殖群体亲本的单倍型多样性(Hd=0.690)结果相似。
D-loop基因是线粒体DNA的非编码区,其碱基替换速率快,一般认为是线粒体基因组上进化最快的部分,它的变异速率为mtDNA完整分子或是其他区域的5~10倍,作为分子钟的进化速率每百万年高达19% [9]。Freeland [30]根据遗传分化系数大小划定了群体的分化程度:低度分化F st=0~0.05,中度分化F st=0.05~0.25,高度分化F st>0.25,这已成为一般接受的群体遗传分化标准。表3中野生群体与养殖群体的遗传分化系数(F st=0.131)说明,野生群体与养殖群体间已经产生了中等程度的遗传分化,且两者间遗传变异存在显著差异(P<0.05)。这与已报道的物种团头鲂 [22]、银鲳 [18]、条石鲷 [21]等的结果相似。发生遗传分化的可能原因主要是养殖群体在育苗中的人为干预以及生存环境的变化。
综上所述,建议在凡纳滨对虾的养殖和繁育工作中,应适当补充不同来源的亲虾并增加亲虾数量,以维持较高水平的遗传多样性,同时实时监测养殖群体的遗传多样性,以防止养殖群体因遗传多样性降低而出现生长缓慢、抗病力下降等 现象。
参考文献
[1]王丹,吴反修,农业农村部渔业渔政管理局,等.中国渔业统计年鉴.2022[M].北京:中国农业出版社,2022.
[2]BUONACCORSI V P,MCDOWELL J R,GRAVES J E.Reconciling patterns of inter-ocean molecular variance from four classes of molecular markers in blue marlin (Makaira nigricans)[J]. Molecular Ecology,2001,10(5):1179-1196.
[3]冯建彬,孙悦娜,程熙,等.我国五大淡水湖日本沼虾线粒体COI基因部分片段序列比较[J].水产学报,2008,32(4):517-525.
[4]王成辉,李思发,刘至治,等.3种中华绒螯蟹群体线粒体 "COⅡ基因序列测定与进化分析[J].水产学报,2008,32(1):8-12.
[5]程起群,马春艳,庄平,等.基于线粒体cyt b基因标记探讨凤鲚3群体遗传结构和进化特征[J].水产学报,2008,32(1): 1-7.
[6]刘红艳,江世贵,苏天凤,等.3个水域黄鳍鲷线粒体DNA "D-loop基因序列多态性研究[J].水产学报,2004,28(4):371-374.
[7]刘红,张海强,蔡生力,等.基于线粒体DNA控制区序列的6个凡纳滨对虾(Litopenaeus vannamei)养殖群体的遗传多样性分析[J].渔业科学进展,2016,37(1):63-73.
[8]CHU K H,LI C P,TAM Y K,et al.Application of mitochondrial control region in population genetic studies of the shrimp Penaeus[J].Molecular Ecology Notes,2003,3(1):120-122.
[9]BROWN W M.The mitochondrial genome of animals[M]//MACINTYRE R J,ed.Molecular Evolutionary Genetics.Boston,MA:Springer US,1985:95-130.
[10]WILLIAMS S T,KNOWLTON N,WEIGT L A,et al.Evidence for three major clades within the snapping shrimp genus Alpheus inferred from nuclear and mitochondrial gene sequence data[J].Molecular Phylogenetics and Evolution,2001,20(3):375-389.
[11]YANG P,ZHANG H,CHEN L, et al. Genetic structure of the "oriental river prawn (Macrobrachium nipponense) from the "Yangtze and Lancang Rivers, inferred from COI gene sequence[J]. Zoological Research, 2008,28(2):113-118.
[12]李延梅,牛栋,张志强,等.国际生物多样性研究科学计划与热点述评[J].生态学报,2009,29(4):2115-2123.
[13]黎中宝,方秀,陈锦,等.大黄鱼(Pseudosciaena crocea)养殖群体遗传多样性的降低[J].海洋与湖沼,2009,40(4):446-450.
[14]HEDGECOCK D,TRACEY M L,NELSON K. Genetics[M]//ABELE L G, ed.The biology of Crustacea.New York:Academic Press,1982:283-403.
[15]ALLENDORF" F W,RYMAN N,UTTER F.Genetic management of hatchery stock[M]//RYMAN N,UTTER F, ed.Population genetics and fishery management.Seattle and London:Washington Sea Grant Publications/University of Washington Press,1987:141-159.
[16]刘若余,夏先林,雷初朝,等.贵州黄牛mtDNA D-loop遗传多样性研究[J].遗传,2006, 28(3):279-284.
[17]LAN H,SHI L M.The origin and genetic differentiation of native breeds of pigs in Southwest China:an approach from mitochondrial DNA polymorphism[J].Biochemical Genetics,1993,31(1):51-60.
[18]彭士明,施兆鸿,侯俊利.基于线粒体D-loop区与COI基因序列比较分析养殖与野生银鲳群体遗传多样性[J].水产学报,2010,34(1):19-25.
[19]SHEN Y B,LI J L,FENG B B.Genetic analysis of cultured and wild populations of Mytilus coruscus based on mitochondrial DNA[J].Zoological Research,2009,30(3):240-246.
[20]韩承慧,马海涛,姜海滨,等.许氏平鲉(Sebastes schlegeli)微卫星标记开发及野生、养殖群体遗传多样性分析[J].海洋与湖沼,2016,47(1):213-220.
[21]肖志忠,肖永双,任桂静,等.条石鲷(Oplegnathus fasciatus)养殖群体与野生群体线粒体控制区序列遗传变异研究[J].海洋与湖沼,2013,44(1):249-254.
[22]唐首杰,李思发,蔡完其.团头鲂野生、驯养、选育3类遗传生态群体遗传变异的线粒体DNA分析[J].中国水产科学,2011,18(3):483-492.
[23]SUNDEN S L F,DAVIS S K.Evaluation of genetic variation in a domestic population of Penaeus vannamei (Boone):a comparison with three natural populations[J].Aquaculture,1991,97(2/3):131-142.
[24]徐田军,王日昕,孙悦娜,等.我国东南沿海日本囊对虾(Marsupenaeus japonicus)4个养殖群体遗传分化及其遗传结构分析[J].海洋与湖沼,2009,40(6):786-792.
[25]VERSPOOR E.Reduced genetic variability in first-generation hatchery populations of Atlantic salmon (Salmo salar)[J]. Canadian Journal of Fisheries and Aquatic Sciences,1988,45(10):1686-1690.
[26]WINANS G A.Genetic variability in Chinook salmon stocks from the Columbia river basin[J].North American Journal of "Fisheries Management,1989,9(1):47-52.
[27]LI Q,PARK C,ENDO T,et al.Loss of genetic variation at microsatellite loci in hatchery strains of the Pacific abalone (Haliotis discus Hannai)[J].Aquaculture,2004,235(1/2/3/4):207-222.
[28]ALLENDORF F W,PHELPS S R.Loss of genetic variation in a hatchery stock of cutthroat trout[J].Transactions of the "American Fisheries Society,1980,109(5):537-543.
[29]陈薇,马凌波,马春燕.凡纳滨对虾亲本和子一代群体的线粒体DNA遗传特征研究[J].水产科技情报,2016,43(3):119-121.
[30]FREELAND J R.Molecular ecology[M].Chichester:John Wiley & Sons,2005,112-116.
Analysis of mitochondrial genetic variation in wild and
cultured populations of Litopenaeus vannamei
FAN Chun1, GAO Xuezhong1, YU Xiaodan2, LIU Hong2, WANG Jiangang1, LI Yun2
(1. Shanghai Fengxian ecological aquaculture service center, Shanghai 201499, China;
2. Shanghai Ocean University, Shanghai 201306, China)
Abstract: To study the maternal genetic background and characteristics of Litopenaeus vannamei, mtDNA D-loop molecular marker technology was used to compare the genetic variation between wild and farmed populations of L. vannamei. The base composition of the two populations was consistent. The content of A+T was 81.52%, which was significantly higher than that of C+G (18.48%). Haplotype diversity (Hd=0.960±0.017) and nucleotide diversity (π=0.104±0.051) in the wild population were significantly higher than those in the cultured population (Hd=0.680±0.075; π= 0.060±0.029). Analysis of molecular variance (AMOVA) revealed that the genetic variation rate between populations was 13.09%, while the genetic variation rate within populations was 86.91%. Significant genetic differentiation was observed between the wild and cultured populations of L. vannamei (F st= 0.131 ,Plt;0.05). The results of this study could provide a theoretical basis for future research on the germplasm resources of L. vannamei.
Key words: Litopenaeus vannamei; wild population; cultured population; mtDNA; genetic diversity

