基于转录组分析花生单粒精播与多粒穴播植株荚果产量差异的分子机制
杨莎,刘珂珂,刘颖,郭峰,王建国,高华鑫,孟静静,张佳蕾,万书波
基于转录组分析花生单粒精播与多粒穴播植株荚果产量差异的分子机制
杨莎,刘珂珂,刘颖,郭峰,王建国,高华鑫,孟静静,张佳蕾,万书波
山东省农业科学院农作物种质资源研究所,济南 250100
【目的】在中国,为保证出苗率及播种质量,田间常采用多粒播种。然而,多粒穴播植株间的竞争往往限制了植株的生长和最终产量。为了解决这一矛盾,团队前期研究建立了单粒精准播种的高产栽培技术。单粒精播技术的节种作用和增产效果结合起来能够带来更大的效益,实现节本增效。挖掘不同种植方式花生叶片、根系及荚果中的差异表达基因,探讨单粒精播栽培技术提高花生荚果产量的分子机制,为进一步推动花生高产高效提供理论依据和技术支撑。【方法】以花生品种花育25号为试材,测定单粒精播和多粒穴播种植产量相关指标,分别取花生开花后30 d倒三叶和主根及侧根、鸡头幼果期花生荚果进行转录组测序,从分子水平阐述不同播种方式花生产量的差异。【结果】与多粒穴播相比较,单粒精播种植方式下花生单株荚果数、单株饱果数、单株果重、经济系数均显著提高。转录组数据组装后,平均每库包含4 430万个读数。通过不同比较组合中差异表达基因、GO和KEGG通路分析,与多粒穴播相比较,单粒精播种植植物叶片中参与赤霉素信号传导、光信号传导等过程的转录因子及光系统Ⅱ放氧复合体、叶绿体膜、氧化还原反应等光合作用相关基因表达量升高;根系中生物胁迫和非生物胁迫诱导的苯丙素代谢途径相关基因包括细胞色素P450基因、氧化还原基因、胁迫反应转录因子和信号调节蛋白显著富集。荚果发育初期淀粉和蔗糖代谢基因显著富集,更加有利于花生荚果发育过程中籽仁充实。【结论】花生叶片中光合相关基因表达上调能够促进光合效率提升,与产量的增加密切相关;单粒精播提高了根系抗生物和非生物胁迫的能力,结合荚果发育初期能量物质相关基因表达上调均有利于地下部花生荚果的发育,提高花生产量。
花生;单粒精播;产量;RNA-Seq;光合作用;苯丙素途径
0 引言
【研究意义】花生(L.)是世界范围内重要的油料作物和经济作物,每年贡献全球产油量的20%及11%的蛋白供给,在农民增收、农业增效及缓解食用油供需矛盾等方面具有举足轻重的地位[1-2]。在耕地紧张和粮油争地矛盾日益突出的情况下,人们逐渐认识到提高单产已成为花生增产的重要有效途径。【前人研究进展】生产中,为保证花生出苗率,人工播种习惯于一穴多粒,少的三粒,多的五六粒,不仅用种量高,一穴多株引起的株距减少及种植密度增大还会造成个体间竞争加剧,植株生长发育不一致,影响花生产量进一步提高[3-4]。虽然目前播种机多是穴播两粒,但因为种子大小不均,也经常存在一穴多粒现象。团队针对传统种植株间竞争排斥现象,提出“单粒精播、健壮个体、优化群体”技术思路,创建出单粒精播高产栽培技术,缓解株间竞争,节省种子20%,增产10%以上[4-5]。然而,单粒精播个体发育及提高花生产量的分子机制尚不清楚。近年来,主要围绕单粒精播提高花生产量的生理机制展开系统研究,比较了单粒精播与双粒穴播花生光合特性、冠层微环境[5]、生物学特性[6]、物质代谢[7]、株间竞争效应[8]、产量构成[3]等方面的差异。【本研究切入点】传统分子生物学方法研究该过程效率低,且难度大,随着高通量测序技术的发展,转录组、蛋白质组、代谢组等基因组分析方法越来越受到重视,通过这些技术的发展可以用来探索农业生产中不同现象的分子机制,为解决科学问题提供了新的途径。花生参考基因组的发表[9-11]促进了人们对花生农学重要现象和遗传改良更好的理解。【拟解决的关键问题】本研究主要通过转录组测序筛选单粒精播和多粒穴播的表达差异基因,揭示单粒精播栽培技术提高花生荚果产量的分子机制,为进一步实现花生高产高效提供理论依据和技术支撑。
1 材料与方法
1.1 试验材料与试验设计
以花生品种花育25号(山东花生研究所提供)为材料,在山东省农业科学院饮马泉基地(36°43′N,117°5′E,中国济南)进行。土壤肥力状况:有机质含量为1.1%(W/W)、碱解氮82.7 mg·kg-1、有效磷36.2 mg·kg-1、有效钾94.5 mg·kg-1、交换性钙14.9 g·kg-1。播种前基施复合肥(N15P15K15)750 kg·hm-2、钙镁磷肥750 kg·hm-2,播种时,用噻虫咯霜灵拌种。其他管理措施同一般高产田。起垄种植,垄宽85 cm,垄长10 m,垄面宽50 cm,垄高10 cm,每垄种2行花生,行距25 cm,每种播种方式各种4垄。设单粒精播穴距10 cm,23.5万穴/hm2,每穴1粒;三粒穴播穴距20 cm,11.8万穴/hm2,每穴3粒;五粒穴播穴25 cm,9.4万穴/hm2,每穴5粒(图1)。开花后30 d,分别取倒三叶、主根和侧根,分别命名为单粒叶(single seed leaf,SL)、三粒叶(three seeds leaf,TL)、五粒叶(five seeds leaf,FL)和单粒根(single seed root,SR)、三粒根(three seeds root,TR)、五粒根(five seeds root,FR),出现鸡头幼果期分别取果实单粒果(single seed pod,SP)、三粒果(three seeds pod,TP)、五粒果(five seeds pod,FP)用于转录组测序。采用3个生物学重复试验,混合后的组织用于试验。
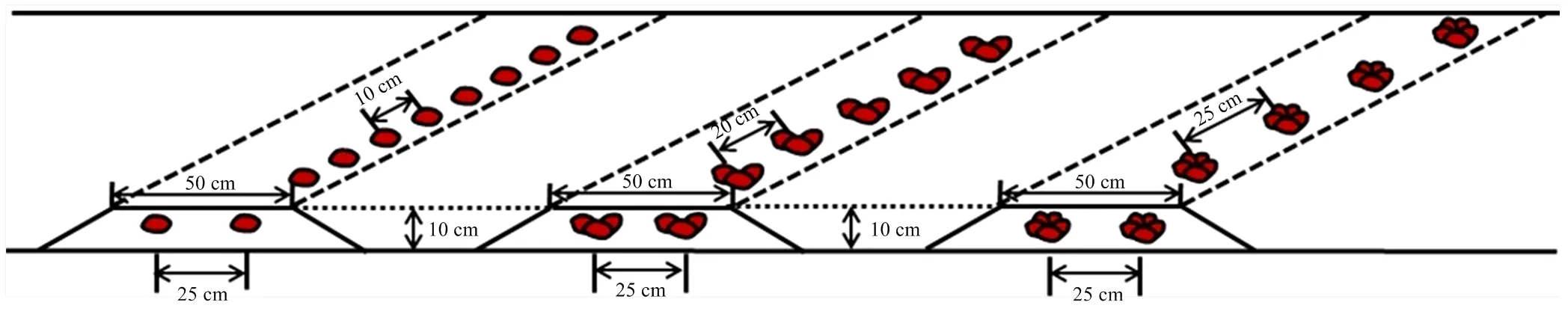
图1 种植模式示意图
1.2 单株产量构成及计量
花生生长至成熟期时选取10株植株,对其单株荚果数、单株饱果数、单株果重进行测定。将荚果和植株干燥至定重,经济系数计算公式为:经济系数=荚果干重/(植株干重+荚果干重)。
1.3 RNA提取和cDNA文库建立
用植物RNA提取试剂盒(Takara,Dalian,China)提取各样品RNA。用Agilent 2100生物分析仪和Agilent RNA 6000 Nano试剂盒(Agilent,Santa Clara,CA,USA)检测RNA的质量和纯度。筛选合格的RNA样本,用寡聚(dT)法分离mRNA,并将其碎片化。然后合成第一链cDNA和第二链cDNA,构建cDNA文库。
使用Illumina NovaSeq 6000进行双端测序,通过CASAVA碱基识别将图像数据转化为序列数据。首先对fastq格式的原始数据进行处理,过滤掉低质量序列后得到高质量数据,用于后续分析。试验原始数据已提交至NCBI(BioProject:PRJNA944737)。使用Hisat2(v2.0.5)将高质量序列与Tifrunner[11]的参考基因组进行匹配。根据CUI等[12]方法对DEG(差异表达基因)、GO(基因本体)和KEGG进行分析。通过TBtools实现基因表达的可视化[13]。在50%的样本中选择FPKM>5的基因进行加权基因共表达网络分析(weighted gene co-expression network analysis,WGCNA),方法参照文献[14]。
1.4 加权基因共表达网络分析
选择FPKM>5的基因进行加权基因共表达网络分析(WGCNA)。此外,由于低表达或无变化基因通常代表噪声,因此,采用绝对中位差(median absolute deviation,MAD)对基因进行筛选。采用软阈值功率26,最小模块大小为60,合并模块阈值为0.30进行模块构建,将模块中的基因根据KEGG分类。
1.5 实时荧光定量PCR(qRT-PCR)
使用Prime Script RT Reagent Kit合成所取样品中的cDNA。由Primer3(https://bioinfo.ut.ee/primer3/)设计基因特异性引物,3次试验重复。反应条件为95 ℃ 5 min;95 ℃ 30 s,40个循环;60 ℃ 30 s。以花生为内参基因。采用2−△△CT法进行相对定量分析[15]。采用SPSS 26.0软件进行显著性分析。
2 结果
2.1 花生荚果产量及测序结果概述
单株荚果数和荚果重是影响花生产量的直接因素。单粒精播处理的单株荚果数、单株饱果数和单株果重均高于三粒及五粒穴播,每穴产量:单粒精播<三粒穴播<五粒穴播;理论总产量:单粒精播>三粒穴播>五粒穴播(表1)。
利用Illumina 2000对不同播种方式的花生幼苗进行转录组测序,构建了27个转录组文库(每个处理有3个文库重复)。经过原始数据过滤、测序错误率检查、GC含量分布检查,获得了179.44 Gb的clean data。每个样品的clean data达到7.47 Gb,Q30百分比平均为92.90%(表2)。表明高通量测序数据是高度可靠的。
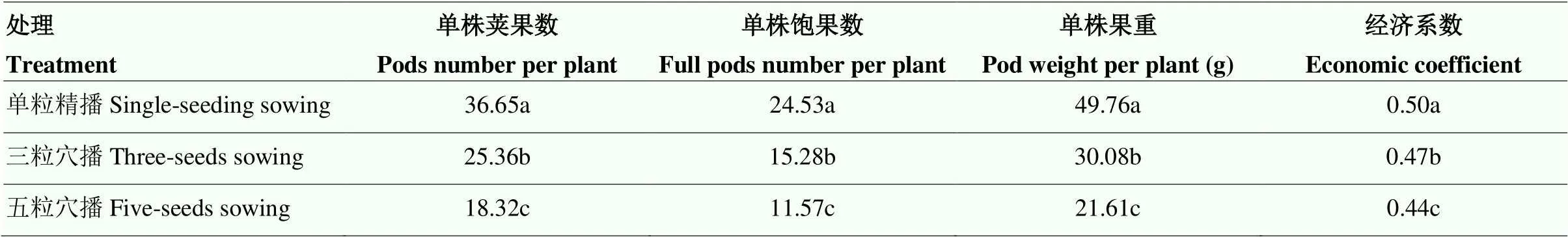
表1 不同种植方式下花生成熟期单株荚果的变化
不同小写字母表示处理间差异显著(<0.05)。下同 Different small letters meant significant difference at<0.05 level. The same as below
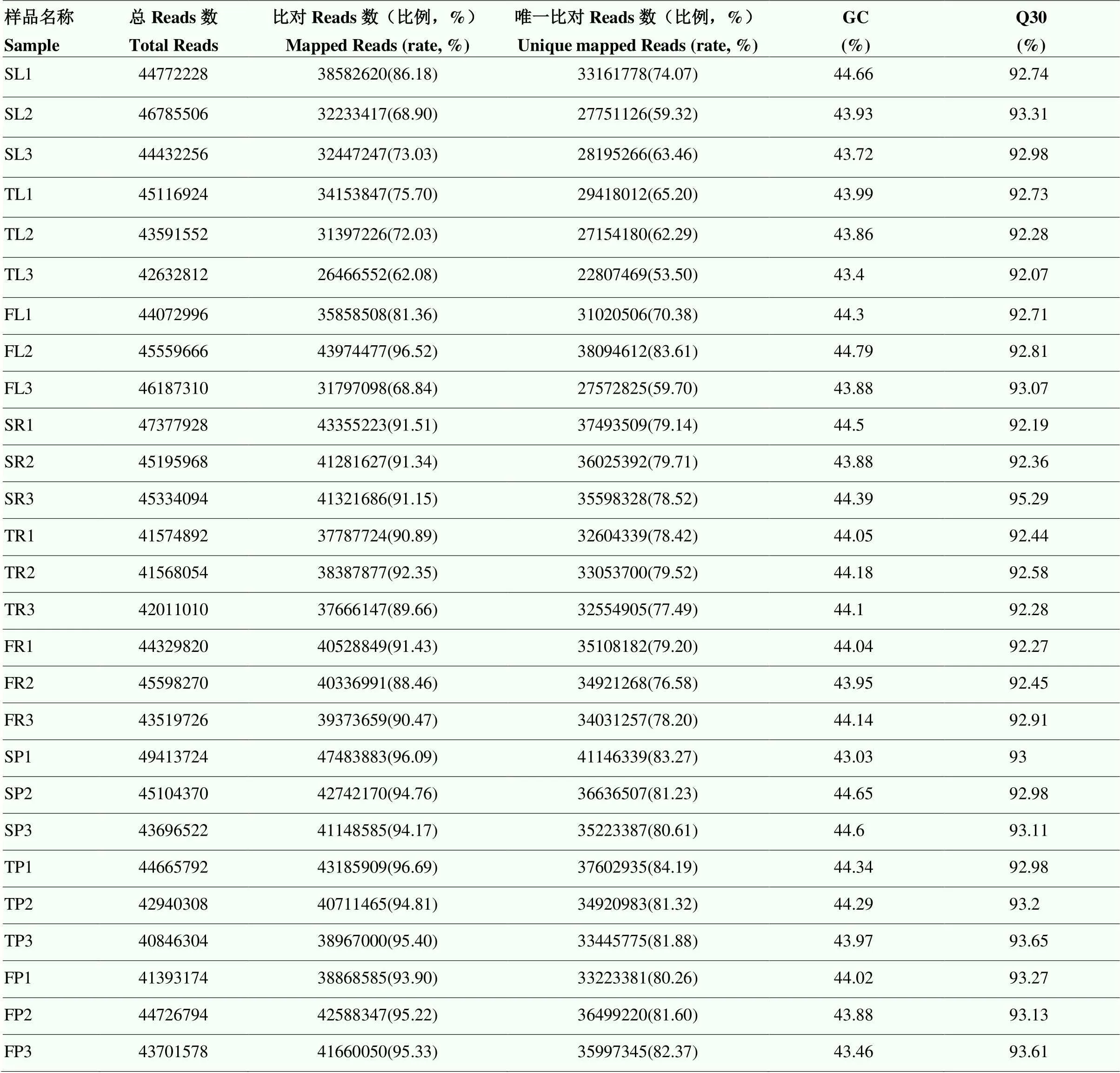
表2 转录组测序基础数据统计分析
SL:单粒叶;TL:三粒叶;FL:五粒叶;SR:单粒根;TR:三粒根;FR:五粒根;SP:单粒果;TP:三粒果;FP:五粒果。下同
SL: single seed leaf; TL: three seeds leaf; FL: five seeds leaf; SR: single seed root; TR: three seeds root; FR: five seeds root; SP: single seed pod; TP: three seeds pod; FP: five seeds pod. The same as below
2.2 不同播种方式下叶片中差异基因富集分析
在测序样本中,根据差异表达水平分析差异表达基因(DEGs)。对所鉴定的基因进行功能注释和富集分析。SL vs TL、SL vs FL、TL vs FL 3对比较组中表达差异基因数分别为599、489和337(图2)。进一步对这些差异基因进行GO分类,分别为生物进程、分子功能和细胞组分3个部分。结果表明,上调DEGs主要富集在光系统Ⅱ放氧复合体(photosystem Ⅱ oxygen evolving complex)、类囊体膜(thylakoid membrane)、氧化还原酶复合体(oxidoreductase complex)及光系统Ⅱ(photosystem Ⅱ);下调DEGs主要富集在核小体组装(nucleosome assembly)、染色质组装(chromatin assembly)、核小体组织(nucleosome organization)、DNA包装(DNA packaging)(表3)。SL vs FL组中,上调基因KEGG通路主要富集在植物-病原体互作(plant-pathogen interaction)、甘油脂代谢(glycerolipid metabolism)、类胡萝卜素合成(carotenoid biosynthesis)等,下调基因KEGG通路主要富集在硫胺素代谢(thiamine metabolism)和2-氧代羧酸代谢(2-Oxocarboxylic acid metabolism)。SL vs TL组中,上调基因的KEGG通路主要富集在甘油脂代谢(glycerolipid metabolism)、卟啉和叶绿素代谢途径(porphyrin and chlorophyll metabolism),而下调基因的KEGG通路主要富集在不同氨基酸代谢途径(arginine and proline metabolism; Cysteine and methionine metabolism)(表3)。转录因子(如MYB、GRAS和WRKY家族)基因在SL vs TL、SL vs FL组中均上调表达。热激蛋白Hsp70及分子伴侣DnaJ的表达量也显著增加,有助于增加光合膜的稳定性(表4)。
2.3 不同播种方式下根中差异基因富集分析
当单粒精播种植模式变为一穴三粒或五粒种植时,花生根系的生长发育过程随之发生变化。SR vs TR、SR vs FR、TR vs FR 3对比较组中表达差异基因数分别为2 024、2 220和1 246(图2)。与三粒及五粒播种种植模式相比较,单粒精播上调基因GO富集主要包括过氧化物酶活性(peroxidase activity)、氧化还原酶活性(oxidoreductase activity)及对氧化胁迫的响应(response to oxidative stress);下调基因同样主要富集在糖脂转运蛋白活性(glycolipid transporter activity)及磺基转移酶活性(sulfotransferase activity)、二糖及低聚糖的生物合成途径(oligosaccharide biosynthetic process)。KEGG通路富集显示,与一穴五粒种植相比较,单粒精播花生根系中上调基因富集在苯丙素生物合成(phenylpropanoid biosynthesis)、MAPK信号途径(MAPK signaling pathway)、植物-病原体互作(plant-pathogen interaction),下调基因主要富集在淀粉和蔗糖代谢(starch and sucrose metabolism)。与一穴三粒种植相比较,单粒精播花生根系中上调基因富集在植物-病原体互作(plant- pathogen interaction)、苯丙素合成(phenylpropanoid biosynthesis)、MAPK信号途径(MAPK signaling pathway)及类黄酮生物合成(flavonoid biosynthesis);下调基因主要富集在剪接体(spliceosome)、嘌呤代谢(purine metabolism)等途径(表3)。相应地,MAPK信号途径中聚酮环化酶(polyketide cyclase)、氨基转移酶Ⅰ类和Ⅱ类(aminotransferase classⅠandⅡ)、蛋白激酶(protein kinase domain)等差异表达基因在单粒精播根中表达上调(表4)。
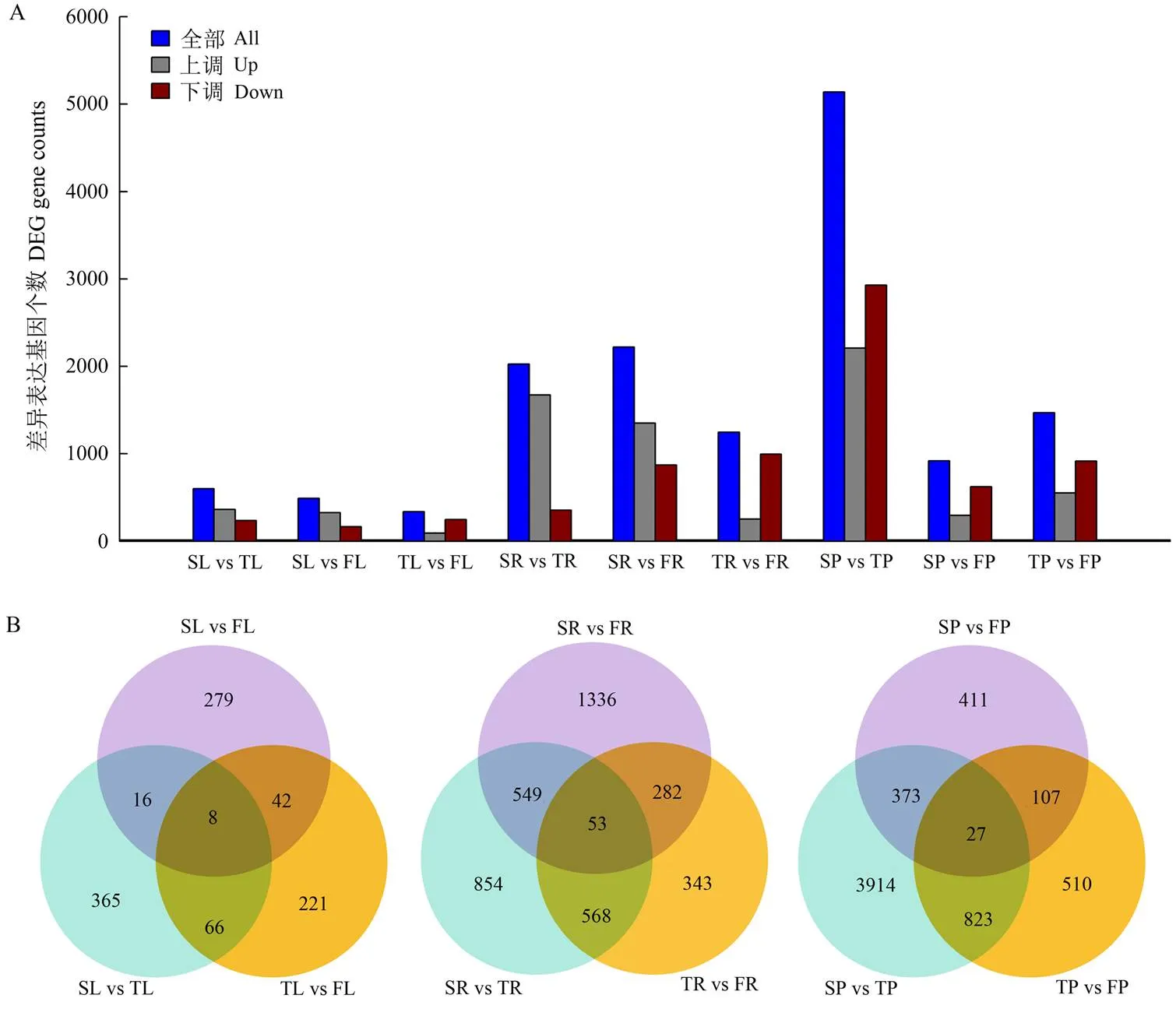
图2 不同播种方式下花生叶片、根系及荚果差异表达基因统计(A)和韦恩图(B)
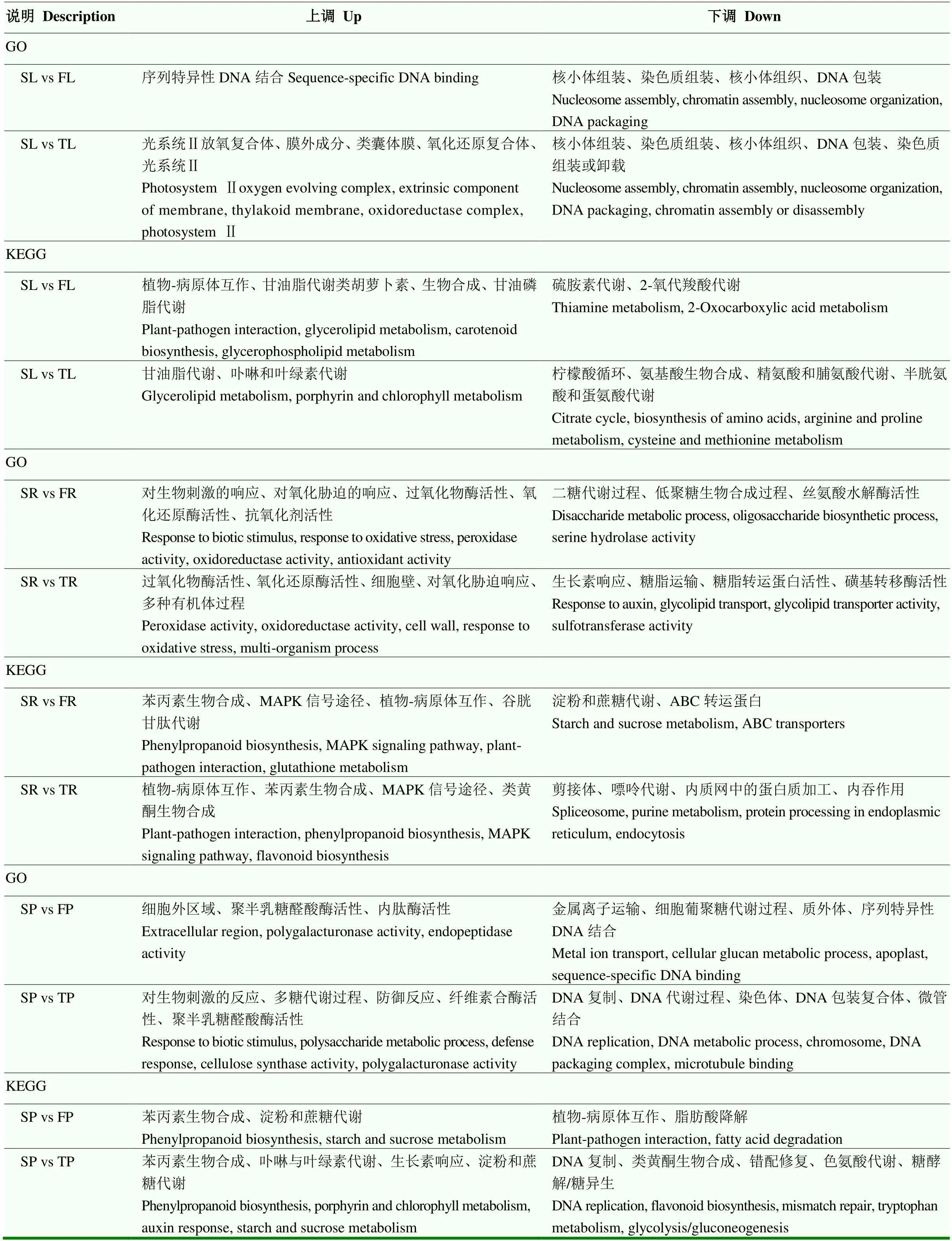
表3 不同播种方式下花生叶片、根及荚果中差异表达基因GO和KEGG富集分析

表4 单粒精播种植叶片、根及荚果中与产量相关差异表达基因列表
2.4 不同播种方式下果中差异基因富集分析
SP vs TP、SP vs FP、TP vs FP 3对比较组中表达差异基因数分别为5 137、918和1 467(图2)。SP vs TP中花生荚果上调基因GO富集在多糖代谢过程(polysaccharide metabolic process)、对生物刺激的反应(response to biotic stimulus)及多聚半乳糖醛酸酶活性(polygalacturonase activity);下调基因GO富集在DNA复制、代谢及组装过程(DNA replication、DNA metabolic process、Chromosome、DNA packaging complex)。SP vs FP单粒精播的花生荚果中GO富集主要包括多聚半乳糖醛酸酶活性(polygalacturonase activity)和内肽酶活性(endopeptidase activity);下调基因GO富集包括金属离子转运(metal ion transport)、质外体(apoplast)及序列特异性DNA结合(sequence- specific DNA binding)(表3);相应地,叶绿体(chloroplast)、生长素相关基因(response to auxin)和大量的泛素相关基因(ubiquitin-protein transferase activity、ubiquitin-like protein transferase activity)表达发生变化(表5)。KEGG通路富集显示,与一穴三粒种植相比较,单粒精播的花生荚果中上调基因主要富集在苯丙素生物合成(phenylpropanoid biosynthesis)、生长素响应(auxin response)及淀粉和蔗糖代谢(starch and sucrose metabolism);下调基因富集在DNA复制、错配修复(DNA replication、mismatch repair)和糖酵解/糖异生(glycolysis/ gluconeogenesis);与一穴五粒种植相比较,单粒精播的花生荚果中上调基因主要富集在苯丙素生物合成(phenylpropanoid biosynthesis)及淀粉和蔗糖代谢(starch and sucrose metabolism),下调基因主要富集在植物-病原体互作(plant-pathogen interaction)和脂肪酸降解(fatty acid degradation)(表3)。
2.5 三粒及五粒种植模式根与果中DEGs分析
花生的根和果均为地下部生长,因此,光照对其生长发育过程不能产生直接影响。与单粒播种相比较,SR vs FR(2 220 DEGs)和SR vs TR(2 024 DEGs)中的相同的DEGs数仅为602个,而大部分DEGs为SR vs FR和SR vs TR中特异表达的,很可能是由于增加种植密度后造成的。有趣的是,这602个DEGs的GO和KEGG富集项主要与不同物质如苯丙素、黄酮类合成途径及氨基酸等代谢途径相关(图3-A)。表明多粒种植均会引起株间竞争,调节植物生长发育及代谢功能的相关物质合成途径关键基因表达发生显著变化。相应地,增加种植密度后SP vs FP(9 180 DEGs)和SP vs TP(5 137 DEGs)中共有DEGs数为400,这400个DEGs的GO项主要富集在葡聚糖代谢途径,GO条目中数量富集最多的为碳水化合物代谢途径;KEGG途径则主要富集在MAPK信号途径(图3-B)。
2.6 根及果中差异关键基因加权基因共表达网络分析
加权基因共表达网络分析(WGCNA)用于挖掘组织特异性模块和与表型相关的关键基因。为了探索不同播种方式下发挥重要作用的关键基因和共表达网络,通过WGCNA分析了样品中的10 299个DEGs。为了构建无标度网络,将最优软阈值设置为11。通过使用函数邻接和tom相似性建立邻接矩阵和tom重叠矩阵。根据动态切割划分模块,总共获得了16个模块,使用标记热图函数来可视化和分析模块与样品之间的关系。相关系数大于0.65且<0.05的模块被定义为样本特异性模块,因此,在6个样本中获得了25个组织特异性模块(图4-A)。棕褐色与SR呈正相关;蓝绿色和浅青色与TR呈正相关;黄绿色和品红色分别与FR和SP呈正相关。TP和FP的特定模块分别为蓝绿色和红色。

表5 SP vs TP中DEGs的GO条目

A:SR vs FR,SR vs TR;B:SP vs FP,SP vs TP
为了深入了解不同种植方式的代谢途径,对上述特定模块上的基因进行了KEGG富集分析。结果表明,SR中三羧酸循环途径丰富富集;在TR中,有机含硒化合物代谢途径及鞘糖脂生物合成途径富集;而花生四烯酸代谢途径在FR中富集(图4-B)。SP特异性模块基因在剪接体、真核生物中的核糖体生物发生和肌醇磷酸代谢等途径中富集;TP特异性模块基因也在DNA复制、生物素代谢、错配修复和核苷酸切除修复等途径中富集;FP特异性模块基因在咖啡因代谢、剪接体和酮体的合成与降解等途径中富集(图4-B)。
2.7 根及果中与苯丙烷类生物合成相关的DEGs
与单粒精播相比较,三粒穴播和五粒穴播下调的差异表达基因均在苯丙烷类生物合成富集。苯丙烷类物质是一类植物的次生代谢产物,在植物发育中起着至关重要的作用。因此,进一步筛选了苯丙烷类物质在不同播种方式调节下根及果中相关基因的表达情况。

图4 加权基因共表达网络分析
当单粒精播种植模式变为一穴三粒或五粒时,下调的苯丙烷类物质相关基因主要包括过氧化物酶、糖基水解酶和细胞色素P450(图5)。根中苯丙烷类物质中有12个过氧化物酶基因在密度增加后表达量降低,相应地,荚果中有9个过氧化物酶基因在密度增加后表达量降低。共有7个糖基水解酶基因被上调,糖基水解酶具有调控植物生长发育和胁迫应答的作用。

图5 花生根及荚果中苯丙烷类途径相关基因表达分析
2.8 差异表达基因的qRT-PCR验证
为验证转录组测序结果中基因表达量的准确性,随机挑选了15个基因进行不同处理下的qRT-PCR验证。将qRT-PCR结果与转录组测序数据比对发现,15个差异表达基因与转录组数据基本一致(图6),同时RNA-seq数据与qRT-PCR结果在0.01水平上线性关系良好,2达到0.9506(图6),表明转录组测序数据的可靠性,可以用来分析不同播种方式条件下花生叶片、根及荚果中的差异表达基因。
3 讨论
3.1 转录组挖掘花生单粒精播与多粒穴播种植的关键差异基因
不同生物和非生物胁迫严重影响全球作物产量和粮食安全[16]。旱、涝、酸化、盐碱等非生物逆境和传统穴播两粒或多粒种植引起的生物逆境,成为制约花生单产突破的瓶颈[17]。近年来,随着测序技术的发展,基因组和转录组广泛应用于农业生产问题的研究。通过转录组深度挖掘花生单粒精播与多粒穴播种植的关键差异基因,全面解析单粒精播增产的分子新机制,为解析花生栽培高产提供有效工具。
3.2 单粒精播能够增加花生叶片光合作用形成“强源”
作物产量的形成实质是源库流协调作用的结果。非生物胁迫通过气孔(气孔关闭)或非气孔(如光系统被破坏和光合作用关键酶活性降低)降低光合作用,导致源强度降低[18-19],从而减少同化物向籽粒中的转运分配,降低产量。在本研究中,单粒精播种植叶片中MYB及WRKY等转录因子家族较一穴三粒和五粒播种表达量增加,这些转录因子在非生物逆境响应过程中发挥重要作用[20-21]。此外,一些参与赤霉素信号传导、光信号传导等过程的转录因子如HSP70、DnaJ和GRAS,在单粒精播种植叶片中表达量上升(表4)。DnaJ蛋白作为分子伴侣,在调节生物体的生长发育和适应环境中起着关键作用[22]。一穴三粒及五粒种植更加加剧了花生叶片对光源的竞争,植株间生长空间不足,受光面积减少。单粒精播叶片中光系统Ⅱ放氧复合体、叶绿体膜、氧化还原反应等相关基因表达上调,有助于提高光合效率(表3)。光合作用为农作物产量的形成提供了主要的物质基础,因此,单粒精播能够促进花生建立强大的群体质量体系,形成“强源”,为干物质的积累提供基础条件。

3.3 单粒精播花生根系中苯丙素代谢途径相关基因表达模式的影响
根系组织的生长发育对花生的产量尤为重要,健壮的根系是作物生长和高产的基础[23]。病虫害等生物胁迫和盐碱、渍涝、机械损伤、干旱等非生物胁迫严重影响花生的生长发育。在本研究中,与三粒及五粒种植相比,单粒精播根系中氧化还原及抗氧化活性物质、苯丙素途径及MAPK信号途径等GO和KEGG富集上调(表3),相应地,苯丙素途径相关基因(过氧化物酶基因、半胱氨酸分泌蛋白家族和细胞色素P450)表达上调(图5)。在拟南芥中,过氧化物酶参与胁迫耐性,并具有清除活性氧(ROS)和阻止DNA损伤的作用[24]。过氧化物酶负调节ABA介导地抑制种子萌发[25],同时在激素介导调节侧根发育中起重要作用[26]。苯丙素是目前研究较多的植物次生代谢类物质,包括类黄酮、花青素、单木质素和单宁。类黄酮由苯丙氨酸通过苯丙素途径合成,而苯丙氨酸则通过莽草酯途径合成。一般苯丙素途径的第二步涉及C4H的活性,C4H是植物中的一种细胞色素P450单加氧酶,催化反式肉桂酸的羟基化生成对香豆酸。这也是类黄酮合成途径中的第一个氧化反应[27]。在白杨和拟南芥中,C4H的表达水平与重要的苯丙素类代谢产物木质素的含量有关。这些苯丙素类物质能够通过参与调控光合作用、营养吸收、调节生长、细胞分裂、维持氧化还原稳态等过程提高植物对生物或非生物逆境的抗性和稳定性[28]。非生物逆境下,苯丙素生物合成途径受到刺激,产生多种具有较强抗氧化潜能的酚类化合物[29]。酚类化合物主要通过清除盐胁迫下植物体内有害的ROS提高植物耐受性[30-31]。参与MAPK级联的基因包括互联的MAPK、MAPKK和MAPKKK,这些级联在植物激素、生物胁迫和非生物胁迫的信号转导中同样发挥重要作用[32]。这些苯丙素类物质及MAPK级联途径在单粒精播种植的花生根系中富集(表3),协助花生提高根系的抗病及抗氧化能力,从而保障花生的优质高产。
3.4 单粒精播对花生荚果中淀粉和蔗糖代谢途径相关基因的影响
糖分作为光合产物以蔗糖的形式输入籽粒,随后经过4种关键酶的催化作用转化为淀粉。淀粉主要在胚乳细胞中储藏,处于不断被贮存同时不断被利用的状态,对果实发育起着物质和能量供应的重要作用。储藏物质一旦无法积累,就会引起种子败育[33]。单粒精播种植荚果中淀粉和蔗糖代谢基因富集有利于花生荚果发育过程中籽仁充实,产量增加(表1和表3)。此外,在DEGs中还发现了大量的叶绿体、生长素相关基因和大量的泛素化蛋白(表5)。因此,推测有2条信号转导途径参与单粒精播花生荚果的发育。一种是从叶绿体及光合作用开始通过泛素化参与调控细胞周期、果实发育等过程;二是通过生物和非生物胁迫的信号调控及物质代谢向下游传递信号。
4 结论
多粒穴播的株间竞争往往限制了植株的生长和最终产量。通过转录组测序发现,该过程涉及光合作用相关基因表达量变化,苯丙素代谢途径相关基因表达发生变化,包括细胞色素P450基因、氧化还原基因、胁迫反应转录因子等。此外,蛋白淀粉和蔗糖代谢基因显著富集,这些通路中基因表达量的改变有助于花生地上部植株生长及地下部荚果发育过程,提高花生产量。
[1] 王才斌. 实施理性栽培, 推进山东花生生产可持续发展. 花生学报, 2018, 47(1): 74-76, 68.
WANG C B. Rational cultivation promotes the sustainable development of peanut production in Shandong. Journal of Peanut Science, 2018, 47(1): 74-76, 68. (in Chinese)
[2] 王在序, 盖树人. 山东花生. 上海: 上海科学技术出版社, 1999: 1-15.
WANG Z X, GAI S R. Shandong Peanut. Shanghai: Shanghai Scientific & Technical Press, 1999: 1-15. (in Chinese)
[3] 张佳蕾, 郭峰, 杨佃卿, 孟静静, 杨莎, 王兴语, 陶寿祥, 李新国, 万书波. 单粒精播对超高产花生群体结构和产量的影响. 中国农业科学, 2015, 48(18): 3757-3766.
ZHANG J L, GUO F, YANG D Q, MENG J J, YANG S, WANG X Y, TAO S X, LI X G, WAN S B. Effects of single–seed precision sowing on population structure and yield of peanuts with super–high yield cultivation. Scientia Agricultura Sinica, 2015, 48(18): 3757-3766. (in Chinese)
[4] LIANG X Y, GUO F, FENG Y, ZHANG J L, YANG S, MENG J J, LI X G, WAN S B. Single–seed sowing increased pod yield at a reduced seeding rate by improving root physiological state of. Journal of Integrative Agriculture, 2020, 19(4): 1019-1032.
[5] 梁晓艳, 郭峰, 张佳蕾, 孟静静, 李林, 万书波, 李新国. 单粒精播对花生冠层微环境、光合特性及产量的影响. 应用生态学报, 2015, 26(12): 3700-3706.
LIANG X Y, GUO F, ZHANG J L, MENG J J, LI L, WAN S B, LI X G. Effects of single-seed sowing on canopy microenvironment,photosynthetic characteristics and pod yield of peanut (), Chinese Journal of Applied Ecology, 2015, 26(12): 3700-3706. (in Chinese)
[6] 张佳蕾, 张雷, 林英杰, 刘颖, 王建国, 郭峰, 唐朝辉, 李新国, 万书波. 单粒精播对花生生物学特性的影响. 中国油料作物学报, 2020, 42(6): 960-969.
ZHANG J L, ZHANG L, LIN Y J, LIU Y, WANG J G, GUO F, TANG Z H, LI X G, WAN S B. Effects of single seed sowing on biological characteristics of peanut. Chinese Journal of Oil Crop Sciences, 2020, 42(6): 960-969. (in Chinese)
[7] 梁晓艳, 郭峰, 张佳蕾, 孟静静, 李林, 万书波, 李新国. 适宜密度单粒精播提高花生碳氮代谢酶活性及荚果产量与籽仁品质. 中国油料作物学报, 2016, 38(3): 336-343.
LIANG X Y, GUO F, ZHANG J L, MENG J J, LI L, WAN S B, LI X G. Effects of single-seed sowing density on characteristics related to yield and quality of peanut (L.).Chinese Journal of Oil Crop Sciences, 2016, 38(3): 336-343. (in Chinese)
[8] 张佳蕾, 郭峰, 苗昊翠, 李利民, 杨莎, 耿耘, 孟静静, 李新国, 万书波. 单粒精播对高产花生株间竞争缓解效应研究. 花生学报, 2018, 47(2): 52-58.
ZHANG J L, GUO F, MIAO H C, LI L M, YANG S, GENG Y, MENG J J, LI X G, WAN S B. Study on relieving inter-plant competition by single seed sowing of high yield peanut. Journal of Peanut Science, 2018, 47(2): 52-58. (in Chinese)
[9] ZHUANG W J, CHEN H, YANG M, WANG J P, Pandey M K, ZHANG C, CHANG W C, ZHANG L S, ZHANG X T, TANG R H, GARG V, WANG X J, TANG H B, CHOW C N, WANG J P, DENG Y, WANG D P, KHAN A W, YANG Q, CAI T C, BAJAJ P, WU K C, GUO B Z, ZHANG X Y, LI J J, LIANG F, HU J, LIAO B S, LIU S Y, CHITIKINENI A, YAN H S, ZHENG Y X, SHAN S H, LIU Q Z, XIE D Y, WANG Z Y, ALI KHAN S, ALI N, ZHAO C Z, LI X G, LUO Z L, ZHANG S B, ZHUANG R R, PENG Z, WANG S Y, MAMADOU G, ZHUANG Y H, ZHAO Z F, YU W C, XIONG F Q, QUAN W P, YUAN M, LI Y, ZOU H S, XIA H, ZHA L, FAN J P, YU J G, XIE W P, YUAN J Q, CHEN K, ZHAO S S, CHU W T, CHEN Y T, SUN P C, MENG F B, ZHUO T, ZHAO Y H, LI C J, HE G H, ZHAO Y L, WANG C C, KAVIKISHOR P B, PAN R L, PATERSON A H, WANG X Y, MING R, VARSHNEY R K. The genome of cultivated peanut provides insight into legume karyotypes, polyploid evolution and crop domestication. Nature Genetics, 2019, 51(5): 865-876.
[10] CHOPRA R, BUROW G, FARMER A, MUDGE J, SIMPSON C E, BUROW M D. Comparisons oftranscriptome assemblers in diploid and polyploid species using peanut (spp.) RNA-Seq data. PLoS One, 2014, 9(12): e115055.
[11] BERTIOLI D J J, JENKINS J, CLEVENGER J, DUDCHENKO O, GAO D Y, SEIJO G, LEAL-BERTIOLI S C M, REN L H, FARMER A D, PANDEY M K, SAMOLUK S S, ABERNATHY B, AGARWAL G, BALLÉN-TABORDA C, CAMERON C, CAMPBELL J, CHAVARRO C, CHITIKINENI A, CHU Y, DASH S, EL BAIDOURI M, GUO B Z, HUANG W, DO KIM K, KORANI W, LANCIANO S, LUI C G, MIROUZE M, MORETZSOHN M C, PHAM M, SHIN J H, SHIRASAWA K, SINHAROY S, SREEDASYAM A, WEEKS N T, ZHANG X Y, ZHENG Z, SUN Z Q, FROENICKE L, AIDEN E L, MICHELMORE R, VARSHNEY R K, HOLBROOK C C, CANNON E K S, SCHEFFLER B E, GRIMWOOD J, OZIAS-AKINS P, CANNON S B, JACKSON S A, SCHMUTZ J. The genome sequence of segmental allotetraploid peanut. Nature Genetics, 2019, 51(5): 877-884.
[12] CUI Y Y, ZHAI Y L, FLAISHMAN M, LI J P, CHEN S W, ZHENG C L, MA H Q. Ethephon induces coordinated ripening acceleration and divergent coloration responses in fig (L.) flowers and receptacles. Plant Molecular Biology, 2021, 105(4): 347-364.
[13] CHEN C J, CHEN H, ZHANG Y, THOMAS H R, FRANK M H, HE Y H, XIA R. TBtools: An integrative toolkit developed for interactive analyses of big biological data. Molecular Plant, 2020, 13(8): 1194-1202.
[14] LANGFELDER P, HORVATH S. WGCNA: an R package for weighted correlation network analysis. BMC Bioinformatics, 2008, 9: 559.
[15] LIVAK K J, SCHMITTGEN T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCTmethod. Methods, 2001, 25(4): 402-408.
[16] MENGISTE T, CHEN X, SALMERON J, DIETRICH R. Thegene encodes an R2R3MYB transcription factor protein that is required for biotic and abiotic stress responses in. The Plant Cell, 2003, 15(11): 2551-2565.
[17] AMIN A B, RATHNAYAKE K N, YIM W C, GARCIA T M, WONE B, CUSHMAN J C, WONE B W M. Crassulacean acid metabolism abiotic stress-responsive transcription factors: a potential genetic engineering approach for improving crop tolerance to abiotic stress. Frontiers in Plant Science, 2019, 10: 129.
[18] 张俊杰, 陈金平, 汤钰镂, 张锐, 曹红章, 王丽娟, 马梦金, 王浩, 王泳超, 郭家萌, KRISHNA SV Jagadish, 杨青华,邵瑞鑫. 花期前后干旱胁迫对复水后夏玉米光合特性与产量的影响. 作物学报, 2023, 49(5): 1397-1409.
ZHANG J J, CHEN J P, TANG Y L, ZHANG R, CAO H Z, WANG L J, MA M J, WANG H, WANG Y C, GUO J M, JAGADISH K, YANG Q H, SHAO R X. Effects of drought stress before and after anthesis on photosynthetic characteristics and yield of summer maize after re-watering. Acta Agronomica Sinica, 2023, 49(5): 1397-1409. (in Chinese)
[19] WANG Y F, GUO Y Y, ZHAO C F, LI H J, ZHANG R H. Exogenous melatonin achieves drought tolerance by improving photosynthesis in maize seedlings leaves. Russian Journal of Plant Physiology, 2021, 68(4): 718-727.
[20] BANERJEE A, ROYCHOUDHURY A. WRKY proteins: signaling and regulation of expression during abiotic stress responses. The Scientific World Journal, 2015, 2015: 807560.
[21] WEI H B, ZHAO H B, SU T, BAUSEWEIN A, GREINER S, HARMS K, RAUSCH T. Chicory R2R3-MYB transcription factors CiMYB5 and CiMYB3 regulate fructan 1-exohydrolase expression in response to abiotic stress and hormonal cues. Journal of Experimental Botany, 2017, 68(15): 4323-4338.
[22] WANG G Z, ZHOU S S, LUO Y, MA C J, GONG Y H, ZHOU Y, GAO S S, HUANG Z C, YAN L L, HU Y, BIAN Y B. The heat shock protein 40 LeDnaJ regulates stress resistance and indole-3-acetic acid biosynthesis in. Fungal Genetics and Biology, 2018, 118: 37-44.
[23] ZHENG Y P, XIN C Y, WANG C B, SUN X S, YANG W Q, WAN S B, ZHENG Y M, FENG H, CHEN D X, SUN X W, WU Z F. Effects of phosphorus fertilizer on root morphology, physiological characteristics and yield in peanut ().Chinese Journal of Plant Ecology, 2014, 37(8): 777-785.
[24] GABER A, OGATA T, MARUTA T, YOSHIMURA K, TAMOI M, SHIGEOKA S. The involvement ofglutathione peroxidase 8 in the suppression of oxidative damage in the nucleus and cytosol. Plant and Cell Physiology, 2012, 53(9): 1596-1606.
[25] ZHAI C Z, ZHAO L, YIN L J, CHEN M, WANG Q Y, LI L C, XU Z S, MA Y Z. Two wheat glutathione peroxidase genes whose products are located in chloroplasts improve salt and H2O2tolerances in. PLoS One, 2013, 8(10): e73989.
[26] PASSAIA G, QUEVAL G, BAI J, MARGIS-PINHEIRO M, FOYER C H. The effects of redox controls mediated by glutathione peroxidases on root architecture in. Journal of Experimental Botany, 2014, 65(5): 1403-1413.
[27] DONG N Q, LIN H X. Contribution of phenylpropanoid metabolism to plant development and plant-environment interactions. Journal of Integrative Plant Biology, 2021, 63(1): 180-209.
[28] WOHL J, PETERSEN M. Functional expression and characterization of cinnamic acid 4-hydroxylase from the hornwortin. Plant Cell Reports, 2020, 39(5): 597-607.
[29] NAIKOO M I, DAR M I, RAGHIB F, JALEEL H, AHMAD B, RAINA A, AHMAD KHAN F, NAUSHIN F. Role and regulation of plants phenolics in abiotic stress tolerance//Plant Signaling Molecules. Amsterdam: Elsevier, 2019: 157-168.
[30] TAÏBI K, TAÏBI F, AIT ABDERRAHIM L, ENNAJAH A, BELKHODJA M, MULET J M. Effect of salt stress on growth, chlorophyll content, lipid peroxidation and antioxidant defence systems inL.. South African Journal of Botany2016, 105: 306-312.
[31] SMIRNOV O E, KOSYAN A M, KOSYK O I, TARAN N Y. Response of phenolic metabolism induced by aluminium toxicity inmoench. plants. Ukrainian Biochemical Journal, 2015, 87(6): 129-135.
[32] WANG L Z, HU W, TIE W W, DING Z H, DING X P, LIU Y,YAN Y, WU C L, PENG M, XU B Y, JIN Z Q. The MAPKKK and MAPKK gene families in banana: identification, phylogeny and expression during development, ripening and abiotic stress. Scientific Reports2017,7(1): 1159.
[33] SEUNG D. Amylose in starch: towards an understanding of biosynthesis, structure and function. The New Phytologist, 2020, 228(5): 1490-1504.
The Molecular Mechanism of Pod Yield Difference between Single- Seeding Precision Sowing and multi-seeds sowing of Peanut Based on Transcriptome Analysis
YANG Sha, LIU KeKe, LIU Ying, GUO Feng, WANG JianGuo, GAO HuaXin, MENG JingJing, ZHANG JiaLei, WAN ShuBo
Institute of Crop Germplasm Resources, Shandong Academy of Agricultural Sciences, Jinan 250100
【Objective】In China, in order to ensure the emergence rate and quality of seedlings, the field often adopts multiple seed seeding. However, inter-plant competition in multi-seeds sowing often limits the growth and eventual yield of subsequent plants. In order to solve this contradiction, the team studied and established the high-yield cultivation technology of single-seed precision seeding. The combination of seed saving and yield increase effect of single-seed precision seeding technology can bring greater benefits and realize cost savings and increased efficiency. the differentially expressed genes in peanut leaves, roots and pods under different planting methods were used to explore the regulatory mechanism of single-seeding precision sowing to improve peanut pod yield, providing theoretical basis and technical support for further promoting peanut high yield and high efficiency. 【Method】Peanut variety Huayu 25 was used as the test material, while the yield related indexes of single-seed sowing and multi-seeds sowing were determined. Inverted three leaves, taproot and lateral root of peanut at 30 days after flowering and peanut pod at young fruit stage of chicken head were selected for transcriptome sequencing, and the yield differences of peanut under different sowing methods were revealed on the molecular level. 【Result】Compared with multi-seeds sowing, the pod number per plant, full fruit number per plant, fruit weight per plant and economic coefficient of single-seed sowing were significantly increased. After the transcriptome data is assembled, each library contains an average of 44.3 million readings. Through the analysis of differentially expressed genes, GO and KEGG pathways in different combinations, it was found that the expression levels of transcription factors, photosystem Ⅱ oxygen-releasing complex, chloroplast membrane, oxidation-reduction reaction and other genes involved in the processes of GA signal and light signal transduction were increased in the leaves of plants under single-seed sowing compared with multi-grain cave seeding. Genes related to phenylpropyl metabolism pathway induced by biological and abiotic stress were significantly enriched in roots, including cytochrome P450 gene, oxidation-reduction gene, stress response transcription factor and signal regulatory protein. The accumulation of starch and sucrose metabolism genes was more conducive to seed kernel enrichment during pod development. 【Conclusion】The up-regulated expression of photosynthetic related genes in peanut leaves at seedling stage could promote the increase of photosynthetic efficiency, which was closely related to the increase of yield. Single-seed sowing improved the ability of root system to resist biological and abiotic stress, and combined with the up-regulation of energy and material related genes in the early stage of pod development, it was beneficial for the development of underground peanut pod and increased peanut yield.
peanut; single seed precision sowing; yield; RNA-Seq; photosynthesis; phenylpropanoid pathway

10.3864/j.issn.0578-1752.2023.22.003
2023-04-30;
2023-06-25
国家重点研发计划(2022YFD1000105-2)、泰山学者工程、山东省重点研发计划(2021LZGC026,2022CXPT031)、山东省农业科学院农业科技创新工程(CXGC2023A20)
杨莎,E-mail:yangsha0904@126.com。通信作者张佳蕾,E-mail:zhangjialei19@163.com。通信作者万书波,E-mail:wanshubo2016@163.com
(责任编辑 李莉)

