大豆转录因子基因GmbHLH130克隆及在干旱胁迫中的功能分析
张斌


摘要: 为了探究大豆GmbHLH130基因在植物干旱胁迫中的调控功能,利用生物信息学分析GmbHLH130与其他物种bHLH家族成员的系统进化关系,检测GmbHLH130基因及其启动子对干旱胁迫的响应,对GmbHLH130蛋白的亚细胞定位和转录激活活性进行分析,最后初步评估GmbHLH130基因过表达拟南芥的耐旱性。结果显示,在进化树中GmbHLH130与拟南芥AtbHLH122进化关系最近;GmbHLH130基因受干旱诱导上调表达;GmbHLH130基因启动子也受干旱诱导激活下游报告基因;烟草叶片瞬时表达分析结果表明GmbHLH130蛋白定位于细胞核,并且具有转录激活活性。此外,GmbHLH130基因过表达拟南芥在干旱处理下的绿色子叶率和根长均显著大于野生型。本研究结果初步证明了大豆GmbHLH130基因在增强植物耐旱方面的功能,为后续探究其参与耐旱性调控的分子机制提供了理论依据。
关键词: 大豆;GmbHLH130;干旱胁迫;耐旱性
中图分类号: S565.1 文献标识码: A 文章编号: 1000-4440(2023)07-1441-08
Cloning and functional analysis of soybean transcription factor GmbHLH130 gene under drought stress
ZHANG Bin
(Hunan University of Science and Engineering/Hunan Provincial Engineering Research Center for Ginkgo biloba, Yongzhou 425199, China)
Abstract: To explore the regulatory function of soybean GmbHLH130 gene in plant drought stress, bioinformatics was used to analyze the phylogenetic relationship between GmbHLH130 protein and bHLH members of other species. The response of GmbHLH130 and its promoter to drought stress was detected, and the subcellular localization and transcriptional activation of GmbHLH130 protein were analyzed. Finally, the drought tolerance of Arabidopsis which overexpressed GmbHLH130 was preliminarily evaluated. The results showed that GmbHLH130 protein was closest to AtbHLH122 protein in the phylogenetic tree. The expression level of GmbHLH130 gene was up-regulated under drought treatment. The promoter of GmbHLH130 gene was also induced by drought stress to activate downstream reporter gene. Transient expression of GmbHLH130 in tobacco leaves indicated that GmbHLH130 was localized in the nucleus and had transcriptional activation activity. In addition, the green cotyledon percentage and root length of GmbHLH130 overexpressed Arabidopsis were all significantly higher than those of wide type under drought treatment. The results of the study preliminarily proved the function of soybean GmbHLH130 gene in enhancing plant drought tolerance, and provided theoretical basis for further investigation of its molecular mechanism involved in drought tolerance regulation.
Key words: soybean;GmbHLH130;drought stress;drought tolerance
植物在生長发育过程中经常会受到多种不利环境因素的影响,如高盐、干旱、高温和低温等,其中干旱胁迫是导致农作物减产的重要原因之一[1]。植物在长期的进化过程中形成了多种应对干旱胁迫的方式,如增加叶片蜡质、气孔关闭等生理响应,关键基因的响应引起植物体内渗透调节物质、抗氧化酶活性增加等分子调控[2]。转录因子在植物干旱胁迫应答过程中发挥重要作用。目前已知能够参与植物干旱胁迫的转录因子家族包括:MYB[3]、bHLH[4]、bZIP[5]、WRKY[6]和AP2/ERF[7]等。转录因子发挥作用的分子机制是其结合到胁迫相关基因启动子的特定顺式作用元件上,进而激活或抑制下游基因的表达,最终防止或减少逆境胁迫对植物的伤害。bHLH家族是被子植物中第二大转录因子家族,其广泛分布于所有的真核生物中。bHLH家族蛋白质能够结合到基因启动子区的E-box或G-box元件上调控基因表达[8]。bHLH转录因子在植物干旱胁迫响应中的功能及分子机制已有较多报道。例如,过表达拟南芥AtbHLH112基因有利于降低植株体内活性氧水平和提高脯氨酸含量,增强转基因拟南芥植株对干旱胁迫的耐受性[4]。拟南芥AtbHLH122在干旱和渗透胁迫响应过程中均发挥重要作用,过表达AtbHLH122基因的转基因拟南芥耐旱性和耐盐性均明显增强[9]。bHLH转录因子AmDEL则可通过介导黄酮类物质的大量累积增强植物耐旱性[10]。密罗木bHLH家族基因MfbHLH38[11]、日本百脉根LjbHLH34[12]等基因均能正调节植物的耐旱性。转录因子bHLH家族成员在植物耐旱性调控过程中的保守功能使其在作物耐旱性育种改良中具有重大价值。
大豆是中国重要的粮油作物,是食用油、植物蛋白和动物饲料的关键原料,但是其生产也受到干旱胁迫的影响。尽管在大豆耐旱机理研究和品种选育方面已有较多报道[13-14],但至今仍无法阐明大豆应对干旱胁迫的分子调控网络。研究结果表明,大豆中有308个bHLH家族成员,其中GmORG3蛋白能够通过减少根對镉的吸收和向地上部的转运增强转基因大豆的耐镉性[15],GmPIB1能够结合到活性氧(ROS)产生关键酶编码基因GmSPOD1启动子上的E-box元件抑制其表达,减少ROS的产生,进而增强对大豆疫霉菌的抗性[16]。此外,GmbHLH57 和GmbHLH300在大豆铁稳态维持方面发挥重要作用[17]。然而,大多数大豆bHLH转录因子家族基因的生物学功能,尤其是在干旱胁迫应答方面的功能和分子机制尚不清楚。本研究前期从大豆中筛选出1个表达受干旱诱导显著上调的bHLH家族基因GmbHLH130,结合生物信息学技术、亚细胞定位、转录激活活性分析及转基因拟南芥相关试验初步探究了其在植物干旱胁迫中的功能。本研究结果将为进一步解析大豆干旱胁迫应答机制和抗旱品种的分子育种提供理论依据。
1 材料与方法
1.1 系统进化树构建
下载已报道的参与植物非生物胁迫(包括干旱胁迫、盐胁迫)调控的bHLH家族成员的氨基酸序列,包括MfbHLH38(MT383747)、GmbHLH300(NP_001354033)、OsbHLH148(AK071734)、VvbHLH1(XP_002270239)、AtbHLH17(At2g46510)、FtbHLH3(KU296217)、ThbHLH1(KM101094)、GmbHLH57(XP_003540203)、OsbHLH035(AK106292)、AtbHLH122(At1g51140)、AtbHLH68(At4g29100)、OsbHLH068(Os04g0631600)、AtbHLH112(AT1G61660)。将整理好的氨基酸序列与GmbHLH130(LOC100812802)序列使用Clustal X软件进行多重比对,利用MAGA-X软件再次分析氨基酸多重比对结果,然后利用邻接法(Neighbor-joining,NJ)绘制系统进化树,参数设置:泊松模型,Bootstrap=1 000。
1.2 大豆种植及模拟干旱处理
试验用栽培大豆品种为Williams 82。将大豆种子在水中浸泡6 h后均匀铺在润湿的滤纸上萌发,待长出胚根(约1.0~1.5 cm)后移至石英砂中,用1/2 Hoagland营养液浇灌,在生长室进行培养,生长环境:温度25 ℃,空气相对湿度60%~70%,长日照(昼16 h/夜8 h)。待幼苗的第一对真叶展开后,挑选株高一致的植株放置在黑暗条件下继续培养24 h。用含有20% 聚乙二醇(PEG)的1/2 Hoagland营养液(干旱处理)浇灌,分别在0 h、3 h、6 h、12 h和24 h取大豆植株的根和叶,液氮速冻后放于超低温冰箱保存,每个时间点、每个组织均包括3组生物学重复。
1.3 总RNA提取和基因表达水平检测
使用RNA提取试剂盒[生工生物工程(上海)股份有限公司]提取大豆根和叶的总RNA,使用Nanodrop 2000测定RNA浓度,根据反转录试剂盒[SuperScript III First-Strand Synthesis SuperMix,赛默飞世尔科技(中国)有限公司]的说明书操作合成cDNA。以合成的cDNA为模板进行半定量PCR分析基因的表达情况,PCR反应总体系为20.0 μl:10.0 μl Taq Mix[翌圣生物科技(上海)股份有限公司],8.0 μl去离子水,正向和反向引物各0.5 μl,cDNA模板1.0 μl,以大豆GmUBI3基因作为内参基因,本研究所用的引物见表1。
1.4 GUS(β-葡萄糖苷酸酶基因)组织化学染色
以GmbHLH130基因起始密码子上游1 500 bp序列作为启动子区,以大豆DNA为模板克隆启动子片段,插入双元表达载体PBI101获得融合质粒PGmbHLH130::GUS。将质粒PGmbHLH130::GUS导入根癌农杆菌GV3101,利用浸花转化法获得转基因拟南芥,并筛选得到T3代纯合子。将在正常MS培养基上培养7 d的拟南芥幼苗分别用去离子水和20% PEG处理24 h,然后取幼苗浸没到GUS染色液[1.44 ml 1 mol/L Na2HPO4、1.06 ml 1 mol/L NaH2PO4、 1.00 ml 100 mmol/L K4Fe(CN)6·3H2O、1.00 ml 100 mmol/L K3Fe(CN)6、500 μl 500 mmol/L乙二胺四乙酸(EDTA)、50 μl TritonX-100、20 mg 5-溴-4-氯-3-吲哚葡萄糖苷(X-Gluc),定容至50.00 ml]中,置于37 ℃,避光染色12 h。倒掉GUS染色液后,用75%乙醇脱色,最后观察GUS染色情况。
1.5 亚细胞定位检测
将不含终止密码子的编码序列(CDS)片段插入pCAMBIA1300-GFP载体,获得重组质粒35S::GmbHLH130::GFP并导入根癌农杆菌GV3101。根据已报道的方法[18],将含有重组质粒的农杆菌菌液注射到生长旺盛的本氏烟草叶片中,以注射35S:: GFP空载体的叶片为对照,继续培养24 h,使用激光共聚焦显微镜观察烟草叶片细胞中的绿色荧光信号。
1.6 转录激活活性检测
克隆GmbHLH130基因全长CDS插入酵母表达载体pGBKT7中,获得重组质粒pGBKT7-GmbHLH130。分别将空载体pGBKT7和重组质粒pGBKT7-GmbHLH130导入酵母菌株Y2H Gold。经PCR检测后,选取阳性单菌落接种到酵母浸出粉葡萄糖液体培养基(YPDA)中,待OD600为0.8~1.0时,抽取菌液分别滴到YPDA、SD/-TRP/-HIS和SD/-TRP/-HIS/+X-α-Gal固体培养基上,5 d后根据菌落的生长情况判断转录因子的激活活性。
1.7 GmbHLH130基因过表达拟南芥表型分析
克隆GmbHLH130基因全长CDS插入双元表达载体pFGC5941的35S启动子下游。利用根癌农杆菌介导的浸花法转化野生型拟南芥,将收获的T0代种子播种在含有5 mg/L草铵膦的MS培养基上筛选抗性植株。此外,提取RNA进行半定量PCR检测外源基因是否成功高表达,选取外源基因高表达的株系,筛选到T3代,获得纯合材料用于初步鉴定GmbHLH130基因的功能。
取同时期收获的野生型(Col-0,WT)和转基因拟南芥种子于75%乙醇中灭菌10 min,在超净台中吹干种子,然后均匀播种于MS培养基和20% PEG处理的MS培养基中,在4 ℃冰箱中春化3 d后移至生长室正常培养7 d,然后观察不同材料的长势并统计绿色子叶率(叶片能正常生长呈现绿色的植株数占播种下去的种子总数比例)。此外,将表面消毒的拟南芥种子播种并经过4 ℃春化后,在生长室垂直放置培养,7 d后观察并测定不同材料的根长。
野生型拟南芥和过表达株系(L1和L2)的种子经75%乙醇表面消毒后,均匀播种于湿润的营养土和蛭石混合(1∶1)基质上,于4 ℃春化3 d后转移至植物培养室,使用自来水浇灌,保证每盆每次浇水量相同。3周后取长势一致的拟南芥用于干旱处理,使其自然干旱,观察野生型和过表达株系的表型变化。
2 结果与分析
2.1 大豆GmbHLH130的系统进化树和氨基酸序列分析
根据已知报道,本研究整理了参与非生物胁迫调控的bHLH家族成员的氨基酸序列,包括MfbHLH38、GmbHLH300、OsbHLH148、VvbHLH1、AtbHLH17、FtbHLH3、ThbHLH1、GmbHLH57、OsbHLH035、AtbHLH122、AtbHLH68、OsbHLH068、AtbHLH112,与GmbHLH130构建系统进化树。结果显示,所选的bHLH家族成员分布在2个不同分支,GmbHLH130与拟南芥AtbHLH112、AtbHLH122、AtbHLH68以及水稻OsbHLH068在同一分支,并且GmbHLH130与AtbHLH122进化关系最近(图1A)。进一步对比氨基酸序列发现,GmbHLH130与AtbHLH122序列相似度为48.18%,但C端的bHLH结构域十分保守(图1B)。
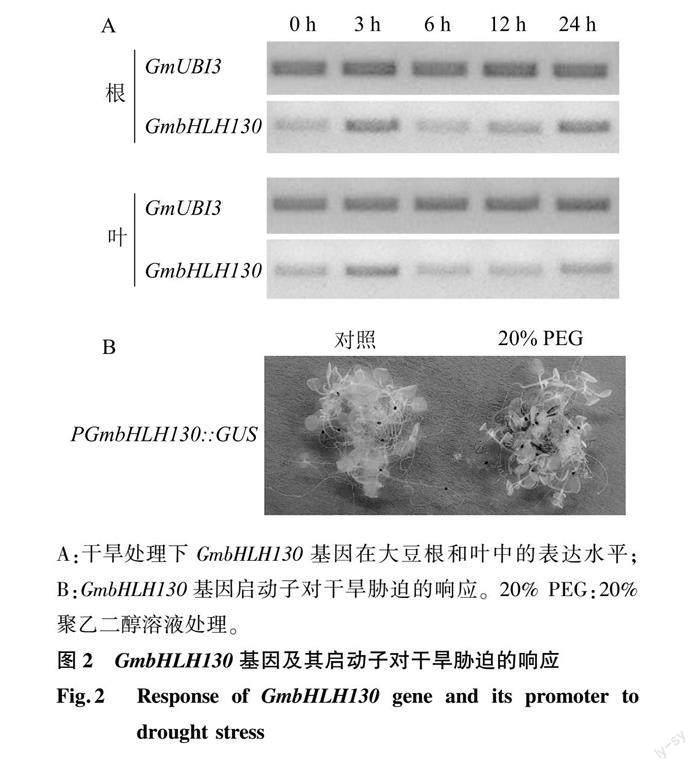
2.2 大豆GmbHLH130基因对干旱胁迫的响应
利用PEG模拟干旱胁迫,检测GmbHLH130基因在大豆幼苗中的表达模式。结果显示,干旱处理3 h时,GmbHLH130基因表达水平在根和叶中均明显增加;但随着处理时间增加,表达水平又恢复至与干旱处理前无明显差异(6 h和12 h),干旱处理24 h时表达水平又增强(图2A)。进一步克隆了GmbHLH130基因起始密码子上游1 500 bp的启动子片段,构建GUS融合载体,并获得相应的转基因拟南芥植株PGmbHLH130::GUS。在MS培养基上生长2周的拟南芥PGmbHLH130::GUS被随机分为2组(各10株),分别用去离子水和20% PEG溶液处理,GUS染色结果显示对照组未观察到蓝色,而处理组观察到明显的蓝色,说明GmbHLH130基因启动子受干旱胁迫诱导激活β-葡萄糖苷酸酶基因的表达,生成的β-葡萄糖苷酸酶与染色液中的底物反应生成蓝色物质(图2B)。
2.3 GmbHLH130蛋白的亚细胞定位
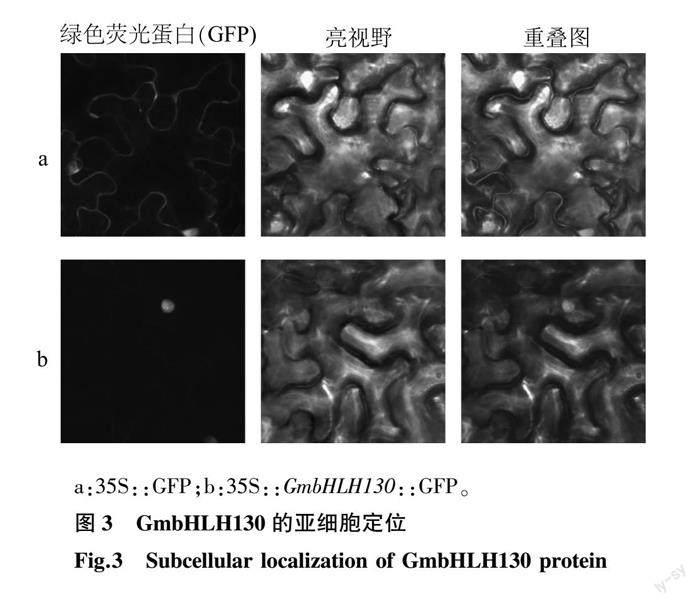
普遍认为转录因子蛋白定位于细胞核内发挥转录调控作用[18]。为了研究大豆转录因子蛋白GmbHLH130的亚细胞定位,将GmbHLH130基因除终止密码子外的CDS插入pCAMBIA1300-GFP载体,构建35S::GmbHLH130::eGFP融合表达质粒。通过根癌农杆菌介导的方法在烟草叶片中进行瞬时表达,以转化pCAMBIA1300-GFP空载体(35S:: eGFP)的叶片为对照。结果显示,35S:: eGFP转化的烟草叶片细胞核和细胞膜上均观察到较强的绿色荧光,而仅在35S::GmbHLH130::eGFP融合质粒注射的叶片细胞核中观察到较强的绿色荧光信号,说明转录因子GmbHLH130蛋白定位在细胞核中(图3)。
2.4 GmbHLH130蛋白具有转录激活活性
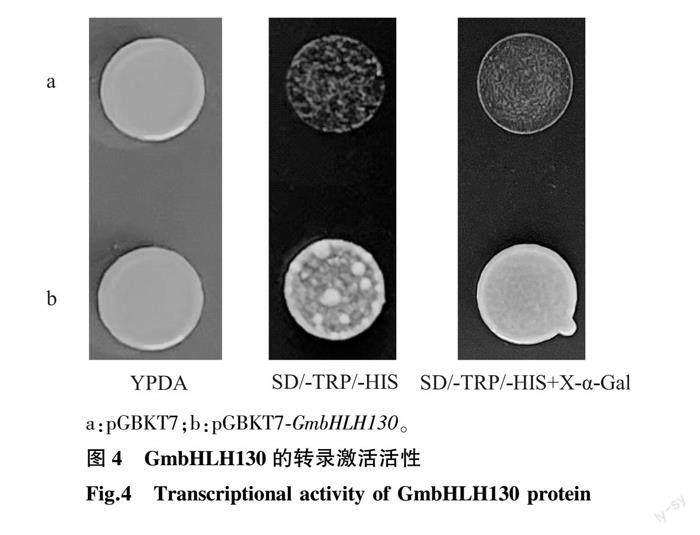
利用酵母双杂交系统验证GmbHLH130转录因子的转录激活活性。结果显示,含有pGBKT7空载体或pGBKT7-GmbHLH130重组质粒的Y2H酵母菌株都能在YPDA培养基上生长。而在SD/-TRP/-HIS营养缺陷培养基上,导入pGBKT7空载体的酵母菌株不能生长,但含pGBKT7-GmbHLH130的酵母可在SD/-TRP/-HIS培養基上生长,且菌斑在含X-α-Gal的SD/-TRP/-HIS培养基上变蓝,说明转录因子GmbHLH130能够激活Y2HGold菌株中HIS、MEL1基因的表达,具有转录激活活性(图4)。
2.5 GmbHLH130基因过表达拟南芥的获得及耐旱性分析
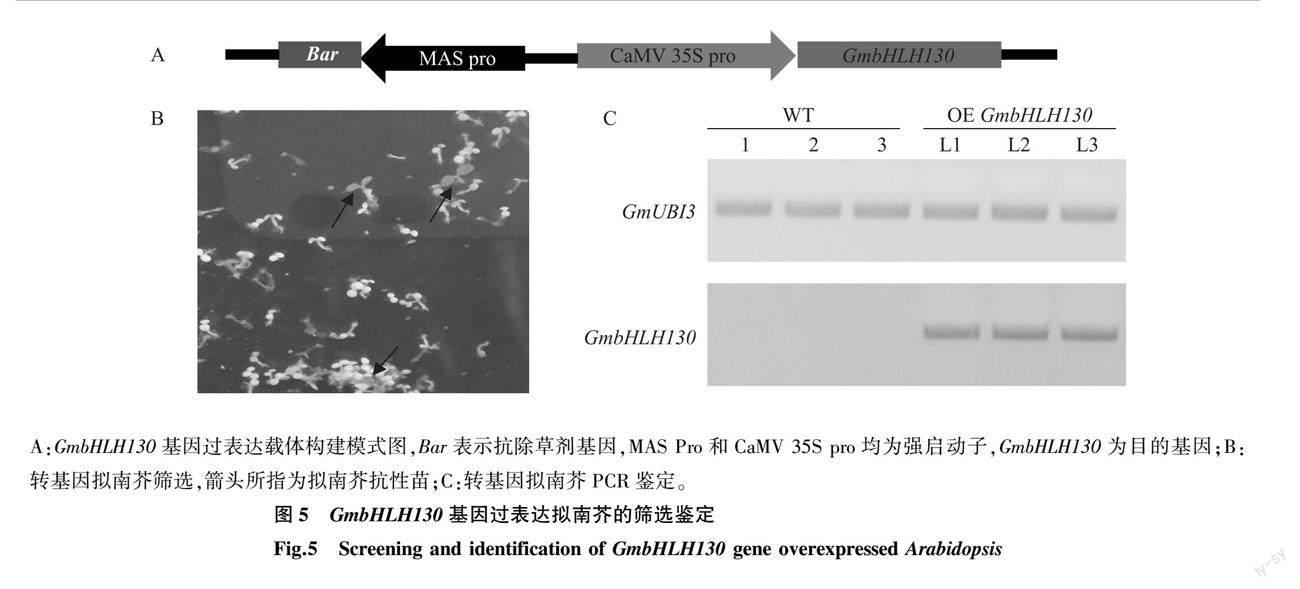
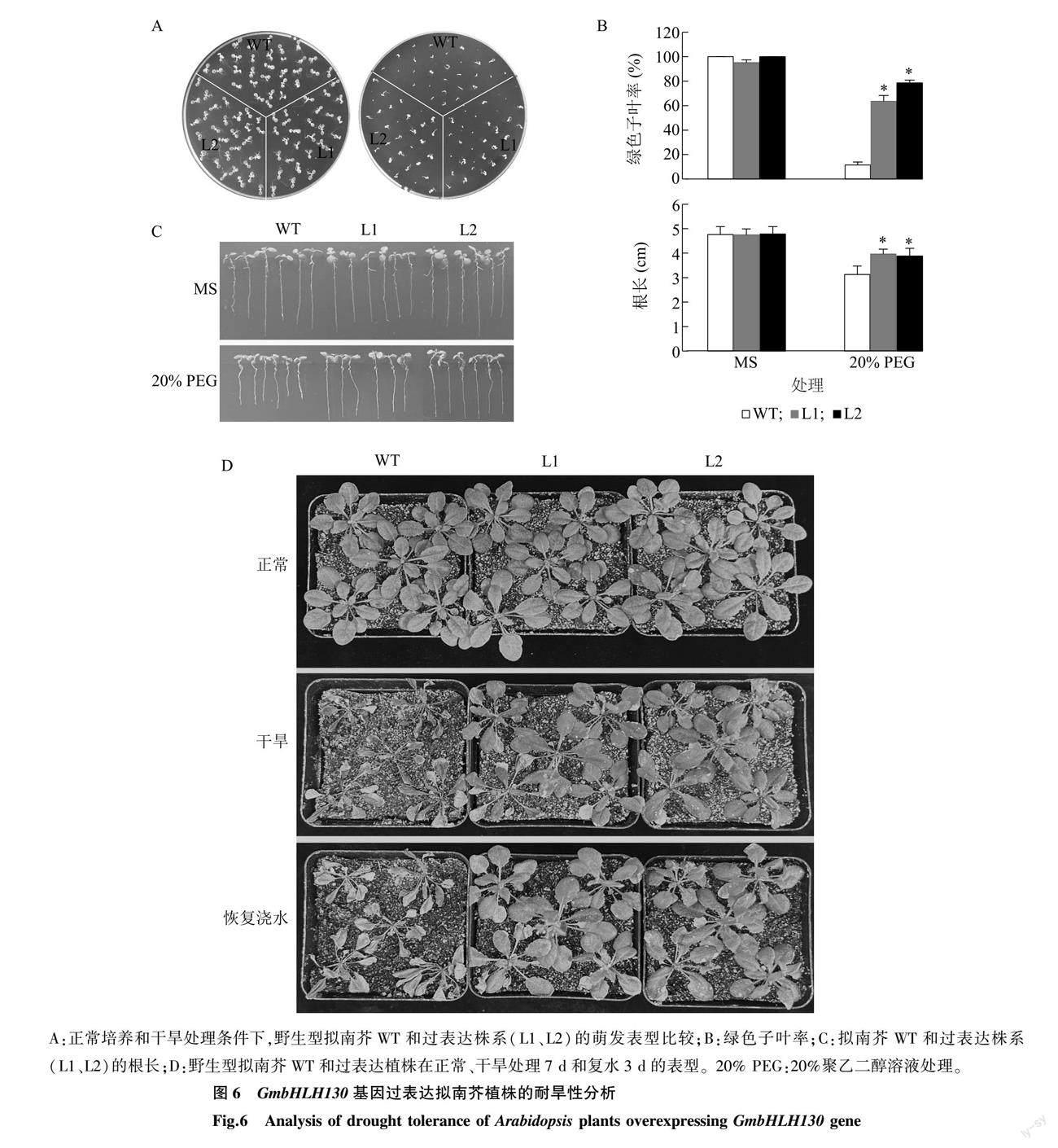
为了验证大豆GmbHLH130基因在植物干旱胁迫响应过程中的功能,本研究构建了该基因的过表达载体(图5A);通过根癌农杆菌介导的浸花法转化,并在含有草铵膦的MS培养基上筛选获得抗性苗(图5B);进一步提取拟南芥野生型和抗性苗的总RNA,半定量PCR检测结果显示,外源基因GmbHLH130在拟南芥抗性苗中成功表达(图5C),说明获得的遗传材料可靠,可用于后续试验。
挑选同时期收获的拟南芥种子,包括野生型(WT)、转基因拟南芥T3代纯合子(2个独立株系L1和L2),均匀播种于正常MS培养基和20% PEG处理的MS培养基上。培养7 d后,观察拟南芥的生长状况,发现正常培养基上WT和转基因拟南芥都能正常生长且子叶几乎都能展开;而在20% PEG处理的MS培养基上野生型种子虽然能够萌发,但是子叶几乎无法正常展开,即受到严重的抑制,过表达株系L1和L2的绿色子叶率则显著高于WT(图6A、图6B)。进一步将在正常MS培养基上萌发2 d的WT、过表达株系L1和L2移至MS培养基和20% PEG处理的培养基上,然后将培养基垂直放置培养7 d,结果显示正常条件下上述材料的根长无明显差异;而在20% PEG处理条件下,L1和L2的根长显著大于WT(图6C)。在盆栽条件下对比过表达GmbHLH130基因植株与WT的耐旱性,发现正常浇水条件下,生长表型无明显差异;而自然干旱导致WT植株枯萎死亡,过表达植株生长虽然受到抑制,但仍然存活;恢复浇水后,WT植株无法复活,而过表达植株叶片变绿、能够继续生长(图6D)。以上结果说明GmbHLH130基因过表达导致转基因拟南芥植株耐旱性增强。
3 討论
研究结果表明,植物bHLH家族基因在植物非生物胁迫逆境响应方面发挥重要作用[4]。甘蓝型油菜BnbHLH122基因在高温、低温、干旱、高盐和渗透胁迫下均显著上调,证明BnbHLH122基因在植株抵御逆境胁迫过程中发挥调节作用[19]。沈方圆等[20]利用全基因组和转录组联合分析在欧洲山杨中鉴定出4个潜在的bHLH抗旱蛋白。本研究通过构建不同植物bHLH家族成员的系统进化树,发现大豆GmbHLH130与拟南芥AtbHLH112、AtbHLH122、AtbHLH68及水稻OsbHLH068在同一分支,并且和AtbHLH122进化关系最近。对基因家族或同源基因进行系统进化分析时,一般认为属于同一亚家族或同一进化分支的成员同源性较高,可能具有类似的生物学功能[21]。鉴于拟南芥AtbHLH122在增强植物耐旱性方面的功能[9],推测进化关系较近的大豆GmbHLH130在干旱胁迫响应和适应过程中可能发挥特定功能。本研究结果显示,20%PEG模拟处理的大豆根和叶中GmbHLH130基因的表达水平都显著上调,且在根中上调更明显,但是随着处理时间延长基因表达出现上下波动,这种干旱诱导表达特性可能与其对干旱胁迫的适应有关[1]。此外,克隆GmbHLH130基因启动子,构建启动子融合GUS报告基因的转基因拟南芥PGmbHLH130::GUS,通过模拟干旱处理和GUS染色显示启动子对干旱胁迫的响应,这一结果也进一步证明GmbHLH130基因参与干旱胁迫调控过程。通常认为转录因子是核定位蛋白,在细胞核中结合到目标基因的特定顺式作用元件调控基因表达[2]。本研究结果显示,大豆GmbHLH130也是核定位转录因子,在Y2H酵母细胞中也能表现明显的转录激活活性,表明GmbHLH130作为激活下游靶基因表达的转录因子发挥作用。
有报道称外源基因在组成型启动子控制下的表达会对转基因拟南芥的正常生长、发育过程产生不利影响[22]。本研究通过农杆菌浸花转化法获得了GmbHLH130基因过表达拟南芥植株。比较拟南芥野生型WT和GmbHLH130过表达株系(L1和L2)在正常培养基和栽培基质上的生长表型,发现过表达植株和WT表型无明显差异,说明大豆基因的异源表达未对拟南芥的生长发育产生不利影响。而在干旱条件下,所有材料的生长均受到抑制,绿色子叶率降低,但是过表达植株的绿色子叶率明显高于WT;此外,模拟干旱条件下,过表达株系的根长也显著大于WT;盆栽自然干旱条件下过表达植株也表现出较WT植株更强的耐旱性。研究结果证明,GmbHLH130基因过表达在不影响植株正常生长过程的前提下增强了拟南芥的耐旱性,这对大豆等作物耐旱性分子育种具有重要意义。本研究筛选到1个受干旱诱导表达上调的bHLH转录因子家族基因GmbHLH130,通过系统进化、亚细胞定位、转录激活活性以及干旱处理的转基因拟南芥表型分析,初步证明了GmbHLH130可增强转基因植株的耐旱性。但是转录因子GmbHLH130参与植物干旱胁迫响应和适应的分子机制仍需进一步研究。
参考文献:
[1] LIANG Y F, MA F, LI B Y, et al. A bHLH transcription factor, SlbHLH96, promotes drought tolerance in tomato[J]. Horticulture Research, 2022, 9: 198.
[2] LIANG B B, WAN S G, MA Q L, et al. A novel bHLH transcription factor PtrbHLH66 from trifoliate orange positively regulates plant drought tolerance by mediating root growth and ROS scavenging[J]. International Journal of Molecular Sciences, 2022, 23(23): 15053.
[3] WANG X P, NIU Y L, ZHENG Y. Multiple functions of MYB transcription factors in abiotic stress responses[J]. International Journal of Molecular Sciences, 2021, 22(11): 6125.
[4] LIU Y J, JI X Y, NIE X G, et al. Arabidopsis AtbHLH112 regulates the expression of genes involved in abiotic stress tolerance by binding to their E-box and GCG-box motifs[J]. The New Phytologist, 2015, 207(3): 692-709.
[5] JOO H, BAEK W, LIM C W, et al. Post-translational modifications of bZIP transcription factors in abscisic acid signaling and drought responses[J]. Current Genomics, 2021, 22(1): 4-15.
[6] KHOSO M A, HUSSAIN A, RITONGA F N, et al. WRKY transcription factors (TFs): molecular switches to regulate drought, temperature, and salinity stresses in plants[J]. Frontiers in Plant Science, 2022, 13: 1039329.
[7] DEBBARMA J, SARKI Y N, SAIKIA B, et al. Ethylene response factor (ERF) family proteins in abiotic stresses and CRISPR-Cas9 genome editing of ERFs for multiple abiotic stress tolerance in crop plants: a review[J]. Molecular Biotechnology, 2019, 61(2): 153-172.
[8] BABITHA K C, RAMU S V, PRUTHVI V, et al. Co-expression of AtbHLH17 and AtWRKY28 confers resistance to abiotic stress in Arabidopsis[J]. Transgenic Research, 2013, 22(2): 327-341.
[9] LIU W W, TAI H H, LI SS, et al. bHLH122 is important for drought and osmotic stress resistance in Arabidopsis and in the repression of ABA catabolism[J]. The New phytologist, 2014, 201(4): 1192-1204.
[10]WANG F B, ZHU H, KONG W L, et al. The Antirrhinum AmDEL gene enhances flavonoids accumulation and salt and drought tolerance in transgenic Arabidopsis[J]. Planta, 2016, 244(1): 59-73.
[11]QIU J R, HUANG Z, XIANG X Y, et al. MfbHLH38, a Myrothamnus flabellifolia bHLH transcription factor, confers tolerance to drought and salinity stresses in Arabidopsis[J]. BMC Plant Biology, 2020, 20(1): 542.
[12]刘 福,陈 诚,张凯旋,等. 日本百脉根LjbHLH34基因克隆及耐旱功能鉴定[J]. 草业学报, 2023, 32(1): 178-191.
[13]VALLIYODAN B, YE H, SONG L, et al. Genetic diversity and genomic strategies for improving drought and waterlogging tolerance in soybeans[J]. Journal of Experimental Botany, 2017, 68(8): 1835-1849.
[14]李文滨,宋春晓,苌兴超,等.干旱胁迫下20个大豆品种抗旱性评价[J].东北农业大学学报, 2019, 50(4): 1-10.
[15]XU Z L, LIU X Q, HE X L, et al. The soybean basic Helix-Loop-Helix transcription factor ORG3-like enhances cadmium tolerance via increased iron and reduced cadmium uptake and transport from roots to shoots[J]. Frontiers in Plant Science, 2017, 8:1098.
[16]CHENG Q, DONG L D, GAO T J, et al. The bHLH transcription factor GmPIB1 facilitates resistance to Phytophthora sojae in Glycine max[J]. Journal of Experimental Botany, 2018, 69(10): 2527-2541.
[17]LI L, GAO W W, PENG Q, et al. Two soybean bHLH factors regulate response to iron deficiency[J]. Journal of Integrative Plant Biology, 2018, 60(7): 608-622.
[18]KU Y S, NI M, MUNOZ N B, et al. ABAS1 from soybean is a 1R-subtype MYB transcriptional repressor that enhances ABA sensitivity[J]. Journal of Experimental Botany, 2020, 71(10): 2970-2981.
[19]唐佳佳,万云宝,王茂林.甘蓝型油菜BnbHLH122基因的克隆、表达模式及胁迫响应分析[J]. 四川大学学报(自然科学版), 2021, 58(2): 180-188.
[20]沈方圆,王岚春,李 校. 欧洲山杨bHLH转录因子家族全基因组分析[J]. 四川大学学报(自然科学版), 2021, 58(3): 179-187.
[21]CHEN Y, ZHU P P, WU S Y. Identification and expression analysis of GRAS transcription factors in the wild relative of sweet potato Ipomoea trifida[J]. BMC Genomics, 2019, 20(1): 911.
[22]LI P, ZHANG B, SU T, et al. BrLAS, a GRAS transcription factor from Brassica rapa, is involved in drought stress tolerance in transgenic Arabidopsis[J]. Frontiers in Plant Science, 2018, 9:1792.
(責任编辑:陈海霞)

