CRISPR基因组编辑技术在传染病致病机理研究和防诊治中的应用
杨臻嵘,周钢桥
综 述
CRISPR基因组编辑技术在传染病致病机理研究和防诊治中的应用
杨臻嵘1,2,周钢桥1,2
1. 中国科学技术大学生命科学学院,生命科学与医学部,合肥 230022 2. 军事科学院军事医学研究院辐射医学研究所,医学蛋白质组全国重点实验室,国家蛋白质科学中心,北京 100850
CRISPR基因组编辑技术在基因操作和传染病研究等方面展现出巨大的应用前景,对于有效控制和治愈传染病具有重要价值。通过其构建的细胞、类器官和动物疾病模型,为探索传染病相关分子机制提供了极大便利。CRISPR筛选技术使得高通量鉴定传染病相关风险因子成为可能。基于CRISPR的新型分子诊断工具为病原体的检测提供了更灵敏和快速的方法。利用CRISPR工具敲入抗性基因或破坏风险基因和病毒基因组,有望实现预防或治疗传染病。本综述讨论了CRISPR基因组编辑技术在疾病模型制备、传染病风险因子筛选、病原体诊断和传染病防治中的应用,以期为后续传染病的研究和防诊治提供参考。
传染病;CRISPR;致病机理;诊断;防治
传染病是由病原体引起的能在人与人、人与动物或动物与动物之间传播的一类疾病。目前我国将法定报告的传染病分为甲、乙、丙三类,另外,还包括按照甲类管理开展应急监测报告的其他传染病,以及按照乙类、丙类管理的其他传染病。传染病的主要传播途径包括接触、空气、水和食物以及虫媒传播等方式。人类活动范围的扩大和频繁的跨境流动等,为病原体的传播和扩散提供了有利条件,尤其当病原体的传播速度较快时,传染病便会爆发成为一种流行病。自21世纪以来,全球已发生过多次传染病大流行,如2003年的严重急性呼吸综合征(severe acute respiratory syndrome,SARS)、2009年的甲型H1N1流感、2012年的中东呼吸综合征(Middle East respiratory syndrome,MERS)、2014年的埃博拉病毒病(Ebola virus disease,EVD),以及2019年的新型冠状病毒肺炎(coronavirus disease 2019,COVID-19)。这些传染病严重危害人类生命健康,并给经济和社会造成巨大伤害。
成簇的规律间隔的短回文重复序列(clustered regularly interspaced short palindromic repeats,CRISPR)与CRISPR相关蛋白(CRISPR-associated proteins,Cas)是源自细菌和古菌中应对外源核酸入侵的免疫防御系统[1],现被开发为基因组编辑的强大工具,广泛应用于生物、医学及环境等各个研究领域。本综述重点关注CRISPR基因组编辑技术在传染病致病机理研究、诊断和防治中的应用,将以多种典型传染病为例,系统讨论该技术在疾病模型制备、传染病风险因子筛选、病原体诊断和传染病防治中发挥的作用,以期为后续传染病的研究和防诊治提供参考。
1 基因组编辑技术简介
1.1 基因组编辑技术发展简史
1.1.1 归巢核酸内切酶
归巢核酸内切酶(homing endonucleases,HEs),又称巨型核酸酶(meganucleases,MNs),是指由内含子或内含肽(inteins)编码的核酸内切酶,可识别并切割14~40 bp的同源等位基因,引起DNA单链或双链断裂,然后利用细胞的同源定向修复(homology- directed repair,HDR)机制(I类内含子和内含肽),或以内含子RNA为模板进行反转录(II类内含子),将编码自身的序列拷贝到靶位点中[2]。可移动内含子及其归巢核酸内切酶的发现可追溯到20世纪70年代。1978年,有学者使用电子显微镜观察到了酿酒酵母()线粒体核糖体DNA(ribosomal DNA,rDNA)中的插入序列[3],随后通过一系列研究,测序和鉴定了多种归巢核酸内切酶[4]。目前至少发现了6个归巢核酸内切酶基因(homing endonuclease genes,HEGs)家族,包括LAGLIDADG、HNH、His-Cys框、GIY-YIG、PD-(D/E)xK和EDxHD[4]。LAGLIDADG家族中,来自酿酒酵母线粒体的I-I和来自莱茵衣藻()叶绿体的I-I归巢核酸内切酶及其变体已广泛应用于基因工程,在哺乳动物细胞[5~9]、细菌[10,11]、植物[12~14]和小鼠[15,16]中实现了定向基因修饰。
1.1.2 锌指核酸酶
锌指核酸酶(zinc-finger nucleases,ZFNs),是指通过将工程化的锌指(zinc-fingers,ZFs)DNA结合域与I核酸内切酶的非特异性切割结构域融合而生成的一种人工核酸酶[17,18]。其DNA结合域通常包含3个连续的锌指蛋白模块,单个锌指蛋白可识别3bp的DNA序列[19]。I的切割结构域需通过二聚化才能发挥切割DNA的作用[20],因此,常使用一对ZFNs来靶向DNA,产生DNA双链断裂(double- strand breaks,DSBs),进而通过非同源末端连接(nonhomologous end-joining,NHEJ)或HDR来修复基因组。利用ZFN,研究人员已成功在果蝇[21,22]、线虫[23]、斑马鱼[24,25]和小鼠[26~29]等物种中实现了基因诱变或靶基因替换。在人类中,已利用ZFN实现了单基因疾病的治疗,例如,敲除艾滋病患者CD4+T细胞中的基因可显著降低血液中的病毒载量[30]。
1.1.3 转录激活因子样效应物核酸酶
转录激活因子样效应物核酸酶(transcription activator-like effector nucleases,TALENs),是由来自黄单胞菌属()的类转录激活因子效应物蛋白(TAL effectors,TALEs)的DNA结合域与I的切割结构域融合而构建得到的一种核酸内切酶[31]。工程化TALEN通常包含多个TALEs模块,每个TALE可识别1个碱基。与ZFN技术类似,常使用一对TALENs来靶向目标基因[32]。TALEN技术已广泛应用于酵母、动植物细胞的基因组编辑[33],并在治疗婴儿急性B淋巴细胞白血病(B-cell acute lymphoblastic leukemia,B-ALL)中实现了对免疫细胞的编辑[34]。
1.1.4 CRISPR基因组编辑技术
CRISPR/Cas系统是细菌中保护其免受外源核酸入侵的免疫防御系统[1],其通过CRISPR向导RNA(guide RNAs,gRNAs)与外源核酸互补配对,然后由Cas蛋白发挥切割作用,从而破坏入侵的外源核酸。CRISPR/Cas系统的发现可追溯到20世纪80年代。1987年,研究人员首次在大肠杆菌()碱性磷酸酶基因附近发现了一种规律间隔的重复序列[35],随后又在多种细菌和古菌基因组中检测到类似的序列[36]。2002年,正式将这种成簇的规律间隔的短回文重复序列命名为CRISPR[37]。CRISPR通常与CRISPR相关蛋白基因一起在微生物基因组中被发现[38]。多项研究分析表明,CRISPR的间隔序列与噬菌体和质粒具有同源性[39~41],且由于Cas蛋白具有预测的核酸酶和解旋酶结构域,研究人员提出CRISPR/Cas系统可能是一种应对噬菌体和质粒入侵的适应性免疫防御系统[42]。2007年,嗜热链球菌()感染烈性噬菌体的实验为CRISPR/Cas介导适应性免疫提供了第一个实验证据[1]。
CRISPR/Cas系统依据发挥效应的Cas蛋白亚基数量、序列相似性、系统发育及基因座位点组织特征划分类型和亚型[43],其可分为两大类:第一类系统通过多个Cas蛋白组成的复合物降解外源核酸,包括I型(如Cas3和Cas8)、III型(如Cas10)和IV型(如Csf1);第二类系统则依靠单个Cas蛋白发挥作用,包括II型(如Cas9)、V型(如Cas12a/Cpf1和Cas12b/C2c1)和VI型(如Cas13a/C2c2和Cas13b)。目前,应用较多的系统有II型的CRISPR/Cas9、V型的CRISPR/Cas12和VI型的CRISPR/Cas13。前者依靠工程化的单链向导RNA(single guide RNAs,sgRNAs)靶向双链DNA(double-strand DNAs,dsDNAs)[44],已广泛应用于基因功能和机制的研究,以及疾病的治疗[38,45],后两者分别靶向切割DNA[46]和RNA[47],但其切割后仍与靶标结合,然后非特异地切割其他单链DNA(single-strand DNAs,ssDNAs)或单链RNA(single-strand RNAs,ssRNAs)分子[47,48]。这种反式切割(tans cleavage)或称为附带切割(collateral cleavage)的特性已被用于开发各种分子诊断工具[49~53]。
此外,研究人员还将无切割活性的Cas9(dead Cas9,dCas9)[54]与逆转录酶(如M-MLV)、胞苷脱氨酶(如APOBEC)、腺嘌呤脱氨酶(如TadA)、转录激活结构域(如SAM、VP64和VPR)、转录抑制结构域(如KRAB)或表观遗传修饰因子(如p300、LSD1、MQ1和TET1)融合,开发出单碱基编辑器[55]、转录激活系统[56]、转录抑制系统[57]和表观遗传编辑系统[58]等,极大地扩展了CRISPR/Cas系统的应用范围。基因组编辑技术发展的时间线总结如图1所示。
1.2 CRISPR与其他基因组编辑技术的比较
归巢核酸内切酶、ZFN和TALEN等基因组编辑工具发挥作用依赖于蛋白质-DNA相互作用,与之相比,CRISPR/Cas系统发挥作用依赖于RNA-DNA或RNA-RNA相互作用,因此具有诸多优势。例如,在操作难度和成本方面,天然的归巢核酸内切酶识别的DNA序列有限,需要改造其DNA结合域以识别更多靶位点,因此操作困难,成本很高;ZFN中串联的每个ZF识别3个核苷酸序列,因此识别不同靶位点需要串联特定的ZFs,操作也十分繁琐,成本较高;TALEN中虽然每个TALE仅识别1个核苷酸序列,但识别特定序列同样需要在蛋白质层面进行设计和改造,操作较繁琐,成本也较高;而CRISPR/Cas系统仅根据靶位点序列设计相应sgRNA即可实现靶向功能,操作较简单,成本较低[59,60]。在多靶点编辑方面,目前只有CRISPR/Cas系统可通过同时表达针对不同靶位点的sgRNA,实现对基因组多位点的敲除、激活或抑制[61]。基因组编辑技术的比较总结见表1。
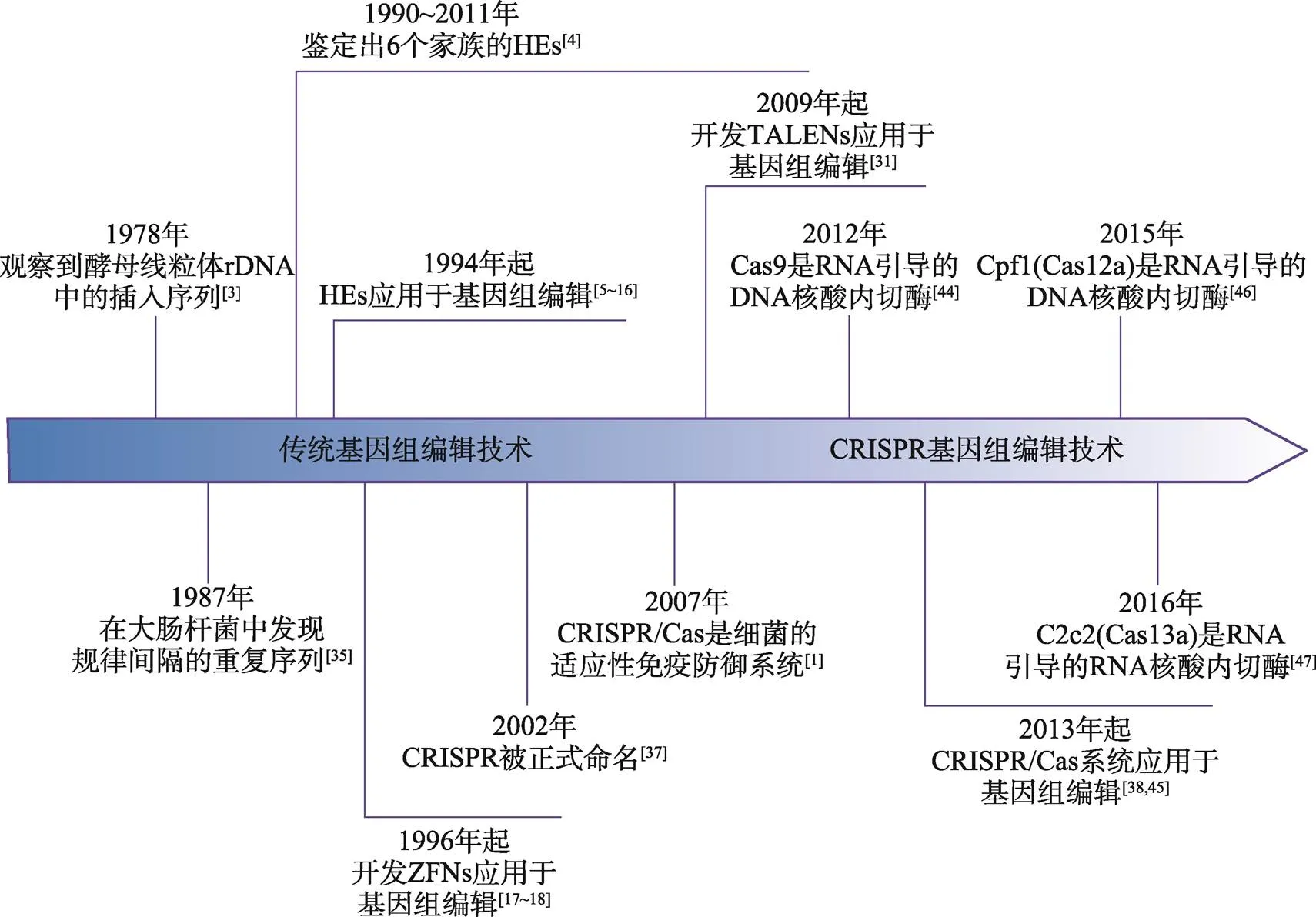
图1 基因组编辑技术发展的时间线
rDNA:核糖体DNA;HEs:归巢核酸内切酶;ZFNs:锌指核酸酶;TALENs:转录激活因子样效应物核酸酶;CRISPR/Cas:成簇的规律间隔的短回文重复序列/CRISPR相关蛋白。
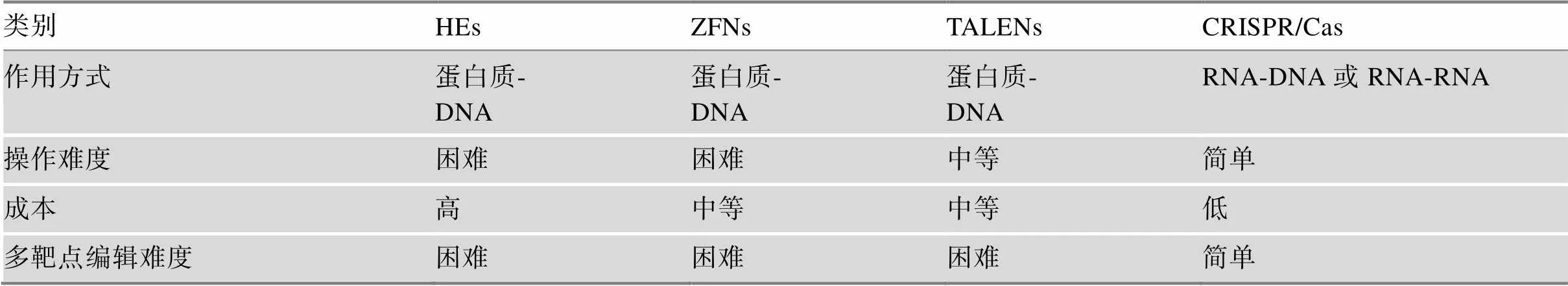
表1 基因组编辑技术的比较
HEs:归巢核酸内切酶;ZFNs:锌指核酸酶;TALENs:转录激活因子样效应物核酸酶;CRISPR/Cas:成簇的规律间隔的短回文重复序列/CRISPR相关蛋白。
2 CRISPR基因组编辑技术在传染病致病机理研究中的应用
2.1 制备疾病模型用于病理研究
在细胞、类器官或动物中采用CRISPR/Cas9系统制备特定的疾病模型,可模拟天然的基因突变。例如,Ye等[62]将CRISPR/Cas9与piggyBac转座子技术[63]结合,首次在诱导多能干细胞(induced pluripotent stem cells,iPSCs)中高效构建了CCR5Δ32的纯合突变。通过模拟天然突变,证明其在体外分化出的单核和巨噬细胞中能够抵抗人类免疫缺陷病毒1型(human immunodeficiency virus type 1,HIV-1)的感染,这为研究和治愈HIV-1感染提供了可靠的细胞模型。
在病毒学研究中,常用各种细胞系作为体外研究模型,但它们与细胞在体内发生的天然反应有一定差异,而类器官模型的建立将能够更好地模拟机体的生理状态,反映病毒感染后体内的病理变化。例如,肠道类器官可用来支持严重急性呼吸综合征冠状病毒2(severe acute respiratory syndrome coronavirus 2,SARS-CoV-2)等病毒的感染,该类器官的一个主要优点就是可以使用CRISPR工具对其进行有效的基因改造,并可与单细胞组学等新技术相结合[64,65]。
为了生成动物模型用于传染病的致病机理研究,常把CRISPR sgRNA和Cas9 mRNA共同显微注射到受精卵中,以修改后代基因组。例如,Sun等[66]利用CRISPR/Cas9敲入技术,将编码hACE2 cDNA的靶向载体、sgRNA和Cas9 mRNA显微注射到C57BL/6小鼠受精卵中,获得了稳定表达hACE2的小鼠品系,用于支持对SARS-CoV-2的感染,以模拟人类感染后的状态,并在肺的Clara细胞和巨噬细胞中发现了强烈的病毒复制。该模型将有助于SARS-CoV-2传播和发病机制的研究,对测试抗SARS-CoV-2的疫苗和疗法也很有价值。
2.2 筛选传染病风险因子及其机制研究
CRISPR筛选(CRISPR screening)是一种高通量基因组筛选技术,其通过将sgRNA文库转导细胞,对大量细胞的不同基因进行敲除、激活或抑制,然后经过特定筛选并获得富集细胞的全基因组DNA,用于扩增sgRNA基因片段,并进行高通量测序,最后对富集或消耗的sgRNA进行生物信息学分析,从而发掘与表型相关的候选基因(图2)。
CRISPR筛选技术被广泛应用于传染病研究,用于发掘与病原体相关的宿主因子。例如,Li等[67]通过全基因组CRISPR敲除筛选,结合MAIC (meta- analysis by information content)算法,发现宿主因子WDR7、CCDC115和TMEM199对于甲型流感病毒进入和调节V型ATP酶组装至关重要。同时发现,mRNA帽甲基转移酶CMTR1对于病毒抢帽(cap snatching)和调节细胞的自主免疫反应是必需的。Hyrina等[68]通过全基因组CRISPR敲除筛选,鉴定到多个参与调节乙型肝炎病毒表面抗原(HBV surface antigen,HBsAg)的宿主因子,例如,发现ZCCHC14锌指蛋白可与TENT4A/B一起稳定HBsAg的表达,这为治疗慢性乙肝提供了新靶点。Mei等[69]使用膜蛋白组(surfaceome)文库进行CRISPR抑制筛选,发现分泌型糖蛋白OLFML3可通过SOCS3抑制宿主细胞I型干扰素信号通路,从而促进鼻病毒(rhinovirus,RV)的感染。近期,在针对SARS-CoV-2的研究中,多个团队在Vero-E6、Huh7、Calu-3和HEK293T等细胞系中揭示了一系列关键宿主因子及其作用机制。例如,Wei等[70]通过全基因组CRISPR敲除筛选发现非组蛋白染色质结合蛋白HMGB1可通过调节ACE2的表达而影响细胞对SARS-CoV-2的易感性。Baggen等[71]通过全基因组CRISPR敲除筛选发现溶酶体膜蛋白TMEM106B能够促进病毒进入细胞。Biering等[72]通过全基因组CRISPR敲除筛选发现接头蛋白AP1G1等成分有利于SARS-CoV-2的感染,同时通过全基因组CRISPR激活筛选发现膜锚定粘蛋白MUC1、MUC4等能够限制SARS-CoV-2进入细胞。Zhu等[73]通过全基因组CRISPR激活筛选鉴定出LDLRAD3和CLEC4G等膜蛋白能够以不依赖于ACE2的方式介导SARS-CoV-2的进入和感染。
在传染病相关病原细菌的研究中,可通过CRISPR筛选鉴定与致病性和耐药性相关的基因。例如,Lai等[74]在被福氏志贺氏菌() M90T ΔvirG突变株感染的THP-1细胞中进行了全基因组CRISPR敲除和抑制筛选,发现抑制TLR1/2信号通路和丙酮酸分解代谢信号通路可限制细胞内病原体的生长和复制,该项研究揭示了福氏志贺氏菌与天然免疫细胞的相互作用及其潜在发病机制,同时也为志贺氏菌病的治疗提供了见解。Bosch等[75]为了定量化表征结核分枝杆菌()的基因脆弱性(gene vulnerability)即靶基因受到攻击时细胞生存受影响的程度,在结核分枝杆菌H37Rv和HN878菌株中进行了全基因组CRISPR抑制筛选,并构建靶基因敲低与病原菌适应度的关系模型,鉴定出了一批高度脆弱的基因,包括参与蛋白质折叠(和)与分泌()、代谢(、、和)、染色体复制(和)和细胞分裂()等过程的基因,为相关药物开发提供了新靶点,同时也为其他细菌性病原体的研究提供了新思路。尽管CRISPR敲除筛选已广泛应用于真核生物,但在细菌等原核生物中由于基因组编辑效率较低,尚未得到大范围应用[76,77]。而Yan等[78]建立了基于CRISPR切割和NHEJ修复途径的基因组编辑方法,能够高效突变原核生物基因组,将该系统应用于结核分枝杆菌,首次在细菌中实现了高通量CRISPR敲除筛选,同时又结合了CRISPR抑制筛选,鉴定出多个与抗结核药物贝达喹啉(bedaquiline,BDQ)敏感性相关的基因,包括参与药物外排的基因以及参与ATP合成的基因、和等[77],该项研究促进了对结核分枝杆菌的基础研究,为抗结核药物的开发提供了理论依据。

图2 CRISPR筛选流程
图绘制网址:https://www.biorender.com。
3 CRISPR基因组编辑技术在传染病诊断中的应用
利用CRISPR/Cas系统能够高度特异性识别靶标核酸分子的特性,研究人员开发了基于CRISPR的新型分子诊断工具,用于病原体核酸的检测(表2)。这类分子诊断工具主要由具有反式切割活性的Cas蛋白(如Cas12和Cas13)、能与靶标核酸特异性结合的gRNA和非靶标报告分子组成。其中,非靶标报告分子通常为两端分别带有荧光基团(如FAM、TEX、Cy5和HEX)和淬灭基团(如BHQ1和BHQ2)的寡核苷酸(ssDNA或ssRNA)。当该系统识别并切割靶标核酸后,将反式切割非靶标报告分子的核酸,使得荧光基团与淬灭基团物理距离变大而发出荧光,从而指示特定核酸的存在(图3)。

表2 CRISPR基因组编辑技术在传染病诊断、预防和治疗中的应用
CRISPR/Cas:成簇的规律间隔的短回文重复序列/CRISPR相关蛋白;ZIKV:寨卡病毒;DENV:登革热病毒;WNV:西尼罗病毒;YFV:黄热病病毒;SARS-CoV-2:严重急性呼吸综合征冠状病毒2;HPV:人乳头瘤病毒;HIV-1:人类免疫缺陷病毒1型;HBV:乙型肝炎病毒;cccDNA:共价闭合环状DNA;HSV-1:单纯疱疹病毒1型;LCMV:淋巴细胞性脉络膜脑膜炎病毒;IAV:甲型流感病毒;VSV:水泡性口炎病毒。
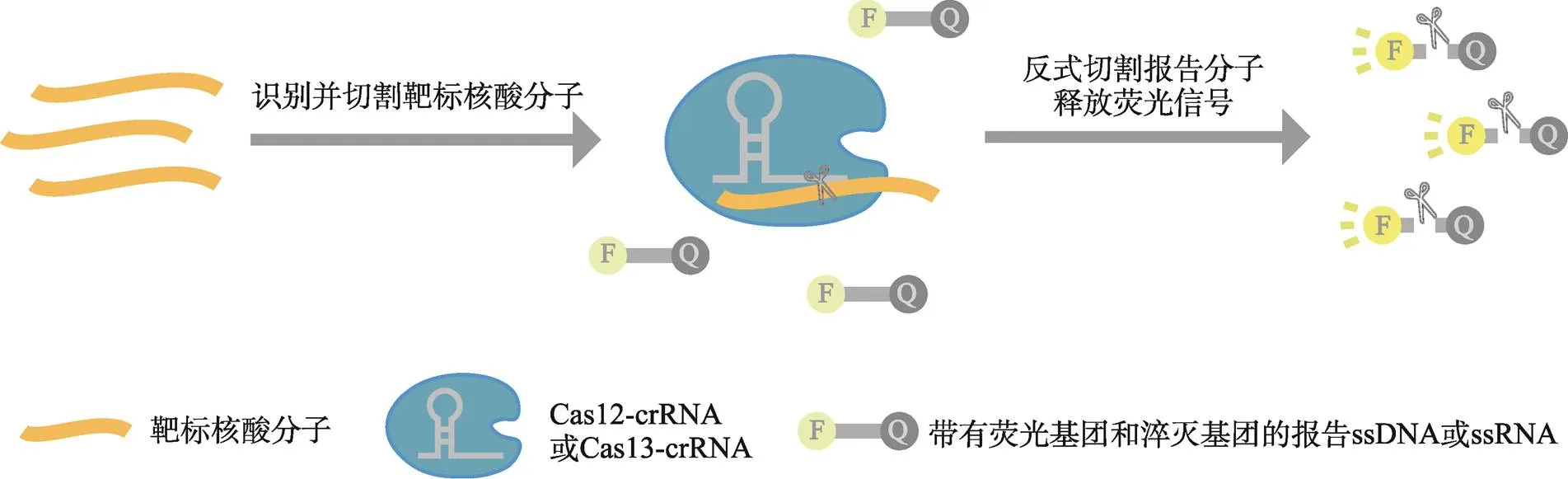
图3 基于CRISPR的诊断工具
crRNA:CRISPR RNA;F:荧光基团;Q:淬灭基团;ssDNA:单链DNA;ssRNA:单链RNA。
例如,2017年,美国麻省理工学院和哈佛大学博德研究所的张锋团队开发了基于CRISPR/Cas13a的核酸检测系统SHERLOCK (specific high-sensitivityenzymatic reporter unlocking),该系统结合了重组聚合酶扩增技术(recombinase polymerase amplification,RPA),能够在恒温下对待测病原体的核酸大量扩增,并获得对应的ssRNA作为底物,若系统能够识别并切割该底物,则接下来将反式切割非靶标报告分子中的ssRNA,从而将淬灭的FAM荧光信号释放出来[51]。该系统能够高灵敏度地检测寨卡病毒(Zika virus,ZIKV)、登革热病毒(dengue virus,DENV)以及多种细菌性病原体,包括大肠杆菌、铜绿假单胞菌()和肺炎克雷伯菌(),且能够区分不同毒株或菌株。2018年,该团队又构建了升级版的SHERLOCK系统,即SHERLOCKv2,它结合了一种对未提取的诊断样品加热来消除核酸酶(heating unextracted diagnostic samples to obliterate nucleases,HUDSON)的技术,能够检测ZIKV、DENV、西尼罗病毒(West Nile virus,WNV)和黄热病病毒(yellow fever virus,YFV),并可区分DENV的4种血清型[79]。升级后的系统能够检测样品中浓度低至0.9×10–18mol/L的ZIKV颗粒,比原系统能识别的病毒最少量缩小了约一半。
2020年,SHERLOCK系统首次被实战用于SARS-CoV-2检测,并取得了不错的效果[80]。同年,张锋团队对基于SHERLOCK技术的SARS-CoV-2检测流程在病毒RNA提取、Cas蛋白选用,如使用来自脂环酸芽孢杆菌()的AapCas12b,以及体系添加物等方面进行了优化[81,82],使其检测灵敏度能达到93.1%,特异性可达98.5%,且能够在1 h以内完成检测。
2018年,美国加州大学伯克利分校的Jennifer Doudna团队开发了基于CRISPR/Cas12a的DNA分子检测系统DETECTR(DNA endonuclease targeted CRISPR trans reporter)[48]。类似的,在系统中加入两端各带有荧光报告基团和淬灭基团的报告ssDNA,当靶标dsDNA或ssDNA存在时,Cas12a识别并切割靶标后,将非特异地切割报告分子的ssDNA,从而释放FAM荧光信号。采用这一系统,研究人员实现了对人乳头瘤病毒(human papillomavirus,HPV)中两种高危亚型HPV16和HPV18的检测。
4 CRISPR基因组编辑技术在传染病预防中的应用
在众多传染病中,病原体的虫媒传播是亟待解决的全球性问题之一,特别是通过蚊子传播病原体,如按蚊传播疟原虫,伊蚊携带登革热病毒、寨卡病毒和黄热病病毒等。现有长效杀虫剂的使用可大大减少疾病的流行,但杀虫剂的过量使用不仅对生态环境造成影响,还可导致耐药性蚊子种群的出现[83,84]。因此,迫切需要强有力的新策略和工具来解决虫媒传播病原体的问题。
已有研究人员从提高蚊子对病原体抗性的角度出发,利用CRISPR/Cas9工具将针对人类恶性疟原虫()的单链抗体基因和敲入斯氏按蚊()印度品系的胚胎中,获得了携带有抗虫基因的后代[85](表2)。此外,还有研究人员从抑制蚊子繁殖的角度出发,使用CRISPR/Cas9工具靶向生育相关的基因,例如对雌性冈比亚按蚊()的基因和雄性埃及伊蚊()的基因进行无效突变(null mutation),使突变蚊子不能产生后代[86,87](表2)。将这些具有抗虫基因或“绝育”的蚊子放归自然,将有望改变蚊子种群的遗传结构,从而减少蚊子传播疾病的能力。
5 CRISPR基因组编辑技术在传染病治疗中的应用
目前,已有多项研究探索了CRISPR基因组编辑技术在临床应用中的可行性(表2)。例如,Xu等[88]首次在造血干祖细胞(hematopoietic stem and progenitor cells,HSPCs)中利用CRISPR/Cas9技术编辑了HIV-1入侵细胞的受体基因,并移植到罹患HIV-1合并急性淋巴细胞白血病的患者中,使患者的急性淋巴细胞白血病完全缓解,且治疗19个月后携带突变的供体细胞在体内仍稳定存活。Dash等[89]通过双重基因编辑,在失活基因的同时切除了整合到基因组上的HIV-1 DNA,使58%受感染的人源化小鼠中前病毒DNA被消除,这为艾滋病的治疗提供了一种新策略。
乙型肝炎病毒(hepatitis B virus,HBV)的基因组DNA进入到细胞核后,可在病毒蛋白和宿主蛋白的作用下形成共价闭合环状DNA (covalently closed circular DNA,cccDNA),从而作为病毒RNA复制的原始模板。只要cccDNA稳定存在,就可以使病毒持续感染,因此,清除肝细胞中的cccDNA对于彻底治愈乙肝非要重要。最近,已有多个团队利用CRISPR/Cas9系统,对受HBV感染的哺乳动物肝细胞进行基因组编辑,靶向切割了HBV的cccDNA,使得HBV的总病毒DNA和cccDNA水平显著下降,病毒复制能力显著降低[90~92]。
采用类似的策略,研究人员成功修饰了受感染细胞中单纯疱疹病毒1型(herpes simplex virus type 1,HSV-1)的基因组,减弱了其毒力,并且未检测到脱靶效应[93,94]。
此外,张锋团队把基于Cas13介导的病毒RNA切割与SHERLOCK快速诊断技术[51]相结合,构建了集诊断与抗病毒治疗于一体的新平台CARVER (Cas13-assisted restriction of viral expression and readout)[95]。实验证明,该系统的Cas13 crRNA能够有效靶向被感染细胞中的淋巴细胞性脉络膜脑膜炎病毒(lymphocytic choriomeningitis virus,LCMV)、甲型流感病毒(influenza A virus,IAV)或水泡性口炎病毒(vesicular stomatitis virus,VSV)的基因组,使得病毒RNA水平显著降低。
6 结语与展望
自从CRISPR基因组编辑技术诞生以来,已被广泛应用于各种物种的基因组编辑,通过其构建的细胞、类器官和动物疾病模型,为研究人员探索传染病相关分子机制提供了极大便利。CRISPR技术使得高通量筛选和鉴定传染病相关的风险因子成为可能。当前,CRISPR技术在传染病诊断和防治研究中也愈发显示出传统方法所不及的优势,SHERLOCK、DETECTR等新型分子诊断工具的开发和应用将提供更灵敏、快速的检测结果。通过靶向宿主易感基因或病毒基因组,携带病原体的蚊虫有望得到控制,HIV-1、HBV等病毒感染患者也有望从根本上得到治愈。
在体外或模式生物中进行CRISPR基因组编辑的操作技术已相对成熟,但要真正应用于临床治疗,还有很多问题需要解决。一是如何保证试验的安全性和有效性。在治疗HIV-1感染患者的案例中[88],研究人员在基础研究方面提高了治疗方法的安全性,并在中断抗逆转录病毒治疗后,持续监测确认无不良免疫反应和脱靶现象,但因为整体基因组编辑效率较低,所以仅被编辑的细胞具有抵抗HIV-1的能力,还不能完全清除体内的病毒。二是如何在体内进行CRISPR/Cas系统的有效递送。病毒载体是进行体内递送的主要手段,目前广泛使用的病毒载体包括慢病毒、腺病毒和腺相关病毒载体,但仍存在一定的局限性,如缺乏组织特异性、引起不良免疫反应和脱靶等问题,而非病毒载体(如脂质颗粒、聚合物颗粒和无机颗粒等)的使用将有望解决这些问题[96,97]。此外,建立合理的生物伦理审查标准和监管机制,对于维护生物伦理、维持生态平衡具有重要意义。例如,经过基因工程修饰的媒介蚊虫,将其放归自然后很可能改变种群基因库,影响种群的进化方向,从而改变整个生态系统,因此,有必要对基因改造后的突变物种进行长期监测。
总之,CRISPR基因组编辑技术的应用加快了对传染病致病机理的研究,为病原体的诊断提供了新的思路,但在传染病防治方面仍有很多工作要做,以实现临床应用的安全有效。
[1] Barrangou R, Fremaux C, Deveau H, Richards M, Boyaval P, Moineau S, Romero DA, Horvath P. CRISPR provides acquired resistance against viruses in prokaryotes., 2007, 315(5819): 1709–1712.
[2] Stoddard BL. Homing endonuclease structure and function., 2005, 38(1): 49–95.
[3] Bos JL, Heyting C, Borst P, Arnberg AC, Van Bruggen EF. An insert in the single gene for the large ribosomal RNA in yeast mitochondrial DNA., 1978, 275(5678): 336–338.
[4] Stoddard BL. Homing endonucleases from mobile group I introns: discovery to genome engineering., 2014, 5(1): 7.
[5] Sargent RG, Brenneman MA, Wilson JH. Repair of site-specific double-strand breaks in a mammalian chromosome by homologous and illegitimate recombination., 1997, 17(1): 267–277.
[6] Cohen-Tannoudji M, Robine S, Choulika A, Pinto D, El Marjou F, Babinet C, Louvard D, Jaisser F. I-I-induced gene replacement at a natural locus in embryonic stem cells., 1998, 18(3): 1444–1448.
[7] Cabaniols JP, Pâques F. Robust cell line development using meganucleases., 2008, 435: 31–45.
[8] Cabaniols JP, Ouvry C, Lamamy V, Fery I, Craplet ML, Moulharat N, Guenin SP, Bedut S, Nosjean O, Ferry G, Devavry S, Jacqmarcq C, Lebuhotel C, Mathis L, Delenda C, Boutin JA, Duchâteau P, Cogé F, Pâques F. Meganuclease-driven targeted integration in CHO-K1 cells for the fast generation of HTS-compatible cell-based assays., 2010, 15(8): 956–967.
[9] Redondo P, Prieto J, Muñoz IG, Alibés A, Stricher F, Serrano L, Cabaniols JP, Daboussi F, Arnould S, Perez C, Duchateau P, Pâques F, Blanco FJ, Montoya G. Molecular basis of xeroderma pigmentosum group C DNA recognition by engineered meganucleases., 2008, 456(7218): 107–111.
[10] Seligman LM, Stephens KM, Savage JH, Monnat RJ. Genetic analysis of theI-I mobile intron homing system in., 1997, 147(4): 1653–1664.
[11] Pósfai G, Kolisnychenko V, Bereczki Z, Blattner FR. Markerless gene replacement instimulated by a double-strand break in the chromosome., 1999, 27(22): 4409–4415.
[12] Puchta H, Dujon B, Hohn B. Two different but related mechanisms are used in plants for the repair of genomic double-strand breaks by homologous recombination., 1996, 93(10): 5055–5060.
[13] Antunes MS, Smith JJ, Jantz D, Medford JI. Targeted DNA excision inby a re-engineered homing endonuclease., 2012, 12: 86.
[14] Djukanovic V, Smith J, Lowe K, Yang MZ, Gao HR, Jones S, Nicholson MG, West A, Lape J, Bidney D, Carl Falco S, Jantz D, Alexander Lyznik L. Male-sterile maize plants produced by targeted mutagenesis of the cytochrome P450-like gene (MS26) using a re-designed I-I homing endonuclease., 2013, 76(5): 888–899.
[15] Rouet P, Smih F, Jasin M. Introduction of double- strand breaks into the genome of mouse cells by expression of a rare-cutting endonuclease., 1994, 14(12): 8096–8106.
[16] Gouble A, Smith J, Bruneau S, Perez C, Guyot V, Cabaniols JP, Leduc S, Fiette L, Avé P, Micheau B, Duchateau P, Pâques F. Efficient in toto targeted recombination in mouse liver by meganuclease-induced double-strand break., 2006, 8(5): 616–622.
[17] Kim YG, Cha J, Chandrasegaran S. Hybrid restriction enzymes: zinc finger fusions toI cleavage domain., 1996, 93(3): 1156–1160.
[18] Cathomen T, Keith Joung J. Zinc-finger nucleases: the next generation emerges., 2008, 16(7): 1200– 1207.
[19] Pavletich NP, Pabo CO. Zinc finger-DNA recognition: crystal structure of a Zif268-DNA complex at 2.1 Å., 1991, 252(5007): 809–817.
[20] Bitinaite J, Wah DA, Aggarwal AK, Schildkraut I.I dimerization is required for DNA cleavage., 1998, 95(18): 10570–10575.
[21] Bibikova M, Golic M, Golic KG, Carroll D. Targeted chromosomal cleavage and mutagenesis inusing zinc-finger nucleases., 2002, 161(3): 1169–1175.
[22] Bibikova M, Beumer K, Trautman JK, Carroll D. Enhancing gene targeting with designed zinc finger nucleases., 2003, 300(5620): 764.
[23] Morton J, Davis MW, Jorgensen EM, Carroll D. Induction and repair of zinc-finger nuclease-targeted double-strand breaks insomatic cells., 2006, 103(44): 16370–16375.
[24] Meng XD, Noyes MB, Zhu LJ, Lawson ND, Wolfe SA. Targeted gene inactivation in zebrafish using engineered zinc-finger nucleases., 2008, 26(6): 695–701.
[25] Doyon Y, McCammon JM, Miller JC, Faraji F, Ngo C, Katibah GE, Amora R, Hocking TD, Zhang L, Rebar EJ, Gregory PD, Urnov FD, Amacher SL. Heritable targeted gene disruption in zebrafish using designed zinc-finger nucleases., 2008, 26(6): 702–708.
[26] Connelly JP, Barker JC, Pruett-Miller S, Porteus MH. Gene correction by homologous recombination with zinc finger nucleases in primary cells from a mouse model of a generic recessive genetic disease., 2010, 18(6): 1103–1110.
[27] Meyer M, de Angelis MH, Wurst W, Kühn R. Gene targeting by homologous recombination in mouse zygotes mediated by zinc-finger nucleases., 2010, 107(34): 15022–15026.
[28] Carbery ID, Ji D, Harrington A, Brown V, Weinstein EJ, Liaw L, Cui XX. Targeted genome modification in mice using zinc-finger nucleases., 2010, 186(2): 451–459.
[29] Cui XX, Ji D, Fisher DA, Wu YM, Briner DM, Weinstein EJ. Targeted integration in rat and mouse embryos with zinc-finger nucleases., 2011, 29(1): 64–67.
[30] Tebas P, Stein D, Tang WW, Frank I, Wang SQ, Lee G, Spratt SK, Surosky RT, Giedlin MA, Nichol G, Holmes MC, Gregory PD, Ando DG, Kalos M, Collman RG, Binder-Scholl G, Plesa G, Hwang WT, Levine BL, June CH. Gene editing ofin autologous CD4 T cells of persons infected with HIV., 2014, 370(10): 901–910.
[31] Christian M, Cermak T, Doyle EL, Schmidt C, Zhang F, Hummel A, Bogdanove AJ, Voytas DF. Targeting DNA double-strand breaks with TAL effector nucleases., 2010, 186(2): 757–761.
[32] Bak RO, Gomez-Ospina N, Porteus MH. Gene editing on center stage., 2018, 34(8): 600–611.
[33] Joung JK, Sander JD. TALENs: a widely applicable technology for targeted genome editing., 2013, 14(1): 49–55.
[34] Qasim W, Zhan H, Samarasinghe S, Adams S, Amrolia P, Stafford S, Butler K, Rivat C, Wright G, Somana K, Ghorashian S, Pinner D, Ahsan G, Gilmour K, Lucchini G, Inglott S, Mifsud W, Chiesa R, Peggs KS, Chan L, Farzeneh F, Thrasher AJ, Vora A, Pule M, Veys P. Molecular remission of infant B-ALL after infusion of universal TALEN gene-edited CAR T cells., 2017, 9(374): eaaj2013.
[35] Ishino Y, Shinagawa H, Makino K, Amemura M, Nakata A. Nucleotide sequence of the iap gene, responsible for alkaline phosphatase isozyme conversion in, and identification of the gene product., 1987, 169(12): 5429–5433.
[36] Mojica FJ, Díez-Villaseñor C, Soria E, Juez G. Biological significance of a family of regularly spaced repeats in the genomes of Archaea, Bacteria and mitochondria., 2000, 36(1): 244–246.
[37] Jansen R, van Embden JDA, Gaastra W, Schouls LM. Identification of genes that are associated with DNA repeats in prokaryotes., 2002, 43(6): 1565–1575.
[38] Wang JY, Doudna JA. CRISPR technology: a decade of genome editing is only the beginning., 2023, 379(6629): eadd8643.
[39] Mojica FJM, Díez-Villaseñor C, García-Martínez J, Soria E. Intervening sequences of regularly spaced prokaryotic repeats derive from foreign genetic elements., 2005, 60(2): 174–182.
[40] Pourcel C, Salvignol G, Vergnaud G. CRISPR elements inacquire new repeats by preferential uptake of bacteriophage DNA, and provide additional tools for evolutionary studies., 2005, 151(Pt 3): 653–663.
[41] Bolotin A, Quinquis B, Sorokin A, Ehrlich SD. Clustered regularly interspaced short palindrome repeats (CRISPRs) have spacers of extrachromosomal origin., 2005, 151(Pt 8): 2551–2561.
[42] Makarova KS, Grishin NV, Shabalina SA, Wolf YI, Koonin EV. A putative RNA-interference-based immune system in prokaryotes: computational analysis of the predicted enzymatic machinery, functional analogies with eukaryotic RNAi, and hypothetical mechanisms of action., 2006, 1: 7.
[43] Makarova KS, Wolf YI, Iranzo J, Shmakov SA, Alkhnbashi OS, Brouns SJJ, Charpentier E, Cheng D, Haft DH, Horvath P, Moineau S, Mojica FJM, Scott D, Shah SA, Siksnys V, Terns MP, Venclovas Č, White MF, Yakunin AF, Yan W, Zhang F, Garrett RA, Backofen R, van der Oost J, Barrangou R, Koonin EV. Evolutionary classification of CRISPR-Cas systems: a burst of class 2 and derived variants., 2020, 18(2): 67–83.
[44] Jinek M, Chylinski K, Fonfara I, Hauer M, Doudna JA, Charpentier E. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity., 2012, 337(6096): 816–821.
[45] Doudna JA, Charpentier E. The new frontier of genome engineering with CRISPR-Cas9., 2014, 346(6213): 1258096.
[46] Zetsche B, Gootenberg JS, Abudayyeh OO, Slaymaker IM, Makarova KS, Essletzbichler P, Volz SE, Joung J, van der Oost J, Regev A, Koonin EV, Zhang F. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system., 2015, 163(3): 759–771.
[47] Abudayyeh OO, Gootenberg JS, Konermann S, Joung J, Slaymaker IM, Cox DBT, Shmakov S, Makarova KS, Semenova E, Minakhin L, Severinov K, Regev A, Lander ES, Koonin EV, Zhang F. C2c2 is a single-component programmable RNA-guided RNA-targeting CRISPR effector., 2016, 353(6299): aaf5573.
[48] Chen JS, Ma EB, Harrington LB, Da Costa M, Tian XR, Palefsky JM, Doudna JA. CRISPR-Cas12a target binding unleashes indiscriminate single-stranded DNase activity., 2018, 360(6387): 436–439.
[49] Broughton JP, Deng XD, Yu GX, Fasching CL, Servellita V, Singh J, Miao X, Streithorst JA, Granados A, Sotomayor-Gonzalez A, Zorn K, Gopez A, Hsu E, Gu W, Miller S, Pan CY, Guevara H, Wadford DA, Chen JS, Chiu CY. CRISPR-Cas12-based detection of SARS-CoV-2., 2020, 38(7): 870–874.
[50] Nguyen LT, Smith BM, Jain PK. Enhancement of trans-cleavage activity of Cas12a with engineered crRNA enables amplified nucleic acid detection., 2020, 11(1): 4906.
[51] Gootenberg JS, Abudayyeh OO, Lee JW, Essletzbichler P, Dy AJ, Joung J, Verdine V, Donghia N, Daringer NM, Freije CA, Myhrvold C, Bhattacharyya RP, Livny J, Regev A, Koonin EV, Hung DT, Sabeti PC, Collins JJ, Zhang F. Nucleic acid detection with CRISPR-Cas13a/C2c2., 2017, 356(6336): 438–442.
[52] Gootenberg JS, Abudayyeh OO, Kellner MJ, Joung J, Collins JJ, Zhang F. Multiplexed and portable nucleic acid detection platform with Cas13, Cas12a, and Csm6., 2018, 360(6387): 439–444.
[53] Iwasaki RS, Batey RT. SPRINT: a Cas13a-based platform for detection of small molecules., 2020, 48(17): e101.
[54] Gasiunas G, Barrangou R, Horvath P, Siksnys V. Cas9-crRNA ribonucleoprotein complex mediates specific DNA cleavage for adaptive immunity in bacteria., 2012, 109(39): E2579–E2586.
[55] Rees HA, Liu DR. Base editing: precision chemistry on the genome and transcriptome of living cells., 2018, 19(12): 770–788.
[56] Konermann S, Brigham MD, Trevino AE, Joung J, Abudayyeh OO, Barcena C, Hsu PD, Habib N, Gootenberg JS, Nishimasu H, Nureki O, Zhang F. Genome-scale transcriptional activation by an engineered CRISPR- Cas9 complex., 2015, 517(7536): 583–588.
[57] Gilbert LA, Larson MH, Morsut L, Liu ZR, Brar GA, Torres SE, Stern-Ginossar N, Brandman O, Whitehead EH, Doudna JA, Lim WA, Weissman JS, Qi LS. CRISPR- mediated modular RNA-guided regulation of transcription in eukaryotes., 2013, 154(2): 442–451.
[58] Hilton IB, D'Ippolito AM, Vockley CM, Thakore PI, Crawford GE, Reddy TE, Gersbach CA. Epigenome editing by a CRISPR-Cas9-based acetyltransferase activates genes from promoters and enhancers., 2015, 33(5): 510–517.
[59] Zhang HX, Zhang Y, Yin H. Genome editing with mRNA encoding ZFN, TALEN, and Cas9., 2019, 27(4): 735–746.
[60] Ren YX, Xiao RD, Lou XM, Fang XD. Research advance and application in the gene therapy of gene editing technologies., 2019, 41(1): 18–27.任云晓, 肖茹丹, 娄晓敏, 方向东. 基因编辑技术及其在基因治疗中的应用. 遗传, 2019, 41(1): 18–27.
[61] McCarty NS, Graham AE, Studena L, Ledesma-Amaro R. Multiplexed CRISPR technologies for gene editing and transcriptional regulation., 2020, 11(1): 1281.
[62] Ye L, Wang JM, Beyer AI, Teque F, Cradick TJ, Qi ZX, Chang JC, Bao G, Muench MO, Yu JW, Levy JA, Kan YW. Seamless modification of wild-type induced pluripotent stem cells to the natural CCR5Δ32 mutation confers resistance to HIV infection., 2014, 111(26): 9591–9596.
[63] Yusa K, Zhou LQ, Li MA, Bradley A, Craig NL. A hyperactive piggyBac transposase for mammalian applications., 2011, 108(4): 1531–1536.
[64] Zhou J, Li C, Liu XJ, Chiu MC, Zhao XY, Wang D, Wei YX, Lee A, Zhang AJ, Chu H, Cai JP, Yip CCY, Chan IHY, Wong KKY, Tsang OTY, Chan KH, Chan JFW, To KKW, Chen HL, Yuen KY. Infection of bat and human intestinal organoids by SARS-CoV-2., 2020, 26(7): 1077– 1083.
[65] Lamers MM, Haagmans BL. SARS-CoV-2 pathogenesis., 2022, 20(5): 270–284.
[66] Sun SH, Chen Q, Gu HJ, Yang G, Wang YX, Huang XY, Liu SS, Zhang NN, Li XF, Xiong R, Guo Y, Deng YQ, Huang WJ, Liu Q, Liu QM, Shen YL, Zhou Y, Yang X, Zhao TY, Fan CF, Zhou YS, Qin CF, Wang YC. A mouse model of SARS-CoV-2 infection and pathogenesis., 2020, 28(1): 124–133.e4.
[67] Li B, Clohisey SM, Chia BS, Wang B, Cui A, Eisenhaure T, Schweitzer LD, Hoover P, Parkinson NJ, Nachshon A, Smith N, Regan T, Farr D, Gutmann MU, Bukhari SI, Law A, Sangesland M, Gat-Viks I, Digard P, Vasudevan S, Lingwood D, Dockrell DH, Doench JG, Baillie JK, Hacohen N. Genome-wide CRISPR screen identifies host dependency factors for influenza A virus infection., 2020, 11(1): 164.
[68] Hyrina A, Jones C, Chen D, Clarkson S, Cochran N, Feucht P, Hoffman G, Lindeman A, Russ C, Sigoillot F, Tsang T, Uehara K, Xie LL, Ganem D, Holdorf M. A genome-wide CRISPR screen identifies ZCCHC14 as a host factor required for hepatitis B surface antigen production., 2019, 29(10): 2970–2978.e6.
[69] Mei H, Zha Z, Wang W, Xie YS, Huang YG, Li WP, Wei D, Zhang XX, Qu JM, Liu J. Surfaceome CRISPR screen identifies OLFML3 as a rhinovirus-inducible IFN antagonist., 2021, 22(1): 297.
[70] Wei J, Alfajaro MM, DeWeirdt PC, Hanna RE, Lu-Culligan WJ, Cai WL, Strine MS, Zhang SM, Graziano VR, Schmitz CO, Chen JS, Mankowski MC, Filler RB, Ravindra NG, Gasque V, de Miguel FJ, Patil A, Chen HC, Oguntuyo KY, Abriola L, Surovtseva YV, Orchard RC, Lee B, Lindenbach BD, Politi K, van Dijk D, Kadoch C, Simon MD, Yan Q, Doench JG, Wilen CB. Genome-wide CRISPR screens reveal host factors critical for SARS-CoV-2 infection., 2021, 184(1): 76–91.e13.
[71] Baggen J, Persoons L, Vanstreels E, Jansen S, Van Looveren D, Boeckx B, Geudens V, De Man J, Jochmans D, Wauters J, Wauters E, Vanaudenaerde BM, Lambrechts D, Neyts J, Dallmeier K, Thibaut HJ, Jacquemyn M, Maes P, Daelemans D. Genome-wide CRISPR screening identifies TMEM106B as a proviral host factor for SARS-CoV-2., 2021, 53(4): 435–444.
[72] Biering SB, Sarnik SA, Wang E, Zengel JR, Leist SR, Schäfer A, Sathyan V, Hawkins P, Okuda K, Tau C, Jangid AR, Duffy CV, Wei J, Gilmore RC, Alfajaro MM, Strine MS, Nguyenla X, Van Dis E, Catamura C, Yamashiro LH, Belk JA, Begeman A, Stark JC, Shon DJ, Fox DM, Ezzatpour S, Huang E, Olegario N, Rustagi A, Volmer AS, Livraghi-Butrico A, Wehri E, Behringer RR, Cheon DJ, Schaletzky J, Aguilar HC, Puschnik AS, Button B, Pinsky BA, Blish CA, Baric RS, O’Neal WK, Bertozzi CR, Wilen CB, Boucher RC, Carette JE, Stanley SA, Harris E, Konermann S, Hsu PD. Genome-wide bidirectional CRISPR screens identify mucins as host factors modulating SARS-CoV-2 infection., 2022, 54(8): 1078–1089.
[73] Zhu SY, Liu Y, Zhou Z, Zhang ZY, Xiao X, Liu ZH, Chen A, Dong XJ, Tian F, Chen SH, Xu YY, Wang CH, Li QH, Niu XR, Pan Q, Du S, Xiao JY, Wang JW, Wei WS. Genome-wide CRISPR activation screen identifies candidate receptors for SARS-CoV-2 entry., 2022, 65(4): 701–717.
[74] Lai Y, Cui L, Babunovic GH, Fortune SM, Doench JG, Lu TK. High-throughput CRISPR screens to dissect macrophage-interactions., 2021, 12(6): e0215821.
[75] Bosch B, DeJesus MA, Poulton NC, Zhang WZ, Engelhart CA, Zaveri A, Lavalette S, Ruecker N, Trujillo C, Wallach JB, Li SQ, Ehrt S, Chait BT, Schnappinger D, Rock JM. Genome-wide gene expression tuning reveals diverse vulnerabilities of.., 2021, 184(17): 4579–4592.e24.
[76] Rousset F, Bikard D. CRISPR screens in the era of microbiomes., 2020, 57: 70–77.
[77] Yan MY, Zheng DD, Li SS, Ding XY, Wang CL, Guo XP, Zhan LJ, Jin Q, Yang J, Sun YC. Application of combined CRISPR screening for genetic and chemical-genetic interaction profiling in., 2022, 8(47): eadd5907.
[78] Yan MY, Li SS, Ding XY, Guo XP, Jin Q, Sun YC. A CRISPR-assisted nonhomologous end-joining strategy for efficient genome editing in., 2020, 11(1): e02364–19.
[79] Myhrvold C, Freije CA, Gootenberg JS, Abudayyeh OO, Metsky HC, Durbin AF, Kellner MJ, Tan AL, Paul LM, Parham LA, Garcia KF, Barnes KG, Chak B, Mondini A, Nogueira ML, Isern S, Michael SF, Lorenzana I, Yozwiak NL, MacInnis BL, Bosch I, Gehrke L, Zhang F, Sabeti PC. Field-deployable viral diagnostics using CRISPR-Cas13., 2018, 360(6387): 444–448.
[80] An JH, Liao XJ, Xiao TY, Qian S, Yuan J, Ye HC, Qi FR, Shen CG, Wang LF, Liu Y, Cheng XY, Li N, Cai QX, Wang F, Chen J, Li GJ, Cai QE, Liu YX, Wang YF, Zhang F, Fu Y, He Q, Tan XH, Liu L, Zhang Z. Clinical characteristics of recovered COVID-19 patients with re-detectable positive RNA test., 2020, 8(17): 1084.
[81] Joung J, Ladha A, Saito M, Segel M, Bruneau R, Huang MLW, Kim NG, Yu X, Li J, Walker BD, Greninger AL, Jerome KR, Gootenberg JS, Abudayyeh OO, Zhang F. Point-of-care testing for COVID-19 using SHERLOCK diagnostics., 2020, doi: 10.1101/2020.05.04. 20091231.
[82] Joung J, Ladha A, Saito M, Kim NG, Woolley AE, Segel M, Barretto RPJ, Ranu A, Macrae RK, Faure G, Ioannidi EI, Krajeski RN, Bruneau R, Huang MLW, Yu XG, Li JZ, Walker BD, Hung DT, Greninger AL, Jerome KR, Gootenberg JS, Abudayyeh OO, Zhang F. Detection of SARS-CoV-2 with SHERLOCK one-pot testing., 2020, 383(15): 1492–1494.
[83] Ranson H, Lissenden N. Insecticide resistance in Africanmosquitoes: a worsening situation that needs urgent action to maintain malaria control., 2016, 32(3): 187–196.
[84] Moyes CL, Vontas J, Martins AJ, Ng LC, Koou SY, Dusfour I, Raghavendra K, Pinto J, Corbel V, David JP, Weetman D. Contemporary status of insecticide resistance in the majorvectors of arboviruses infecting humans., 2017, 11(7): e0005625.
[85] Gantz VM, Jasinskiene N, Tatarenkova O, Fazekas A, Macias VM, Bier E, James AA. Highly efficient Cas9-mediated gene drive for population modification of the malaria vector mosquito., 2015, 112(49): E6736–E6743.
[86] Kyrou K, Hammond AM, Galizi R, Kranjc N, Burt A, Beaghton AK, Nolan T, Crisanti A. A CRISPR-Cas9 gene drive targeting doublesex causes complete population suppression in cagedmosquitoes., 2018, 36(11): 1062–1066.
[87] Chen JY, Luo JJ, Wang YJ, Gurav AS, Li M, Akbari OS, Montell C. Suppression of female fertility inwith a CRISPR-targeted male-sterile mutation., 2021, 118(22): e2105075118.
[88] Xu L, Wang J, Liu YL, Xie LF, Su B, Mou DL, Wang LT, Liu TT, Wang XB, Zhang B, Zhao L, Hu LD, Ning HM, Zhang YF, Deng K, Liu LF, Lu XF, Zhang T, Xu J, Li C, Wu H, Deng HK, Chen H. CRISPR-edited stem cells in a patient with HIV and acute lymphocytic leukemia., 2019, 381(13): 1240–1247.
[89] Dash PK, Chen C, Kaminski R, Su H, Mancuso P, Sillman B, Zhang C, Liao SR, Sravanam S, Liu H, Waight E, Guo LL, Mathews S, Sariyer R, Mosley RL, Poluektova LY, Caocci M, Amini S, Gorantla S, Burdo TH, Edagwa B, Gendelman HE, Khalili K. CRISPR editing of CCR5 and HIV-1 facilitates viral elimination in antiretroviral drug-suppressed virus-infected humanized mice., 2023, 120(19): e2217887120.
[90] Kennedy EM, Bassit LC, Mueller H, Kornepati AVR, Bogerd HP, Nie T, Chatterjee P, Javanbakht H, Schinazi RF, Cullen BR. Suppression of hepatitis B virus DNA accumulation in chronically infected cells using a bacterial CRISPR/Cas RNA-guided DNA endonuclease., 2015, 476: 196–205.
[91] Ramanan V, Shlomai A, Cox DBT, Schwartz RE, Michailidis E, Bhatta A, Scott DA, Zhang F, Rice CM, Bhatia SN. CRISPR/Cas9 cleavage of viral DNA efficiently suppresses hepatitis B virus., 2015, 5: 10833.
[92] Li H, Sheng CY, Wang S, Yang L, Liang Y, Huang Y, Liu HB, Li P, Yang CJ, Yang XX, Jia LL, Xie J, Wang LG, Hao RZ, Du XY, Xu DP, Zhou JJ, Li MZ, Sun YS, Tong YG, Li Q, Qiu SF, Song HB. Removal of integrated hepatitis B virus DNA using CRISPR-Cas9., 2017, 7: 91.
[93] Russell TA, Stefanovic T, Tscharke DC. Engineering herpes simplex viruses by infection-transfection methods including recombination site targeting by CRISPR/Cas9 nucleases., 2015, 213: 18–25.
[94] Lin CL, Li HH, Hao MR, Xiong D, Luo Y, Huang CH, Yuan Q, Zhang J, Xia NS. Increasing the efficiency of CRISPR/Cas9-mediated precise genome editing of HSV-1 virus in human cells., 2016, 6: 34531.
[95] Freije CA, Myhrvold C, Boehm CK, Lin AE, Welch NL, Carter A, Metsky HC, Luo CY, Abudayyeh OO, Gootenberg JS, Yozwiak NL, Zhang F, Sabeti PC. Programmable inhibition and detection of RNA viruses using Cas13., 2019, 76(5): 826–837.e11.
[96] Wilbie D, Walther J, Mastrobattista E. Delivery aspects of CRISPR/Cas for in vivo genome editing., 2019, 52(6): 1555–1564.
[97] Niu XR, Yin SM, Chen X, Shao TT, Li DL. Gene editing technology and its recent progress in disease therapy., 2019, 41(7): 582–598.牛煦然, 尹树明, 陈曦, 邵婷婷, 李大力. 基因编辑技术及其在疾病治疗中的研究进展. 遗传, 2019, 41(7): 582–598.
The application of CRISPR genome editing technologies in the pathogenesis studies, diagnosis, prevention and treatment of infectious diseases
Zhenrong Yang1,2, Gangqiao Zhou1,2
The CRISPR genome editing technology shows great application prospects in gene manipulation and infectious disease research, and is of great value for effective control and cure of infectious diseases. It has been utilized to generate specific disease models in cells, organoids and animals, which provide great convenience for research into the molecular mechanisms associated with infectious diseases. CRISPR screening technology enables high-throughput identification of risk factors. New molecular diagnostic tools based on CRISPR offer a more sensitive and faster method for detecting pathogens. The use of CRISPR tools to introduce resistance genes or to specifically destroy risk genes and virus genomes is intended to help prevent or treat infectious diseases. This review discusses the application of CRISPR genome editing technologies in the construction of disease models, screening of risk factors, pathogen diagnosis, and prevention and treatment of infectious diseases, thereby providing a reference for follow-up research in pathogenesis, diagnosis, prevention and treatment of infectious diseases.
infectious diseases; CRISPR; pathogenesis; diagnosis; prevention and treatment
2023-07-31;
2023-10-06;
2023-10-24
广州实验室应急攻关项目(编号:EKPG21-19)资助[Supported by the Emergency Key Program of Guangzhou Laboratory (No. EKPG21-19)]
杨臻嵘,博士研究生,专业方向:细胞生物学。E-mail: yangzr@mail.ustc.edu.cn
周钢桥,博士,研究员,研究方向:医学遗传学与基因组学。E-mail: zhougq114@126.com
10.16288/j.yczz.23-206
(责任编委: 谢建平)