PIWI interacting RNA通路相关基因作为肠癌生物标志物的研究*
张 伟, 王 莹, 温珍平
(1.北京大学肿瘤医院内蒙古医院,内蒙古 呼和浩特 010020;2.赤峰学院附属医院;3.响水县人民医院)
癌症已是仅次于缺血性心脏病的全球第二大死亡原因,很可能在2060年成为人类第一大死亡原因[1]。结直肠癌(CRC)是临床中常见的消化道恶性肿瘤,发病率呈上升趋势,患者初诊时多处于进展期。因此,迫切需要寻找新的诊断和治疗靶点来降低CRC的死亡率。piRNA于2006年在哺乳动物系统中被发现并正式定义为与PIWI特异性相互作用的小型非编码RNA,其长度为26~31 nt,存在于生殖细胞和体细胞中且具有重要功能,例如抑制转座因子和基因表达的表观遗传调控,与Argonaute蛋白的PIWI亚家族特异性结合形成piRNA沉默复合物(piRNA induced silencing complex,piRISC)[2]。piRISC可参与调控靶序列,在转录或转录后降解并通过调节转座子的活性来控制其他基因的表达[3]。随着新一代测序技术的发展,piRNA被发现在CRC中发挥着重要作用。2018年欧洲学者Vychytilova-Faltejskova等[4]研究指出:与健康人群相比,在癌症患者的血清中piRNA显著降低。根据PIR-5937和PIR-28876的水平,可以高度敏感和特异地区分癌症患者和健康人群。此外,这两种piRNA在Ⅰ期疾病患者中具有不错的诊断性能,并且比结肠癌目前使用的生物标记物CEA和CA19-9表现出更高的灵敏度[4]。也有研究表明:结直肠癌患者血清中的Pir-020619和Pir-020450会持续升高,但二者在肺癌、乳腺癌和胃癌患者血清的表达无明显变化而且它们联合应用具有显著的诊断价值,敏感性达72.50 %。因此,这两种piRNA可能作为结直肠癌特异性循环肿瘤标志物,适用于癌前病变的早期筛查,有助于降低结直肠癌的发病率和死亡率[5]。然而,piRNA通路相关基因在CRC中的功能仍不清楚。本研究通过构建piRNA通路相关基因风险预后模型,探索piRNA通路相关基因在CRC肿瘤中的诊断和预后价值。
1 材料与方法
1.1临床数据获取 TCGA(https://genomecancer.ucsc.edu/)是人类基因组计划的大型免费肿瘤数据库,包含癌症患者的RNA序列和临床和病理信息。通过R下载结直肠肿瘤患者的RNAseq数据和临床信息,最终将647例CRC肿瘤样本和51例正常肠样本用于后续分析。GEO(http://www.ncbi.nlm.nih.gov/geo)是一个存储遗传数据的开放平台[6]。从GEO获得三个数据集GSE211496(GPL2857)、GSE74602(GPL6104)和GSE89076(GPL16699)。GEO2R被用于识别GEO中的差异基因(DEG),在此处用于识别CRC和健康的肠组织样本之间的差异基因[7]。对P值(adj.P)进行调整以控制错误发现率并保持假阳性可能性与重要基因检测之间的平衡,倍数变化(FC)阈值设置为≥2和P≤0.01认为差异具有统计学意义。
1.2piRNA通路相关基因的获取 PathCards数据库(https://pathcards.genecards.org/)是人类生物通路及其注释的集成数据库[8]。在PathCards数据库中可检索到唯一的piRNA通路集,并从中可获取piRNA通路中的基因。GEPIA2数据库能够对相似基因检测、查询肿瘤差异表达谱或病理分期等分析[9]。利用GEPIA2的“相似基因检测”模板,通过计算P值和关联系数R获得了与piRNA通路蛋白相关的基因。最后将获得的piRNA通路相关基因与GSE56355、GSE74602、GSE89076三者的DEG取交集,筛选得到piRNA通路相关的基因。
1.3预后模型构建和验证 LASSO(least absolute shrinkage and selection operator)方法是一种压缩估计。它通过构造一个函数得到一个较为精炼的模型,设定一些系数为零,使得它可压缩一些系数[10]。采用glmnet包对所有基因进行LASSO Cox回归降维分析,通过计算每个患者的风险评分可以得到组成最优模型的基因组。风险评分由相对基因表达水平乘以回归系数的线性组合计算得出。回归系数通过多次Cox分析获得,代表基因的相对权重。对数秩检验用于生存分析比较上述两组或多组之间的生存差异,进行了timeROC分析判别预测模型的准确性。
1.4网络数据库 STRING数据库用于分析所发现的模型基因之间相互作用的分子过程[11]。目标基因是PPI网络中的节点,连接它们的线表示各自之间的相互作用,连接的强度由线的数量表示。利用cytoHubba和MCODE在Cytoscape软件(http://cytoscape.org/)中建立的截止度标准,确定了中枢基因,并将其划分为在网络中起关键作用的基因。使用Cytoscape软件对相关的相互作用进行可视化。TIMER2数据库(http://timer.cistrome.org/)是一个进行肿瘤免疫评估的综合性网站,用于自动分析和可视化免疫入侵水平和一系列变量之间的关联[12]。因此,通过使用TIMER2“Immunogene”模块检测POLR2F 和 PAICS的表达与免疫浸润的关系。此外,通过Human Protein Atlas(www.proteinatlas.org)检测了POLR2F及PAICS蛋白在肠癌组织和正常肠组织中的表达
1.5KM、ROC曲线及临床样本资料分析 利用TCGA数据进行Kaplan-Meier(KM)生存分析和单因素/多因素cox比例风险回归分析。通过survival包分析去除了对照/正常组的TCGA数据,survminer包用于可视化,实施 OS 生存分析并绘制KM曲线,借以估计 在风险评估模型中piRNA 通路相关基因的表达对预后的影响。采用卡方检验评估差异表达与患者临床特征的关系。此外,通过pROC包用于分析处理TCGA有临床信息的数据并去除重复样本,ggplot2包用于可视化,绘制了ROC曲线来评估诊断意义。
1.6细胞培养 CRC细胞系(SW480,HCT116)和正常肠上皮细胞系(NCM460)在10 %胎牛血清(北京全式金生物公司)的DMEM高糖(赛默飞世尔科技有限公司)培养基中37 ℃和 5 % CO2培养;使用酶消法分离纯化获得正常原代肠上皮成纤维细胞(NF)、原代肠癌相关成纤维细胞(CAF)和原代正常肠上皮细胞(N)。CAF和NF在10 %胎牛血清(北京全式金生物公司)的F-12(赛默飞世尔科技有限公司)培养基中37 ℃和5 % CO2培养。原代肠上皮细胞N在10 %血清的DMEM高糖培养基中37 ℃和5 % CO2培养;所有细胞每2~3 d进行一次传代,所有原代细胞均经免疫荧光法鉴定细胞特异性蛋白进行验证。
1.7RNA提取和定量实时PCR 用TRIzol试剂(赛默飞世尔科技有限公司)提取总RNA。使用PrimeScriptTMRT试剂盒(北京全式金生物公司)逆转录cDNA。qPCR使用SYBR Green Real-time PCR Master Mix(北京全式金生物公司)进行。POLR2F、PAICS和U6的引物序列如下(5′-3′):POLR2F正向GCTCCAGATTGCGATGTGTG和反向GGATCTTTCGGGCCTTGAGTT;PAICS正向GCAGGGTTGTAGTGTTGATGG和反向GCCACTGCCACAAATACAGTAG;U6正向CTCGCTTCGGCAGCACA和反向AACGCTTCACGAATTTGCGT。
1.8统计分析 以上所有TCGA数据处理均使用v4.0.3版R软件(R 2020)执行。使用SPSS软件(22.0版;IBM,Armonk,NY,USA)分析所有实验数据。首先,检验方差的正态性和同质性,当满足正态性和方差齐性时,多组数据比较采用ANOVA,两组比较采用Student's-test。P<0.05为差异具有统计学意义。
2 结果
2.1CRC中差异表达的piRNA通路相关基因的鉴定 使用PathCards数据库得到了29个piRNA通路基因,结合GEPIA2和GEO数据库得到了10个piRNA通路相关基因,在包含来自TCGA的647个CRC和51个正常肠组织标本中发现这39个piRNA通路相关基因中36个有差异表达(图1),横坐标为基因名称,纵坐标为基因的表达水平;蓝色代表正常肠组织,红色代表肠癌组织;P值在最上方标注。这其中包括27个上调(HSP90AA1、TDRKH、HENMT1、ASZ1、PIWIL1、PIWIL2、POLR2A、POLR2C、POLR2D、POLR2E、POLR2F、POLR2G、POLR2H、POLR2I、POLR2J、POLR2K、POLR2L、CDCA5、MCM7、MTHFD1L、MCM2、CHEK1、CSE1L、DKC1、PUS7、PPAT和PAICS)和9个下调(MOV10L1、DDX4、MAEL、MYBL1、PLD6、TDRD1、TDRD6、TDRD9和TDRD12)piRNA通路相关基因。

图1 piRNA通路相关基因的表达
2.2piRNA通路相关基因预后模型的构建 本研究建立了预后风险模型,通过LASSO Cox回归分析识别了8个 piRNA 通路相关基因[图2(A)]。风险评分如下所示(图2B):LASSO Cox回归,选定特征的系数由lambda参数显示,横坐标代表自变量 lambda 的值,纵坐标表示自变量的系数;使用LASSO Cox回归模型绘制了部分似然偏差与log(λ)的关系。由此将这8个基因作为预后模型的整体并进行下一步评估。风险评分建模公式为:
Riskscore=(-0.0761)×CHEK1+(-0.1336)×PAICS+(-0.0047)×CSE1L+(-0.175)×HENMT1+(-0.2514)×TDRD6+(-0.5797)×POLR2F+(0.3863)×POLR2J+(0.5152)×POLR2H
2.3piRNA 通路相关基因预后风险模型的评估 基于中位风险评分,所有患者在图3A中被分为高、低风险组,图中蓝色为低风险,红色为高风险; 图曲线由患者生存风险从低到高的散点图构成;它们代表了患者生存时间、存活状态等情况;B图为风险模型在数据集中的KM生存曲线分布,其中不同组间通过log rank进行检验,HR(High risk)代表高风险组相对于低风险组样本的风险系数,若HR>1代表该模型为危险模型,若HR <1则代表该模型为保护模型;Medain time代表高危组和低危组两组生存率在50%时对应的时间(即中位生存时间)。构建的KM曲线显示:与CRC低危组相比,CRC高危组Overall Survival (OS) 明显降低(HR=1.799,P=0.0013)。图C中不同样本Riskscore对应的生存时间和生存状态散点分布,点图可显示单个 CRC 患者的 OS 状态,通过对患者的 OS 风险评分进行排名来分析它们的分布;E图为该模型中包含基因的表达热图,热图显示了训练组中风险基因的表达水平。最后ROC 曲线进一步验证了预后风险模型的性能(图3D),该曲线分别显示1年0.624、3年0.669和5年0.670的曲线下面积(AUC 值)。其中AUC值越高,说明该模型预测能力越强,图中AUC值的结果表明基于 piRNA 通路相关基因特征的预后预测模型呈中等性能。通过以上对预后模型的评估证明了基于 piRNA 通路相关基因的预后预测模型的稳定性。
2.4piRNA通路相关基因的生存和ROC曲线分析 图4的KM曲线中评估患者总生存数,纵坐标为生存率,横坐标为生存时间;蓝色代表基因的低表达组,红色代表高表达组;实线代表生存曲线,双虚线间代表95 %置信区间。风险评估模型中8个 piRNA 通路相关基因的表达与预后生存的关系被阐述:CHEK1(P=0.001)、CSE1L(P=0.033)、HENMT1(P=0.005)和 PAICS(P=0.001)的高表达可预测较好的 生存性;POLR2F(P=0.001)的高表达的肠癌患者有较低的生存概率 ;然而,TDRD6(P=0.317), POLR2H(P=0.083)和POLR2J(P=0.291)的表达与OS没有显着相关性。在图5的ROC 曲线中估计8个 piRNA 通路相关基因在 CRC 中的诊断作用,横坐标 X轴为1-特异性,也称为假阳性率(误报率),X轴越接近零准确率越高;纵坐标Y轴称为敏感度,也称为真阳性率(敏感度),Y轴越大代表准确率越好。根据曲线位置,整个图被划分为两部分,曲线下方部分的面积被称为AUC(Area Under Curve),用来表示预测准确性,AUC值越高,也就是曲线下方面积越大,说明预测准确率越高。曲线越接近左上角(X越小,Y越大),预测准确率越高 ,来自 TCGA 的72 对 CRC 组织和正常肠组织被用作来生成该曲线。结果表明,在预测癌和非癌的结局上,TDRD6(AUC=0.543)的预测能力有较低准确性;POLR2F (AUC=0.761)、HENMT1(AUC=0.872)、POLR2J(AUC=0.813) 有一定的诊断准确效果;CHEK1(AUC=0.927)、CSE1L(AUC=0.975)、POLR2H(AUC=0.939)和PAICS(AUC=0.950) 预测能力有较高准确性。总体而言,结果表明POLR2F、CHEK1、CSE1L、HENMT1和 PAICS这5个piRNA 通路相关基因的表达与 CRC 患者的预后和诊断显著相关,可用作 CRC 患者预后的生物标志物和 CRC 的诊断靶点。

图4 piRNA通路相关基因的KM曲线
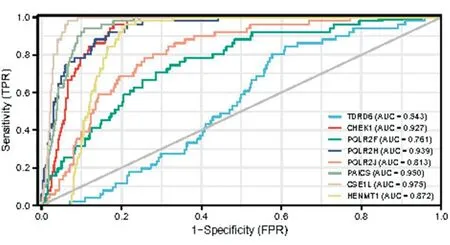
图5 候选基因的受试者工作曲线
2.5PPI网络图和枢纽基因 图6A是通过对筛选到的9个piRNA相关基因构建的PPI网络,代表着基因间的相互关系,每个圆代表不同的基因,两个圆间的线条数量越多代表基因间相互作用越强,结果表明POLR2J、POLR2H、POLR2F、PPAT、PAICS为最具相关性的基因。图6B是利用Cytoscape软件 中cytoHubba和Module 挑选到的3个枢纽(Hub)基因,颜色越深(红)代表基因间相互作用越强:POLR2J、POLR2F和PAICS,其中POLR2F和PAICS是枢纽基因中最具调控作用的两个基因。考虑基因间最密切的相互作用和之前的结果中发现POLR2F和PAICS都在肠癌中兼具诊断及预后价值,最后,将它们鉴定为piRNA相关基因中的关键基因,因此,选择 POLR2F 和 PAICS 进行进一步研究。
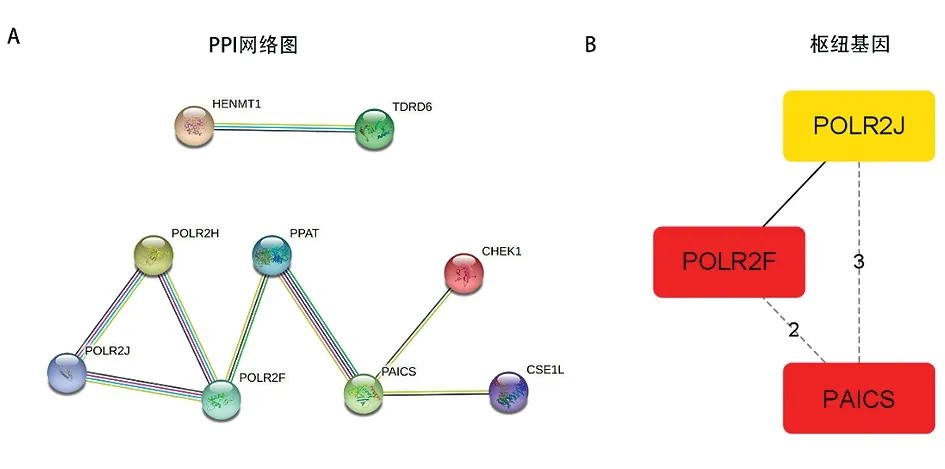
图6 PPI网路图及枢纽基因筛选
2.6CRC患者中POLR2F 和 PAICS的表达水平 在图1 中利用TCGA 数据集数据已初步评估了POLR2F和PAICS在肠癌组织中的表达水平,为进一步验证表达情况,在图7中探究了PAICS和POLR2F在细胞水平的表达,纵坐标为基因的表达水平,横坐标上的柱图为两基因在不同细胞间的表达,依次是原代肠上皮细胞(N)、正常肠上皮细胞系(NCM460)和肠癌细胞系(SW480、HCT16),图8中原代肠上皮细胞经免疫荧光鉴定PCK,E-Cadherin阳性表达,代表成功获取原代正常肠上皮细胞。PAICS组中,N组的平均水平为(1.686±1.497),NCM460组的平均水平为(5.285±5.239),SW480组的平均水平为(532.315±212.168),HCT116组的平均水平为(56.995±20.239);在POLR2F组中,N组的平均水平为(1.525±1.681),NCM460组的平均水平为(47.506±44.991),SW480组的平均水平为(596.505±210.898),HCT116组的平均水平为(324.61±241.864);在PAICS亚组中,纵向分组(group)之间观测变量的差异具有统计学意义(F=17.402,P=0.001);在POLR2F亚组中,纵向分组(group)之间观测变量的差异具有统计学意义(F=8.691,P=0.007)。结果显示POLR2F和PAICS表达水平在CRC组织中显著上调(P<0.001)。在RT-qPCR实验检测到与正常肠上皮细胞系NCM460和原代肠上皮细胞N相比,肠癌细胞系SW480都表现出POLR2F(P<0.05)和PAICS(P=0.001)高表达水平,但是在HCT116细胞中未发现具有统计学意义的差距。此外,使用来自HPA数据库中免疫组化(IHC)的结果,发现结直肠癌标本中POLR2F及PAICS蛋白的表达明显强于正常肠标本。图9分别为POLR2F和PAICS在正常肠组织及肠癌组织中蛋白表达水平IHC图,部分表达点使用蓝色箭头标出。结果进一步表明,POLR2F和PAICS在肠癌中为高度表达。

图7 PAICS和POLR2F在细胞水平表达
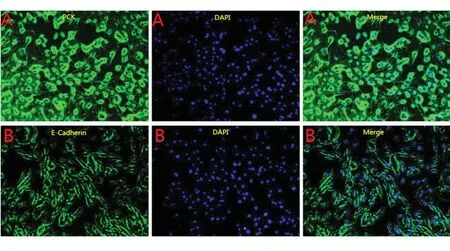
图8 原代正常肠上皮细胞鉴定
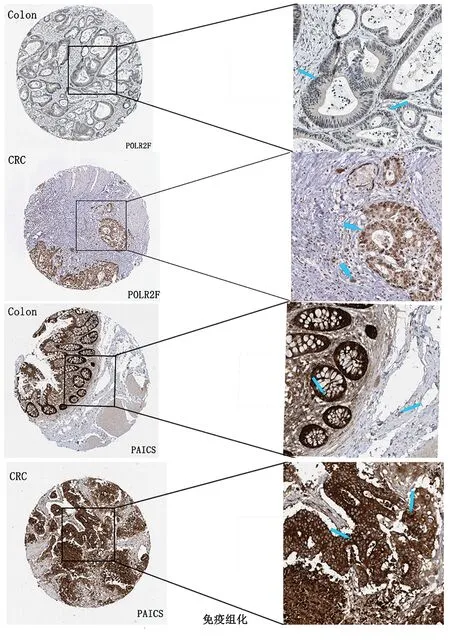
图9 肠癌中POLR2F和PAICS在蛋白水平表达的免疫组化
2.7与CRC中POLR2F和PAICS表达相关的临床特征 为了根据临床数据进一步确定POLR2F和PAICS的潜在作用,回顾了TCGA CRC患者的临床数据。临床病理参数包括患者的TNM分级、病理阶段、年龄、BMI、神经和淋巴浸润(表1)。POLR2F和PAICS与淋巴浸润水平均有显著相关性,此外,POLR2F的表达与年龄(P=0.044)和BMI(P=0.006)显著相关。PAICS表达与N分期(P=0.004)和神经浸润(P=0.017)显著相关。在CRC患者中,POLR2F和PAICS表达与T分期M分期及病理分类之间的差异也不显著。进一步研究了CRC患者POLR2F和PAICS蛋白表达水平与上述临床病理特征的相关性。结果表明,POLR2F和PAICS表达水平可能与肿瘤的淋巴浸润有关。
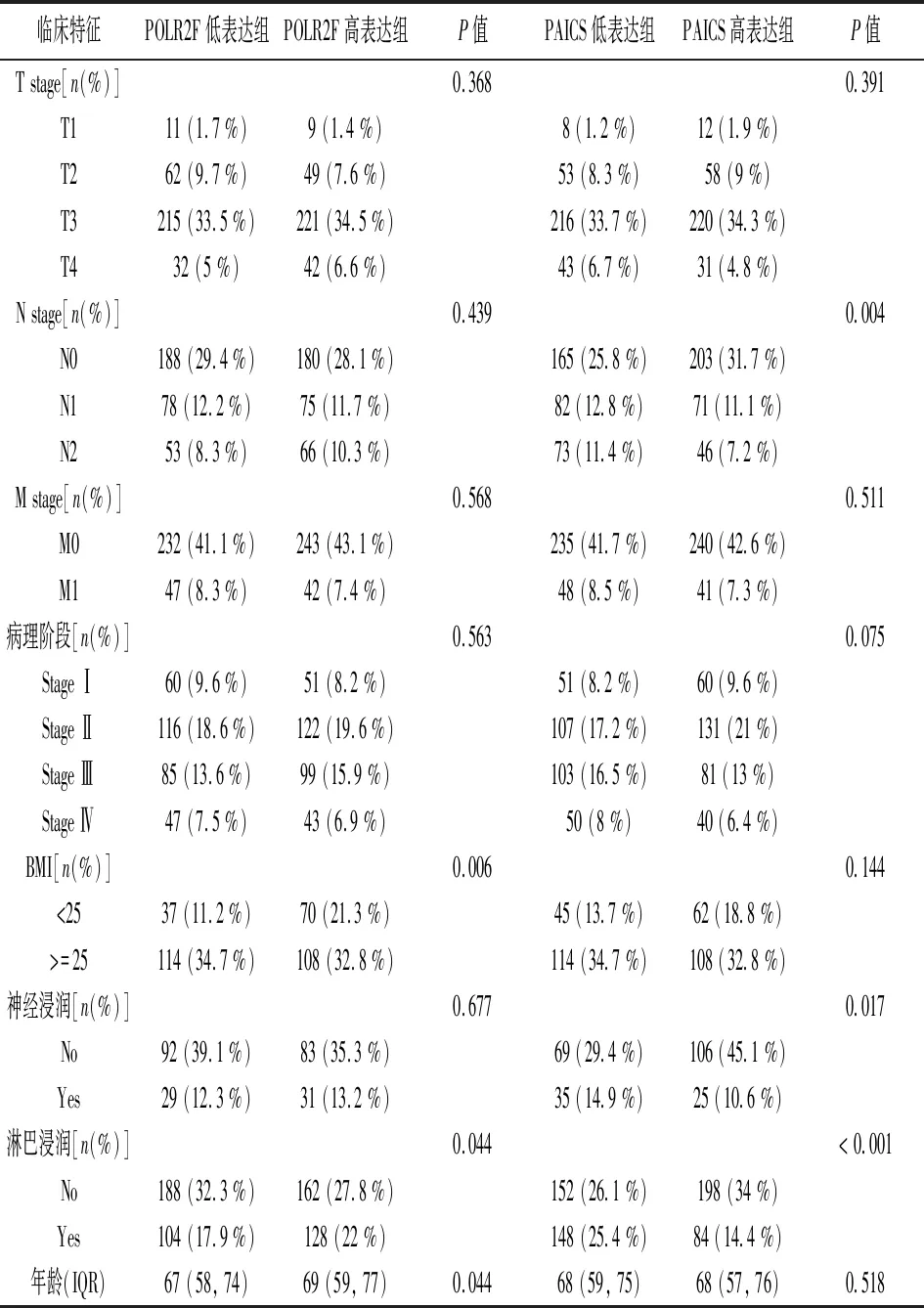
表1 POLR2F 和 PAICS 表达相关的临床特征
2.8POLR2F 和 PAICS 表达与肿瘤免疫相关性 POLR2F 和 PAICS表达均与淋巴浸润相关,这可能与肿瘤免疫及肿瘤微环境有关。但POLR2F与肿瘤免疫的相关性较弱,因此只研究了PAICS在肿瘤微环境中的潜在效能。在图10得到了CRC中PAICS表达与肿瘤浸润性免疫细胞(TIC) 、免疫检查点之间的关系热图。热图中纵坐标代表不同的免疫细胞,每个小图右上角的*号代表P值(*P<0.05,**P<0.01,***P<0.001)。结果表明: B细胞、 CD4T 细胞、NK细胞、CD4T 细胞、巨噬细胞, 肥大细胞、单核细胞、髓源性抑制细胞、辅助性T细胞和树突状细胞在结直肠癌中与PAICS的表达水平呈有显著相关。由于POLR2F没有被发现在结肠和直肠肿瘤中同时存在肿瘤浸润性免疫细胞,所以为进一步评估PAICS在肠癌免疫微环境中的作用,检测了它在原代肠癌相关成纤维细胞和原代正常肠成纤维细胞的表达,图11为基因PAICS在免疫微环境中成纤维细胞表达的箱式图,纵坐标为PAICS的表达水平,横坐标为CAF和NF两种细胞,结果显示PAICS在原代肠癌相关成纤维细胞中显著降低(P<0.001),表明其在肠癌免疫微环境中可能存在抑制肿瘤的重要作用。

图10 PAICS与结肠癌(COAD)和直肠癌(READ)免疫微环境中细胞关系热图
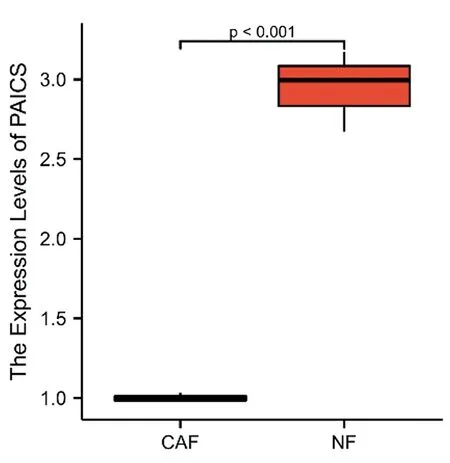
图11 PAICS在CAF/NF的表达
3 讨论
目前,随着分子诊断技术的进步,CRC的诊断率已大大提高。然而,由于缺乏早期临床症状和敏感的生物标志物,仍有大量患者未能获得最佳治疗。多数肠癌研究都集中在mRNA上,但非编码RNA也是肿瘤发生的重要参与者。除了公认的microRNA,piRNA也在人类癌细胞和体细胞中被检测到,并且有人认为piRNA比以前更广泛地控制基因表达[13]。piRNA作为种类最为丰富的小分子非编码RNA,它们的异常表达也是体外和体内癌细胞增殖、凋亡、侵袭和迁移的关键参与者[14]。然而,很少有研究关注piRNA通路相关基因的机制及其在CRC中的作用。因此,本文探索了哪些piRNA通路相关基因可用作CRC患者的新型有效预后生物标志物。在这项研究中,在利用生物信息学分析了CRC39个piRNA通路相关基因的表达,使用其中的8个基因建立了预后风险模型。最后选择了POLR2F和PAICS在细胞水平进行了进一步验证。
磷酸核糖氨基咪唑羧化酶和磷酸核糖氨基咪唑琥珀酰甲酰胺合成酶(PAICS)是催化嘌呤生物合成途径中的重要组成部分[15]。RNA聚合酶Ⅱ、Ⅰ和Ⅲ亚基F(POLR2F)是基因编码RNA聚合酶Ⅱ的第六大亚基,该聚合酶负责在真核生物中合成信使RNA[16]。PAICS和POLR2F在许多肿瘤中过度表达,如膀胱癌[17-18]、宫颈癌[19-20]等;在结肠癌中PAICS和POLR2F被认为与肿瘤预后相关,其中PAICS会参与肿瘤生长和结直肠癌的转移[21-22],且靶向PAICS对侵袭性结直肠癌具有治疗效果[15]。
通过分析发现与正常肠上皮细胞相比,POLR2F和PAICS在肠癌样本中高表达;在RT-qPCR实验进一步验证了POLR2F和PAICS在肠癌细胞系SW480中上调。此外,PAICS表达较高的CRC患者预后较好,而POLR2F表达较高的CRC患者预后较差,在探索CRC与肿瘤免疫浸润细胞相关性中发现:PAICS可能存在改善肿瘤微环境的作用,这一发现已初步在微环境中的癌相关成纤维细胞里得到验证。结合文献及实验结果,初步推断POLR2F和PAICS除了可作为预测因子和诊断标志物外,还可能参与了CRC肿瘤微环境的免疫反应,这表明piRNA通路相关基因在肠癌研究甚至肿瘤研究中具有相当意义。
这项研究也有一些局限性:只是通过数据库和部分实验初步预测了POLR2F和PAICS的作用,未在组织和机制水平探索,且尚缺乏有关PAICS的功能实验,仍需进一步研究评估piRNA及其相关基因在CRC中的表达水平和功能,为早期诊断和精准靶向治疗提供参考。
综上所述,本研究首先发现piRNA通路相关基因在CRC中具有不同的表达水平,然后确定了piRNA通路相关基因风险模型可能是CRC的独立预后因素。此外,通过piRNA通路相关基因POLR2F和PAICS进行进一步研究,研究结果表明,POLR2F和PAICS的表达降低可能有助于预测CRC患者的不良预后,并且在研究二者与免疫微环境的关系发现PAICS可能促进CRC中的肿瘤免疫细胞浸润。这项研究表明,piRNA通路相关基因可以作为预测和诊断肠癌的整体,其中POLR2F和PAICS可能是其中最主要靶点。

