利用线粒体序列比较分析梭鲈鸭绿江和乌伦古湖群体的遗传结构
孙志鹏,鲁翠云,那荣滨,郑先虎
(中国水产科学研究院黑龙江水产研究所,农业农村部淡水水产生物技术与遗传育种重点实验室,淡水鱼类育种国家地方联合工程实验室,黑龙江 哈尔滨 150070)
梭鲈(Sander lucioperca)体肥肉厚、营养丰富、肉质细嫩、味道鲜美,素有“淡水鱼王”之称,俗称十道黑、牙鱼,属鲈形目(Perciformes)、鲈科(Percidae)、梭鲈属。梭鲈肌肉粗蛋白水平(20.99%)和总氨基酸含量显著高于加州鲈(Micropterus salmoides)(18.63%)和鳜(Siniperca chuatsi)(18.90%)[1-4],粗脂肪水平(0.63%)显著低于加州鲈(4.58%)和鳜(2.56%),是具有广阔发展前景的淡水养殖对象。梭鲈在我国主要分布于新疆额尔齐斯河水系、伊犁河水系和黑龙江水系[5]。1992 年,新疆福海县水产技术推广站成功地人工繁殖出梭鲈,随后推广到全国十几个省区[6]。目前,许多天然水体,如黑龙江、兴凯湖和乌苏里江在渔业调查中均发现了梭鲈[5,7,8]。本研究团队在鸭绿江发现有梭鲈群体存在,且具有较大的资源量,其来源不详。乌伦古湖作为最早开展梭鲈繁殖的水体,是我国梭鲈资源的主要产地和供给地。本研究比较分析了梭鲈鸭绿江群体与乌伦古湖群体的遗传差异。
线粒体DNA(Mitochondria DNA,mtDNA)具有结构简单、突变速度快、单倍型随机缺失、较稳定的母系遗传等特点,已被广泛应用于研究群体遗传结构和物种进化等[9,10]。线粒体细胞色素b(Mitochondria cytochrome b,Cyt b)基因和线粒体DNA 控制区(又称D-环区,control region displacement loop,DLoop)是常用的线粒体标记基因[11-13]。Cyt b 基因进化速度适中,其变异主要为转换和颠换,能较好地显示不同科、属、种间的进化关系,常用于构建物种系统发育关系[14,15]。D-loop 区处于非编码区,构特殊,富含A-T 碱基,是线粒体基因组中进化速度最快的区域和研究鱼类近缘种间进化关系和种群多样性的重要分子标记[16-18]。目前国内有关梭鲈主要分布群体的遗传多样性研究较少,本研究结合这2种标记的特点,比较分析了资源记录最早的乌伦古湖梭鲈群体及本研究调查发现的鸭绿江梭鲈群体的遗传结构,有助于了解其资源状况,为梭鲈种质资源保护及繁育利用提供依据。
1 材料与方法
1.1 材料
梭鲈样品分别于2021 年6—8 月取自新疆乌伦古湖(XJ,48 尾)和鸭绿江丹东段(YL,62 尾),剪取部分鳍条用无水乙醇固定后,-20 ℃保存备用。
1.2 基因组DNA 提取
参照基因组提取试剂盒(天根生物)使用说明,从梭鲈鳍条组织中提取基因组DNA,用NanoDropTM8000 分光光度计检测所提取DNA 的浓度及纯度,稀释至50 ng/μL,4 ℃保存备用。
1.3 PCR 扩增与测序
使用引物L14724 和H15149 扩增梭鲈Cyt b基因部分序列[19]。根据梭鲈线粒体基因组全序列(GenBank 收录号:KP125333)[20]设计D-loop 引物、S-Dloop1F 和S-Dloop1R。引物由苏州金唯智生物科技有限公司合成(表1)。Cyt b 基因序列和D-loop控制区的PCR 反应体系均为25 μL,其中模板DNA 2 μL(50 ng/μL)、混合PCR 缓冲液buffer 18 μL(10 mmol/L Tris-Cl(pH8.0)、50 mmol/L KCl、1.5 mmol/L MgCl2、200 μmol/L dNTP)、上下游引物(10 μm/L)各0.6 μL、Taq DNA 聚合酶1.5 U,其余体积用去离子水补充。扩增反应在ABI 9700 型PCR 仪上完成,反应程序为:94 ℃预变性5 min;94 ℃变性30 s,退火温度复性45 s,72 ℃延伸1 min,30 个循环;72 ℃延伸7 min。PCR 反应结束后,取少量产物经8%聚丙烯酰胺凝胶电泳检测合格后,由上海生工生物工程技术服务有限公司纯化后正向测序。

表1 扩增梭鲈Cyt b 和D-loop 区序列所使用的引物Tab.1 Primers used to amplify Cyt b gene and D-loop sequences of mtDNA in pikeperch Sander lucioperca
1.4 序列数据分析
测序后,将序列输入Clustal X 软件[21]进行序列的对位排列,加以人工校对,截取相同长度的序列用于群体遗传分析。用DnaSP v 5 软件[22]统计变异位点类型和数目及简约信息位点数,计算单倍型数、单倍型多样性(haplotype diversity,Hd)、核苷酸多样性(nucleotide diversity,π),识别每个群体的单倍型组成;进行Tajima's D 和Fu's Fs 的中性检验。用MEGA 7.0 软件[23]分析序列的碱基组成和转换/颠换值,用Kimura 2-Parameters 方法计算群体内和群体间的遗传距离,以梭鲈(KP125333)作为参考序列,以加拿大梭鲈(S.canadensis)(MH301082)作为外群绘制基于邻接法(Neighbor-Joining,NJ)[24]的单倍型发生关系图,采用Bootstrap(1 000)检验分子系统树各分支的置信度。用PopART 软件基于TCS 算法,绘制单倍型网络图。用Arlequin 3.11 软件[25]中的分子方差分析(AMOVA)计算群体间及群体内的遗传变异组成,以及群体间的遗传分化系数(Fst)。
2 结果与分析
2.1 梭鲈Cyt b 基因序列和D-loop 区序列特征
将所测得的序列与梭鲈参考线粒体基因组序列(KP125333)进行比对,去除引物序列及低质量序列,截取425 bp 的Cyt b 基因序列和479 bp 的Dloop 区序列用于分析群体遗传多样性。Cyt b 基因共检测到保守位点423 个,变异位点2 个,其中单变异位点1 个,简约信息位点1 个,定义了3 种单倍型(HapA~HapC,表2)。测得的序列中A、T、C、G 的碱基组成分别为26.55%、30.82%、27.30% 和15.33%,其中A+T 含量(57.37%)高于C+G 含量(42.63%),碱基明显偏向A+T,转换/ 颠换值R=14.625。D-loop 区共检测到保守位点453 个,变异位点26 个,其中单变异位点19 个,简约信息位点7 个,定义了17 种单倍型(Hap1~Hap17,表3)。测得的序列中A、T、C、G 的碱基组成分别为32.40%、34.24%、18.38%和14.98%,其中A+T 含量(66.64%)明显高于C+G 含量(33.36%),碱基组成具有明显的偏倚性,转换/颠换值R=0.988。

表2 梭鲈群体Cyt b 基因序列变异位点Tab.2 Variable loci of Cyt b gene sequence in Sander lucioperca

表3 梭鲈群体D-loop 基因序列变异位点Tab.3 Variable loci of D-loop sequence in pikeperch Sander lucioperca
2.2 梭鲈群体单倍型分析
在110 个梭鲈Cyt b 基因序列中,检测到3 个单倍型。单倍型HapA 为2 群体共享单倍型,出现频次最高,分布于92 个样本中,占总数83.64%;其次为HapC(17 个)占总数15.45%,为XJ 特有单倍型;HapB 为YL 特有单倍型,只有1 个。NJ 聚类图显示,HapA、HapB 与梭鲈参考序列KP125333 聚为一支,再与HapC 聚为一大支,加拿大梭鲈作为外群独立为一支(图1)。单倍型网络图显示,HapB 和HapC以HapA 为中心,各有1 个变异位点(图2)。

图1 梭鲈群体基于Cyt b 基因序列的3 个单倍型分布及NJ 聚类图Fig.1 Distribution of three haplotypes and NJ dendrogram based on Cyt b gene of mtDNA in Sander lucioperca
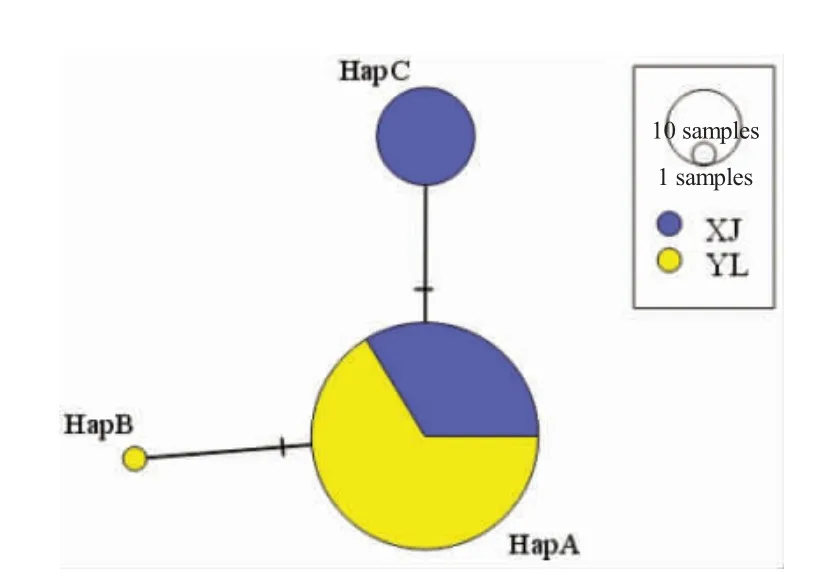
图2 梭鲈基于Cyt b 基因的单倍型网络图Fig.2 Haplotypes network in pikeperch Sander lucioperca based on Cyt b gene of mtDNA
87 个梭鲈D-loop 序列中检测到17 个单倍型。单倍型Hap1 数量最多(65 个),占总数74.71%,为2 群体共享单倍型;其他单倍型均为1~3 个不等,Hap13~17 为XJ 特有单倍型,剩余Hap2~12 为YL特有单倍型。NJ 聚类图显示,大多数单倍型(Hap1、Hap10、Hap3、Hap7、Hap6、Hap15、Hap4、Hap8、Hap2、Hap9、Hap11、Hap12 和Hap5)与梭鲈参考序列KP1 25333 聚为一支,再与Hap14、Hap13 和Hap16 聚为一大支,Hap17 独立为一支,加拿大梭鲈作为外群独立为一支(图3)。单倍型网络图以Hap1 为中心呈现放射状结构,Hap3、Hap4、Hap5、Hap11、Hap15、Hap17 与Hap1 相比只变异1 个位点,其他单倍型变异位点较多,YL 群体与XJ 群体除Hap1 共享外,单倍型分隔明显(图4)。

图3 梭鲈群体基于D-loop 序列的17 个单倍型分布及NJ聚类图Fig.3 Distribution and NJ dendrogram of 17 haplotypes in pikeperch Sander lucioperca base d on D-loop sequence of mtDNA

图4 梭鲈基于D-loop 序列的单倍型网络图Fig.4 Haplotypes network in Sander lucioperca based on D-loop sequence of mtDNA
2.3 梭鲈群体遗传多样性
2 群体Cyt b 基因序列的单倍型数(nh)均为2个,总体单倍型多样性(Hd)为0.279±0.049,核苷酸多样性(π)为0.000 66±0.000 12,YL 的单倍型多样性和核苷酸多样性明显低于XJ。Tajima's D 和Fu’s Fs 检验结果都显示,2 群体差异均不显著,符合中性突变,说明群体保持稳定(表4)。

表4 梭鲈群体基于Cyt b 基因和D-loop 序列的遗传多样性Tab.4 Genetic diversity estimates in pikeperch Sander lucioperca based on Cyt b gene and D-loop sequence of mtDNA
2 群体D-loop 序列的单倍型数(nh)为6~12个,总体单倍型多样性(Hd)为0.442±0.068,核苷酸多样性(π)为0.002 25±0.000 52,YL 的单倍型多样性和核苷酸多样性略高于XJ。Tajima's D 中性检验及Fu’s Fs 检验结果显示,YL 差异不显著,符合中性突变,群体稳定;而XJ 差异显著且均为负值,表明群体曾经历过快速扩张;2 群体D-loop 序列总体极显著偏离中性突变(表4)。
2.4 梭鲈群体间遗传分化与遗传距离
梭鲈群体Cyt b 基因的35.44%的遗传变异来自群体间,遗传分化系数(FST)为0.354 38,表明群体遗传分化程度极大(FST>0.25),达到显著水平。梭鲈群体D-loop 序列的群体间遗传分化系数(FST)为0.025 82,分化程度很大,群体内遗传变异(97.42%)远大于群体间(2.58%),遗传分化也主要来自群体内,与Cyt b 结果一致(表5)。Cyt b 基因的群体间遗传分化程度稍大于D-loop 序列。

表5 基于线粒体Cyt b 基因和D-loop 序列的AMOVA 分析结果Tab.5 The result of AMOVA based on Cyt b gene and D-loop sequence of mtDNA
利用Cyt b 基因序列和Kimura 2-Parameters 方法计算的2 群体之间的遗传距离显示,YL 与XJ 的遗传距离为0.000 873,YL 和XJ 群体内遗传距离分别为0.000 076 和0.001 102;利用D-loop 序列分析显示,2 群体间遗传距离为0.002 269,YL 和XJ 群体内遗传距离分别为0.002 422 和0.001 919(表5)。D-loop 序列分析2 群体间遗传距离大于Cyt b基因序列。鸭绿江群体内遗传距离大于群体间,也提示这个群体可能是一个混合来源的群体。
3 讨论
本研究运用序列测定技术,分析鸭绿江和乌伦古湖2 个梭鲈群体的Cyt b 基因和D-loop 区序列特征,发现梭鲈这两个基因都有高(A+T)含量、低(G+C)值含量的特点,与杜景豪等[26]对日本囊虾(Penaeus japonicus)线粒体Cyt b 和D-loop 基因序列的研究结果相符,也与孔杰等[27]对施氏鲟(Acipenser schrencki)和西伯利亚鲟(Acipenser baerii)线粒体Cyt b 和D-loop 基因序列的研究结果相符。Dibattista 等[28]研究发现,脊椎动物线粒体都具有高A+T值低G+C值特点。胡玉婷及王桢璐等[29,30]发现碱基中A+T 的含量越高,线粒体DNA 的进化优势越明显。本研究中,Cyt b 基因和D-loop 区序列的鸭绿江梭鲈和乌伦古湖梭鲈A+T 含量分别为57.41%、57.30%(Cyt b)和66.69%、66.60%(D-loop),均明显高于G+C 含量,鸭绿江与乌伦古湖相比,碱基含量没有明显差异,表明鸭绿江与乌伦古湖梭鲈群体进化较为同步。本研究中2 群体D-loop 区的A+T 含量均明显大于Cyt b 的A+T 含量,表明碱基组成有偏倚性,说明本研究梭鲈群体的D-loop 比Cyt b 基因在进化中更具优势。吴静等[31]发现。生物体进化过程中颠换是不断积累的,因此转换/颠换的比值逐渐减小,一般认为当比值小于2 时,基因序列突变已经达到饱和状态。本研究结果显示,鸭绿江梭鲈D-loop 的转换/颠换比值为0.504,表示其突变已经达到饱和。
遗传多样性可有效反应生物适应生存能力,遗传多样性越高,该生物体的遗传育种潜力越丰富[32]。单倍型多样性(Hd)与核苷酸多样性(π)是衡量一个群体mtDNA 遗传多样性的重要指标,核苷酸多样性能体现各种mtDNA 单倍型在群体中所占的比例,能更准确地表现一个群体的多态程度,π 值越高表明遗传多样性越高。Ichikawa-Seki 等[33]研究发现,当一个群体核苷酸多样性指数在0.001 5~0.004 7时,该群体的遗传多样性较低。本研究发现,鸭绿江及乌伦古湖梭鲈群体遗传多样性较低。Grant 等[34]根据单倍型多样性和核苷酸多样性之间的关系将群体的扩张事件分为4 种类型:第一类为低单倍型多样性和低核苷酸多样性(Hd<0.5,π<0.5%);第二类为高单倍型多样性和低核苷酸多样性(Hd>0.5,π<0.5%);第三类为低单倍体多样性和高核酸多样性;第四类为高单倍体多样性和高核酸多样性。鸭绿江及乌伦古湖梭鲈群体遗传多样性类型应属于第一类型。此种类型表明群体最近发生了瓶颈效应或由单一、少数系群发生了奠基者效应[35]。梭鲈鸭绿江群体及乌伦古湖群体均受到奠基者效应的影响,群体遗传多样性较低。
本研究结果显示,Cyt b 基因的种群间变异率35.44%明显低于种群内变异率64.56%,D-loop 基因种群内变异率97.42%也明显高于种群间变异率2.58%。表明变异主要来自于种群内,可能是由不同水系不同批次引入梭鲈造成。一般通过Tajima's D检验及Fu’s Fs 检验来检测种或种群内部的自然选择作用。本研究基于D-loop 基因序列Tajima's D 检测及Fu’s Fs 检验结果均显示,乌伦古湖群体为负值且显著偏离中性检验(P<0.01),说明该群体在近期历史阶段出现过种群快速扩张事件。Heather 等[36]根据群体间遗传分化系数(FST)大小划定了群体的分化程度(高度分化,FST>0.25;中度分化,0.05<FST≤0.25;低度分化,0<FST≤0.05),作为群体遗传分化标准被广泛认可。本研究中,Cyt b 基因的群体间遗传分化系数(FST)为0.35438,大于0.25,表明两个梭鲈群体分化程度较高。鲁翠云等[37]利用COI 基因分析了国内外8 个群体梭鲈的遗传结构,在FST判定方面发现国内群体除黑河群体外,XJ 群体和YL 群体并无明显分化,主要是由于参照不同导致,在与国外群体相对比时,FST相对减小。
本研究结果提示,鸭绿江群体部分可能来源于逃逸、放生或养殖弃养等,不完全来源于乌伦古湖群体,且鸭绿江及乌伦古湖梭鲈群体分化程度较高,遗传多样性较低。本研究所得到的梭鲈种群遗传结构特征对于其人工繁育及遗传多样性的监测具有重要意义。

