氨基酸序列分析揭示苯丙氨酸/酪氨酸解氨酶在陆生植物中的演化
盛超雷,原 野,庞力豪,卢宝荣
(复旦大学 生命科学学院 生物多样性与生态工程教育部重点实验室,上海 200438)
陆生植物体内的苯丙氨酸解氨酶(Phenylalanine Ammonia Lyase,PAL,E.C.4.3.1.5),在酶解苯丙氨酸,最终合成木质素和黄酮类等关键次生代谢产物的过程中,具有至关重要的作用[1-2]。通常,PAL仅以苯丙氨酸为底物,催化苯丙氨酸脱氨合成反式肉桂酸再进一步合成对香豆酸,从而参与上述次生代谢产物的生物合成[3]。因此,PAL对苯丙烷途径(phenylpropanoid pathway)的正常运转有至关重要的影响[1]。如果缺乏PAL,就会影响植物的正常生长,同时还会降低植物对病原体的抗性[4]。PAL 广泛存在于高等植物中,并在藻类、真菌和细菌中也有分布,但是尚未在动物中发现[5-6]。PAL对催化底物(苯丙氨酸)具有高度的特异性要求[7],然而在极少数高等植物,例如玉米(Zeamays)和绿竹(Bambusaoldhamii)中,也偶见有类似酪氨酸解氨酶(Tyrosine Ammonia-Lyase,TAL)的活性报道,认为TAL 能够以酪氨酸为底物,直接催化酪氨酸合成对香豆酸[8-9]。
在对二穗短柄草(Brachypodiumdistachyon)的最新研究中,发现了一种能同时催化苯丙氨酸和酪氨酸的新型脱氨酶,即苯丙氨酸/酪氨酸解氨酶(Phenylalanine/Tyrosine Ammonia-Lyase,PTAL,E.C.4.3.1.25)[10-11]。研究表明,PTAL可以在苯丙烷途径中同时催化苯丙氨酸和酪氨酸这两种底物脱氨,参与下游木质素和黄酮类等次生代谢产物的合成,而且PTAL催化酪氨酸的过程与植物中近一半的木质素合成相关[10]。显然,在陆生植物演化过程中产生的双功能解氨酶PTAL,对植物芳香族氨基酸代谢和相关次生产物的高效合成均有重要作用。但是,除了二穗短柄草等少数物种以外,其他植物类群中是否也存在PTAL? 如何准确鉴定包含PAL和PTAL的植物? 对这些问题几乎还没有研究。
与PAL相比较,双功能的PTAL是苯丙烷途径中能够更为高效地利用底物的脱氨酶,相关通路也是苯丙烷途径中相对较为高效的通路[10-11],因为PTAL 不只是丰富了可利用底物的来源,而且还可以催化酪氨酸脱氨而直接合成对香豆酸,比PAL 催化苯丙氨酸脱氨后先合成反式肉桂酸,然后再合成对香豆酸,减少了一步反应。因此,深入分析PAL家族在陆生植物中的变异及演化式样,有助于揭示PTAL 的起源、产生机制以及对陆生植物演化的意义。由于PAL基因家族在DNA 序列水平上的突变类型很多且复杂,直接利用DNA 序列构建系统发育树,难以准确反映其在陆生植物中的演化过程。Watts等[7]和Louie等[12]曾经探索利用PAL与PTAL氨基酸序列第123位点的差异(H/F)作为预测该解氨酶是否具有苯丙氨酸和酪氨酸底物偏好性,但本研究前期的分析结果表明,利用该氨基酸差异位点鉴定PAL 与PTAL的准确率约为90%。因此,亟需一种更为精确的方法来鉴定包含PAL与PTAL的植物。
基于PAL氨基酸序列分析,可以排除编码基因DNA 序列中的无义突变等复杂事件,因此推测,利用PAL氨基酸全长序列构建的陆生植物不同类群系统发育树,能够更接近已知的这些类群间的系统关系。联合一个以上氨基酸序列位点的差异,能够更精准鉴定PAL和PTAL,而且通过蛋白质高级结构同源建模,能够分析PAL和PTAL氨基酸序列中的这些关键位点变异是否提高了对酪氨酸底物结合的能力。利用更精准鉴定PAL和PTAL的方法,就可以在系统发育树上准确标记出包含PTAL的植物种类。基于此,本研究拟回答以下科学问题: 1) 利用PAL氨基酸序列构建的系统发育树,是否能够较为准确反映陆生植物各类群已知的系统关系? 2) 植物从PAL演化为PTAL的过程中,氨基酸序列发生了哪些关键变异? 3) 利用这些关键变异是否可以准确鉴定包含PTAL的植物? 4) 哪些类群的植物中已经演化出了PTAL?
1 材料与方法
1.1 实验材料
用于本研究的植物共35个具有全基因组序列的物种,包括藓类(2个)、蕨类(3个)、裸子(3个)和被子植物(双子叶7个和单子叶19个),在构建系统发育树时,苔类的地钱植物用作为外类群(表1)。所有物种的氨基酸序列均来自不同的全基因组氨基酸序列库(表1)。

表1 用于本研究的植物物种及其全基因组氨基酸序列基本信息Tab.1 Plant species used in this study with information on their complete genomic amino acid sequences
1.2 PAL同源氨基酸序列鉴定及其系统树构建
为了确定PAL的氨基酸序列变异是否能反映陆生植物各大类群的系统关系,在不同植物的全基因组蛋白质数据集中,以栽培稻PAL 的氨基酸序列作为参照,通过BLASTp(NCBI blastp v2.12.0,https:∥ftp.ncbi.nlm.nih.gov/blast/)进行同源检索[13]。BLASTp结果过滤标准: 同源性大于40%,相似氨基酸占查询序列长度的比例大于40%,相似氨基酸占BLASTp结果序列长度的比例大于40%。检索后的氨基酸序列在NCBI CD-Search(https:∥www.ncbi.nlm.nih.gov/cdd/)上验证,确保每个序列都含有完整的苯丙氨酸解氨酶结构域(NCBI accession: PLN02457),并舍弃没有完整结构域的序列及重复序列[14]。
使用MEGAX(Molecular Evolutionary Genetics Analysis,v10.2.6)[15]软件,基于最大似然法(Maximum Likelihood,ML)构建系统发育树,参数选择JTT模型(Jones-Taylor-Thornton model),并用Uniform Rates来表明比对序列在所有位点突变速率相同,Bootstrap验证1000次,当bootstrap值大于80%时,认为该分支真实存在。基于PAL氨基酸全长序列,以苔类植物地钱为外类群,构建包括不同类群植物,如藓类植物、蕨类植物、裸子植物和被子植物(双子叶和单子叶)的系统发育树。
1.3 氨基酸序列比对及PTAL的鉴定方法
利用MEGAX 软件中的Clustal W 工具,对典型PAL 和PTAL 的氨基酸序列进行多重序列比对和鉴定[16]。序列比对结果中的氨基酸残基位置以栽培稻的OsPTAL 作为参照。典型的PAL 氨基酸序列材料来源包括拟南芥(AtPAL,AT2G37040)、银杏(GbPAL,Gb_25608)、地钱(MaPAL,Mapoly0070s0071)、二穗短柄草(BdPAL,Bradi3g49270)、玉米(ZmPAL,Zm00001d051163)、绿竹(BoPAL,AAR24505)、柳枝稷(PvPAL,Pavir.1NG356400)和栽培稻(OsPAL,Os02g41650);而典型的PTAL 氨基酸序列材料来源包括二穗短柄草(BdPTAL,Bradi3g49250)、玉米(ZmPTAL,Zm00001d017274)、绿竹(BoPTAL,ADE08261)、柳枝稷(PvPTAL,Pavir.1NG356200)和栽培稻(OsPTAL,Os02g41630)[10]。
在比对鉴定过程中,具有L121氨基酸残基的鉴定为PTAL,具有I121氨基酸残基的鉴定为PAL,再结合H123氨基酸类型来提高鉴定PTAL的准确率。本研究对35个物种(表1)所有PAL和PTAL序列都进行了鉴定。
1.4 蛋白质三维结构同源建模与分子对接预测
利用SwissDock分子对接平台(http:∥www.swissdock.ch/)计算蛋白质与小分子底物之间的对接[17]。靶蛋白是基于同源建模得到的PAL/PTAL 蛋白质结构,小分子底物为酪氨酸(ZINC entry:266964)。蛋白质结构及分子对接结果的可视化和美化标记使用UCSF Chimera X 1.4(http:∥www.rbvi.ucsf.edu/chimerax)进行。
在蛋白质三维结构同源建模与分子对接预测实验中,选择栽培稻PTAL(Os02g41630)和PAL(Os02g41650)编码的氨基酸序列作为研究对象,基于同源建模算法在SWISS-MODEL 平台(https:∥swissmodel.expasy.org/)[18],同时以高粱PAL酶(PDB accession: 6AT7)[19]的蛋白质结构为模板,进行栽培稻PTAL和PAL高级结构的预测[20]。选用栽培稻作为研究对象,主要考虑到PTAL蛋白研究在高粱、二穗短柄草、玉米和竹子中已有一定报道[8-10,19],作为重要经济作物栽培稻的研究却为空白,亟需了解。另外,最新发表的高粱PAL蛋白结构与栽培稻同源性很高[19],因此参照高粱的PAL 蛋白结构模拟栽培稻PAL和PTAL高级结构准确性较高。
2 结果与分析
2.1 利用PAL氨基酸序列构建的陆生植物系统发育树
利用苯丙氨酸解氨酶基因家族的氨基酸全长序列并以地钱(苔类)为外类群,构建了陆生植物代表类群,包括藓类、蕨类、裸子植物和被子植物共15个物种的系统发育树。结果表明,基于PAL 氨基酸序列构建的植物系统发育树,其系统发育的拓扑结构总体与传统认知的陆生植物各类群间系统关系相吻合(图1)。
图1 基于PAL氨基酸全长序列构建的陆生植物系统发育关系Fig.1 Phylogenetic relationships of terrestrial plants based on full length of PAL amino-acid sequences
在此系统树中,藓类植物在最基部,依次演化形成蕨类植物、裸子植物和被子植物(包括双子叶和单子叶植物)。其中,藓纲的小立碗藓(Pp)与中位泥炭藓(Sphmag)形成最近缘的类群,蕨类植物和裸子植物的某些家族成员相互交错,形成几个系统发育关系不同的分支,而被子植物则表现出从裸子植物北美乔柏(Thupl)和银杏(Gb)演化而来。被子植物表现为一个明显的单系,而无油樟(AmTr)是被子植物中较为原始的类群。值得注意的是,被子植物的物种明显划分为单子叶和双子叶植物两支(图1)。上述结果表明,PAL氨基酸全长序列所反映的陆生植物演化关系基本与传统认知的陆生植物系统发育样式大体一致,特别是在被子植物中,单子叶与双子叶植物的PAL氨基酸序列之间存在明显分化。上述植物系统发育关系的结果,为进一步分析PAL和PTAL氨基酸序列在不同植物中的关键差异,以及利用其序列差异探究PAL和PTAL植物类群的演化关系奠定了基础。
2.2 PAL和PTAL氨基酸序列中的差异位点分析
通过比对陆生植物中含有PAL的8种代表性物种(拟南芥、地钱、银杏、二穗短柄草、玉米、绿竹、柳枝稷、栽培稻)和含有PTAL 的5种代表性物种(二穗短柄草、玉米、绿竹、柳枝稷、栽培稻)的氨基酸全长序列,发现上述物种的PAL和PTAL 均存在3,5-二氢-5-亚甲基-4H-咪唑-4-酮(3,5-dihydro-5-methylidine-4Himidazol-4-one,MIO),即Ala-Ser-Gly(ASG)三肽活性中心——PAL酶活性的重要结构域(图2),表明供试物种的比对序列中均包含了解氨酶活性主要区域。此外,比对分析结果进一步表明,供试PAL 和PTAL代表性物种的氨基酸序列中,在I121L、F123H、A250S、Q301S、I365V、L373V、D420E和P427T 这8个位点存在不同差异(图2)。

图2 8种PAL(a)和5种PTAL(b)植物的氨基酸序列差异位点示意图Fig.2 Diagram of residue differences in amino-acid sequences between eight PAL (a) and five PTALs (b) plant species
PAL代表性物种的氨基酸序列具有典型的I121、F123、A250、Q301、I365、L373、D420和P427残基,而PTAL代表性物种的氨基酸序列具有L121、H123、S250、S301、V365、V373、E420和T427残基(图2)。二穗短柄草、玉米、绿竹、柳枝稷、栽培稻同时存在PAL 和PTAL,其PAL 和其他物种的PAL 序列相似(图2)。然而,在个别供试物种的PAL 氨基酸序列中存在非典型变异或反常变异。例如,银杏GbPAL存在S250和V373(图2)。
本研究通过以上分析,发现I121L和F123H 两个位点相对稳定,可以作为关键差异位点的组合,用以区分PAL和PTAL氨基酸序列。对于任意陆生植物的PAL与PTAL氨基酸序列,可以采用如下方法进行鉴定: 在大多数情况下,具有L121 氨基酸残基的鉴定为PTAL,具有I121 氨基酸残基的可鉴定为PAL;在少数仅用121位点无法准确鉴定的情况下,可以联合123位点进行鉴定,即具有H123氨基酸残基的为PTAL,具有F123氨基酸残基的为PAL,联合121和123位点,在本实验中检测的准确率达到100%(图2)。
上述基于氨基酸序列的差异以及121和123两个位点组合快速鉴定PAL 和PTAL 的方法,为准确鉴定更多物种的PAL和PTAL氨基酸序列,并进一步厘清包含PAL和PTAL陆生植物之间的系统发育关系,以及为探索PTAL产生双功能的原因和演化意义奠定了基础。
2.3 基于PAL/PTAL氨基酸序列的蛋白质结构同源建模
为探究PAL和PTAL氨基酸序列关键差异位点(121和123)可能对其功能差异造成的影响,了解PTAL产生双功能的可能的原因,本研究采用基于氨基酸序列的蛋白质同源建模方法,预测栽培稻PAL和PTAL的蛋白质高级结构,并模拟其与催化底物酪氨酸(Tyr)的分子对接,比较PAL和PTAL氨基酸序列上的关键差异位点I121L和F123H 发生双变异,是否会改变其对结合底物的能力。
栽培稻PTAL蛋白与高粱PTAL的氨基酸序列相似度达90.84%。同源建模结果预测OsPTAL为同源二聚体(图3(a))。其中MIO 结构域含有PAL 酶家族的保守序列和Ala-Ser-Gly三肽活性中心,是PAL脱氨活性的决定性结构域。而本研究关注的I121L 和F123H 关键差异位点,与PAL 活性中心(MIO)的空间距离较近,L121距离MIO 活性中心约17.7Å,而H123距离MIO 活性中心约为11.4Å(图3(b))。因此,PAL在121和123位的氨基酸残基发生变异,可能会影响PAL/PTAL 的蛋白质高级结构,进而改变与酪氨酸底物结合的能力。

图3 基于栽培稻PTAL氨基酸序列的蛋白质高级结构预测结果Fig.3 Prediction of protein conformation based on amino-acid sequence of OsPTAL
通过对栽培稻PTAL和PAL氨基酸序列引入I121L和F123H 双变异(图4),再进行蛋白质高级结构预测,并模拟与酪氨酸底物进行分子对接状况。结果显示,双功能的PTAL 发生氨基酸关键差异位点双变异后,原本在L121残基附近能够结合酪氨酸(Tyr,图4(b))的结构,变得再无法结合酪氨酸(图4(c))。反之,单功能的PAL原本在I121附近无法结合酪氨酸(图4(e)),而当发生了L121I和H123F双变异之后,在L121残基处就变得能够结合酪氨酸(Tyr,图4(f))。
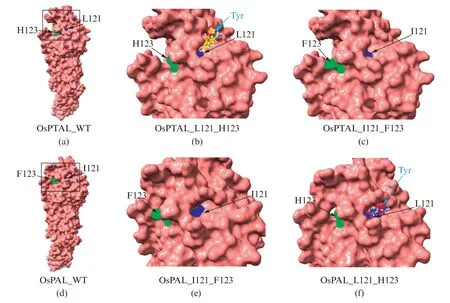
图4 栽培稻PAL和PTAL蛋白质与酪氨酸小分子模拟对接结果Fig.4 Molecular docking results of OsPAL and OsPTAL with tyrosine
上述蛋白质预测结构与模拟分子对接结果,可以说明栽培稻PAL 和PTAL 之间的氨基酸序列关键位点I121L和F123H 残基的差异可能引起蛋白质结构改变,进而改变在L121残基处与酪氨酸底物结合的能力。该结果有助于进一步揭示PTAL出现双功能的原因与机制。
2.4 基于关键氨基酸位点鉴定系统发育中含有PTAL的被子植物种类
为了确定含有PTAL物种在被子植物中的分布,本研究利用PAL氨基酸全长序列,以地钱(Mapoly)为外类群,构建了25属共26个被子植物物种的系统发育树,并在该系统发育树鉴定含有PAL 和PTAL的物种。结果表明,所有被选取的被子植物物种均包含在一个单系中(图5,见 第560页)。利用PAL 和PTAL氨基酸全长序列上两个关键差异位点联合鉴定的方法,对系统发育树中鉴定为含有PAL 和PTAL的物种进行标记(图5)。标记的结果表明含有PTAL氨基酸序列的所有物种聚集为独立一支(图5,绿色支),均为单子叶植物;并且这些物种也同时含有PAL,形成另一个独立的支(图5,蓝色支),也即是说,含有PTAL的物种也同时含有PAL的氨基酸序列。上述同时含有PAL和PTAL的单子叶植物能与其他仅含有PAL的物种分开,支持率为100%(图5,黑色支)。进一步分析还表明,含有PTAL 的物种,其分支的基部类群是夏威夷拟苇(Joac.05G060400)和阔叶原禾(Pl04g11030、Pl02g21590),分别属于禾本目的拟苇科和禾本科,该分支的其余物种均为禾本科植物。以上系列结果,验证了基于PAL 和PTAL氨基酸序列两个关键差异位点联合的鉴别方法,可以准确鉴定包含PTAL 的被子植物,有利于确定其在演化树上的系统发育地位,为深入地揭示PTAL在陆生植物的演化式样具有重要意义。

图5 26种被子植物的系统发育关系以及含有PAL/PTAL的物种Fig.5 Phylogenetic relationships of 26 angiosperm species and those containing PAL/PTAL
3 讨论
本研究基于苯丙氨酸解氨酶基因家族的氨基酸全长序列,构建了陆生植物各大类群,包括藓类、蕨类、裸子植物和被子植物(包括双子叶和单子叶)的系统发育树。分析结果显示,以PAL 氨基酸全长序列构建的系统发育树,能够较为准确地反映陆生植物类群之间的总体演化关系,并与目前已知的植物演化式样基本一致[21]。在这个以地钱(苔类)为外类群的系统树中,藓类在系统发育树的基部,依次演化出了蕨类植物、裸子植物以及被子植物的双子叶和单子植物,其中蕨类和裸子植物类群之间出现部分交叉,反映了PAL演化的复杂性。这一结果与前人的相关研究发现,即裸子植物PAL基因在演化中出现多样化,并且保留了一组不同的PAL基因,分布在3个主要分支中的结果基本一致[22],可能是由于PAL的分支既起源祖先物种,在其衍生物种中又产生了基因复制[22]。本研究结果还表明,被子植物的PAL是从裸子植物中一个较演化的分支而来。因此,基于PAL 氨基酸全长序列分析陆生植物各大类群的演化关系是比较可靠的,能够排除掉PAL基因DNA 序列中的大量无义突变所带来的“噪音”以及基因树拓扑结构的不稳定性(结果未展示),从而更客观地反映陆生植物的总体演化关系,为本研究锁定PAL 和PTAL氨基酸序列的研究,以此分析PAL/PTAL基因家族在植物中的演化奠定了基础。
前人研究发现了二穗短柄草具有双功能的PTAL,即它的PTAL 既能催化苯丙氨酸,也能催化酪氨酸,这对于研究陆生植物苯丙烷途径的演化具有重要的意义[10-11,23]。但是,对于如何利用氨基酸序列来准确鉴定含有PAL或PTAL的植物种类,仍缺乏准确和有效的方法。这些不足,限制了对含有PTAL植物的快速有效鉴定,无法了解PTAL植物在陆生植物各大类群中的分布,也限制了对PTAL在陆生植物演化过程中变异的深入了解。上述问题的进一步解答,可以为揭示苯丙烷途径的演化及其应用开辟新的视野。
本研究以栽培稻为参照,选取了8种具有代表性的植物,共13条氨基酸序列,比较了典型的PAL 和PTAL氨基酸全长序列,发现PAL 和PTAL 在I121L、F123H、A250S、Q301S、I365V、L373V、D420E 和P427T 位点的氨基酸残基上存在显著差异。其中,I121L和F123H 两个关键差异位点的氨基酸残基非常稳定,联合使用这两个关键差异位点,可以作为有效鉴定含有PAL 和PTAL 植物的方法,在本研究已经分析过的物种中,准确率可达100%。此外,本研究中还发现了另一些候选位点,例如Q301S、I365V 和P427T 等,它们可以在未来鉴定更多包含PAL和PTAL 植物种类或样本中,作为重要的补充参考位点,进一步优化含有PAL和PTAL植物的鉴定方法。
过去研究表明,F123H 是一个关键的氨基酸残基变异,能够改变PAL/PTAL 结合酪氨酸的能力。例如,对高粱[19]、竹子[9]、拟南芥[7]等物种的PAL进行H/F氨基酸的人工变异,会带来酶-底物偏好性的转变。I121L是本研究首次提出的另一个关键位点,进一步通过PAL和PTAL 蛋白质同源建模和与酪氨酸的分子对接分析,证明I121L和F123H 的变异均会影响PAL/PTAL 的121位点与酪氨酸的结合能力。因此,由这两个位点残基的变异引起121位点与酪氨酸的结合能力,可能是PAL在演化过程中导致其功能产生改变的关键原因。这些结果为探究陆生植物在演化过程中,其PAL如何从只能够结合并催化苯丙氨酸变异为同时能够结合并催化酪氨酸(PTAL)的可能机制提出了新见解。另外,其他的候选位点,例如Q301S、I365V 和P427T等,也可以在进一步探究和分析不同类群植物PTAL 双功能酶活性产生的机制中发挥作用。
在上述结果的基础上,本研究进一步利用121和123这两个关键位点,对更大群体的被子植物是否包含PTAL进行了鉴定,也即具有L121类型的植物种类均被鉴定为含有PTAL,当121位氨基酸残基出现L/I以外的变异类型,例如V121,就通过结合F123H 位点来进一步确认。基于该方法,对本研究对涉及的全部35个物种的所有PAL/PTAL氨基酸序列进行了鉴定,准确率达到了100%。进一步分析发现,所有鉴定为含有PTAL的被子植物,在构建的系统发育树中形成一个独立的支,仅包含禾本科和拟苇科物种。基于在演化上,禾本科和拟苇科植物的关系非常近,而它们与双子叶植物和本研究中仅包含PAL的其他单子叶植物亲缘关系都相对更远[24-25]的事实,我们推测利用该鉴定PTAL 物种的方法,有利于未来引入更多的类群和物种来深入研究PAL/PTAL 的起源与演化,并深入探究PTAL 的起源、苯丙烷途径高效生物合成以及与植物适应性演化的关系。同时,还可以通过进一步结合其他候选位点的辅助应用来进一步优化更大植物类群的PAL/PTAL精确鉴定方法。
包括木质素和黄酮类在内的次生代谢产物是植物生存与繁殖过程中不可缺少的物质[19],其生物合成效率的提高在陆生植物的演化中极为重要,因而与苯丙烷途径相关的PAL也在不断发生变化。与上述次生代谢产物合成相关的苯丙烷途径,从单功能的PAL脱氨过程演化到双功能的PTAL 脱氨过程,应该是陆生植物在苯丙烷途径演化过程中的一次重要突破。本研究结果表明,具有PTAL 的被子植物只包含在禾本目两个科即禾本科和拟苇科的物种。也即是说,至少在禾本科和拟苇科这两个近缘的科的植物中都演化出了能够同时催化苯丙氨酸和酪氨酸脱氨的双功能酶PTAL。显然,禾本科和拟苇科植物在合成木质素和黄酮类等次生代谢产物方面,发生了重大的变异。那么,在禾本目中还有那些科的植物也演化出了双功能PTAL 呢?
根据目前认知的禾本目系统发育关系[24-25],二柱草科与禾本科的亲缘关系最为接近,而拟苇科与上述两个科的亲缘关系也非常近(图6)。由于拟苇科植物类群只有1属2种,其中夏威夷拟苇中已经出现了PTAL,因此本研究推测为拟苇科也应该出现了PTAL。基于前人已发表的这三个科的系统发育关系[24-25]以及本研究获得的结果,我们进一步推测,至少在禾本科、二柱草科和拟苇科的共同祖先中就已经演化出了双功能的PTAL(图6)。由于目前尚无法获得二柱草科和其他近缘科物种的样本或PAL/PTAL氨基酸序列,上述假设需要进一步的实验来得到证实。但根据本研究检测的结果,禾本目的凤梨科(菠萝Ananascomosus)和莎草科(康藏嵩草Carexlittledalei)等物种中均不存在PTAL。因此,我们推断并不是所有禾本目植物中都演化出了PTAL,而禾本目中的其他植物类群(科)中,例如须叶藤科(Flagellariaceae)和帚灯草科(Restionaceae)是否也已经出现了PTAL,还需要进一步的实验来予以证实。
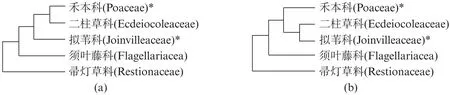
图6 禾本目中禾本科与4个近缘科的系统发育关系以及含有PTAL的状况(修改自Soltis等和Givnish等[24-25])Fig.6 Phylogenetic relationship of Poaceae and the four closely related families in Poales that contain PTAL(modified from Soltis et al.and Givnish et al.[24-25])
在禾本科、拟苇科和二柱草科这三个近缘类群中,后两者的科下物种数量较少,但禾本科是目前世界上物种数最多的科之一,大约包含771个属,12000多个物种,广泛分布于全球各地,也被认为是演化最成功的被子植物类群之一[26-27]。禾本科植物家族的繁荣及其物种、形态特征和生态环境的多样性,可能得益于苯丙烷代谢途径中关键酶,即能够同时催化苯丙氨酸和酪氨酸的PTAL的起源。首先,PAL被普遍认为是特异性利用苯丙氨酸的解氨酶,近年研究对禾本科植物PAL 家族中存在双功能酶PTAL 的重要发现,揭示了这些植物种类能够同时利用苯丙氨酸和酪氨酸参与苯丙烷代谢途径,扩大了可利用底物来源。其次,PTAL的出现,使植物可以利用酪氨酸直接合成对香豆酸,相较于通过苯丙氨酸脱氨合成反式肉桂酸,再合成对香豆酸,进而合成木质素和黄酮类等次生代谢产物,可以缩短反应路径。这两个重要改变,能够大大提高含有PTAL植物种类的苯丙烷代谢途径中代谢产物的合成速率。此外,PTAL 的功能还与禾本科植物独特的细胞壁结构密切相关,改变了植株的机械支撑能力和抗胁迫能力[10-11]。综上,PTAL的产生与演化,可能大大提高了禾本科植物的生存和适应能力,使其物种丰富、形态多样和分布更广泛。
具有PTAL的禾本科是一个与人类密切相关的植物类群,其中包含了许多对人类生存至关重要的农作物。对禾本科等植物类群中的双功能PTAL起源和演化机制的深入研究,不仅能够揭示苯丙烷代谢途径的演化过程,更高效地利用芳香族氨基酸合成木质素(生物能源)和黄酮类化合物(生物制药)等植物重要的次生代谢产物,还能通过调控细胞壁的组成和结构,培育具有抗倒伏、抗病、抗虫等特殊性状的植物品种。另外,通过揭示PTAL的产生机制以及在植物中的演化式样和意义,确定PAL与PTAL的关键差异位点并证实其变异所引起的、与酪氨酸底物结合能力的改变,将有利于未来通过对苯丙烷途径相关基因的遗传工程改造,提高木质素和黄酮类等化合物的产量,使其为人类造福。

