燕麦PRR基因家族鉴定与表达分析
南金生,王跃飞,安江红,3,刘 娜,温欣燕,王梦旭,王媛媛,强 泽,韩 冰
(1.内蒙古农业大学麦类种质创新利用自治区高等学校重点实验室,内蒙古呼和浩特 010018;2.内蒙古自治区农牧业生态与资源保护中心,内蒙古呼和浩特 010010;3.内蒙古自治区农牧业科学院,内蒙古呼和浩特 010031)
燕麦是禾本科燕麦属(AvenasativaL.)一年生草本植物,是一种世界性的粮饲兼用作物[1]。光是植物的重要环境因子,参与多个生理过程,包括光形态建成、避荫反应、种子萌发、开花和衰老[2-3]。植物通过自身的光响应系统,能够准确及时地响应外界光信号的变化[4-5]。昼夜长短影响植物开花的效应叫做光周期现象,Garner和Allar在1920年研究发现日照长度是调控烟草成花的重要因素,由此提出光周期现象[6]。根据开花结实对昼夜长短的不同响应,将植物划分为长日照、短日照和日中性植物等类型。
植物开花是一个受外部环境和内部基因调节的复杂生物学过程。在拟南芥中,CONSTANS/CONSTANSLIKE/TOC1基因家族成员参与光周期开花调控、光信号途径和生物钟调控等过程[7-8]。CCT家族包括CMF(CCT MOTIF FAMILY)亚家族、COL(CONSTANS-like)亚家族和PRR亚家族[9]。氨基端的PRR/RLD(receiver like domain)结构域和羧基端的CCT结构域是伪应答蛋白调节(pseudo-response regulators,PRRs)基因家族结构的显著特征[10-12]。N端响应调节接收结构域和参与ASP磷酸化信号转导途径的接收域极度相似,C端CCT结构域与酵母血红素激活蛋白(HAP2)的NF-YA2DNA结合区相似,CO(CONSTANS)的CCT结构域能够与其他HAP蛋白结合[13-14]。拟南芥中鉴定出了PRR1(TOC1)、PRR3、PRR5、PRR7和PRR9基因[15-17],水稻中有5个直系同源PRR基因,分别为OsPRR1/OsTOC1、OsPRR37、OsPRR59、OsPRR73和OsPRR95[18]。
传统的燕麦是长日照作物,当日照时长低于14 h时,传统的春夏播燕麦材料(下文统称普通燕麦材料)不能抽穗开花[19]。为了提高燕麦产能,扩大适种范围,创制光周期不敏感材料成为育种工作亟待解决的问题。前期通过野生燕麦与栽培燕麦品种的杂交创制出一批对光周期不敏感的种质资源,在海南自然短日照条件下可以正常生长发育。前期通过转录组测序挖掘出了与光周期不敏感相关的差异表达基因,并预测了燕麦光周期不敏感模型[20];一个昼夜光周期的表达量分析表明,PRR家族成员在普通燕麦材料和光周期不敏感材料中表达量变化趋势、表达峰值出现时间和表达量均存在差异,这些基因可能通过响应昼夜节律变化进而影响燕麦光周期敏感性[21]。本研究根据转录组数据筛选出31个与燕麦光周期不敏感相关PRR家族基因的转录本,进行蛋白理化性质分析、结构预测、进化分析及光周期的表达分析;进一步在全基因组水平挖掘到2个AsPRR基因家族成员,利用生物信息学方法全面分析其染色体定位、基因结构、进化关系、启动子区域的顺式作用元件及其表达模式,为揭示光周期不同敏感性燕麦材料在响应短日照胁迫下的分子机制奠定基础。
1 材料和方法
1.1 材料与处理
供试材料包括普通燕麦红旗2号(本实验室保存)和之前创制的光周期不敏感材料gp012。对供试材料进行短日照处理,控制日照时长为12 h,播种和遮光方法参考杨晓虹等[22]的研究。
1.2 燕麦PRR基因家族成员的鉴定
转录组水平:根据COG_class_annotation、GO_annotation、KEGG_annotation、KOG_class_annotation、Pfam_annotation、Swissprot_annotation、eggNOG_class_annotation和nr_annotation的分类和注释结果筛选出与植物开花调控、昼夜节律相关的转录本。
基因组水平:根据GrainGenes网站(https://wheat.pw.usda.gov/GG3/graingenes_ downloads/oat-ot3098-pepsico)公布的燕麦基因组数据库,利用已经鉴定的9条拟南芥(Arabidopsisthaliana)PRR家族蛋白序列的保守结构域构建隐马尔可夫模型(Hidden Markov Models,HMM),以此模型搜寻Avenasativa的所有编码蛋白序列,使用blastp(ncbi-blast-2.10.1+)进行比对,找出Avenasativa蛋白序列中的所有潜在的PRR家族序列。对获得的候选序列利用软件pfamscan(v1.6)和Pfam A(v33.1)数据库对目标序列进行结构域注释,确定同时含有PF06203、PF00072结构域的序列作为最终的PRR序列。
1.3 生物信息学分析
利用BLAST在线分析基因同源性;利用DNAMAN同源序列比对;利用ProtParam工具预测蛋白质基本理化性质;利用NPS@server在线工具预测蛋白质二级结构。使用在线分析软件GSDS(http://gsds.cbi.pku.edu.cn/index.php)预测基因结构;用MEME软件(V5.0.5,http://meme.nbcr.net/meme)分析保守motif。用鉴定出来的AsPRR蛋白与拟南芥、小麦、大麦、玉米、高粱和黑麦草等的PRR蛋白家族序列构建NJ树。用mafft(V7.427)进行多序列比对,然后用MEGA(MEGA7.0)软件进行NJ树的构建,Bootstrap设为1 000。截取基因结构上游2 kb区域作为启动子调节序列,利用(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)预测启动子上面的TF结合位点。在基因启动子物理图谱中标记和显示结合位点的位置。利用在线软件(http://www.softberry.com/berry.phtml? topic=protcomppl&group=programs&subgroup=proloc)对AsPRR家族成员进行亚细胞定位预测。根据AsPRR基因在染色体上的位置,利用网站http://mg2c.iask.in/mg2c_v2.0/绘制染色体物理位置图。利用SignalP-4.1工具进行共线性分析。
1.4 RNA的提取与cDNA合成
使用Transzol Up Plus试剂盒提取试验材料叶片的总RNA,以超微量紫外分光光度计对RNA进行浓度定量测定,1%琼脂糖凝胶电泳检测RNA完整性和浓度,并反转录为cDNA。
1.5 引物设计
利用Primer5.0软件,设计qRT-PCR所需引物,试验所用引物由北京六合华大有限公司合成(表1)。

表1 qRT-PCR所用引物Table 1 Primer of the qRT-PCR analysis
1.6 qRT-PCR
以cDNA作为模板,以燕麦Actin为内参基因,进行qRT-PCR分析。反应体系20 μL: cDNA 1 μL,正反向引物各0.5 μL(10 μmol·L-1),2×TransStart Tip Green qPCR SuperMix 10 μL, ddH2O 8 μL。反应程序:95 ℃预变性1 min;按照下列循环参数进行扩增反应:95 ℃ 15 s,退火温度15 s,72 ℃ 10 s, 40个循环;60 ℃ 30 s,20 ℃ 10 s。每个待测基因均进行3次技术重复及3次生物学重复。采用2-ΔΔCT法分析数据。
2 结果与分析
2.1 燕麦PRR家族成员理化特征分析
转录组测序数据中功能注释为PRR基因的转录本共31个,包含6个PRR基因,分别是PRR1(7个)、PRR3(3个)、PRR6(1个)、PRR37(6个)、PRR73(12个)和PRR95(2个)。筛选获得上述6个注释为PRR基因的最长转录本,其中PRR1-7编码513个氨基酸,PRR3-2编码703个氨基酸,PRR3-2编码253个氨基酸,PRR37-2编码434个氨基酸,PRR73-2和PRR73-6均编码767个氨基酸,PRR95-1编码594个氨基酸(表2)。
2.2 燕麦PRR蛋白家族成员结构域及建模
利用在线预测工具分析31个PRR基因编码蛋白序列二级结构。结果表明,无规则卷曲所占比例最大,其次是α螺旋,延伸链与β-转角所占比例较小(图1)。
PRR1-5、PRR1-7、PRR3-2、PRR3-3、PRR73-2、PRR73-6、PRR95-1等7个基因编码产物含有REC结构域和CCT结构域,PRR1-2、PRR73-1编码产物不包含保守结构域。同时含有REC和CCT结构域的序列中,REC结构域的氨基酸数目有79和104两类,CCT结构域的氨基酸数目也有43和44两类,PRR1-2和PRR73-1编码蛋白均不含有REC和CCT结构域(图2)。
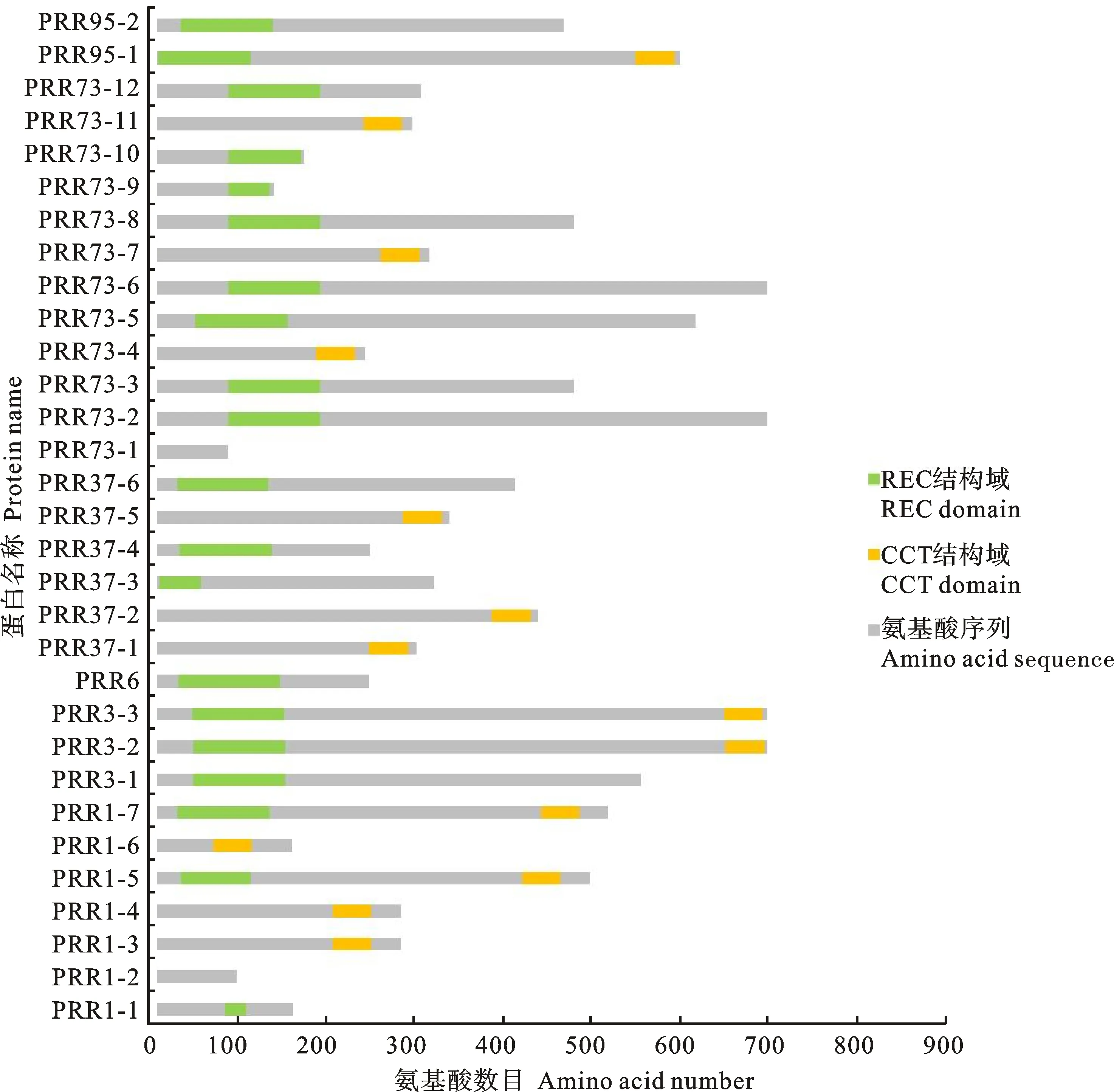
图2 燕麦 PRR 家族成员蛋白保守结构域分析
2.3 燕麦与其他物种PRR蛋白的进化分析
为了研究燕麦PRR家族成员的进化关系,使用MEGA7.0对燕麦PRR家族成员与其他物种的PRR蛋白构建系统发育树(图3)。结果表明,PRR1与大麦(Hordeumvulgare)TOC1、小麦(Triticumaestivum)TOC-A1/TOC-B1/TOC-D1、山羊草(Aegilopstauschii)PRR1亲缘关系较近;PRR3与山羊草、大麦的PRR3亲缘关系较近;PRR37与山羊草亲缘关系最近;PRR73与山羊草、大麦的PRR73亲缘关系较近;PRR95与黑麦草(Loliumrigidum)的亲缘关系最近。
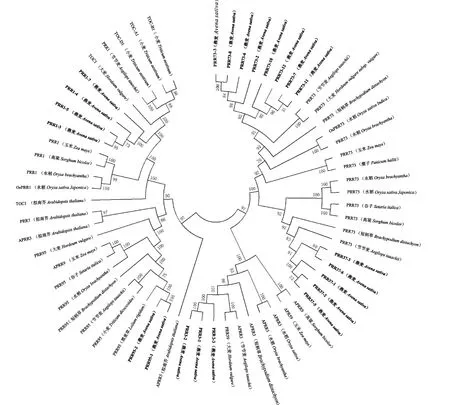
图3 燕麦与其他物种的PRR蛋白的进化分析
2.4 燕麦AsPRR基因家族成员的全基因组鉴定
根据拟南芥PRR蛋白构建隐马可夫模型对燕麦蛋白数据库进行搜索,同时具有PF06203和PF00072结构域的才认定为AsPRR蛋白。结果得到2个AsPRR家族成员,分别定位在4A和5C染色体上,其中AsPRR1位于4A染色体的中部,AsPRR2位于5C染色体的末端(表3)。共线性分析结果表明二者之间不存在共线性。AsPRR1和AsPRR2基因分别编码703和621个氨基酸,其编码蛋白分子量分别为76.5和68.6 kDa,等电点分别为8.65和6.52,蛋白不稳定指数分别为47.23和54.07,GRAVY值分别为-0.657和-0.756,结果表明AsPRR1和AsPRR2为不稳定的疏水蛋白。经亚细胞定位预测分析,AsPRR1定位在细胞外,而AsPRR2定位在细胞核中。

表3 燕麦AsPRR基因家族成员信息Table 3 Information of AsPRR gene family members in oat
2.5 燕麦AsPRR家族的基因结构和蛋白的motif结构分析
经过基因结构预测发现AsPRR1和AsPRR2均包含9个外显子和8个内含子(图4)。为了研究燕麦AsPRR序列的保守性,利用MEME软件进行蛋白序列分析,预测到15个保守motif(图5)。motif1、motif2和motif3最为保守,而motif6、motif8、motif10和motif13等基序在两个基因中的位置不同。
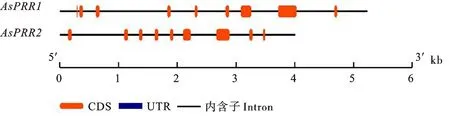
图4 燕麦AsPRR基因结构分析

图5 燕麦AsPRR家族蛋白的motif分析
2.6 燕麦AsPRR蛋白家族系统进化
为了明确燕麦AsPRR基因家族与拟南芥、水稻、小麦、大麦、黑麦草与山羊草PRR家族间的进化关系,利用PRR蛋白与燕麦AsPRR蛋白序列构建系统进化树(图6),结果发现AsPRR1与黑麦草的APRR3关系最近,AsPRR2与黑麦草PRR95关系最近。
2.7 燕麦AsPRR基因顺式作用元件分析
截取基因结构上游2 000 bp区域作为启动子调节序列,分析顺式作用元件的分布。图7展示了排在前15的元件,不同元件用不同颜色表示,其中包括转录起始相关作用元件TATA-box和CAAT、生长发育作用元件O2-site、激素响应元件ABRE以及光响应元件G-box等,而MYC元件可能参与调控花发育、花器官的形态发生和开花时间。
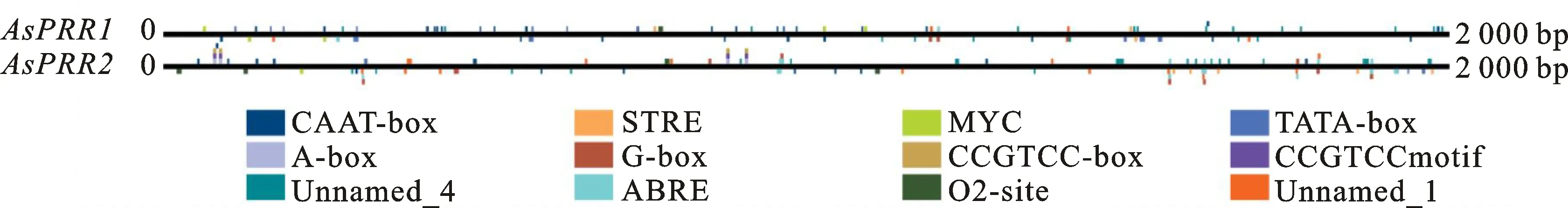
图7 AsPRR基因启动子区域的顺式作用元件分布
2.8 燕麦AsPRR基因表达分析
短日照条件下,AsPRR基因在gp012和红旗2号的幼穗和叶片中不同时期的表达模式不同。红旗2号幼穗发育滞留在枝梗分化期,AsPRR1和AsPRR2在穗(图8a和图8b)和叶(图8c和图8d)的表达量均呈现升高趋势;gp012可以完成整个穗分化过程,AsPRR1和AsPRR2在穗和叶的表达量均呈现先升高后降低再升高的趋势(图8a-d)。AsPRR1和AsPRR2在gp012的初生期幼穗中表达量与红旗2号差异不显著,在叶片中的表达量差异极显著;AsPRR1和AsPRR2在gp012的伸长期幼穗中表达量与红旗2号差异极显著,叶片中AsPRR1的表达量在两种材料的伸长期差异不显著,而AsPRR2的表达量差异极显著。随着生长发育至枝梗分化期,AsPRR1和AsPRR2在gp012穗中的表达量显著降低,叶片中AsPRR1和AsPRR2的表达量于伸长期就已经开始出现显著降低。AsPRR2基因在gp012的不同组织、不同时期中的表达量均高于AsPRR1(图8f和图8h),AsPRR2基因在红旗2号穗中的表达量高于AsPRR1(图8e),AsPRR1和AsPRR2在红旗2号叶中的表达量随着发育阶段的不同而不同(图8g)。以上结果表明,AsPRR1和AsPRR2基因参与燕麦光周期途径,并发挥调控作用。
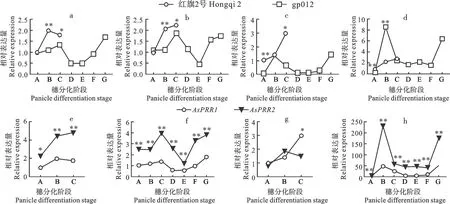
a是AsPRR1基因于穗部的表达量;b是AsPRR2基因于穗部的表达量;c是AsPRR1基因于叶片的表达量;d是AsPRR2基因叶片的表达量;e是基因于红旗2号穗部的表达量;f是基因于gp012穗部的表达量;g是基因于红旗2号叶片的表达量;h是基因于gp012叶片的表达量;A~G分别为初生期、伸长期、枝梗分化期、小穗分化期、小花分化期、雌蕊分化期和四分体期;*和**分别表示0.05和0.01水平显著性
3 讨论
3.1 多物种PRR家族基因研究及其保守结构域
伪应答蛋白调节家族(pseudo-response regulators,PRRs)是生物钟核心振荡器成员,它在维持生物钟稳定的调控、开花时间的调控、光合作用和氧化应激反应等过程中发挥着非常重要的作用[23]。目前在拟南芥与水稻中PRR家族基因对光周期开花途径的调控机制研究成果较多,在大麦、小麦、高粱和大豆等也发现了PRR家族基因。PRR家族基因还可通过调控ABA等方式影响植物抗逆性[14],对植物生物量的积累有重要影响。目前,燕麦PRR基因家族的研究较少,其调控机制尚未明确,有待进一步研究。
REC结构域中的磷酸激酶磷酸化位点能识别来自双组分信号转到系统的信号,在生物应答调节方面发挥重要作用[15],CCT结构域能介导蛋白质之间的互作,能够响应环境的节律变化。拟南芥、大麦、小麦、水稻、高粱等植物中发现的PRR基因也是同时含有REC与CCT结构域,有关保守结构域之间具体的互作关系尚未明确。
3.2 PRR基因在多个物种中参与生理调控
在拟南芥中,PRR基因是生物钟的核心组分,参与生物钟循环,调控下游基因表达[25]。野生和栽培大豆杂交产生的重组近交种群中表征了两个生长期数量性状基因座Gp11和Gp12,携带它俩的品系往往具有更短的生长期和更高的GmFT2a和GmFT5a表达[25]。PRR5、PRR7和PRR9均可以通过CONSTANS依赖性途径控制开花时间[26],PRR7是通过抑制其他蛋白的表达来调控光周期开花的关键基因[27]。水稻中的PRR37在其开花过程中充当中央阻遏物。大麦的Ppd-H1(HvPRR37)[28]在长日照下能调控HvCO-like基因的表达,而短日照无明显影响[29]。高粱中SbPRR37在长日照条件下会出现早上和晚上的表达峰并通过直接抑制SbEhd1的转录来抑制成花素FT(FLOWERING LOCUS T)的表达,另一方面能够激活CO的表达来间接抑制FT的表达,从而抑制高粱开花;在短日照条件下,SbPRR37在晚上的表达峰消失,对高粱开花基因的抑制减弱。大豆中GmPRR37基因在短日照条件下对开花时间没有明显的影响,而在长日照条件下,GmPRR37下调促进开花FT同系物GmFT2a和GmFT5a的表达,上调抑制开花的FT同系物GmFT1a的表达[30]。除影响开花外,PRR基因还有其他功能。例如,OsPRR73通过降低表达来响应水稻干旱胁迫[31]。冷胁迫会刺激水稻OsPRR1的响应,促进OsPRR37、OsPRR73、OsPRR59和OsPRR95基因的表达[23]。PRR5基因表达受ABA抑制,但prr5突变体的种子发芽率显著高于野生型,且主根长于野生型[33]。小麦TaPRR37与小麦抽穗期和株高显著关联,OsPRR37也可以参与控制水稻抽穗期、穗粒数和株高等性状[34]。
燕麦PRR基因家族参与光周期途径,gp012在短日照条件下可以完成全生育期的发育过程,AsPRR1和AsPRR2基因在短日照条件下表达量下调,解除了对开花调控基因FT的抑制,促使gp012能够正常进入小穗分化期发育阶段。
3.3 PRR的短串联重复序列
AsPRR基因在编码区域存在多处短串联重复序列。编码区域的大多数串联重复序列变异可能会引起错义突变、同义突变甚至移码突变,产生新型的蛋白质,从而引起基因功能的增加或者丢失。外显子区域的微卫星序列变异使外显子区域序列的多样化,可能会影响基因转录和翻译。植物中的串联重复序列在外显子区域密度较低,但它们的消失可能引起剪辑改变、外显子改组和点突变,从而改变基因表达进而极可能导致表型的改变[35]。基因间隔区域的串联重复序列变异通常不会直接影响基因表达,但可通过影响染色质组织结构来调控基因表达从而间接影响基因的功能[36],如在着丝粒区域产生折叠而形成特殊的二级结构或者更高的结构。
4 结论
燕麦光周期敏感性与穗发育从枝梗分化期向小穗分化期的转化有关,短日照条件下,AsPRR基因在小穗分化期的下调表达促进了gp012在开花;普通燕麦材料与光周期不敏感性材料的PRR基因在UTR、外显子和内含子区域均存在差异。

