小麦超氧化物歧化酶基因家族鉴定及盐胁迫下响应锌钾的表达分析
杨林林韩敏琦高嘉杨胜敏
(1. 北京农业职业学院,北京 102442;2. 北京清河水利建设集团有限公司,北京 100192)
活性氧(ROS)是细胞代谢产生的羟基自由基(·OH)、超氧阴离子自由基(O2·-)和过氧化氢(H2O2)等一系列副产物[1]。 ROS 是生物体所必需的信号分子,影响着细胞分化、凋亡及胞间物质传导。 正常条件下,细胞内ROS 的产生和清除处于动态平衡状态;然而,植物生长过程中经常会受到极端温度、水分、盐分、重金属离子等各种胁迫,这往往会导致ROS 过量累积[2]。 ROS 的过量积累会诱导氧化应激,破坏膜脂、蛋白质、核糖核酸和光合色素,严重时甚至导致细胞死亡[3]。 为了消除ROS 毒性并保护细胞免受氧化应激带来的损伤,植物已经进化出有效的机制来减轻损害[4],如一些抗氧化酶,包括超氧化物歧化酶(SOD)、抗坏血酸过氧化物酶(APX)、过氧化氢酶(CAT)、过氧化物酶(POD)等,可有效清除植物体内过量的ROS,以维持植物体内正常的生理代谢[5]。
SOD 是抗氧化防御系统的首位限速酶,可以通过催化O2·-歧化为H2O2和O2以减少ROS 造成的损害[6]。 生物体含有一系列SOD 同工酶,均属于金属蛋白酶。 根据催化位点金属辅因子的不同,SOD 可分为四种类型,即Cu/Zn-SOD(CSD)、Mn-SOD(MSD)、Fe -SOD(FSD) 和Ni -SOD(NSD)[7]。 FSD 和MSD 主要存在于低等植物中,CSD 主要存在于高等植物中,NSD 存在于链霉菌、蓝细菌和海洋生物中[8]。 这些SOD 分布在细胞的不同区域,在应对氧化应激中起关键作用。前人研究发现,FSD 存在于线粒体和叶绿体中,MSD 主要存在于线粒体和过氧化物酶体,CSD 主要存在于叶绿体和细胞质,而NSD 主要存在于细胞质中[9]。 目前,已对许多植物的SOD基因家族成员进行了分子解析,包括拟南芥(Arabidopsis thaliana)、丹参(Salvia miltiorrhiza)、玉米(ZeamaysL.)、烟草(Nicotiana tabacumL.)、水稻(Oryza sativaL.)等,结果表明,不同物种的SOD基因家族组成不同,同一物种的不同基因型间SOD组成亦存在一定差异;此外,同一类型的SOD基因在不同物种中作用也不尽一致[7,10]。
小麦(Triticum aestivumL.)是世界上广泛种植的谷类作物之一,含有高比例的碳水化合物、蛋白质、矿质营养及膳食纤维,是全球85%以上人类和牲畜的主要食物原材料[11]。 然而,小麦产区往往处于干旱或半干旱环境,长期灌溉和施肥使得土壤盐渍化加剧,但现推广的大多数小麦品种对土壤盐的耐受性较低、敏感性较强[12],这在一定程度上致使小麦产量和品质显著下降。 小麦SOD基因分析可为其抗性遗传改良提供重要信息,然而到目前为止,在全基因组水平上检测小麦SOD(TaSDs) 基因家族组成的研究较少。 本研究以‘西农979’为试材,对小麦SOD基因家族进行全基因组鉴定,并综合分析TaSDs的系统发育关系、基因组内的分布、基序组成、不同组织中的表达谱以及盐胁迫下TaSDs响应锌钾的功能作用,以期为进一步研究小麦抗逆性奠定基础。
1 材料与方法
1.1 供试材料
试验于2021年12月在北京农业职业学院技术示范区庞各庄实验站进行。 供试小麦品种为‘西农979’,种子来自中国农业科学院。 供试氯化钠(NaCl)、七水硫酸锌(ZnSO4·7H2O)、氯化钾(KCl)均购自北京索莱宝化学生物试剂有限公司。
1.2 小麦SOD 基因家族成员的鉴定和系统发育分析
1.2.1 小麦SOD 基因的鉴定小麦的核苷酸、蛋白质序列和基因注释(General Feature Format Version 3, GFF)信息从IWGSC 公共数据库(https://wheat-urgi.versailles.inra.fr/Seq-Repository/Assemblies)获取,并从Pfam 数据库(pfam.sanger.ac.uk/)下载小麦SOD 的DAN-binding 结构域信息(PF00199),将其结构域信息保存为隐藏马尔可夫格式(HMM)。 使用HMMER 3.0 搜索工具对HMM 进行初步检索与筛选[13]。 使用NCBI 保守域数据库(https://www.ncbi.nlm.nih.gov/Structure/cdd/)对HMM 进行进一步筛选,借助Pfam数据库(http:/ /pfam.xfam.org/)再次筛选以去除冗余、缺失或不正确的序列,得到最终的SOD 蛋白质家族。 按照小麦基因命名指南(http:/ /wheat.pw.usda.gov/ggpages/wgc/98/Intro.htm/)所述的分类方法对小麦SOD 蛋白质家族进行命名与分类,以得到TaSDs基因家族信息。
1.2.2 TaSDs 理化性质表征分析采用ExPASy(https://web. expasy. org/protparam/) 和 WoLF PSORT 在线工具(https://www.genscript.com/wolfpsort.html)预测TaSDs的理化特性和亚细胞定位;根据各TaSDs在基因组注释中的位置,使用Map-Inspect 软件检测TaSDs的染色体分布;根据Savadi 等[14]所述方法对TaSDs进行基因重复分析。
1.2.3 TaSDs 系统发育分析为了进一步阐明TaSDs与其他物种SOD基因家族(SDs)之间的进化关系,下载并比较分析了拟南芥(Arabidopsis thaliana)、玉米(Zea maysL.)、谷子(Setaria italica)和水稻(Oryza sativaL.)的SOD 蛋白序列。 使用MUSCLE 进行全长蛋白质比对[15],并借助MEGA-X 在线软件(https://www. megasoftware.net/)采用邻接法构建系统发育树,Bootstrap 设置为1 000,其余参数为默认值。
1.2.4 TaSDs 染色体定位及共线性分析从IWGSC 公共数据库下载得到小麦的GFF3 基因组注释文件,借助MapInspect 软件将TaSDs一一定位到染色体上,并获得每条染色体的基因密度谱。借助TBtools 软件将物种内和不同物种之间的染色体定位和基因重复进行可视化分析。
1.2.5 TaSDs 的保守结构域、基序和启动子序列分析TaSDs 蛋白的保守域采用NCBI (https://www.ncbi.nlm.nih.gov/) 保守域搜索工具(CD Search)进行检测,使用GSDS 2.0 在线工具(http:/ /gsds.gao-lab.org/)检测TaSDs家族成员的内含子-外显子结构,使用MEME 在线软件(https://meme-suite.org/meme/)鉴定TaSDs的保守蛋白基序,使用TBtools 软件可视化TaSDs的保守结构域。 为了进一步鉴定TaSDs启动子区域的推定顺式调节元件,借助TBtools 软件获得TaSDs基因的起始密码子上游2 000 kb 序列(2-kb 5'),采用PlantCARE 数据库(http:/ /bioinformatics. psb.ugent.be/webtools/plantcare/html/)进一步分析预测TaSDs的顺式调控元件。
1.3 TaSDs 基因家族响应不同盐胁迫类型的表达分析
1.3.1 供试小麦培养方法小麦种子采用0.5%NaClO 进行表面灭菌10 min,用去离子水冲洗数次并浸泡6 h,然后放置于铺垫润湿滤纸的培养皿中,28 ℃培养箱暗处理催芽48 h。 催芽结束后,先用1/4Hoagland 营养液培育4 d, 然后用1/2Hoagland 营养液[16]培育3 d,之后用150 mmol/L NaCl 处理小麦幼苗,1 h 后进行相应Zn、K 处理,其中,Zn、K 目标离子均为200 mmol/L,对照(CK)则加入无菌水。 培养期间,光周期为10 h/14 h(昼/夜),温度为26 ℃。 每处理设置3个生物学重复。 将小麦植株根系、地上部分开后立即用液氮冷冻,并储存于-80 ℃用于RNA 提取。
1.3.2 小麦RNA 的提取 将小麦样品(250 mg)在液氮中快速研磨,按照Plant RNAiso Plus 提取试剂盒(Omega)相关说明提取小麦总RNA,采用Nano Drop 2000 紫外分光光度计(Nano Drop 2000,USA)检测RNA 浓度,采用2%葡萄糖检验RNA 质量。
1.3.3 小麦RT-qPCR 分析将质检合格的RNA用带有gDNA Eraser (TaKaRa RR047Q,北京宝日医生物技术有限公司)的PrimeScript RT 试剂盒逆转录为cDNA。 将cDNA 用无菌水稀释100 倍,作为后续RT-qPCR 的模板。 相关引物(表1)采用Premier 5.0 软件设计,以Actin(Gene ID:AB181991)作为内参基因。 RT-qPCR 反应体系与反应程序参照刘佳月等[17]所述,使用Bio-Rad(Bio-Rad Laboratories Inc., USA)的C1000 Touch PCR 热循环仪和CFX Connect 实时检测系统进行RT-qPCR 分析,每个处理进行3个复孔。 用2-ΔΔCt法计算基因的相对表达量。 使用Origin 2020 软件绘图。
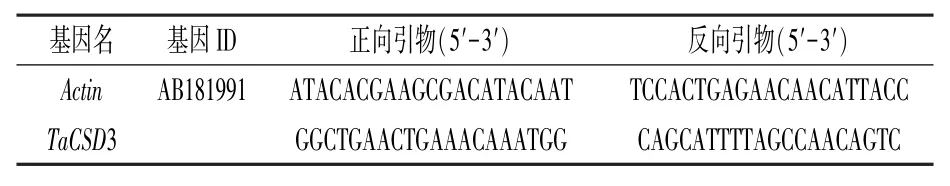
表1 小麦基因的qRT-PCR 引物序列信息
2 结果与分析
2.1 小麦SOD 基因家族理化性质特征分析
经Blast 序列比对、除冗余和蛋白结构域筛选后,共挖掘出10个SOD家族成员,按照基因在染色体上的初步预测位置及命名指南,将其分别命名为TaCSD1~TaCSD4、TaFSD1 ~TaFSD4、TaMSD1 ~TaMSD2。 由表2 可知,小麦SOD基因家族蛋白质长度152 ~382 aa,分子量在15.17 ~42.87 kDa之间;等电点为5.33 ~8.84,其中仅TaFSD2、TaFSD3 为碱性蛋白(等电点大于7),其余均为酸性蛋白。 亚细胞定位预测显示,TaCSD1、TaCSD2 蛋白位于细胞质中,TaCSD3、TaCSD4、TaFSD4 则位于叶绿体中,其余5个TaSDs 均位于线粒体中。

表2 小麦SOD 基因家族理化性质
2.2 TaSDs 基因系统发育关系和分类
为了进一步研究双子叶植物和单子叶植物中SOD 蛋白的系统发育关系,基于37个全长蛋白质序列的比对构建系统发育树,其中包括小麦的10个序列、玉米的3个序列(ZmFSD1、ZmCSD1、ZmCSD2)、谷子的8个序列(SiCSD1、SiCSD2、SiCSD3、SiCSD4、SiFSD1、SiFSD2、SiFSD3 和SiMSD)、水稻的8个序列(cCuZn-SOD1、cCuZn-SOD2、CuZn-SOD-L、pCuZn-SOD、CuZn-SOD-CCh、Fe-SOD3、Fe-SOD2 和Mn-SOD1)、拟南芥的8个序列(AtCSD1、AtCSD2、AtCSD3、 AtFSD1、 AtFSD2、 AtFSD3、 AtMSD1 和AtMSD2)。 根据系统发育树,这37个SOD 蛋白可分为两组,Ⅰ组为Fe/MnSODs,Ⅱ组为Cu/ZnSODs,与它们所包含的结构域类型一致(图2)。Ⅰ组包含19 种SOD 蛋白,可进一步分为3个亚组;在Ⅱ组中,18 种SOD 蛋白分为4个亚组。 聚集在同一亚组中的大多数SOD 蛋白具有较为相同的亚细胞定位(表2),例如,在线粒体的FSDs(TaFSD1、TaFSD2、TaFSD3)、MSDs(TaMSD1、TaMSD2)分别聚为a、c 亚群。 此外,在每个亚组中,我们发现TaSDs 与玉米、谷子及水稻的关系比拟南芥更密切。

图1 5个物种SOD 家族系统发育分析

图2 TaSDs 基因家族编码蛋白的基因结构与保守基序分析
2.3 TaSDs 基因结构及保守结构域分析
2.3.1 TaSDs 基因结构与保守基序分析使用GSDS 2.0 在线工具分析了35个SOD基因组(图2A)的结构信息,即所有序列中的外显子-内含子数量和分布。 由图2B 可知,外显子和内含子的数量在单子叶和双子叶植物的基因之间存在较大差异,外显子的数量为1 ~9 不等。 水稻、高粱、玉米和小麦的SOD基因有1~8个外显子,拟南芥和油菜的SOD基因有7~9个外显子,而所有TaSDs都具有1~7个内含子和2~7个外显子。
为了更好地了解SOD基因家族,使用NCBI保守域数据库对SOD 蛋白的保守域进行分析,并构建所有SOD 蛋白结构的示意图(图2C),可见,所有SOD 蛋白均包含一个超氧化物歧化酶核心结构域(PF00199,Superoxide dismutase)和一个超氧化物歧化酶免疫反应结构域(PF06628,Superoxide dismutase-rel),且来自不同物种的SOD基因具有高度的序列和基因结构相似性,揭示了SOD基因家族成员之间的进化仍具有保守性。
2.3.2 TaSDs 基因保守蛋白质基序分析由图3可知,CSD 的所有保守结构域的bits 值皆大于1,表明TaSDs 蛋白中的8个氨基酸(甘氨酸、亮氨酸、组氨酸、天冬氨酸、丝氨酸、苏氨酸、天冬酰胺和脯氨酸)基序均为保守结构蛋白质基序。 类似的,FSD 包括8个保守氨基酸(缬氨酸、脯氨酸、酪氨酸、丙氨酸、亮氨酸、谷氨酸、丝氨酸和组氨酸),而MSD 则包括FSD 和铁基的保守金属结合结构域DVWEHAYY。 以上结果表明,小麦SOD基因家族中的不同亚家族(CSD、FSD、MCD)含有其特有的氨基酸序列组成,且均包括一系列高度保守的活性位点残基,这些残基可能在催化离子(如Fe2+、Zn2+)的序列特异性结合中发挥作用。

图3 TaSDs 基因保守蛋白质基序分析
2.4 TaSDs 基因的染色体定位
为了确定TaSDs基因在染色体上的位置及相关基因扩增片段区域,基于小麦基因组数据库IWGSC 的最新数据,以97%的序列映射到染色体以及无法映射到任何染色体的为锚定支架,结果(图4)显示10个TaSDs基因中,除TaFSD4外,其余9个SOD基因均可被定位到小麦染色体上,且该9个基因不均匀地分布在8个不同染色体臂的远端区域;其中,TaCSD3和TaMSD1聚集成串联重复事件区域,但这些串联基因在任何其他亚基因组中都没有同源拷贝。 此外,TaSDs基因家族扩增和重复之间的关系显示,TaFSD2、TaFSD3、TaFSD1为同源基因;TaCSD2、TaCSD1、TaCSD4为同源基因,且与TaFSD2、TaFSD3、TaFSD1构成不同的同源基因组。 这些结果表明TaSDs家族的进化是由片段复制和基因串联事件驱动的。
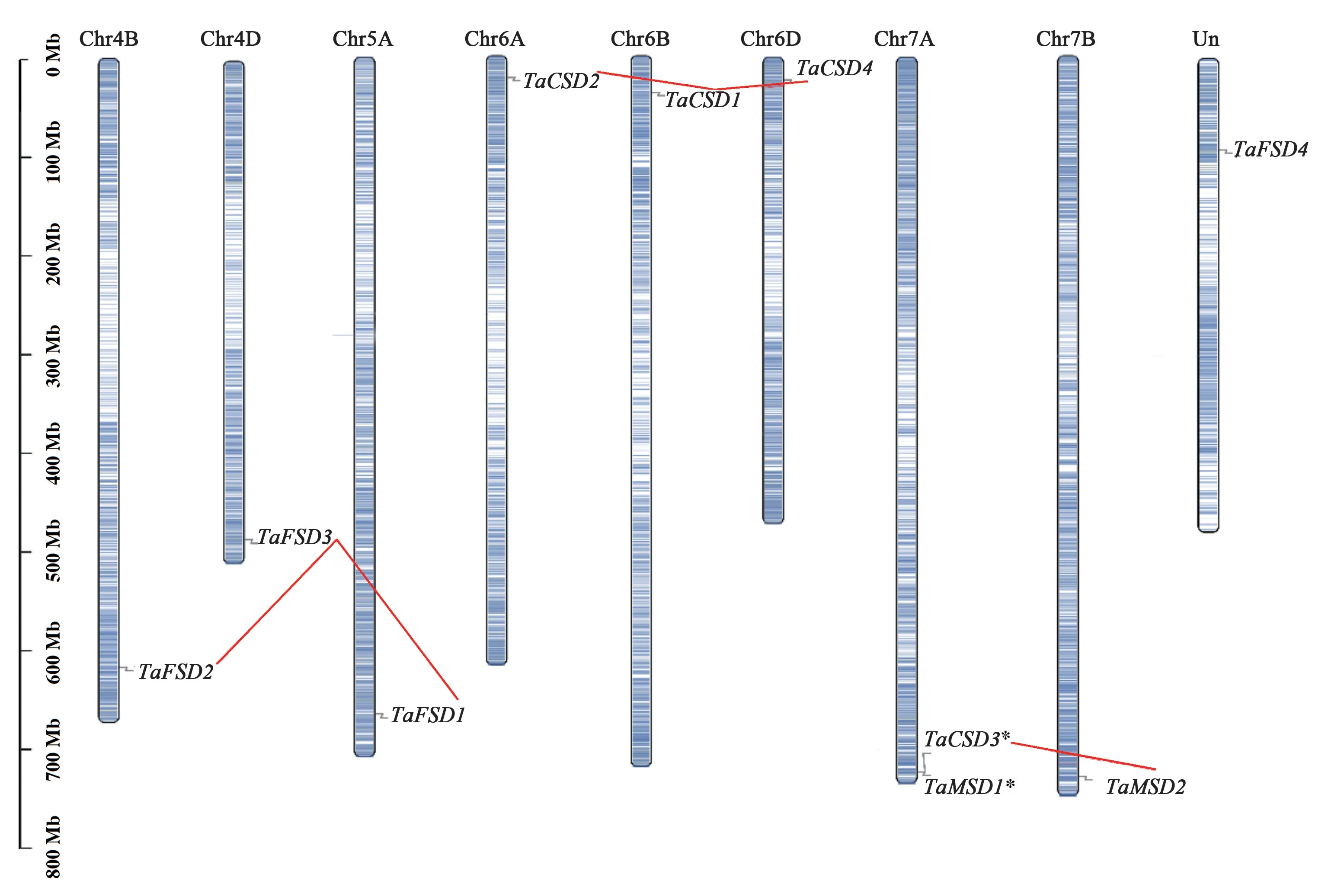
图4 TaSDs 基因的染色体定位
2.5 TaSDs 基因启动子中的顺式元件分析
结果(图5)表明,除一些光照、细胞分化调控等基本核心元件外,TaSDs 还含有激素反应元件(ABA、GA、SA、Me-JA)以及缺氧或厌氧诱导元件等,这些顺式作用元件是非生物应激反应的关键组成部分。 例如,TaCSD1、TaCSD4含有与冷胁迫相关的顺式元件,而TaFSD3、TaFSD4、TaCSD4、TaMSD2含有水杨酸(SA)反应元件;TaFSD2、TaCSD2、TaCSD3的启动子特异地含有一个与分生组织表达相关的元件,表明其可能与分生组织的发育有关。 此外,在大多数启动子区域内发现了一些转录因子结合位点,如MYB 结合位点,表明TaSDs基因可能受MYB 转录因子的调节。 总的来说,同一类别的TaSDs基因可能具有不同的作用方式,并且不同类别的基因也可能具有协同作用。

图5 TaSDs 基因启动子的顺式元件分析
2.6 TaSDs 基因在盐胁迫下的表达模式分析
由图6 可知,盐胁迫(NaCl)和无盐胁迫对照(CK)条件下,TaFSD4、TaMSD2、TaFSD3在小麦地上部(shoot)和根系(root)中均具有较高的表达水平,而TaFSD1均不表达。TaCSD2仅在盐胁迫0、12、24 h 的小麦根系中高表达。TaMSD1、TaCSD4、TaCSD1均在小麦根系中表达。 特别的,TaCSD3、TaFSD2在盐胁迫12、24 h 的根系、地上部中高表达,而在盐胁迫0 h 及对照的根系、地上部不表达或极低表达,初步表明这两个基因可能参与了盐胁迫的调控,应在后续研究中予以重视。
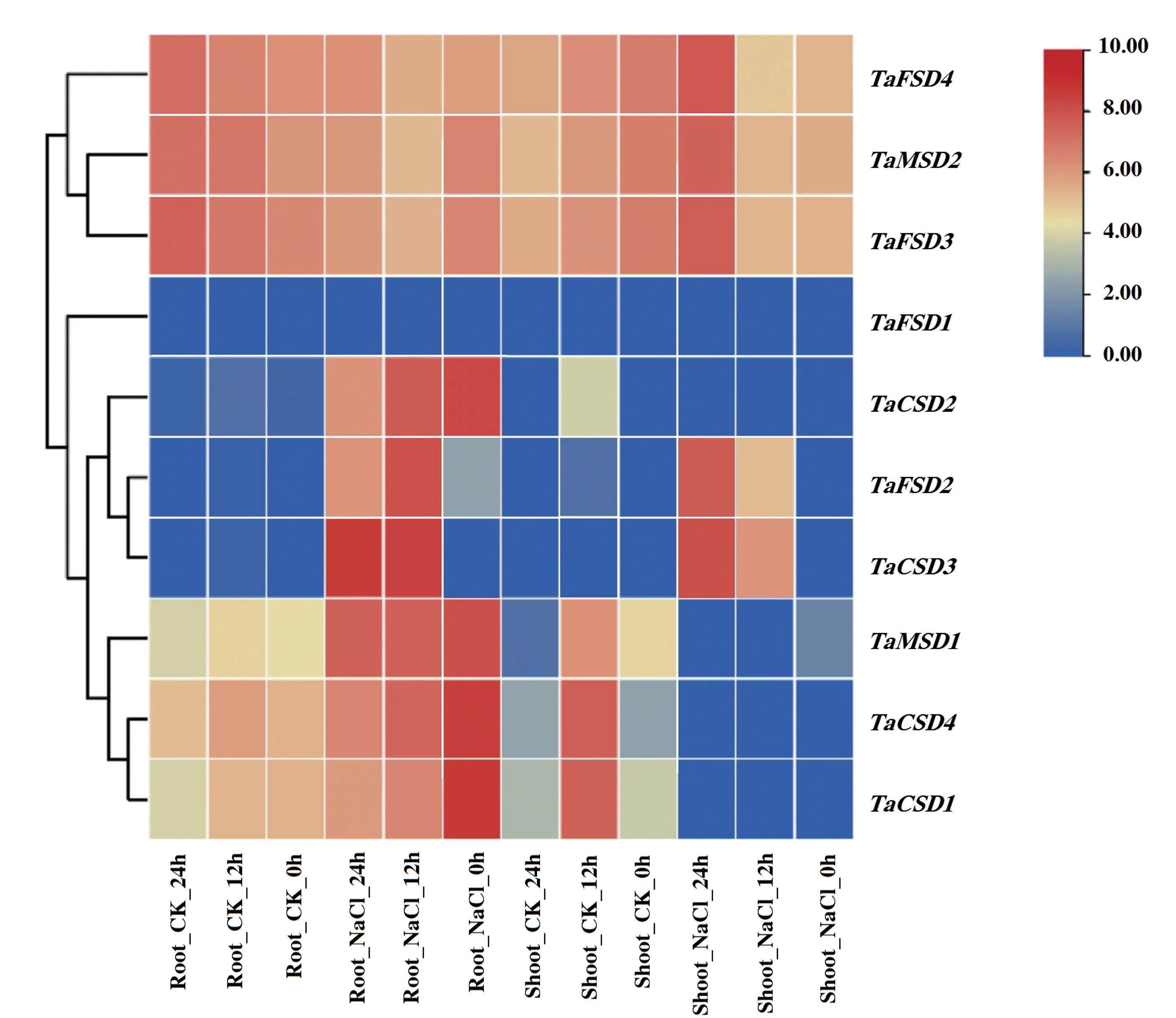
图6 TaSDs 基因在盐胁迫和对照小麦不同部位的Heatmap 分析
2.7 TaCSD3 基因在盐胁迫下及响应锌钾的表达分析
根据TaSDs表达谱分析结果,选择可能参与盐胁迫调控的TaCSD3,进一步研究其在盐胁迫下的表达水平及响应Zn、K 的表达情况。 由图7 可知,无论在无盐胁迫处理(CK)还是无盐胁迫处理下的相关锌钾处理(Zn、K、Zn+K)中,TaCSD3在小麦根系和地上部中均几乎不表达;而在150 mmol/L NaCl 处理下,TaCSD3表达水平较CK 显著升高,增施Zn、K 后其表达水平进一步显著提高,在同时增施Zn 和K 处理下最高,即无论是在根系还是地上部中各处理表现为NaCl<NaCl+K<NaCl+Zn<NaCl+Zn+K,且TaCSD3在根系的表达水平明显高于地上部。TaCSD3在不同组织中的表达模式与转录组测序结果基本一致,进一步证实了上述TaSDs基因表达数据的可靠性,同时表明TaCSD3是响应盐胁迫的指示性SOD基因,同时受Zn、K 正向调控。

图7 TaCSD3 基因在盐胁迫下响应锌钾的表达分析
3 讨论与结论
小麦是世界第二大粮食作物,对保障人类粮食安全具有重要意义[11]。 然而,小麦生长发育及产量易受到土壤盐渍带来的不利影响。 在植物防御系统中, SOD 在清除细胞内过量的活性氧自由基中发挥着重要作用[6-8,18]。 已在多种植物物种中进行了SOD家族基因的研究,鉴定小麦SOD基因并确定其在盐胁迫响应中的功能可为小麦抗盐育种提供优良的基因靶点。 本研究基于pfam搜索和蛋白质Blast 对比,在小麦品种‘西农979’中鉴定出10个SOD基因(TaSDs),包括4个Cu/Zn-SODs 基因(TaCSDs)、4个Fe-SODs 基因(TaFSDs)和2个Mn-SODs 基因(TaMSDs);这些蛋白质长度在152 ~382 aa,分子量在15.17 ~42.87 kDa,且多为酸性蛋白,主要分布于线粒体、叶绿体、细胞质中。SOD基因的数量因植物而异,水稻、高粱和番茄中往往更少,这种差异可能归因于基因重复(串联和节段重复)[7,19]。
系统发育分析表明,5个物种的37个SOD 蛋白可以分为Fe/Mn-SODs 和Cu/Zn-SODs 两组,其中,前者包含19个SOD 蛋白,可进一步分为3个亚组;后者包含18个SOD 蛋白,可分为4个亚组。 此外,来自单子叶植物(小麦、玉米、谷子和水稻)和双子叶植物(拟南芥)的SOD 更倾向于分散分布,且单子叶植物的SOD 更易聚集在Fe/Mn-SODs 组中,表明这些基因可能共享共同的祖先基因。 然而,一些小麦SOD 与其旁系同源蛋白以外的其他物种的直系同源物表现出密切的系统发育关系,表明其与这些直系同源物可能起源于共同的祖先,并在早期基因组变异后分化。
基因结构已被确定为基因家族进化的代表性标志之一[20]。 本研究结果表明,TaSDs的外显子和内含子在单子叶植物和双子叶植物之间亦存在差异,所有TaSDs都具有1~7个内含子和2~7个外显子。 前人研究也表明,一个SOD基因的祖先拷贝有7个内含子[21]。 可见,TaSDs基因在进化过程中经历了外显子和内含子的变化,相应的TaSDs含有超氧化物歧化酶结构域。 此外,TaSDs的不同亚家族(CSD、FSD、MCD)有各自特有的氨基酸序列组成,包含一系列高度保守的活性位点残基。 以上结果表明这些基因在进化过程中是保守的。
基因串联重复事件经常发生在植物进化过程中,与片段重复事件共同被认为是增加基因家族多样性的关键机制,而基因重复在SOD基因的多样化扩展中亦起着至关重要的作用[22]。 本研究中,除TaFSD4外,其余9个SOD基因均被定位到8个不同的染色体上;发现了串联重复的SOD基因对,即TaCSD3与TaMSD1,该两基因在任何其他亚基因组中均没有同源拷贝。 一般而言,单子叶植物基因应在亚基因组中同时具有3个同源拷贝[7];然而TaCSD3、TaMSD1仅为两个,这可能是小麦α-型亚基在染色体之间发生易位以及进化过程中基因组多倍化和基因复制的结果[23]。
基因表达受许多参与各种途径的顺式作用元件和反式作用因子的相互作用调节[8]。 前人研究已经证实,一些SOD基因可被不同的元件途径所诱导,例如寒冷、高温、激素[21,24-25]等。 本研究结果表明,TaSDs基因启动子包含各种应激反应元件,如冷反应、厌氧诱导元件;一些TaSDs启动子还包括MYB 结合位点,表明这些SOD基因可能受MYB 转录因子的调节;此外,一些TaSDs基因启动子中还发现了一些响应分生组织表达、细胞周期调控以及光、激素(ABA、SA、GA、Me-JA、IAA) 反应的元件,表明TaSDs基因可能通过调节活性氧代谢网络参与植物生长和细胞分化。
矿质养分在提高植物的盐胁迫耐受性方面起着重要作用。 钾(K)是一种常量营养元素,在调节植物生理生化中起着重要作用,K+与Na+往往共用同类运输蛋白,因此增加对K+的摄取可以降低对Na+的吸收,从而避免盐诱导的氧化应激和相关细胞损伤[26]。 锌(Zn)是重要的微量营养物质,是Cu/Zn-SODs 亚家族编码蛋白特殊位点的重要催化物质之一[27],能最大限度地减少盐碱化的有害影响,降低NADPH 氧化酶的活性以减少活性氧的产生,从而减轻膜脂过氧化[28]。 Zn、K在基因调控、蛋白质合成、降低氧化损伤和DNA转录中均起着关键作用[26,29]。 本研究中,基于RNA 转录组的Heatmap 分析显示,TaCSD3基因可能参与盐胁迫的调控;进一步的RT-qPCR 分析显示,TaCSD3在无盐胁迫下不表达,而在盐胁迫下显著上调表达,且受Zn、K 的诱导进一步上调表达,在NaCl+Zn+K 处理下表达水平最高。 表明TaCSD3是影响盐胁迫的靶向SOD基因,同时受Zn、K 正向调控。 本研究结果为未来研究小麦SOD 蛋白在生物过程中的功能奠定了基础,也为小麦在盐胁迫育种方面的改良提供了理论依据。

