磁共振酰胺质子转移模态的胶质瘤IDH 基因分型识别:基于深度学习的Dual-Aware框架
楚智钦,屈耀铭,钟涛,梁淑君,温志波,张煜
1南方医科大学生物医学工程学院,2广东省医学图像处理重点实验室,广东 广州 510515;3南方医科大学珠江医院放射科,广东 广州510282
脑胶质瘤是发病率最高的中枢神经系统原发恶性肿瘤[1]。2016年世界卫生组织在脑胶质瘤病理分类中首次加入了基因分型[2],IDH基因分型被确认为脑胶质瘤分类的基础生物标志物,其突变状态的准确识别关系到脑胶质瘤的确诊、治疗方案规划和预后判断[3-6]。在临床中,对脑胶质瘤标本进行病理检测是基因分型的金标准,但肿瘤标本需要通过穿刺或手术切除等有创方式获取,有一定机率导致患者出现神经功能缺损后遗症,给患者的正常生活造成严重影响[7,8]。现代影像学技术的快速发展和普及对临床中脑胶质瘤患者的辅助治疗和预后诊断具有重要意义。
多序列MRI是最常规的用于辅助分析脑胶质瘤的医学影像技术,其中T1CE模态图像在临床中常被用于脑胶质瘤基因分型的影像学判断[9]。然而,T1CE模态需要对病人进行造影剂注射,一方面会对患者造成有创性损伤;另一方面,部分对造影剂过敏的患者无法完成增强扫描检查,造成T1CE模态的缺失。因此,Zhou等[10]于2003年首次提出基于磁共振APT成像技术,这是一种无创的细胞分子水平磁共振技术。APT模态成像对肿瘤的信号强度跟肿瘤恶性程度呈正相关,且IDH野生型的APT信号强度要高于IDH突变型的APT信号强度,它的优点是无创检测内源性,无需注射外源性对比剂。Jiang等[11]利用APT成像首次在术前检测胶质瘤中IDH的突变状态。
当前预测IDH基因分型的方法主要包括基于影像组学的机器学习、深度学习或两者的结合。基于影像组学机器学习的方法从MR影像中提取高维特征,包括肿瘤强度、形状、纹理等,然后基于这些特征训练机器学习模型,用于预测分子标记、肿瘤等级或患者存活率等[12-14]。近年来,基于深度学习的方法因其可以自动提取特征、具有更好的分类精度和鲁棒性较好等特点,也被用于IDH基因分型预测[15-17]。此外,也有将两者结合的方法用于IDH基因分型,例如基于放射组学特征构建深度神经网络,以提高预测性能[18-21]。尽管已经有众多用于IDH基因分型的方法,但上述方法大多是基于FLAIR、T1CE、T1、T2模态构建的。因此,本文将根据APT模态设计深度学习框架,验证其在IDH基因分型任务中的有效性,以替代T1CE模态实现无创的辅助诊断。
此前有关APT模态IDH基因分型的研究大都是基于二维数据构建的,没有考虑肿瘤的空间结构[11,22]。在本研究中,我们基于APT模态的图像特点,使用三维APT影像数据构建Dual-Aware深度学习框架用于IDH基因分型。针对不同患者的肿瘤存在形状、大小各异的情况,而APT模态的分子成像特性使得肿瘤区域附近存在一定的衍生区域。为了充分利用APT图像信息,我们设计Multi-scale Aware模块,用于多尺度信息的提取,并采用空间注意力机制对其进行融合,以增加全局信息优化分类;针对APT模态的肿瘤区域缺乏明显的边缘轮廓这一问题,我们设计Edge Aware模块以增强边缘特征,进一步区分病变和正常组织区域,以提高模型对病变区域的敏感性。
1 材料和方法
1.1 实验数据
本文实验数据来自珠江医院,共包括118例胶质瘤患者的3D MR图像,其中包括50个IDH突变型,68个IDH野生型,每一例患者都采集了三个模态的MR图像,分别是FLAIR模态、T1CE模态、APT模态,其中图像的平面尺寸分别为432×432、480×480、512×512,切片的数量为9~30层。所有图像均以DICOM格式存储,并由经验丰富的放射诊断医生使用ITK-SNAP 软件(http://www.itksnap.org)勾画感兴趣目标区域(ROI)。
1.2 数据预处理
图像预处理的步骤如图1所示。由于采集参数设置不一致,部分病人的不同模态图像存在不同尺寸,无法直接用于深度学习分类网络的训练,需要预处理手段将图像数据设置为同样尺寸。根据数据的尺寸分布特点,文中将平面尺寸统一为480×480,并采用双线性插值方式进行图像缩放;纵向尺寸大于18层的将上下限切片截去缩小至18层,小于18层的将切片数向上下两侧补零层至18层,最终所有图像的尺寸统一为480×480×18。所有模态图像都通过配准工具包Advanced normalization tools(ANTs)[23]进行刚性配准,使得所有数据处于统一的坐标空间,以进行后续实验。
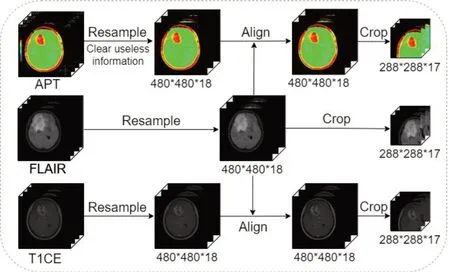
图1 图像预处理步骤Fig. 1 Preprocessing of theAPT modality,FLAIR modality,and T1CE modality.
对于整个3D 脑影像体积而言,胶质瘤肿瘤区域的体积相对较小,其余的大体积非病灶区域对IDH基因分型而言属于无用噪声,不利于该任务的深度学习框架的训练。因此,本研究遍历所有病人的肿瘤区域,确定图像的边界从而得到一个初始的bounding box。遍历所有病人得一个最大体积的bounding box,以覆盖不同尺寸的肿瘤区域及周边区域。最终,根据所遍历的胶质瘤肿瘤区域体积,设置的bounding box 的尺寸为288×288×17,并以此为裁剪目标区域对图像进行裁剪,裁剪使用以下的定位算法。
基本定位的算法:(1)确定肿瘤感兴趣区域的初始bounding box的几何中心坐标(x0,y0,z0);(2)以x坐标为例,如果几何中心的x轴坐标x0距离x轴边界的距离小于288/2,如果其距离x轴左侧较近,则所选取的x坐标最终区域为[0,287],如果其距离x轴正边界较近,则所选取的x坐标最终区域为[192,479];如果几何中心的x轴坐标x0距离x两侧边界的距离大于288/2,则按照中心区域往两侧扩展,则所选取的x坐标的最终区域为[x0-144,x0+143];(3)坐标y、z的确定方式同上。通过上述算法对肿瘤区域进行定位后可以得到最终参与构建分类模型的3D数据。此外,所有数据已完成归一化处理以加速模型收敛,提升模型的精度和鲁棒性。
1.3 模型
本研究基于APT 模态图像特点,构建了Dual Aware框架(图2),包括Edge Aware模块和Multi-scale Aware模块。其中Multi-scale Aware模块针对肿瘤尺寸多变的特点,深度挖掘多尺度信息,并通过空间注意力机制对多尺度信息进行融合,从而增加模型对不同尺寸肿瘤特征的提取能力;Edge Aware模块针对APT模态肿瘤区域边缘不明显的特点,深度挖掘边缘信息,从而增加模型对肿瘤的边界特征信息的挖掘能力,提高分类的精度。
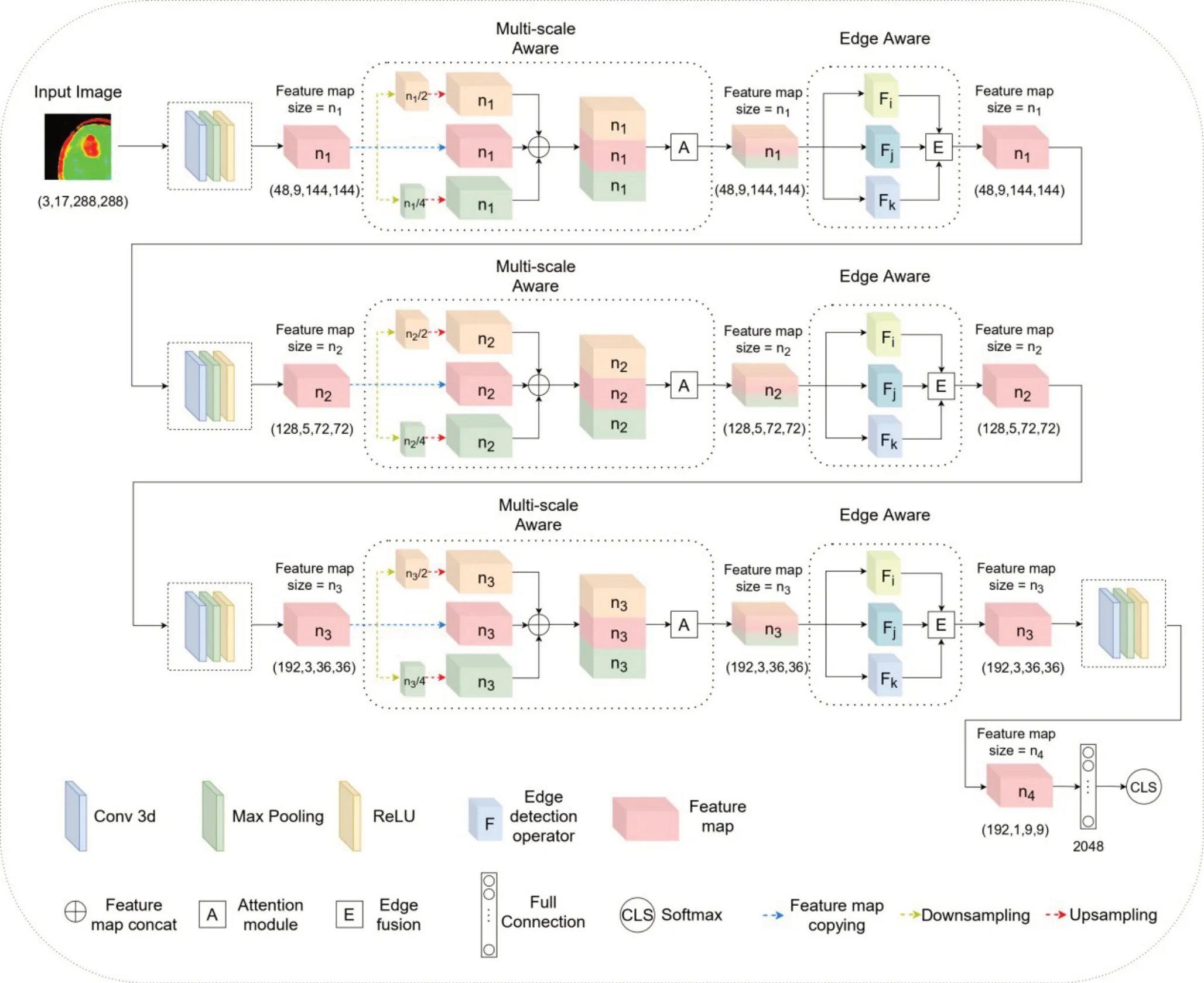
图2 网络框架图Fig. 2 Frame network.
如图2所示为本研究所构建的Dual Aware框架,该框架以3D 图像作为输入,通过3 个层级进行特征提取。每个层级包含如下结构:首先经过一次卷积、池化和修正线性单元进行初步的特征提取;然后输入至Multi-scale Aware模块进行多尺度特征提取,多尺度特征将通过空间注意力机制进行信息融合;再输入至EdgeAware模块,通过三个三维边缘检测卷积算子进行边缘特征的提取,然后进行边缘特征融合。层级之间的下采样尺度为1/2和1/4。经过3个层级的特征提取后,特征被输入至卷积、池化和修正线性单元进行特征维度控制,最后输入至全连接层,通过softmax得到二分类的概率,按照最大概率决定分类结果。
下文将详细介绍本框架所包含的各个模块,其中Multi-scale Aware 模块及其中 的Attention Module 见1.3.1,EdgeAware模块中的Edge fusion见1.3.2。
1.3.1 Multi-scaleAware模块
现实世界中,观测对象的不同特征独立存在于特定的空间范围上,在不同的尺度上观察,对象会有不同的表现形式。在计算机视觉任务中,深度学习模型需要在合适的尺度上提取图像的特征。然而,由于不同图像中目标对象的尺寸不一,不可能事先定义一个最优的尺度去分析图像,因此,需要在多个尺度下去考虑图像内容。多尺度特征在CNN网络中已经展现出很好的效果,被广泛应用在自然图像领域[24,25],在医学图像任务中也错的了不错的结果[26,27]。
针对APT模态中不同患者肿瘤区域大小形状各异,且附近存在一定的衍生区域的特点,我们在模型中构建了Multi-scale Aware 模块。在Multi-scale Aware模块中,输入特征将被下采样卷积至输入尺寸的1/2和1/4,然后通过上采样卷积恢复为输入尺寸,从而完成对多尺度特征的提取。考虑到对多尺度特征直接进行逐像素叠加可能会造成特征信息的冗余,我们引入了3D attention module来进行信息融合。许多研究证明了注意力机制的有效性[28-30],与常规特征融合策略相比,它可以将局部特征与其对应的全局特征进行依赖性权重计算,并自适应地突出相互依赖的特征信息,从而选择性对来自不同类型输入的有用信息进行加权,并抑制相应的噪声信号。在模型中我们构建了基于3D的attention module,建立了基于3D的全局依赖,提高了有效特征的整体权重。
在图3中,首先输入的特征图X(D×H×W×C)通过三个1×1×1的卷积得到三个特征F1、F2和F3用于权重计算,之后三个特征经过展开得到D×H×W×C0的矩阵M1、M2和M3,注意力机制将使用这三个矩阵,定义如下:
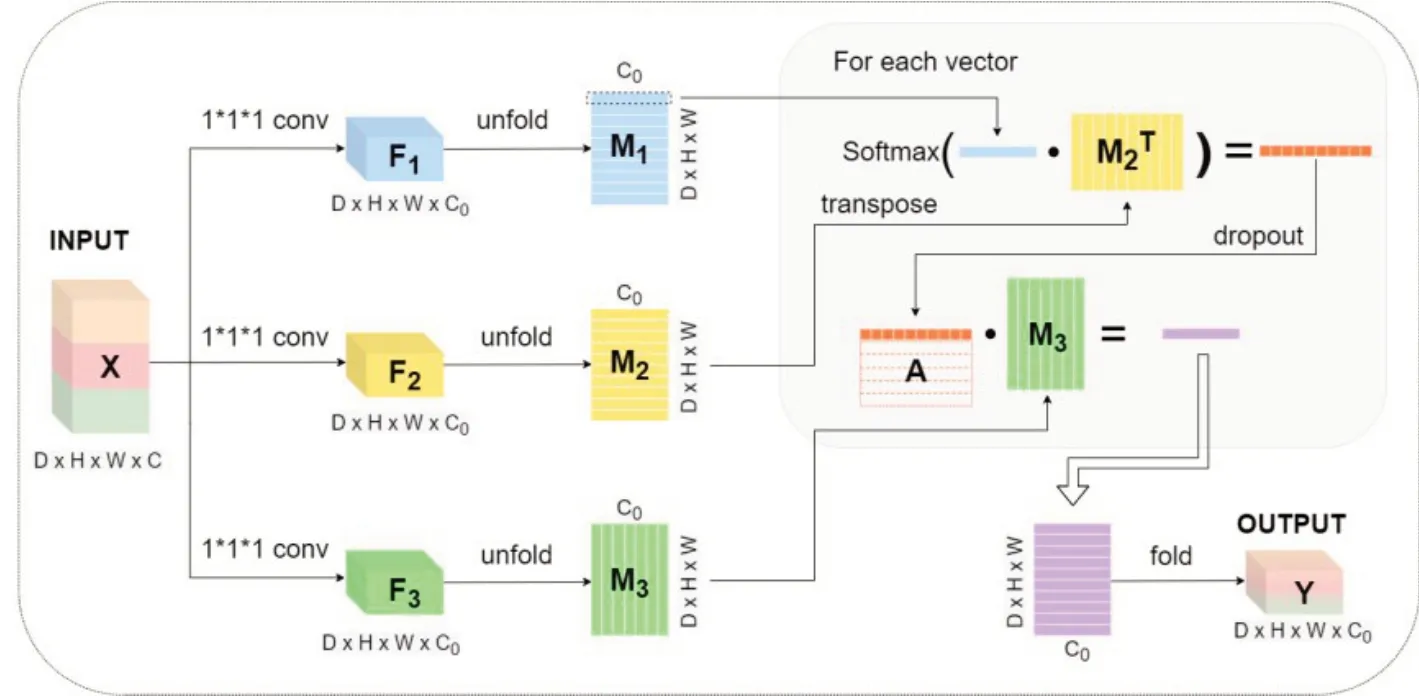
图3 Multi-scale模块中的Attention模块细节Fig. 3 Detail ofAttention module in the Multi-scale module.
其中注意力机制矩阵A的维度为(D×H×W×C)×(D×H×W×C),输出的维度是D×H×W×C0。为了方便描述,以矩阵M1中的一个向量为例,该向量表示某一空间点在C0个维度中的所有特征值,在注意力模块中,该向量将按顺序与M2T中的所有向量进行交互,其中每两个向量之间的相乘为相应的向量产生一个标量权重。这个标量权重是一个加权和,表示一个空间点与所有其他点之间的关系,权重将通过softmax 进行归一化。两个空间点的特征越相似,表示它们之间存在越大的权重系数。对M1中的所有向量重复此过程,最终生成(D×H×W)×(D×H×W)三维空间注意矩阵A。然后,矩阵A和矩阵M3之间将通过矩阵乘法进行特征加权,从而得到加权后的输出特征矩阵,最终该加权特征矩阵将被折叠为空间注意力加权特征图Y。可以看到,每个位置的结果特征Y是所有位置的特征权重。因此,它具有全局上下文信息,并根据空间注意力图选择性地聚合相似的语义特征,有效融合不同类型的特征,从而提高语义特征一致性。
1.3.2 Edge Aware模块 边缘检测可以反映MR图像的灰度变化,并反映了MR图像中不同组织之间的边界,因此对边缘进行提取可以有效捕捉图像内容的纹理结构,有助于将模型对特定病变区域的特征提取。特别是在APT模态中,肿瘤区域对比其他模态图像具有更模糊的边缘轮廓,对其边缘信息进行预先挖掘有助于区分病变和正常组织,有助于更好地描绘异常区域的轮廓[31,32],从而提高模型对肿瘤病变区域的特征提取能力。
如图4所示,输入的特征图X通过EdgeAware模块分别与3个sobel卷积核Fi、Fj、Fk进行卷积,得到3个边缘特征图。3个边缘特征图通过以下方式合并到一起得到最终的特征图Y,计算过程如下:
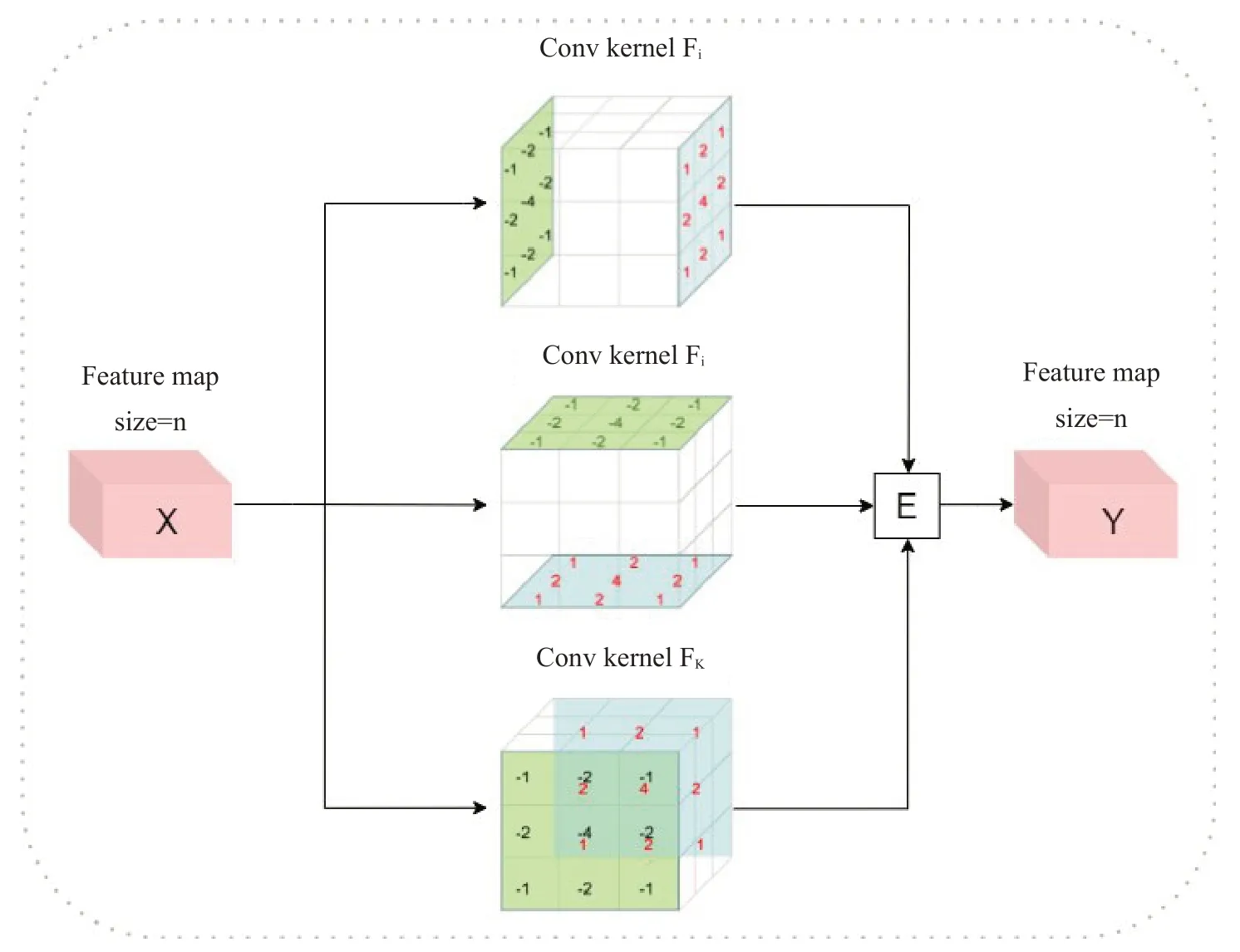
图4 三维sobel算子包括三个卷积核Fi、Fj、Fk,每一个卷积核的大小均为3×3×3。三维卷积核中间没有标注数字的位置均代表着0Fig. 4 The three-dimensional sobel operator includes 3 convolution kernels Fi,Fj,and Fk,and the size of each convolution kernel is 3×3×3.The positions without numbers in the middle of the three-dimensional convolution kernel represent 0.
如图5 所示,以输入模态的原始图像为例,经过EdgeAware模块处理后,病变区域的边缘轮廓被有效提取,特别是APT模态中的代谢变化和生理病理信息,形成了特定的轮廓,有助于模型对其特殊的边缘信息和纹理信息的进一步挖掘,有助于模型更精确区分病变和正常组织,从而提升分类的精度。
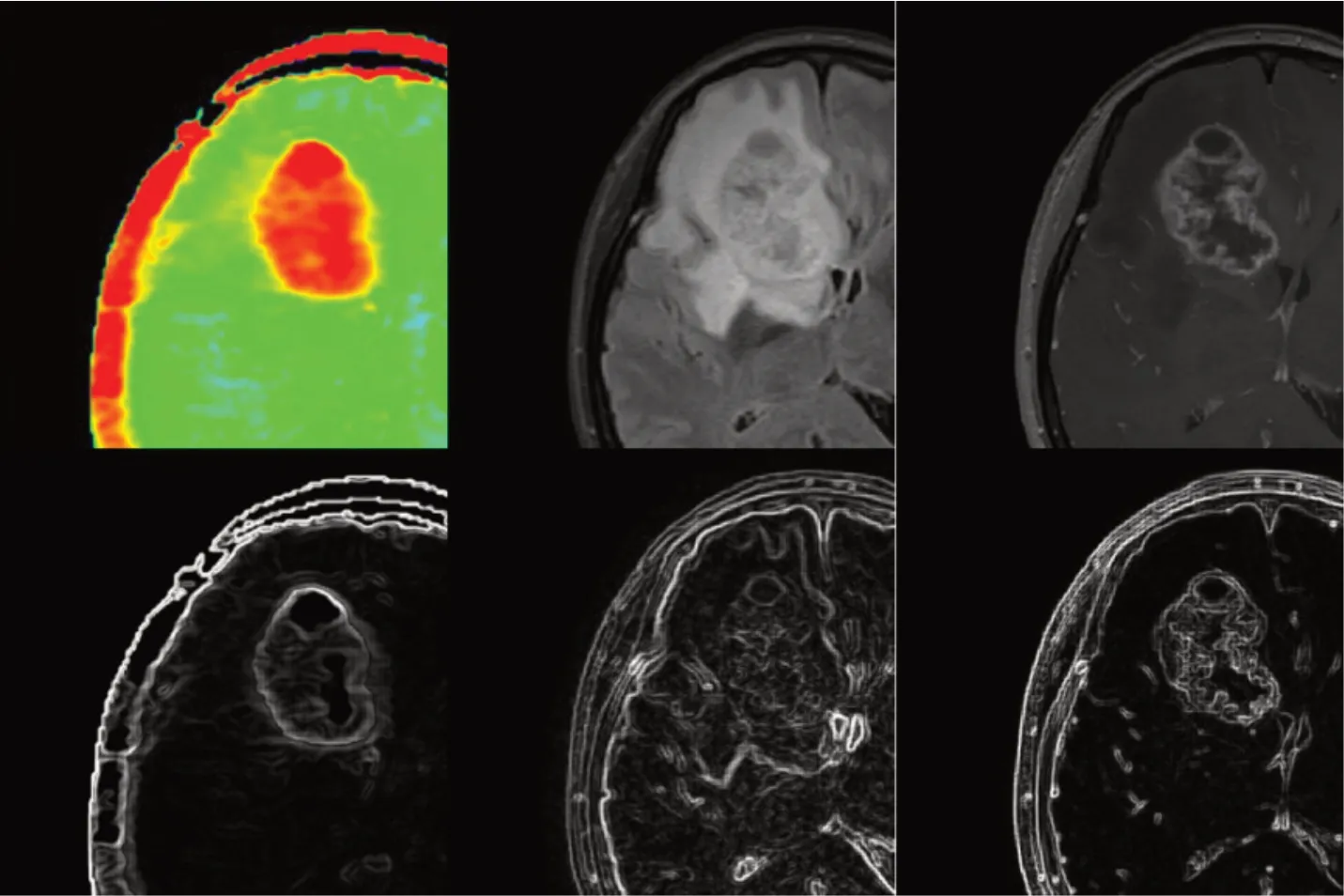
图5 使用sobel边缘检测前后的影像,左、中、右分别为APT 模态、FLAIR 模态、T1CE模态Fig. 5 Image before and after edge detection with sobel.The left,middle and right are APT modality,FLAIR modality,and T1CE modality,respectively.
1.4 实验设置
本研究所使用数据共包括118例脑胶质瘤患者的3D 多模态MR图像,其中包括50例IDH突变型,68例IDH野生型。所有的实验结果均采取四折交叉验证,对4次结果取平均,从而避免单次验证容易造成的偶然性误差。数据分折的情况如表1所示。
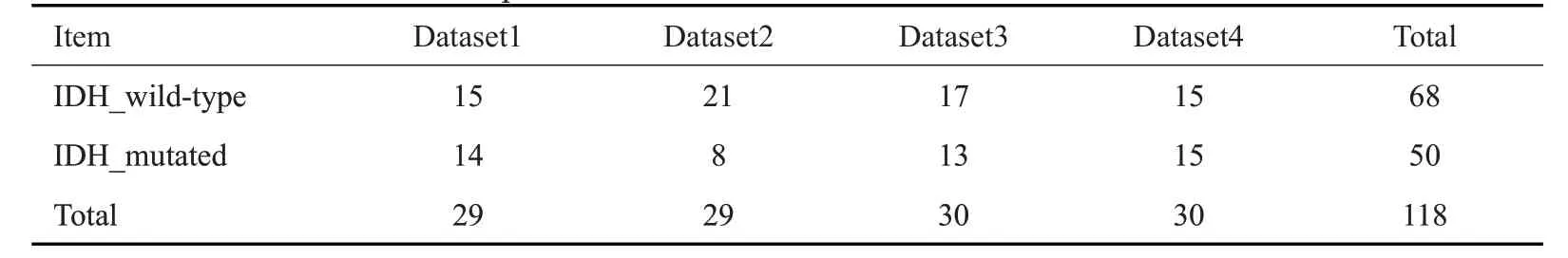
表1 四折交叉验证分折结果Tab.1 Four-fold cross-validation split results
模型使用11GB Nvidia RTX2080Ti GPU进行相关计算和构建,采用了PyTorch深度学习框架。模型训练中,学习率设为10-4,批大小为2,损失函数为交叉熵,损失函数的优化方法为Adam算法[33]。
模型的图像输入尺寸设定为(288,288,17),每一层级的特征尺寸n1、n2、n3分别是9×144×144、5×72×72、3×36×36,特征通道数分别为48,128,192。即整个框架中图像和特征在每一层级的尺度变化为:(2,x,17,288,288)->(2,48,9,144,144)->(2,128,5,72,72)->(2,192,3,36,36),其中x为输入模态的维度,当输入模态为T1CE和FLAIR时x为1,当输入模态为APT时,因其为RGB彩色图像,x为3。
2 结果
为验证所构建模块对Backbone的改进作用,我们基于APT模态的基因分型设计了消融研究(Ablation study)。APT模态在Backbone及使用不同的Aware模块改进下对应的准确率、AUC、特异性和敏感性的结果(表2),其中表中的APT 的结果为Backbone(即AlexNet),M 为Backbone 基础上加入了Multi-scale Aware 模块,E 为Backbone 基础上加入了Edge Aware模块,Dual为Backbone基础上同时嵌入了两类Aware模块。表2的结果显示,无论加入了哪种Aware模块,改进后的分类模型的分类性能均高于Backbone,证明了单个Aware 模块对模型的有效改进;在准确率方面,APT+Multi-scale Aware+Edge Aware的分类准确率最大值、最小值和平均值明显优于其它框架;在AUC值方面,APT+Multi-scale Aware+Edge Aware稍微优于其它网络框架,而APT+Multi-scale Aware 和APT+Edge Aware 基本一致;在特异性方面,APT+Multi-scale Aware+Edge Aware明显优于其他网络框架;在敏感性方面,APT+Multi-scale Aware+Edge Aware优于其它网络框架,而APT+Multi-scaleAware和APT+EdgeAware基本一致。
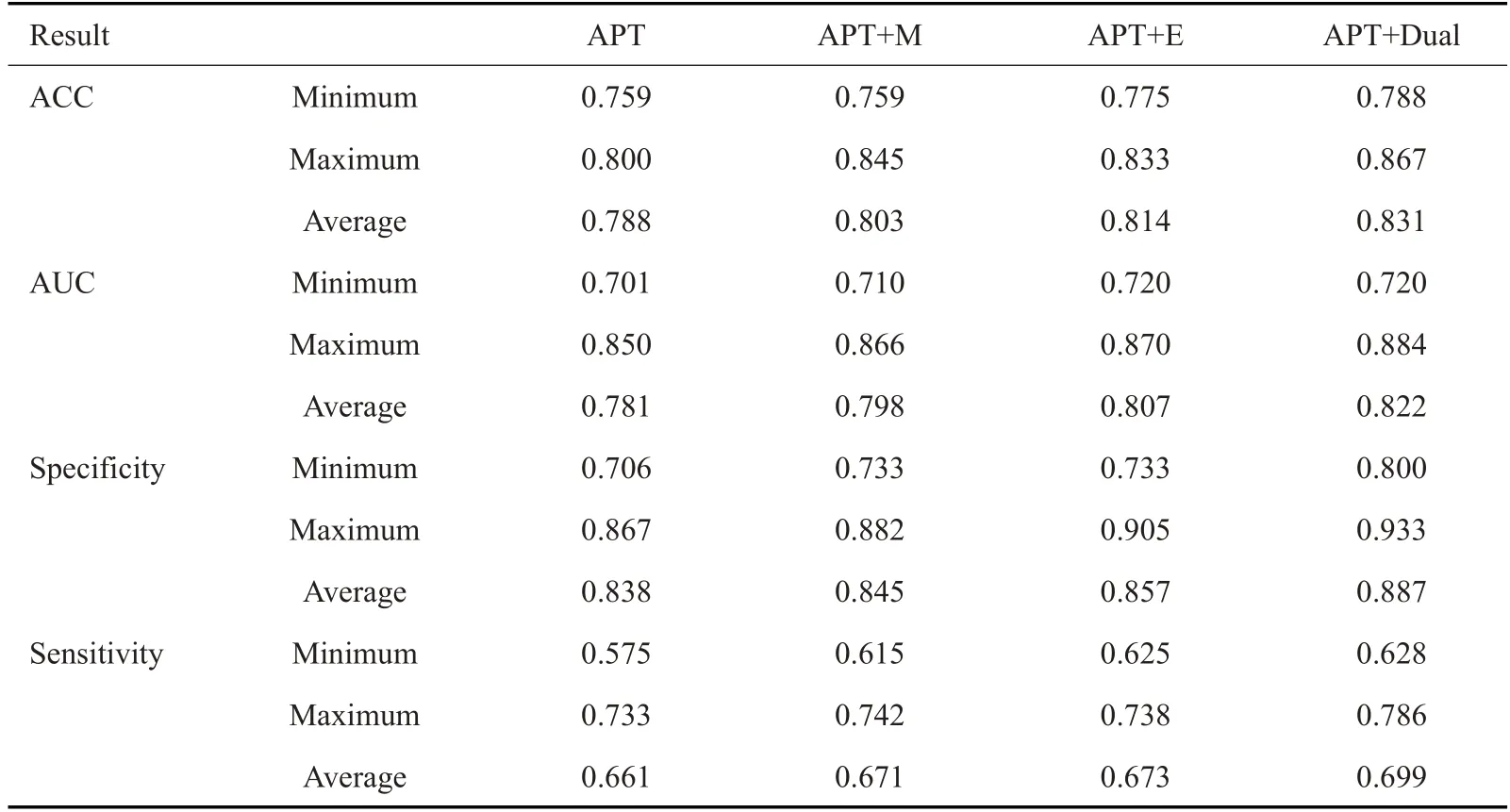
表2 APT模态使用Aware模块分类的实验结果Tab.2 Experimental results ofAPT modality classification using aware module
此外,为了验证APT模态对比其他两类常用模态(即T1CE和FLAIR模态)在IDH基因分型任务中的有效性,我们对比了三者分别在Backbone和Dual-Aware框架中的性能表现,对应的准确率、AUC、特异性和敏感性结果如表3 所示。结果显示,尽管在Backbone中,APT模态对比T1CE模态和FLAIR模态没有太大的性能优势,但是在Dual-Aware框架中,APT模态的整体性能要高于T1CE模态和FLAIR模态。在准确率方面,APT+Dual-Aware的分类准确率最大值、最小值和平均值明显优于FLAIR模态和T1CE模态,T1CE模态要优于FLAIR模态;在AUC值方面,APT+Dual-Aware也明显优于FLAIR模态和T1CE模态,而T1CE模态要优于FLAIR模态;在特异性方面,APT+Dual-Aware明显优于FLAIR模态和T1CE模态,而T1CE模态和FLAIR模态表现基本一致;在敏感性方面,APT+Dual-Aware优于FLAIR模态,却略差于T1CE模态。
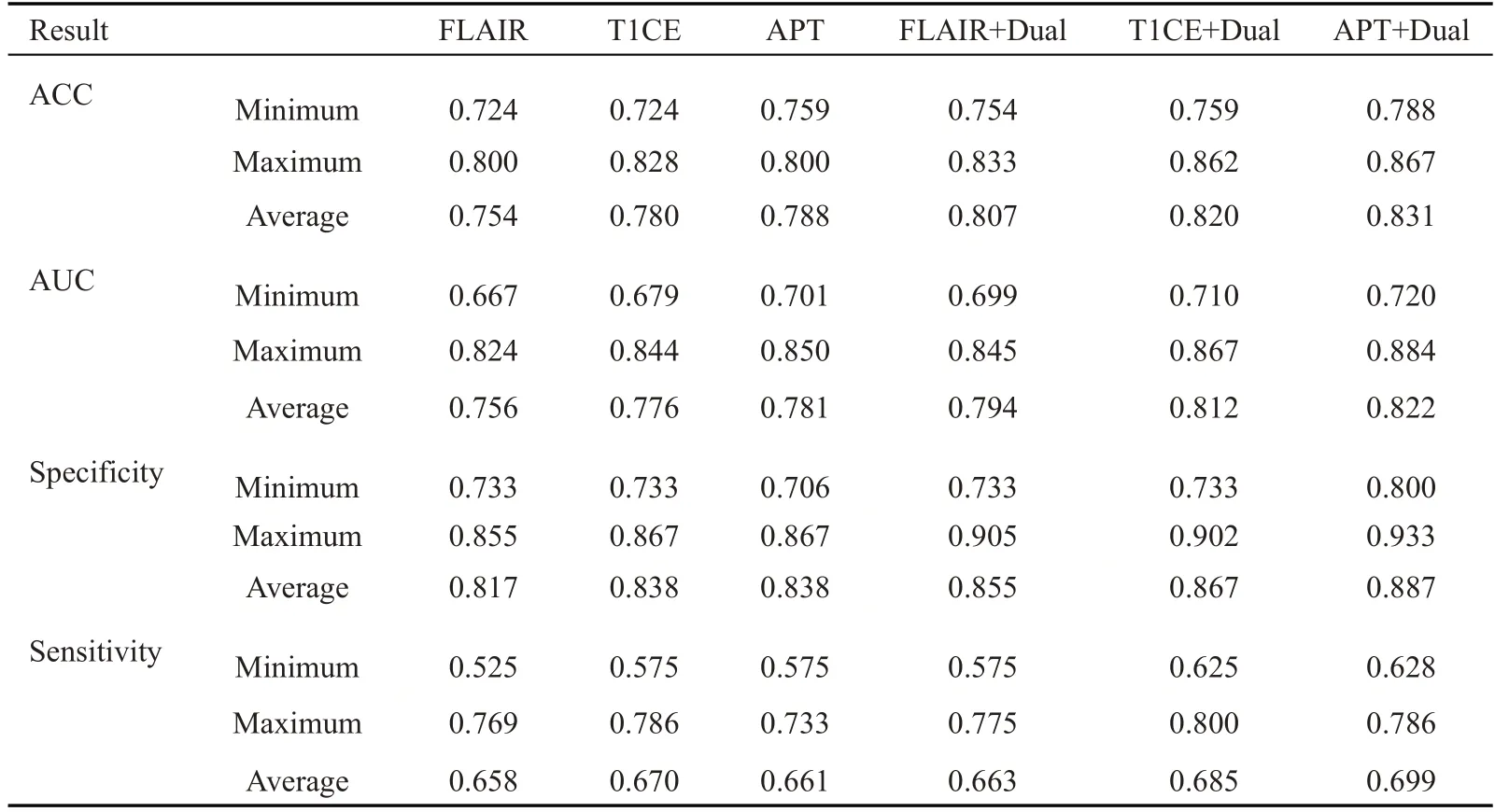
表3 APT、T1CE和FLAIR模态使用Dual-Aware模块分类的实验结果Tab.3 Experimental results ofAPT,T1CE and FLAIR modality classification using dual-aware module
3 讨论
面对日益增长的医疗需求,人工智能在临床问题中的运用得到迅速发展,为一些临床问题提供了可行的解决方案,有利于提升临床诊断效率,基于深度学习的诊断辅助工具也成为了当前临床研究的热点。胶质瘤临床治疗方案的制定所面临的最基本的问题是相关基因型的确认[34]。目前临床实践中通常使用穿刺或手术切除等有创的方式获取病灶组织进行基因测序,检测成本相对较高。现代医学成像技术和人工智能方法的结合与发展,为临床中实现无创的基因分型诊断提供了可能[35]。
相较于其他磁共振成像模态,基于APT 模态的IDH基因分型相关研究相对较少。然而,我们已经了解到APT信号与IDH基因突变状态之间存在相关性[36],这与最近的研究使用APT成像预测神经胶质瘤中IDH突变状态是一致的[11]。Han等[37]通过使用影像组学和支持向量机等方法进一步验证了APT在IDH基因分型的有效性。虽然之前的研究多采用2D APT成像,但我们的研究中采用了3D APT成像序列。在常规临床应用中,我们通常使用三维数据进行图像读出,与2DAPT成像相比,3D APT能够更充分地呈现肿瘤的空间信息和全局特征。在一些研究中,ROI是通过手动放置的方式进行选取[38,39],通常是通过目视检查将ROI手动放置在肿瘤实体的部分,这种手动放置的方式容易导致误差的出现。本研究通过医生直接勾画完整肿瘤的ROI进行分析,3D APT成像覆盖了整个肿瘤区域。深度学习方法对于图像特征的提取更加鲁棒,不受病变位置和范围的影响,因此为我们对胶质瘤的综合分析提供了可靠的支持。在之前的研究中[11,12,37,39],大多数是在特定级别的胶质瘤上进行IDH基因分型,而本文则是对所有胶质瘤患者进行IDH基因分型,且取得了很好的分类性能。
本研究创新地提出了Dual-Aware深度学习框架,基于三维APT模态用于IDH基因分型。考虑到肿瘤形态多变的特点,我们构建了Multi-scale Aware模块,使用多尺度信息加上空间注意力机制对特征图进行融合,能够获得更加丰富的肿瘤多尺度形态及纹理信息,增加全局信息用于优化分类,从而提高准确率。另外,针对APT模态的肿瘤区域缺乏明显的边缘轮廓这一局限性,我们构建了Edge Aware模块以增强边缘特征,进一步区分病变和正常组织区域,提高了模型对病变区域的敏感性。
尽管本研究所构建框架在APT模态的脑胶质瘤IDH基因分型任务中取得了良好的性能,但依然有改进空间。进一步提升该框架性能可从以下两个方面进行:(1)数据集的扩增:深度学习方法通常需要多站点、大容量样本充分训练网络,以增加模型的泛化性和鲁棒性。本研究所用数据集的容量相对较小,且都来自同一个合作医院。后续将从多个医院收集数据,以增加数据容量,提高数据的多样性。(2)多模态融合:后续将尝试通过多模态的信息融合以提升IDH基因分型的预测准确性。
综上所述,本文基于APT 模态图像特点构建的Dual-Aware框架验证了APT模态在胶质瘤IDH基因分型中的有效性,有望在临床上避免对病人进行造影剂的注射,从而帮助医生提高临床实践的准确率和效率。

