水稻OsRIP基因家族的全基因组鉴定及其表达分析
张 琪,刘 爽,胡艳娟,王晓雪
(沈阳农业大学水稻研究所,沈阳 110161)
在自然环境中,植物始终面临着很多与它们生存相关的生物与非生物胁迫。为了应对逆境,植物进化出不同的机制来调节自身的生长发育[1-6]。泛素(ubiquitin)是小分子量的蛋白质,含有76个氨基酸。蛋白质的泛素化(ubiquitination)是指在一系列酶的催化下,将泛素分子连接到靶标蛋白质的赖氨酸残基上的过程,是蛋白质翻译后修饰的重要形式,被多泛素化的蛋白质通过26S蛋白酶体降解[7]。这一过程在调节植物生长发育中扮演重要角色,同时也是调控植物对生物、非生物胁迫响应的重要机制之一[5,7-8]。水稻中的RMD Interacting Proteins(OsRIP)基因家族含有Really Interesting New Gene(RING)-HC 结构域和SEVEN IN ABSENTIA(SINA)结构域,是典型的E3泛素连接酶[9-10]。鉴定和分析水稻中RIP基因家族成员和编码蛋白质的理化特性、结构域和进化关系,分析其在激素和非生物胁迫处理后的表达模式,对深入研究水稻RIP基因家族在激素信号转导及非生物胁迫响应中的作用具有重要的理论和实践意义。
蛋白质的泛素化(ubiquitination)是指在E1泛素激活酶(E1 ubiquitin-activating enzyme)、E2泛素结合酶(E2 ubiquitin-conjugating enzyme)、E3泛素连接酶(E3 ubiquitin ligase)的催化下,将泛素分子连接到蛋白质的赖氨酸残基上,对蛋白质进行翻译后修饰的过程。被多泛素化的蛋白质通过26S 蛋白酶体(26S proteasome system)降解,被称为泛素化介导的26S蛋白酶体降解途径[7]。E3泛素连接酶是决定底物特异性的关键酶,它能够将泛素分子与靶蛋白的赖氨酸残基结合,从而降解、调控靶蛋白[11-13]。目前在植物中已发现数百种E3泛素连接酶,共分为四种类型,分别为RING 泛素连接酶、homologous to E6-AP carboxy terminus (HECT)泛素连接酶、U-box 泛素连接酶以及Cullin-RING ligases (CRLs)泛素连接酶[7,14-15]。其中RING 型E3 泛素连接酶数量最多,在调节种子萌发、开花期、激素响应过程中发挥重要作用[16-18],研究发现,Delayed Seed Germination 1 (OsDSG1) 是一种C3HC4 型RING 泛素连接酶,通过与Rice ABA Insensitive 3(OsABI3)相互作用,调节水稻生长发育。osdsg1 基因功能缺失突变体的种子萌发延迟,耐盐和耐旱能力显著增强[19]。水稻中的SINA 型E3泛素连接酶Flowering-Related RING Protein 1(FRRP1)与Rice Histine 2B(OsH2B)互作,通过影响Heading date 3a(Hd3a)的表达来调控水稻开花期[20]。近年来,RING型E3泛素连接酶在各种非生物胁迫响应中发挥的作用被广泛研究[21-25]。研究发现,含有RING 结构域的E3 泛素连接酶Salt-induced RING finger protein 4 (OsSIRP4) ,可以使Peroxisomal membrane protein 11-1(OsPEX11-1)蛋白多泛素化降解,对盐胁迫起负向调节作用,过表达OsSIRP4 的转基因植株表现出对盐敏感的表型[26]。C3HC4 型RING 泛素连接酶RING finger protein v6(OsRFPv6)通过调控Na+离子吸收影响水稻的耐盐性,是盐胁迫的正向调节因子[12,26]。
SINA型E3泛素连接酶是RING型泛素连接酶的成员,它的特点是N端含有一个RING结构域,负责结合E2蛋白分子,对SINA 型E3 泛素连接酶活性具有决定性作用;C 端含有一个SINA 结构域,在动物和植物中高度保守[27]。SINA型E3泛素连接酶在调控植物的生长发育、逆境响应过程中发挥重要作用[21,28-29]。香蕉(Musa acuminata)中的SINA 型E3 泛素连接酶MaSINA1 通过调节INDUCER of CBF Expression 1(MaICE1)的稳定性而对冷胁迫起着负向调节作用[30]。在玉米和水稻中,Salt-and Drought-Induced RING finger 1(SDIR1)超表达转基因株系耐旱性增强,进一步研究发现SDIR1 通过调节其靶蛋白SDIR1-Interacting Protein 1(SDIRIP1)的稳定性来调控对盐和干旱的反应[13]。E3 泛素连接酶在调控植物的生长发育、逆境响应过程中发挥重要作用,但对水稻中具有E3 泛素连接酶活性的OsRIP基因家族的系统鉴定及在逆境响应过程中的作用未见详细报道。本研究鉴定了水稻RIP基因家族的所有成员,分析OsRIP 蛋白质的理化性质、蛋白质的结构、保守基序、进化关系、分析OsRIP基因的染色体定位,共线性,启动子顺式作用元件、表达模式以及在非生物胁迫响应中的表达模式,为深入研究OsRIP在水稻生长发育和逆境响应过程中的生物学功能奠定基础。
1 材料与方法
1.1 水稻材料与处理
用75%的酒精处理沈农9816(SN9816)种子1 min,在30 ℃的条件下浸种、催芽;将发芽的种子在14 h光照、10 h黑暗的条件下培养2周。然后,将培养2周的幼苗分别用20%聚乙二醇(polyethylene glycol,PEG)、150 mmol·L-1NaCl、100 mmol·L-1甘露醇(mannitol)、用100 μM 脱落酸(abscisic acid, ABA) 和100 μM 茉莉酸(jasmonic acid,JA)处理幼苗,在处理后0,6,12 h后分别采样,-80 ℃保存备用。
1.2 方法
1.2.1 水稻OsRIP家族成员的全基因组鉴定 在Ensembl Plants数据库(http://plants.ensembl.org/index.html)下载各物种全基因组数据。利用保守结构域预测网站Pfam(http://pfam.xfam.org/)和SMART(http://smart.embl.de/)进行结构域分析,下载基因的隐马尔可夫模型,在不同物种的全基因组蛋白序列数据中检索,运用BLAST对数据库进行比对,将所得到的序列合并去掉重复,获取RIPs基因家族的备选成员,阈值设定为E<1e-5。将所有鉴定得到的候选基因序列提交到InterPro数据库(http://www.ebi.ac.uk/interpro/)、SMART数据库以及NCBI CDDsearch数据库(https://www.ncbi.nlm.nih.gov/cdd/)验证蛋白保守结构域。
1.2.2OsRIP基因家族的特征分析 利用ExPASy Proteomics Server网站(http://web.expasy.org/protparam/)在线分析6个基因的氨基酸数、相对分子量、理论等电点、不稳定系数[31]。利用亚细胞定位预测网站PSORT Prediction(http://psort1.hgc.jp/form.html/)进行蛋白定位分析。利用GSDS 2.0(http://gsds.gao-lab.org/)绘制基因结构图。利用Mapchart软件工具进行OsRIP基因染色体位置可视化分析。
1.2.3 保守基序分析 为了鉴定RIP蛋白家族中的保守基序,使用在线分析网站MEME(http://meme-suite.org/)分析了OsRIP基因家族基序,基序数量设置为8个,其余参数默认。
1.2.4 系统发育分析 将基因家族成员用MEGA软件进行序列比对,以最大释然法(Maximum Likelihood)构建进化树。用进化树美化软件EvolView(https://www.evolgenius.info/evolview-v2/#login)生成进化树。
1.2.5 顺式作用元件分析 基于水稻基因组数据,使用TBtools软件提取OsRIP基因家族上游2000 bp(起始密码子ATG之前)序列作为顺式作用元件分析序列。使用在线分析网站PlantCARE(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)进行顺式作用元件的分析[32]。
1.2.6 表达模式分析 用在光照14 h和黑暗10 h条件下生长2周龄的沈农9816(SN9816)的幼苗,在胁迫和激素处理前后采样。提取样品的总RNA,最后反转录为cDNA,进行quantitative RT-PCR (qRT-PCR)。qRT-PCR所用引物如表1。微阵列数据来自水稻表达谱数据库RiceXPro(https://ricexpro.dna.affrc.go.jp/)。用50 μM ABA 和100 μM JA处理水稻四叶老苗0,1,3,6,12 h;进行3次生物重复,用处理后的幼苗进行微阵列分析。用TBtools绘制热图[33]。
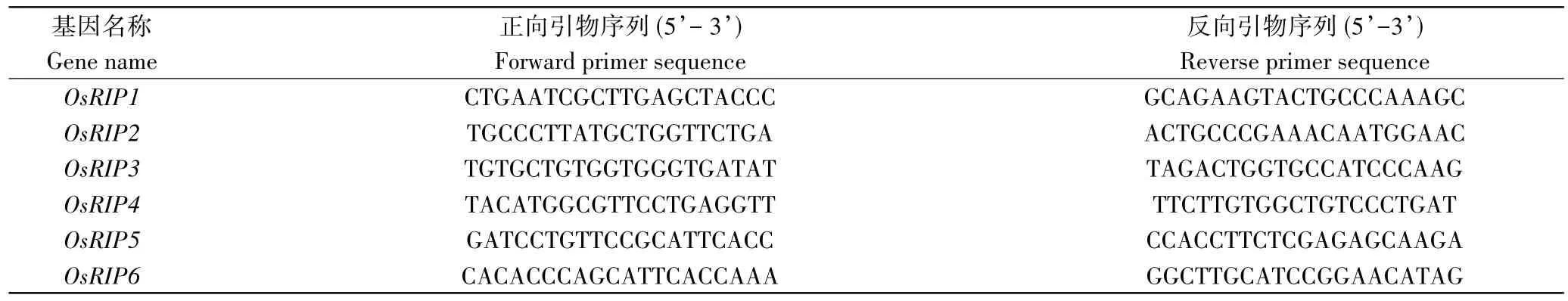
表1 引物列表Table 1 The list of primer sequences
2 结果与分析
2.1 水稻中OsRIP基因家族的鉴定及蛋白的理化性质分析
OsRIP1 是E3 泛素连接酶,具有SINA 保守的结构域[9]。为了鉴定水稻中OsRIP基因家族的成员,本研究利用有保守的SINA 结构域的OsRIP1,检索水稻全基因组,共有6 个编码SINA 结构域的基因,进一步利用Inter-Pro、SMART 和NCBI数据库对水稻中OsRIP 蛋白家族进行了鉴定,结果发现,在水稻中共存在6个OsRIP 蛋白,分别命名为OsRIP1~6(图2A)。
本研究利用ExPASy Proteomics Server 网站分析了OsRIP家族蛋白质的氨基酸数、相对分子量、理论等电点(isoelectronic point,PI)、不稳定系数,并通过PSORT Prediction 网站预测了该蛋白家族的亚细胞定位。结果表明,OsRIP 理论等电点在6.26~7.51,其中只有OsRIP5 的PI 值大于7,为碱性蛋白质,其余5 个成员的PI 值小于7,为酸性蛋白质(表2)。不稳定系数除OsRIP6 外均大于40,表明该基因家族编码的蛋白大多为不稳定蛋白。OsRIP家族CDS 编码区的长度在906~1086 bp,氨基酸(amino acid,aa)数量在301~361 aa,相对分子量在34.1~39.2 kDa。亚细胞定位预测的结果为OsRIP1、OsRIP2、OsRIP3、OsRIP4、OsRIP6 定位在细胞质中,而OsRIP5 在细胞核、细胞质和质膜上均有分布(表2)。
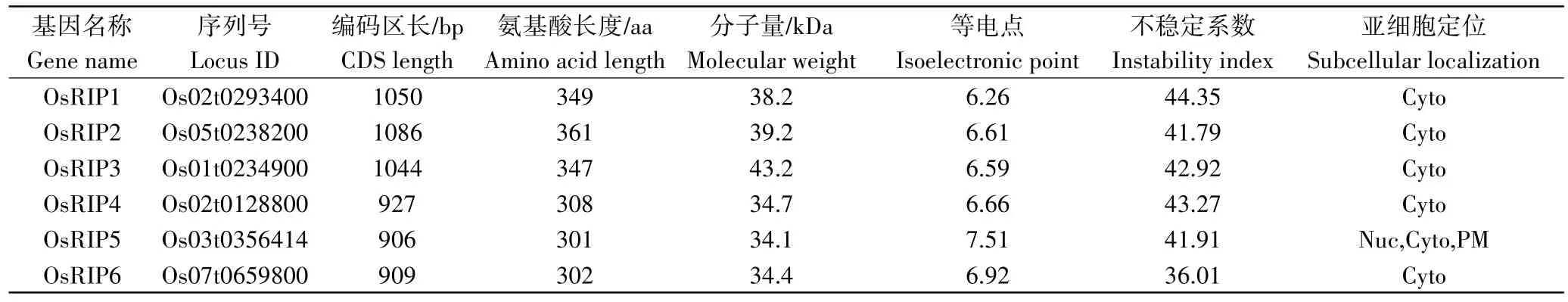
表2 水稻OsRIP基因家族成员及其编码蛋白的理化性质Table 2 OsRIP gene family members and physicochemical properties of the encoded proteins in rice
2.2 OsRIP基因家族的染色体定位
利用Mapchart 软件分析了OsRIP基因在染色体上的位置,结果显示,在水稻的12条染色体上只有1,2,3,5,7 染色体上分布着OsRIP基因,在2 号染色体上有Os-RIP1和OsRIP4两个基因,他们之间的间隔为9.6 Mb,并不是串联重复基因。在1,3,5,7号染色体分别只含有1个OsRIP基因(图1)。

图1 OsRIP基因在染色体中的定位Figure 1 Location and distribution of OsRIP genes in chromosomes
2.3 基因结构分析
基因结构的多样性是研究基因家族功能和进化的重要依据。本研究利用MEGA-X 软件绘制水稻中Os-RIP基因家族的系统发育进化树,并将这6 个蛋白分为了3 个亚家族,其中OsRIP1,OsRIP2,OsRIP3 为第Ⅰ亚家族,OsRIP5、OsRIP6为第Ⅱ亚家族,OsRIP4为第Ⅲ亚家族(图2A)。本研究利用GSDS网站分析了基因结构进行,发现基因家族成员含有3~4 个外显子和2~3 个内含子(图2B)。与系统发育进化树相结合分析发现其内含子与外显子的分布可能与进化关系有关,第Ⅰ亚家族均含有2 个内含子,3 个外显子;而第Ⅱ、Ⅲ亚家族含有3个内含子和3个外显子(图2A,图2B)。
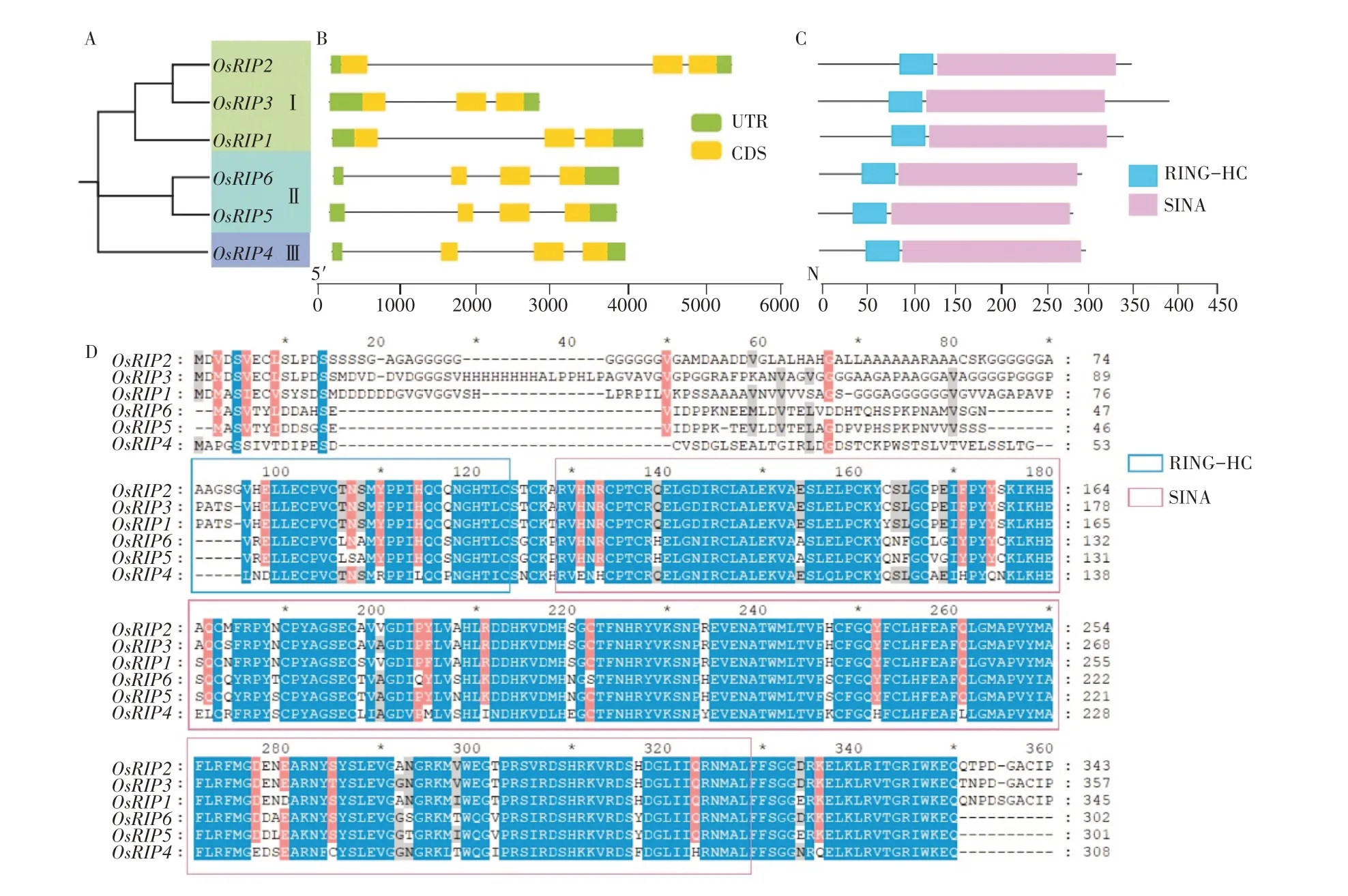
图2 OsRIP家族基因结构和保守结构域分析Figure 2 Analysis of gene structure and conserved domains of OsRIP family
不同的蛋白质结构域通常具有不同的功能。本研究利用Pfam 数据库分析了OsRIP 蛋白质的结构域,结果发现,所有OsRIP 蛋白质C 端含有SINA 结构域,N 端为RING-HC 结构域(图2C)。OsRIP 蛋白序列比对的结果表明,SINA和RING-HC结构域在水稻中是保守的(图2D)。
2.4 水稻OsRIP蛋白保守基序分析
本研究利用MEME 网站分析了OsRIP基因家族保守基序(motif),分析发现,OsRIP 蛋白质共有8个基序,含有15~50 aa 的氨基酸序列(表3)。

表3 OsRIP蛋白的基序信息Table 3 The motifs information of OsRIP proteins
为进一步揭示OsRIP蛋白进化与保守基序的关系,本研究利用TBtools绘制了OsRIP系统进化、保守基序分析图。结果表明,亲缘关系越近的蛋白,motif的数量和位置越相似。OsRIP1、OsRIP2、OsRIP3蛋白亲缘关系更近,它们均含有8 个motif,且分布位置、顺序相对一致,N 端均为Motif 8,C 端均为Motif 7(图3)。Motif 7、Motif 8为第Ⅰ亚家族OsRIP1、OsRIP2、OsRIP3 特有的。OsRIP4、OsRIP5、OsRIP6 只有6 个Motif,OsRIP5、OsRIP6 在同一亚家族,motif 的种类、数量和位置完全一致。Motif 1、Motif 2、Motif 3、Motif 4、Motif 5 及Motif 6 在OsRIP 家族所有成员中均存在。以上结果表明OsRIP基因家族Motif高度保守。
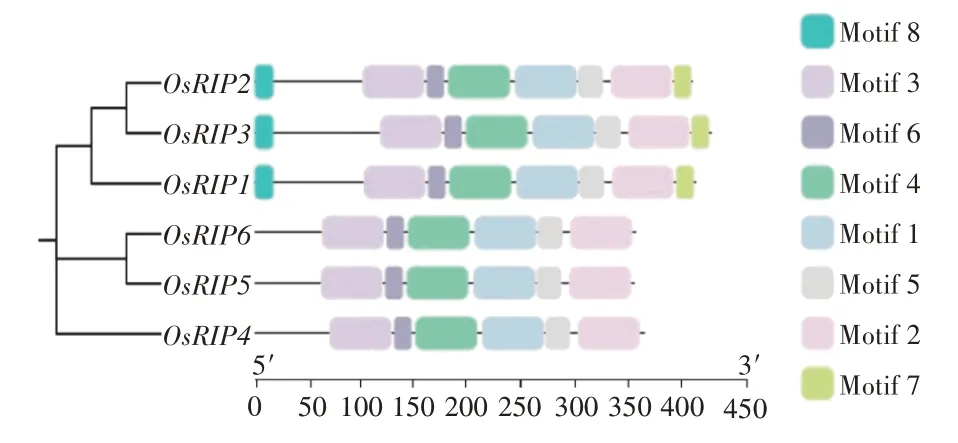
图3 OsRIP基因家族保守基序Figure 3 Conserved motif of OsRIP gene family
2.5 进化分析
本研究在Ensembl Plants 数据库下载了水稻、玉米(Zea mays)、大豆(Glycine max)、拟南芥(Arabidopsis thaliana)、大麦(Hordeum vulgare) 和小麦(Triticum aestivum) 的全基因组数据。利用保守结构域预测网站Pfam 和SMART 分析OsRIP1 结构域,下载基因的隐马尔可夫模型,在不同物种的全基因组蛋白序列数据中检索,运用BLAST对数据库进行比对,阈值设定为E<1e-5。将所有鉴定得到的RIP 候选基因序列提交到InterPro 数据库、SMART数据库以及NCBI数据库验证蛋白保守结构域,每个蛋白都包含SINA 这一保守的结构域。结果发现,RIP 蛋白在不同物种中的数量分别为:玉米中10 个,大豆中40 个,大麦中27 个,小麦中72 个,拟南芥中17 个。为了分析RIP 家族各成员之间的进化关系,利用MEGA软件构建了一个系统发育进化树。水稻中的OsRIP5、OsRIP6 分布在第Ⅱ组,OsRIP4、OsRIP1、OsRIP2、OsRIP3在第Ⅲ组,但OsRIP4与OsRIP1、OsRIP2、OsRIP3距离较远(图4)。通过统计与水稻中OsRIP基因在同一组中的其他物种中的同源基因的数量发现,小麦、玉米中同源基因的数量最多,分别为15个和8个。大麦中的同源基因有4个,而拟南芥中有2个,且距离较远。这些结果表明,水稻中OsRIP基因和玉米、小麦中的同源基因进化关系较近,而与拟南芥中的同源基因进化关系较远。
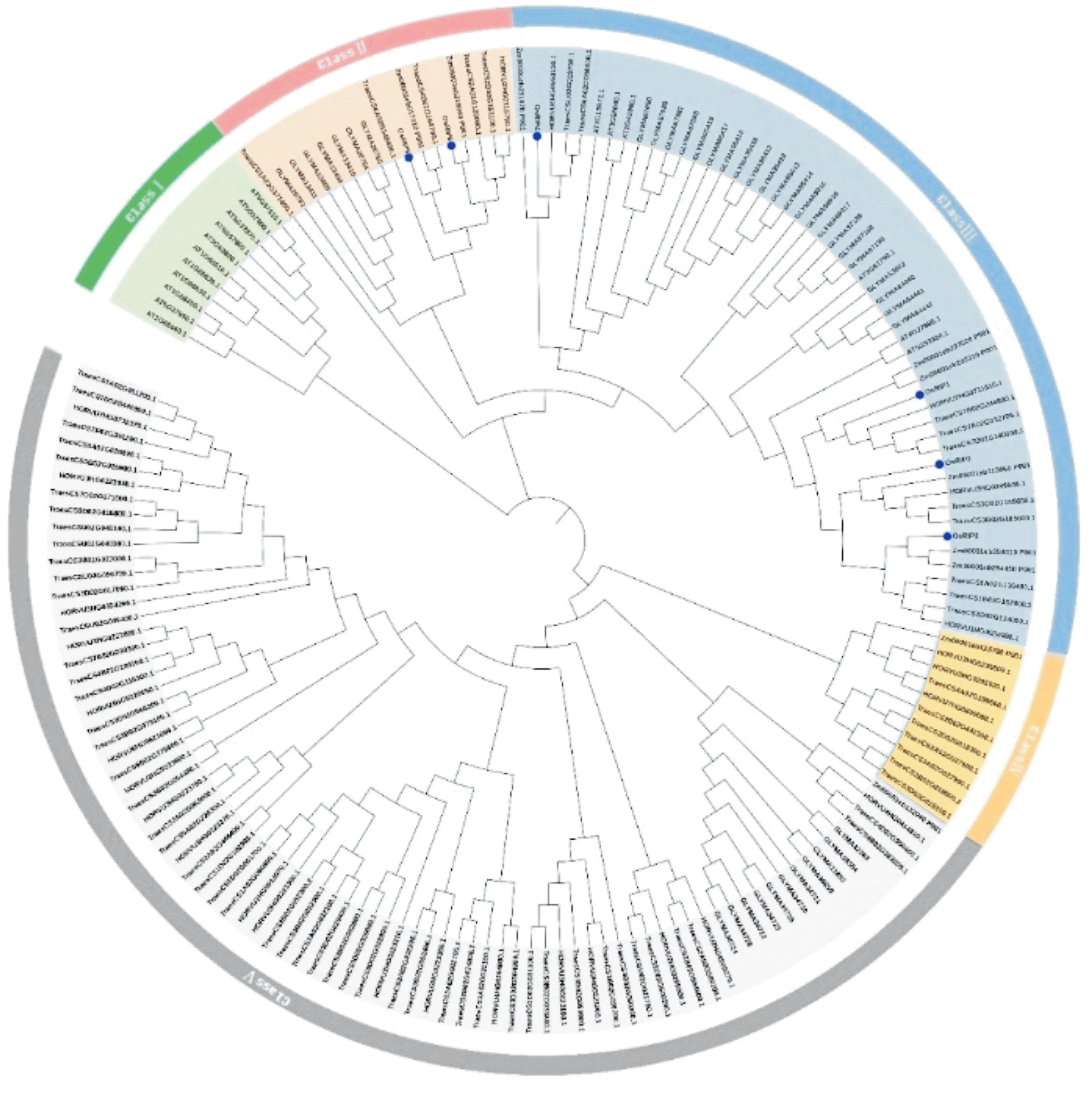
图4 RIP基因家族系统发育树Figure 4 Phylogenetic trees of RIP gene family
2.6 共线性分析
为了进一步研究RIP蛋白在水稻中系统进化机制,分析水稻和其他物种中RIP的线性关系,本研究分析了水稻中OsRIP 与玉米、小麦、大麦、大豆、拟南芥的基因共线性。结果显示,水稻中的OsRIP 蛋白与玉米之间存在最多的同源基因共线对,一共为9 个;与大麦之间存在5 个共线对,与大豆和小麦之间分别存在3 个共线对(图5)。说明玉米、大麦、小麦、大豆的RIP蛋白和水稻中的OsRIP进化关系更近。与拟南芥之间不存在共线对,也就是说RIP 蛋白在水稻和拟南芥这两个物种间进化关系较远。进一步分析发现,1 号染色体上的OsRIP3 与其他物种间都存在共线对(图5),这表明在进化过程中,RIP3在RIP家族中有重要作用。
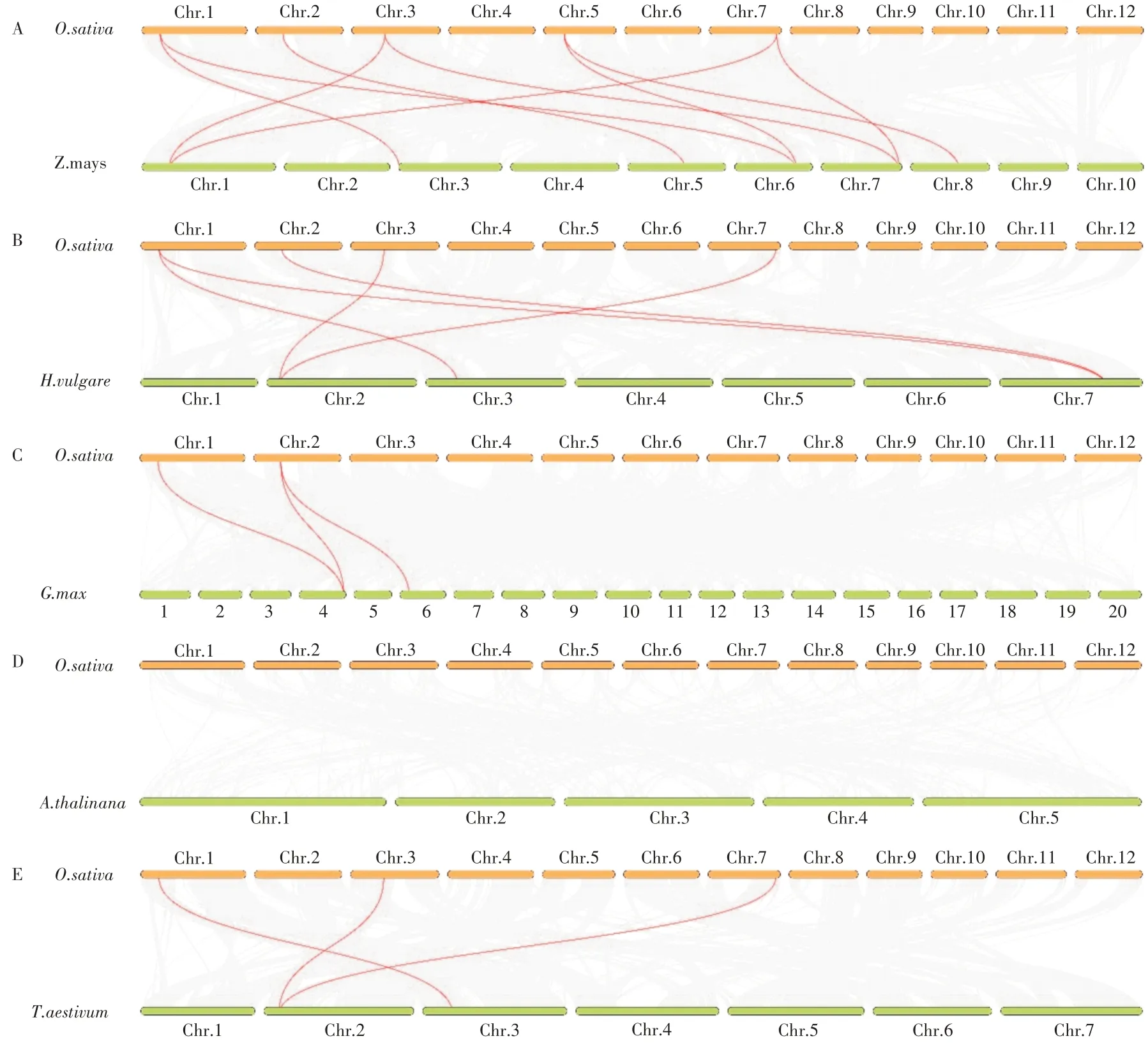
图5 水稻、玉米、大麦、小麦、大豆、拟南芥中RIP家族基因的共线性关系Figure 5 Syntenic relationships of RIP gene family in rice, maize, barley, wheat, soybean,Arabidopsis.
2.7 顺式作用元件分析
在调控基因功能的过程中,启动子活性起着至关重要的作用,为了解RIP 的调控机制,本研究分析了Os-RIP基因家族启动子区域的顺式作用元件。选取OsRIP上游2000 bp 的序列,用PlantCARE 网站分析其顺式作用元件,结果表明,在所有OsRIP基因的启动子区都发现了逆境相关的转录因子结合元件,如厌氧诱导元件、低温响应元件(图6A),这表明RIP家族可能受非生物胁迫诱导表达。此外,在OsRIP基因的启动子区还发现了光响应元件、脱落酸(ABA)响应的顺式元件ABA response element (ABRE)和CGTCA-motif 和TGACG-motif 茉莉酸甲酯(MeJA)响应的顺式元件(图6A)。
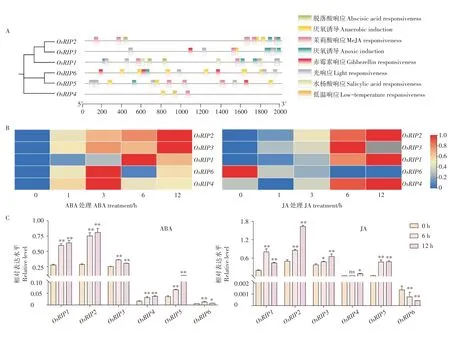
图6 OsRIP启动子顺式作用元件分析(A)及ABA、JA处理下的表达模式(B~C)Figure 6 The cis-acting elements in the promoters of OsRIP(A) and expression of OsRIP under ABAJA treatment(B~C)
为了研究OsRIP是否被ABA、MeJA 激素诱导表达,本研究以用100 μM ABA 和100 μM JA 处理水稻幼苗,进行qRT-PCR。结合RiceXPro 数据库的数据和qRT-PCR 数据,结果表明,OsRIP1-5在ABA、JA 处理6 h 和12 h 被诱导表达,OsRIP6在ABA 处理时表达水平上调,而JA 处理时表达水平下调,这表明OsRIP6被ABA 诱导表达,被JA抑制表达(图6)。
2.8 OsRIP基因表达模式分析
基因表达的特异性通常与其功能相关,为了研究OsRIP基因家族在水稻生长发育过程中的作用,本研究利用RiceXPro 数据库中的微阵列数据检测了OsRIP基因的表达模式。结果发现,OsRIP基因在所有器官和组织中都有表达,未见明显的组织特异性表达(图7)。但是,OsRIP家族不同的基因表达水平差异较大,其中Os-RIP1、OsRIP2与OsRIP3在根中表达水平较高;OsRIP1与OsRIP4和OsRIP6在茎中表达水平较高;OsRIP3、Os-RIP4和OsRIP6在花器官中有较高水平的表达。对不同时期OsRIP基因的表达水平分析发现,OsRIP1、Os-RIP2、OsRIP3、OsRIP4在播种后的48~69 d,表达水平均处于整个生育期相对高的水平,OsRIP6在播种后83 d达到表达水平最高值。由于OsRIP基因的顺式作用元件中含有光响应元件,对其昼夜的表达模式进行了分析,结果发现,在给光后10 hOsRIP基因表达水平最高,这一结果表明OsRIP基因能够被光诱导表达(图7C)。

图7 OsRIP在各个器官(A)、各个生育时期(B)及昼夜节律的表达模式(C)Figure 7 Expression of OsRIP in different organs(A), developmental stages(B) and natural photoperiod(C)
参与调控非生物胁迫的基因往往被非生物胁迫诱导表达,因此,本研究进一步检测了OsRIP基因家族在150 mmol·L-1NaCl、20%PEG、100 mmol·L-1甘露醇等胁迫下的表达模式。结果发现,OsRIP1-5均能够被盐、PEG、甘露醇不同程度的诱导表达。其中,在盐处理下,OsRIP5被诱导表达水平最高,在PEG 和甘露醇模拟干旱处理下,OsRIP4被诱导表达水平最高。而OsRIP6与其他成员的表达模式不同,只有在PEG 处理6 h 被诱导表达,在12 h时表达水平降低,而盐、甘露醇胁迫处理后均使OsRIP6的表达被抑制(图8)。
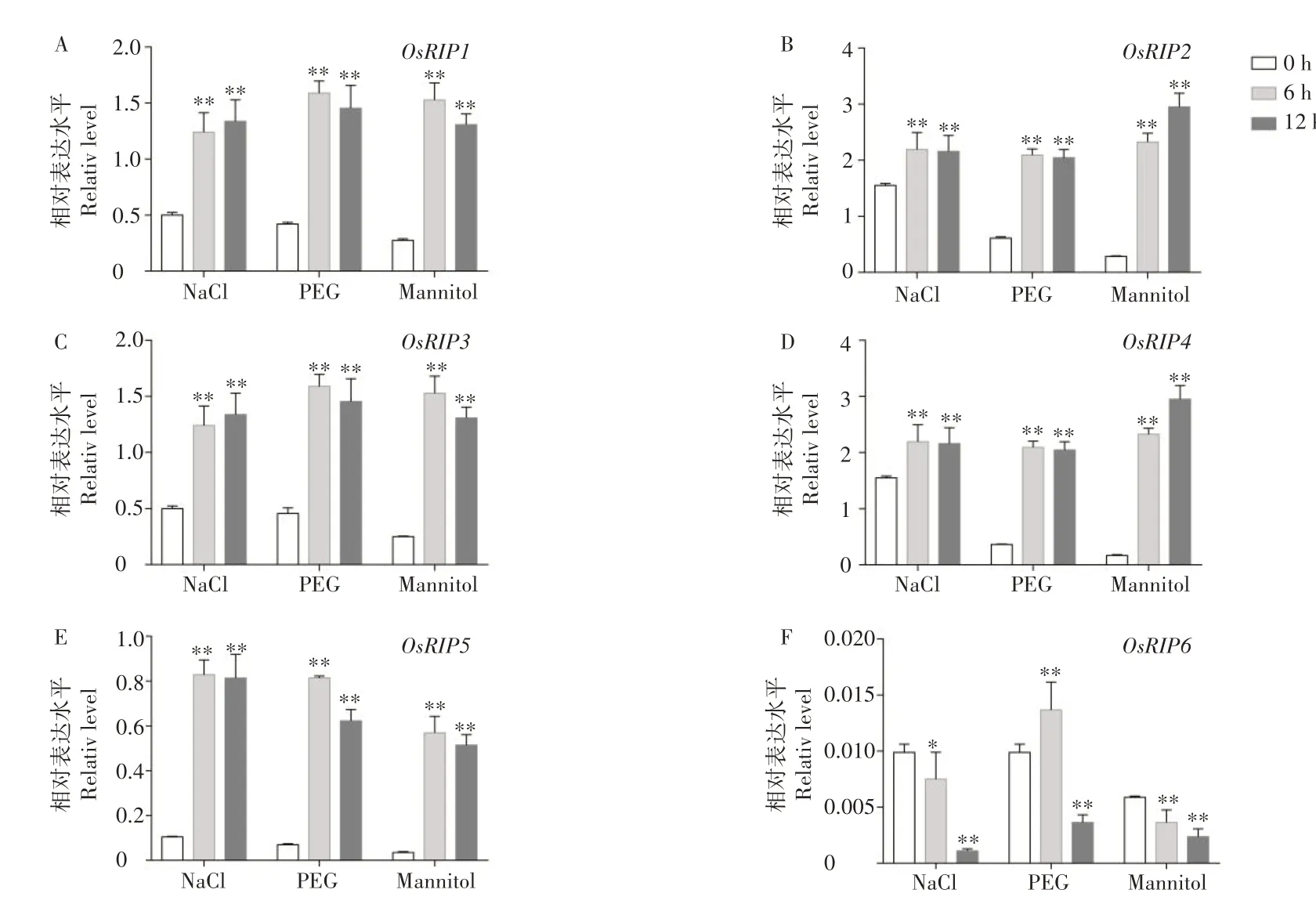
图8 OsRIP在盐、PEG、甘露醇处理下的表达模式Figure 8 The expression of OsRIP under NaCl,PEG,mannitol treatment
3 讨论与结论
3.1 水稻中的OsRIP的功能具有多样性
泛素蛋白酶系统积极参与调节植物对非生物胁迫的响应,RING型E3泛素连接酶调控植物对干旱胁迫、盐胁迫等非生物胁迫的抗性。本研究是在水稻基因组中,鉴定到6个具有SINA结构域的OsRIP基因,对其基因结构和结构域分析结果显示,该基因家族的内含子数量在2~3 个。有研究表明内含子的数量与在发育过程中环境的适应能力相关,没有内含子或内含子较少是植物进化的结果[34-35],OsRIP基因家族的内含子数量不同,表明RIP 在进化上的进程和生物学功能可能不同。其染色体定位结果表明,6 个基因分布在5 条染色体上,没有串联重复基因,这一结果表明OsRIP可能起源于不同的祖先,具有进化的多样性。有研究表明OsRIP5(OsIDS1)调控水稻的抗旱性[10],而另一个研究则证实了OsRIP1-6的六重突变体表现出严重的生长发育缺陷的表型[9]。这些结果均说明了OsRIP家族在水稻中的功能具有多样性。
3.2 光、ABA、JA及非生物胁迫诱导OsRIP的表达
基因的时空表达通常与基因功能相关[36],OsRIP家族的基因在水稻的各个时期以及各个器官均有表达,表明其基因家族可能参与调控水稻发育的多个进程。启动子顺式作用元件的分布和类型决定了基因的表达与功能[37],为了更多的了解调控OsRIP表达的机制,对启动子顺式作用元件进行了分析,结果显示ABRE、G-Box、ARE、CGTCA-motif 等响应光、激素和非生物胁迫的顺式作用元件,在OsRIP的启动子区中大多都有存在,因此猜测OsRIP可能与光响应、激素响应及胁迫的耐受性相关。结合转录组数据及定量RT-PCR结果发现OsRIP家族中ABA、JA 激素处理能够诱导OsRIP1~5基因的表达;同时OsRIP1~5也能够被盐、甘露醇、PEG 诱导表达,这一结果表明OsRIP1-5可能参与调节植物对干旱和盐胁迫的耐受性。而昼夜表达节律的结果表明该家族所有成员都能被光诱导表达,在10 h出现了表达高峰。
3.3 RIP在进化上具有保守性
水稻是重要的粮食作物,同时也是单子叶植物的主要模式植物。共线性分析的结果表明,水稻与玉米、大麦等单子叶植物之间存在更多的共线性,而与拟南芥之间没有共线性,这一结果说明RIP蛋白在单子叶植物中的进化是相对保守的。在多物种的系统发育树中,一个亚群中的基因具有相似的功能。在本研究中,利用水稻和其他物种的氨基酸序列对RIP家族进行了系统发育分析,结果表明,单子叶和双子叶的同源基因在同一组上,MEME 分析结果显示,RIP 蛋白有8个保守的motif,在系统发育树同一个亚家族的蛋白,motif越相似。以上结果表明,RIP蛋白比单子叶和双子叶植物的差异更原始。
在水稻的全基因组中鉴定到6个RIP基因,分布在5条染色体上。编码区长度在906~1086 bp之间,在水稻中,OsRIP基因家族所有成员结构域非常保守,均含有SINA 和RING 结构域。通过分析水稻与其他物种的共线性发现,与玉米存在最多的共线对,进化关系更近,与拟南芥不存在共线对,进化关系较远。水稻RIP家族基因在不同组织器官、不同时期的表达模式略有差异但均有表达;OsRIP6在JA、盐处理、甘露醇处理的条件下呈现出明显的下调表达,而OsRIP1~5在ABA、JA、盐处理、PEG处理、甘露醇处理后均呈现明显的上调表达,表明Os-RIP1~5能够积极参与调控激素和逆境胁迫的响应。

