大蒜2个中性/碱性转化酶基因AsNI的克隆与表达分析
周倩怡,黄思杰,田 洁
(1.青海大学 农林科学院,青海省高原种质资源研究与利用实验室,青海 西宁 810016;2.生态环境部 南京环境科学研究所,江苏 南京 210042)
蔗糖转化酶是植物体内蔗糖代谢的关键酶,高等植物体内的蔗糖需要通过水解为己糖才能被吸收利用[1]。蔗糖从光合活性组织(主要为成熟的叶片)运输到非光合作用的库(如花、果实、根),进而被降解成为己糖等其他衍生物,用于各种生物过程[2]。转化酶催化蔗糖的裂解将蔗糖转化为葡萄糖和果糖,在植物的初级代谢中起着关键作用[1],蔗糖转化酶不仅参与了植物的生长发育,还可以作为信号物质调节植物对非生物胁迫的抗性[3]。根据发挥作用的最适的pH,可分为酸性转化酶(Acid invertase,AI)和中性/碱性转化酶(Netural/alkaline invertase,NI)[4]。在之前的研究中,酸性转化酶的功能及调控机制已经被广泛报道,如酸性转化酶可以调控碳同化物库器官的发育和库强调节[5],由于中性/碱性转化酶活性不稳定,难以纯化,导致人们对其功能知之甚少。最近的研究中,人们发现中性/碱性转化酶在植物的生长发育过程中同样具有至关重要的作用。目前,已经鉴定并从芒果[6]、番茄[7]、石斛[8]、木薯[9]等植物中克隆出中性/碱性转化酶。Liu等[10]克隆得到3个橡胶中性/碱性转化酶基因(HbNI),其中HbNI2表现出NI基因功能,其动力特征与蔗糖浓度呈正相关。在茶树中鉴定出的中性/碱性转化酶在非生物胁迫下提高了植株的抗性[11]。
大蒜(AlliumsativumL.)是百合科葱属植物,富含多种营养和药用价值。蔗糖是大蒜糖积累的主要形式[12],其含量不仅是影响大蒜的风味品质的重要因素,也是植株抗逆性的体现[13]。乐都紫皮大蒜是青海特有品种,具有喜冷凉、产量高、品质好、商品性高的特点,但青海年平均温度低,降水量少,会影响大蒜幼苗的生长发育[14]。目前,关于中性/碱性氧化酶对提高大蒜的抗逆作用研究较少,对大蒜的中性/碱性氧化酶基因的克隆及胁迫下表达模式也鲜有报道。本研究以乐都紫皮大蒜为试材,利用TA克隆法对AsNI进行克隆并进行生物学信息分析,分析这2个基因在不同胁迫处理下不同组织的表达模式,为探明逆境胁迫下大蒜AsNI基因的分子响应机制提供了一定的理论基础。
1 材料和方法
1.1 试验材料
本试验以乐都紫皮大蒜为材料,播种于栽培基质配比为草炭∶珍珠岩∶蛭石=2∶1∶1的盆钵中,置于光照培养箱中进行培养。设置培养条件昼/夜:25 ℃/15 ℃,14 h/10 h;相对湿度70%;光照强度:300 μmol/(m2·s)。培养至大蒜苗高为12 cm左右时,进行分组培养处理:
正常培养(CK):培养条件不变,相对湿度保持70%,定期浇水保持土壤湿润;干旱胁迫处理(DT):通过停止浇水进行人工控水[15],保持空气相对湿度25%~30%,土壤湿度为45%左右,光周期及光照强度不变;低温胁迫处理(CT): 将大蒜幼苗放置在昼/夜:4 ℃/4 ℃,14 h/10 h的培养条件下培养,相对湿度保持70%,定期浇水保持土壤湿润,光周期及光照强度不变。
分别取对照、低温胁迫处理(4,12,24 h)、干旱胁迫处理(1,6,9 d)大蒜根、假茎、鳞茎、叶片用于RNA的提取和cDNA的合成,上述采集样品立即用液氮速冻,于-80 ℃保存。
1.2 试验方法
1.2.1 总RNA的提取及cDNA的合成 将采集的胁迫处理不同时期的大蒜组织材料的总RNA的提取利用TRNzol Universal(TRNzol Universal,DP424)总RNA提取试剂进行。采用1%琼脂糖凝胶和TGem微量分光光度计(OSE-260,天根生化科技有限公司)分别检测RNA完整性和质量。cDNA的合成按照HonorTMⅡ 1st Strand cDNA Synthesis SuperMix for qPCR试剂盒进行。合成的cDNA放在-20 ℃冰箱进行保存备用。
1.2.2 大蒜AsNI基因的克隆 从大蒜基因组数据检索得到2条中性/碱性转化酶基因序列,据此设计克隆引物AsNI1-F(5′-ATGGCTTTAAGGGCTTCGCTCAC-3′)、AsNI1-R(5′-T CTAACCTGGCCAAGATCCTCCAT-3′)和AsNI2-F(5′-ATGGCTTTAAGGGCTTCGCTCA-3′)、AsNI2-F(5′-CTAACCTGGCCAAGATCCTCCA-3′)。PCR扩增模板为乐都紫皮大蒜假茎cDNA。PCR扩增条件:94 ℃预变性5 min;94 ℃变性30 s,56 ℃退火30 s,72 ℃延伸2 min 30 s,进行35个循环;72 ℃延伸10 min。1.0%琼脂糖凝胶电泳回收产物,连接pMD19-T载体,转化大肠杆菌感受态,选取单克隆进行菌液PCR验证,阳性克隆送至北京六合华大基因科技有限公司测序。
1.2.3 序列分析 蛋白质的理化性质通过Ex PaSy(https://web.expasy.org/protparam/)在线网站进行预测分析,跨膜结构域利用TMHMM(https://services.healthtech.dtu.dk/service.php?TMHMM-2.0)在线网站分析。亚细胞定位和信号肽预测利用Softberry(http://www.softberry.com/berry.phtml?topic=protcomppl&group=programs&subgroup=proloc)和SignalP 3.0(https://services.healthtech.dtu.dk/service.php?SignalP-3.0)进行。利用SOPMA(https://npsa-prabi.ibcp.fr/cgi-bin/secpred_sopma.pl)和Phyre 2(http://www.sbg.bio.ic.ac.uk/phyre2/html/page.cgi?id=index)分别进行蛋白质二、三级结构的预测。利用NetPhos 3.1 Server(https://services.healthtech.dtu.dk/service.php?NetPhos-3.1)和NetNGlyc-1.0 Server(https://services.healthtech.dtu.dk/service.php?NetNGlyc-1.0)预测AsNI蛋白的磷酸化位点和糖基化位点。利用MEME(https://meme-suite.org/meme/)在线分析蛋白基序的保守性;使用 STRING网站(https://cn.string-db.org/)预测蛋白的相互作用网络。在NCBI数据库中获得拟南芥等其他植物的NI序列,使用MEGA 6.0对系统进化树进行测试和编辑,生成报告图形。利用PlantCARE(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)预测AsNI启动子序列的顺式作用元件。利用PlantRegMap网站(http://plantregmap.gao-lab.org/binding_site_prediction.php)预测转录结合位点。
1.2.4 实时荧光定量PCR 根据荧光定量试剂盒(Unique AptamerTMQpcr SYBR®Green Master Mix)在Roche LightCycler®480 ℃上进行实时荧光定量PCR。参考Liu等[16]的研究,以大蒜CYP基因为内参基因,其引物序列为CYP-F(5′-AAGGACGAGAACTTCATC-3′)、CYP-R(5′-TCAATATCTCTCACCACTTC-3′);根据克隆得到序列设计引物AsNI1-F(5′-GGCGGTTATTTTGTTGGA-3′)、AsNI1-R(5′-ATAGATTGCTCGGGTGTA-3′)、AsNI2-F(5′-ATCTCCAGCCTGCTCACA-3′)、AsNI2-R(5′-TCCCATTTCTCTTCCATT-3′)。
1.3 数据分析
采用2-ΔΔCt计算目的基因的相对表达水平[17]。显著性差异采用SPSS的Duncan法(P<0.05)进行分析。图表利用Origin 2021软件进行绘制。
2 结果与分析
2.1 大蒜转化酶基因AsNI的克隆
以乐都紫皮大蒜的cDNA为模板,使用特异性引物AsNI1-F、AsNI1-R和AsNI2-F、AsNI2-R进行 PCR扩增,得到一条长度约为500 bp和一条长度约为1 200 bp的目的条带(图1),片段大小与预期一致。测序结果表明,AsNI1基因开放阅读框(Open Reading Frame,ORF)全长522 bp,编码173个氨基酸(图2),AsNI2基因ORF全长1 203 bp,编码400个氨基酸(图3)。AsNI1基因与AsNI2基因ORF序列的相似性为26.25%。

*.终止密码子。图3同。

图3 大蒜AsNI2基因的核苷酸序列及其编码的氨基酸序列
2.2 大蒜AsNI基因氨基酸序列分析
2.2.1 大蒜AsNI蛋白的理化性质分析及亚细胞定位预测 利用ExPaSy-ProtParam等对AsNI1与AsNI2基因编码的蛋白质的理化性质进行分析(表1)。结果表明,2个蛋白均为亲水性蛋白,均不稳定,AsNI1有18个磷酸化位点,AsNI2含有41个磷酸化位点;AsNI1未检测到糖基化位点,AsNI2含有一个糖基化位点;亚细胞预测定位表明2个蛋白均位于细胞质。

表1 大蒜AsNI的蛋白质理化性质
2.2.2 大蒜AsNI的蛋白结构分析 利用SOPMA对蛋白进行蛋白质二级结构预测(图4,5),结果表明,AsNI1二级结构包含的α-螺旋、延伸结构、β-转角和无规则卷曲分别为43.00%,15.00%,6.25%和35.75%;AsNI2则分别包含45.09%,12.27%,2.31%和39.88%。利用Phyre 2软件进行同源建模,并通过PyMOL软件分析蛋白三级结构,其结果显示AsNI1蛋白的三维结构由9个α螺旋和2个β折叠所构成;AsNI2蛋白的三维结构由15个α螺旋和4个β折叠所构成。

蓝色.α螺旋;绿色.β转角;黄色.无规则卷曲;红色.延伸结构。图5同。

图5 AsNI2蛋白二级(A)、三级(B)结构预测
2.2.3 大蒜AsNI蛋白的保守结构域分析 利用MEME在线分析AsNI蛋白基序的保守性(图6),结果表明,AsNI1和AsNI2均含有10个保守基序列,但结构相差较大,其中二者的3,9,10号基序相同且位置一致,AsNI1的4号与AsNI2的6号、5号与7号、7号与8号基序相同,但位置存在差异。通过NCBI网站的BlastP的程序对AsNI蛋白进行功能域分析,结果表明(图7),AsNI1蛋白在1—172氨基酸位点间有Glyco_hydro_100结构域,AsNI2蛋白在1—399氨基酸位点存在Glyco_hydro_100结构域,此结构域是植物中性/碱性转化酶的保守结构域。

图6 AsNI蛋白的保守基序分析
2.2.4 大蒜AsNI的多序列对比和系统进化分析 利用NCBI BlastP对AsNI与其他植物的NI氨基酸序列进行同源性对比,选取相似度较高的几个物种进行多重比较分析(图8),AsNI1与AsNI2氨基酸序列的相似性为25.75%,AsNI1与拟南芥的相似性分别为86.71%;AsNI2与铁皮石斛、野生稻、姜、玉米、高粱、黑麦草和短柄草的相似性分别为84.50%,85.50%,84.50%,85.25%,85.50%,86.00%和85.25%;表明大蒜与其他物种的AsNI的氨基酸序列具有较高的同源性,两端与其他蛋白没有相似性,且N端比其他蛋白少约110个氨基酸残基。
利用MEGA 6.0软件中的Neighbor-Joining法将大蒜与其他植物的NI构建同源进化树(图9)。结果表明,AsNI1与拟南芥属于同一分支,在进化关系上最为接近,AsNI2与铁皮石斛、野生稻和姜同属一个大分支,亲缘关系较为接近,AsNI1与AsNI2属于不同分支,亲缘关系较远。
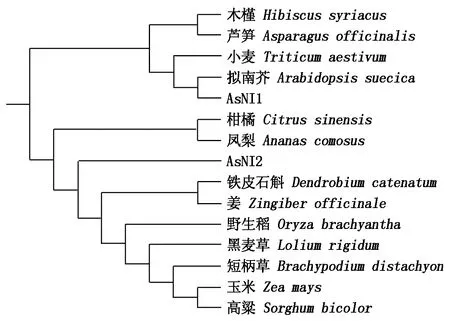
图9 大蒜与其他作物NI氨基酸序列的系统进化树
2.3 大蒜AsNI的蛋白互作网络预测
利用STRING网站,以拟南芥为模板构建大蒜AsNI的蛋白互作网络(图10)。结果表明,AsNI1和AsNI2均与参与糖代谢的细胞壁转化酶作用,但AsNI2与3种细胞壁转化酶作用,多于AsNI1。此外,AsNI1也和参与氨基酸磷酸化的丝氨酸/苏氨酸蛋白激酶(AT1G29720)、聚腺苷酸结合蛋白(PAB2、短柄草(XP_003558048.1∶222-620)、凤梨(OAY70851.1)、柑橘(KAH9692069.1)、高粱(XP_002465359.1∶227-625)、姜(XP_042399050.1∶235-634)、芦笋(XP_020247438.1)、木槿(XP_039002564.1)、拟南芥(KAG7589826.1∶297-469)、铁皮石斛(AVA07409.1∶212-610)、小麦(XP_044375374.1)、野生稻(XP_006645848.2∶240-637)、黑麦草(XP_047072902.1∶223-621)、玉米(XP_008655058.1∶226-624)。
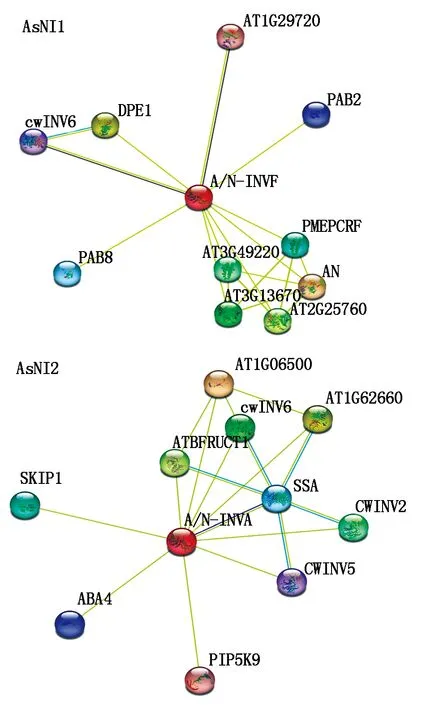
图10 大蒜AsNI1和AsNI2的蛋白互作网络
PAB8)和4-α-葡聚糖转移酶(DEP1)相互作用。AsNI2与蔗糖合酶(SSA)、参与脱落酸形成的脱落酸缺乏酶(ABA4),糖介导的根系发育调控过程中的1-磷脂酰肌醇-4-磷酸5-激酶(PIP5K9)也相互作用。
2.4 大蒜AsNI基因启动子序列分析及转录因子结合位点预测
利用PlantCARE在线网站对2个AsNI基因进行启动子元件预测分析,结果表明AsNI基因不仅包含典型的CAAT-box和TATA-box,还包含其他重要元件,部分重要元件见表2。AsNI1和AsNI2基因启动子均具有多种逆境响应元件,但调控元件的种类和数量存在差异,如AsNI1和AsNI2的启动子序列都含有干旱诱导的MYC结合位点,但数量上AsNI2比AsNI1多1个;AsNI2含有3种不同的逆境响应元件,而AsNI1只含有1种,且数量上少于AsNI2;AsNI1含有7种激素调节元件,AsNI2含有2种,但彼此之间的元件类型不同。
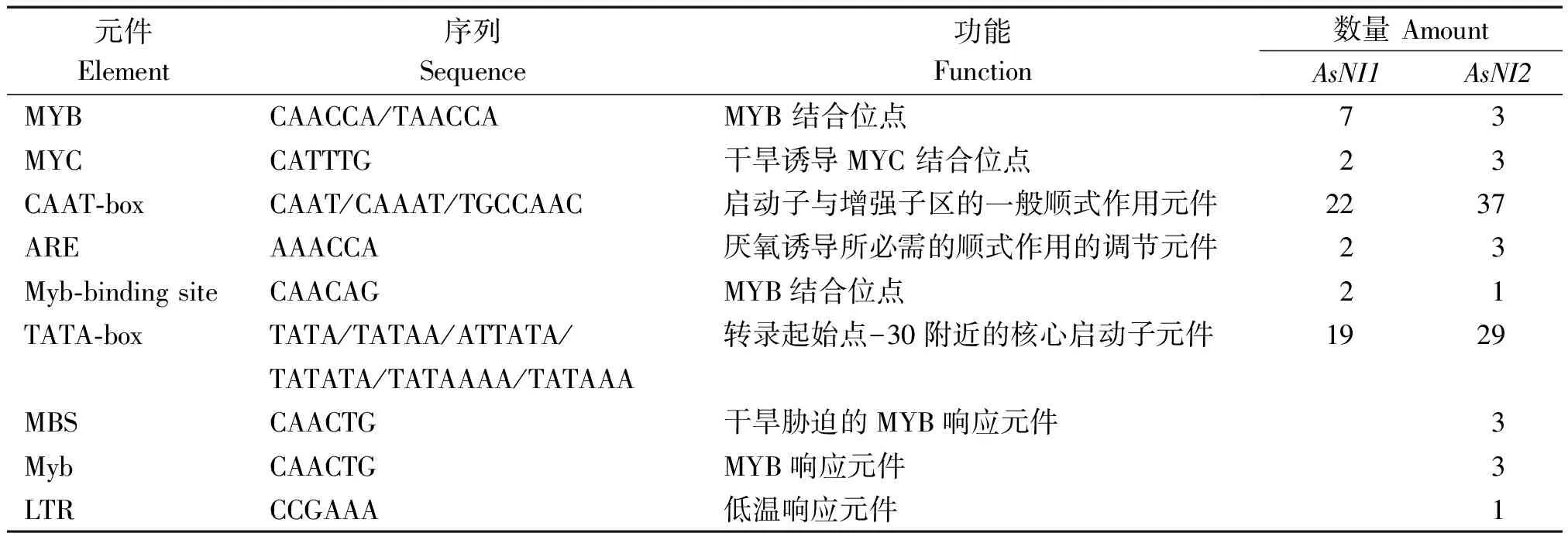
表2 AsNI基因启动子部分顺式作用元件
通过PlantRegMap在线网站以拟南芥为模板预测AsNI启动子的转录因子结合位点。结果表明,AsNI1和AsNI2分别可以与31,28个转录因子家族具有结合位点,与顺式作用元件预测结果相符,AsNI1和AsNI2均可以参与植物逆境调控的转录因子结合,如MYB、NAC、ERF等。其中,AsNI1与MYB家族的结合位点出现频率最高,出现了28次。预测发现,AsNI1和AsNI2基因启动子的正负链均分布有上述转录因子,推测该启动子具有丰富的调控机制,控制基因的表达,并通过调节不同转录因子的转录起始参与行使不同的功能。
2.5 大蒜AsNI基因在不同组织和逆境条件下的表达分析
通过qRT-PCR对AsNI在大蒜不同部位的表达模式进行分析(图11),结果表明,正常生长条件下,AsNI在大蒜的4个组织(根、假茎、鳞茎和叶片)中均有表达,其中AsNI1基因在根中表达量最高,分别比鳞茎、假茎和叶片高4.17,5.00,14.29倍,而AsNI2基因在鳞茎中表达量最高,分别比根、假茎和叶片高2.29,5.09,1.89倍。

不同小写字母表示不同处理间差异显著(P<0.05)。
低温胁迫下,大蒜不同组织AsNI1和AsNI2的表达模式差异显著(图12)。与胁迫前(CK 0 h)相比,低温处理除了使根系AsNI1的表达量在胁迫12 h显著提高以外,其他3个组织在整个处理期间均显著下调。与之不同,低温处理能够显著提高各部位AsNI2的表达量。同时,低温下根、假茎、鳞茎、叶AsNI2的最大表达量分别比对照高4.75,2.18,3.16,14.51倍,即叶片AsNI2对低温胁迫的响应较其他组织更强。综上可得,低温胁迫对AsNI2的诱导强于AsNI1,其中以叶片AsNI2对低温逆境的响应更为明显。

图12 AsNI基因在低温胁迫处理下不同组织相对表达水平
干旱胁迫对大蒜各组织AsNI1和AsNI2表达的影响具有显著差异(图13)。干旱处理下,AsNI1除了在假茎和鳞茎于胁迫6~9 d显著高于对照以外,其他组织表达并未受到显著上调表达。而对于干旱胁迫的AsNI2,其表达量在4个组织中均出现1个或多个显著高于对照的时间点。其中,根、假茎、鳞茎、叶AsNI2的表达量分别于干旱处理1,1,6,1 d达到峰值,较对照高出5.01,1.06,1.41,1.10倍,即根系AsNI2对干旱胁迫的响应明显强于其他组织。由此可知,与AsNI1相比,干旱胁迫对AsNI2的诱导更明显;与其他组织相比,根系AsNI2对干旱逆境的响应更强。

图13 AsNI基因在干旱胁迫处理下不同组织相对表达水平
3 结论与讨论
植物在生长发育的过程中常常会遭受低温、干旱、盐碱等非生物胁迫,这会影响植物的正常生长发育以及农作物的生产力,因此,植物进化出多种调节机制来抵抗非生物胁迫[18]。研究表明,可溶性糖具有清除逆境胁迫下植物体内产生的ROS的功能[10],蔗糖作为主要的碳水化合物来源,运输到库器官后,在酶的作用下降解为己糖,供应库器官的生长,如果实、根和块茎[19],蔗糖降解产生的己糖是合成纤维素、蛋白质、果聚糖、淀粉和抗氧化物质所必需的[3]。转化酶介导的蔗糖代谢不仅参与了植物的正常生长,还响应多种非生物胁迫[20]。玉米在中等水分胁迫下,其液泡转化酶的活性会增强,表达量升高并促进己糖的积累[21]。Qi等[22]研究发现,NaCl和PEG处理下,甘薯根和叶中的中性/碱性转化酶表达量增加。因此,转化酶是植物生长发育和逆境响应的关键酶。本研究利用TA克隆法从大蒜基因组克隆了2个中性/碱性转化酶基因,分别编码173,400个氨基酸,2个基因的ORF和氨基酸序列差异较大,相似性分别为26.25%和25.75%。多重序列对比发现,AsNI蛋白两端与其他蛋白没有相似性,且N端比其他蛋白少约110个氨基酸残基;AsNI1与拟南芥的相似性为86.71%,AsNI2与铁皮石斛等物种的相似度为85.25%~86.00%,表明不同物种的中性/碱性转化酶序列具有较高保守性。与本研究结果类似,Shen等[23]克隆了7个辣椒中性/碱性转化酶基因,发现各基因编码的蛋白质的氨基酸残基数存在较大差异,从286—655个氨基酸残基不等;钱文俊等[24]在分析茶树中性/碱性转化酶基因CsINV10的氨基酸序列时发现,CsINV10与其他物种的在氨基酸序列在N端和C端的同源性较低,但在中间的450个氨基酸具有较高的保守性。通过保守结构域分析发现这2个基因均含有糖苷水解酶100(GH100)家族典型的Glyco_hydro_100结构域;亚细胞定位预测2个基因均位于细胞质中,因此,AsNI1和AsNI2较大可能是细胞质中的中性/碱性转化酶基因。蛋白质理化性质结果表明,AsNI2含有糖基化位点,而AsNI1未检测到糖基化位点。糖基化对蛋白质起重要的修饰作用,可以通过糖基化调节蛋白质功能,因此,推测2个基因的功能有所差异。通过构建蛋白互作网络预测发现,2个基因可能参与不同的生化过程,AsNI1可能主要参与蛋白质的磷酸化过程,而AsNI2可能参与糖代谢过程。
植物调控抗逆相关基因的表达依赖基因启动子中特定的顺式作用元件[25]。王运林等[9]克隆了木薯中性/碱性转化酶基因MeNINV1的启动子序列,发现其序列上存在激素相关响应顺式作用元件,进而利用荧光定量证明外源激素调控MeNINV1基因的表达。杨梅等[26]鉴定并分离出水稻中一个受干旱胁迫诱导的基因Oshox24,实时荧光定量结果和GUS活性检测均表明该基因强烈受到干旱胁迫的诱导并上调表达,其启动子Oshox24P是干旱诱导型启动子,积极响应植物的抗旱应答。通过对AsNI基因启动子序列的顺式作用元件预测分析,发现2条基因的启动子区域除了包含 TATA-box 和 CAAT-box 等核心元件外,还包含逆境响应元件等。核心启动元件TATA-box是决定基因转录起始的选择,含有TATA-box的基因对转录起始较为敏感,CAAT-box调节转录起始的频率,可以增强启动子的强度[27],因此,可以推测AsNI的启动子具有正常的转录功能;此外AsNI1和AsNI2含有不同种类及数量的逆境响应元件,推测AsNI1和AsNI2具有不同的逆境调控机制。启动子上的转录因子结合位点是转录开始的关键部位,研究表明,转录因子结合位点具有保守性,相似的序列就可以结合同样的转录因子[28-29]。预测结果表明,AsNI1和AsNI2的启动子有多种转录因子结合位点,表明AsNI1和AsNI2可行使不同的基因功能。
在胁迫下,植物通过提高转化酶基因的表达水平,促进蔗糖的水解,以此来缓解逆境损伤[30]。在本研究中,AsNI1和AsNI2在不同胁迫下具有不同的表达模式。低温胁迫下,根系中AsNI1的表达量显著提高,AsNI2在叶片中的响应更为明显;干旱胁迫下,AsNI1在假茎和鳞茎中表达量显著增加,AsNI2则以根系的响应最为强烈,且在2个胁迫下,AsNI2基因的响应程度均强于AsNI1。低温破坏细胞膜的稳定性[31],植物光合作用的主要部位在膜结构上,低温会降低光合相关酶的活性,抑制光合作用[32]。因此,低温胁迫下叶片通过上调基因的表达以缓解逆境损伤。植物会通过合成渗透物质来抵抗胁迫,中性/碱性转化酶通过将蔗糖分解为葡萄糖和果糖,参与调节渗透平衡,减轻逆境胁迫对植物的损伤。根系是植物应对干旱胁迫的关键器官[33],当植株遭受干旱胁迫时,根部加快可溶性糖的合成,并运输至库器官,弥补光合产量的降低[34],缓解逆境损伤。
本研究利用生物信息学的方法分析了2条AsNI基因的序列,综合对比序列特征发现AsNI1与AsNI2在编码氨基酸个数、糖基化位点个数、蛋白互作网络、启动子顺式作用元件类型及转录因子结合位点等方面存在差异,因此,推测二者可能在逆境响应中表现出不同的功能;通过分析不同逆境胁迫下2个基因表达的变化趋势,发现低温和干旱处理下AsNI2的响应程度均强于AsNI1。其中,低温胁迫主要诱导叶片AsNI2的表达,干旱胁迫以诱导根系AsNI2的表达为主。通过本研究结果表明,AsNI参与大蒜的逆境响应,为解析AsNI响应大蒜逆境胁迫的分子机制提供了理论基础。

