坛紫菜洞头传统养殖品系和新品系遗传多样性分析
吴晓雯 王铁杆 刘颖 张鹏
摘 要:为探究坛紫菜(Neoporphyra haitanensis)温州洞头传统养殖品系与两个新品系(SW-81,HR-5)的遗传差异,利用CO I和rbcL基因片段对这3个品系共60株个体进行了遗传多样性分析研究。经过比对分析,得到398 bp的CO I基因序列和488 bp的rbcL基因序列等长同源序列,联合分析长度为886 bp,所有获得的序列中T、C、A、G 4种碱基的相对含量分别为35.100%、14.220%、34.320%和16.360%;变异位点共6个,其中单变异位点5个,简约信息位点1个;3个品系的核苷酸多样性为0.000 30±0.001,单倍型多样性为0.249±0.074;60株个体定义了6个单倍型(Hap1~Hap6),其中Hap3是核心单倍型,占全部个体的88.600%;单倍型构建的NJ系统发育树表明,各品系存在基因交流,尚未发生遗传分化。研究结果表明,坛紫菜新品系的遗传多样性较低,因此应加大种质保护和选育范围,防止种质退化。建议在坛紫菜育苗生产过程中采用多个品系联合应用,以适应多变的海洋环境。
关键词:坛紫菜;新品系;CO I基因;rbcL基因;遗传多样性
坛紫菜(Neoporphyra haitanensis)隶属于红藻门(Rhodophyta)、红藻纲(Rhodophyceae)、红毛菜科(Bangiaceae)、紫菜属(Neoporphyra)[1],其味美价廉,富含多种人体必需氨基酸、维生素和微量元素,是我国主要的经济藻类,在浙江、福建等地大规模养殖。根据《2021中国渔业统计年鉴》[2],浙江省坛紫菜养殖面积已达1.5万hm2,鲜紫菜产量在7.5万t以上。坛紫菜产业的发展对浙江省水產品发展、近海岸环境改善和东海渔场振兴有着积极的作用。
在育种技术的不断进步与选育工作的逐步推进下,坛紫菜育种工作已卓有成效。SW-81是经过紫外人工诱变的野生型突变坛紫菜新品种,具有产量高、品质好、耐高温且壳孢子放散量大等优点。HR-5是红色杂交品系,具有叶状体薄、生长快等优良特性。刘颖等[3]通过扩增片段长度多态性(AFLP)分子标记技术对浙江省台州市玉环市箬笠礁海域中的坛紫菜养殖品系与新品系“浙南3号”进行了比对,发现不同品系间存在一定的基因交流。测序技术的流行和成熟使提供更为准确的核苷酸序列结果成为可能,内转录间隔区ITS-5.8S rDNA和RUBISCO技术[4-6]均在藻类研究中有了应用。线粒体和叶绿体分子标记技术等也得到了应用。线粒体的细胞色素氧化酶亚基I基因(CO I基因)变异较大,是研究种群遗传变异较好的标记;光合作用第一关键酶——核酮糖1,5-二磷酸羧化酶/加氧酶(rubisco)的大亚基由叶绿体基因组编码,简称rbcL基因。CO I基因和rbcL基因经常被作为分子条形码标记,用于评估遗传多样性,揭示隐藏物种的多样性或揭示藻类的种群结构[7-8]。
本研究采用CO I和rbcL基因对坛紫菜温州洞头地区传统养殖品系和两个新品系的遗传多样性进行分析,旨在了解目前该地区坛紫菜传统养殖品系与两个新品系的遗传多样性差异,并评估坛紫菜新品系的遗传背景,为选育更优的新品系和育苗生产提供理论依据。
1 材料和方法
1.1 试验材料
于2020年分别采集温州洞头坛紫菜传统养殖品系、新品系SW-81和HR-5的新鲜叶状体17、18、25株,洗净晾干后,密封保存于-20 ℃冰箱,备用。
1.2 试验方法
1.2.1 坛紫菜DNA提取
用双蒸水再次冲洗试验用坛紫菜叶状体后,用吸水纸吸干,按照操作说明用DN 14-植物基因组DNA快速提取试剂盒(北京艾德莱生物科技有限公司,DN 1401)提取总DNA,经1%琼脂糖凝胶电泳检测DNA质量,再利用微量分光光度计(Nano-400)测定DNA的质量浓度,于-20 ℃分管保存。
1.2.2 PCR扩增和测序
本试验引物序列参考NCBI相关序列:CO I-F:GATGCTGTACCCGGTAGAT,CO I-R:GTTGTAATTGTTTAGCTGTTTTT,rbcL-F:GACTCCAACAGCAAACATCTAG,rbcL-R:TTAATAYCTAGCTCCTTCAGGC。引物由生工生物工程(上海)有限公司合成。
本试验PCR扩增反应选用25 μL反应体系,包括:超纯水9.5 μL,上、下游引物各1 μL,DNA模板1 μL,2×A8 FastHiFi PCR Master(北京艾德莱生物科技有限公司)12.5 μL。PCR反应程序为:95 ℃预变性3 min;95 ℃变性10 s,CO I基因引物49 ℃、rbcL基因引物52.5 ℃,退火15 s,72 ℃延伸30 s,35个循环;最后72 ℃再延伸5 min。每次反应均设置阴性对照,以检验是否有污染。PCR产物经质量分数为1%的琼脂糖凝胶电泳检测,确定PCR产物为目的基因条带后,送至杭州擎科生物科技有限公司进行纯化及双向测序,以确保序列的可靠性。
1.3 数据处理
将获得的原始序列放在DNAstar 5.0软件包中的Seqman软件中进行拼接,并结合峰图进行人工校对,确保每个位点都准确无误。两组数据(CO I和rbcL)经过NCBI的BLAST验证,所得的序列均为目的基因。采用DnaSP 5.0[9]分析多态位点数、单倍型多样性和核苷酸多样性,统计单倍型[10]。采用MEGA 6.0[11]分析碱基组成,计算简约信息位点数、单突变位点,根据K2P两参数模型(Kimura-2-Parameter,K2P)[12]计算群体间的遗传距离以及单倍型遗传距离,采用邻接法(neighbor-joining,NJ)构建系统发育树。系统发育树各个节点支持度计算采用bootstrap方法,进行1 000次自检。利用PopART 1.7[13]构建单倍型网络图,探讨本试验研究的坛紫菜单倍型谱系关系。利用DnaSP 5.0中的Tajimas D值推断物种基因序列的变异类型[14]。使用Arlequin 3.5[15]进行分子方差分析(analysis of molecular variance,AMOVA)。
2 结果
2.1 碱基组成
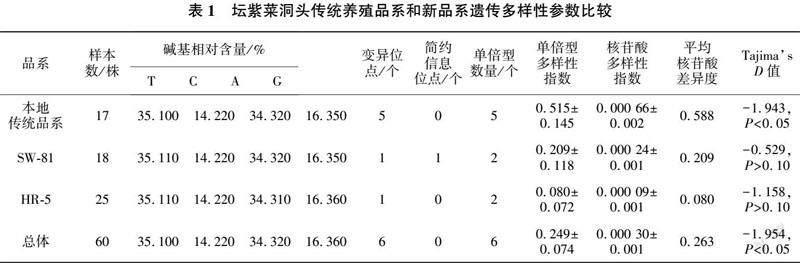
本试验共获得120条序列,经过比对去除测序起始两端的部分序列,得到398 bp的CO I基因序列和488 bp的rbcL基因序列等长同源序列。3个群体CO I基因序列的T、C、A、G 4种碱基的相对含量分别为37.940%、13.570%、33.920%、14.570%,C+G含量为28.140%,符合线粒体组成特征。上传至NCBI数据库,获得的序列号为ON533891~ON533895。rbcL基因的T、C、A、G 4种碱基的相对含量分别为32.790%、14.760%、34.640%、17.810%,上传至NCBI数据库,获得的序列号为ON533896~ON533898。联合分析长度为886 bp,所有获得的序列中,T、C、A、G 4种碱基的相对含量分别为35.100%、14.220%、34.320%、16.360%(见表1)。
2.2 遗传多样性分析
CO I基因序列有394个保守位点,4个变异位点,其中单变异位点4个,简约信息位点0个,定义了5个单倍型,单倍型多样指数为0.130±0.059,核苷酸多样性指数为0.000 34±0.002,平均核苷酸差异度为0.133,Tajimas D值为-1.844(P<0.05)。
rbcL基因序列保守位点485个,变异位点2个,其中单变异位点1个,简约信息位点1个,定义了3个单倍型,单倍型多样性指数为0.128±0.057,核苷酸多样性指数为0.000 27±0.001,平均核苷酸差异度为0.130,Tajimas D值为-1.191(P>0.10)。
CO I和rbcL基因联合遗传多样性分析结果见表1。由表1可见,坛紫菜洞头传统养殖品系的单倍型多样性和核苷酸多样性均高于两个新品系,新品系HR-5的单倍型多样性和核苷酸多样性均最低。
2.3 遗传距离分析
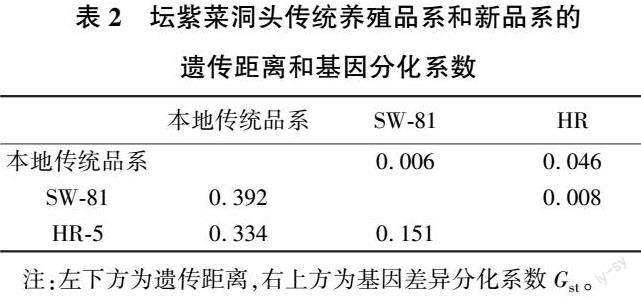
坛紫菜洞头传统养殖品系和两个新品系的遗传距离见表2。由表2可见,坛紫菜洞头传统养殖品系与新品系SW-81的遗传距离大于与新品系HR-5的遗传距离,SW-81和HR-5的遗传距离较小。
2.4 群体遗传结构
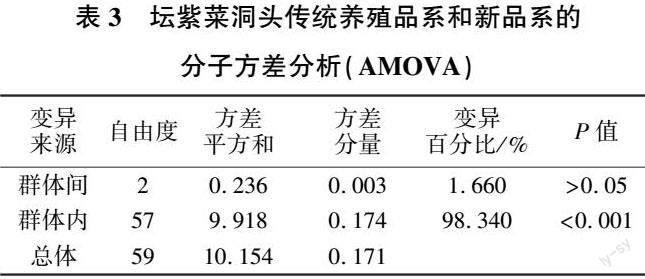
分子方差分析(AMOVA)的结果见表3。由表3可见,坛紫菜洞头传统养殖品系和新品系的遗传变异大多来自群体内,表明研究的坛紫菜群体的遗传分化主要表现为群体内分化(FST=0.017,P>0.05)。群体间的遗传分化见表2。
Tajimas D值通常用于推断物种基因序列的变异类型。所有个体,无论是CO I基因(Tajimas D值为-1.844,P<0.05)、rbcL基因(Tajimas D值为-1.191,P>0.10),还是 CO I和rbcL基因的联合分析(Tajimas D值为-1.954,P<0.05),结果显示,Tajimas D值均为负值(见表1),坛紫菜群体在进化过程中发生了瓶颈效应,说明环境的选择作用对有害变异进行了清除。
6个变异位点定义了6个单倍型(Hap1~Hap6),其中Hap3为核心单倍型,占全部个体的88.600%。本地传统养殖坛紫菜品系的单倍型数量多于其他两个品系定义的单倍型数量(见表1)。单倍型网络图见图1。其他单倍型均由Hap3衍生出来。单倍型网络图没有出现明显的地理谱系结构。
以条斑紫菜(Pyropia yezoensis)(CO I:KF561997.1;rbcL:AB818919.1)和紫菜(Pyropia fucicola)(CO I:KF561997.1;rbcL:KJ776837.1)为外群,构建坛紫菜单倍型NJ系统发育树(见图2)。
由系统发育树可以看出,3个品系的种群尚未形成明显的谱系现象。
3 讨论
3.1 堿基组成
线粒体DNA的一个突出特点是鸟嘌呤(G)和胞嘧啶(C)相对含量低。在已经测定的藻类线粒体DNA中,G+C的相对含量范围为13.300%~53.200%,平均值为38%。但大多数藻类的G+C相对含量集中在20%~40%[16]。本研究中,3个坛紫菜群体的G+C相对含量为28.140%,该结果符合藻类线粒体DNA的组成特征,如红毛菜纲中红藻(Cyanidioschyzon merolaede)的G+C相对含量为27%,红藻门中皱波角叉菜(Chondrus crispus)的G+C相对含量为27%,紫色紫菜(Porpgyra purpurea)的G+C相对含量为33%[16]。
3.2 遗传多样性
物种或种群的遗传多样性一般来源于长期进化过程中积累的丰富遗传变异。一个物种的遗传多样性越丰富,其对生存环境的适应能力越强,而物种的适应能力越强,其进化潜力也会越大。遗传多样性不仅是生物多样性的内在表现形式,还是物种保持进化潜能的基本条件,对生物多样性的形成、发展或者维持有重要的意义[17-18]。单倍型多样性是衡量群体遗传变异的重要指标之一。本次检测的60株坛紫菜个体只获得了6个单倍型,表明3个坛紫菜养殖品系的遗传多样性水平较低。群体单倍型多样度是评价该群体多态程度的重要指标之一,数值越大,群体遗传多样性越高。本研究3个群体的核苷酸多样性为0.000 30±0.001,单倍型多样性为0.249±0.074,出现了高单倍型多样性和低核苷酸多样性的现象,符合Grant等[19]提出的海洋类生物具有较高的单倍型多样性和较低的核苷酸多样性的模式。温州洞头传统养殖品系的单倍型多样性和核苷酸多样性最高,其次为新品系SW-81,新品系HR-5最低。刘颖等[3]利用AFLP技术研究发现,台州玉环地区坛紫菜传统养殖品系的多态位点比例高于新品系“浙南3号”,这可能是因为玉环本地传统养殖品系的种菜来源多样且范围大,而新品系“浙南3号”选育品系的种菜来源范围较小,所以玉环品系的遗传多样性较高。由此可见,本研究与之前的研究结果是相吻合的,即坛紫菜种群遗传多样性整体处于较低水平;人工培育新品系的遗传多样性低于本地传统养殖品系,本地养殖品系的遗传多样性又低于野生品系[20]。
坛紫菜是一种喜浪海藻。野生坛紫菜大多分布于高潮带,在受风浪冲击的外海礁石上生长特别旺盛[21]。潮间带受潮汐影响,微生境条件恶劣,该区域生物会受到温度、盐度和干露等条件的考验,而野生坛紫菜的遗传多样性程度较高,可以更好地适应变化多样的生存环境。坛紫菜新品系是选自于某株特殊材料,经过诱变、裂解、扩繁培养后得到的纯系种质,该方法使得新品系的遗传多样性较低。在为农业生产选育优良品种时,一方面需确保稳定高产,另一方面则应扩大选育材料来源,以提高种质的遗传多样性,确保其适应性更强。
本研究发现,3个坛紫菜品系间的遗传分化水平较低,这与刘颖等[3]的研究结果相吻合。坛紫菜大部分为雌雄异体,繁育类型主要为异交,而且生活史中的果孢子及壳孢子都可以通过海流的运动进行长距离传播。我国沿海同时存在南向和北向两种主要的洋流模式,一是黑潮的支流,沿我国台湾北上入东海,即台湾暖流;二是季节性的浙闽沿岸流,在冬季和夏季分别自北向南和自南向北流动,促使不同海区间的联系加强[22]。温州洞头海域受江浙沿岸流、台湾暖流、地表径流等3大洋流的影响,使得坛紫菜果孢子和壳孢子可以随着洋流漂流,促进坛紫菜不同养殖群体间发生基因交流,从而减少了群体间的遗传分化[20]。另外,目前坛紫菜养殖品系间相互混杂,也增加了品种间基因交流的机会[3]。
4 结论
本研究使用线粒体CO I和叶绿体rbcL基因片段对坛紫菜温州洞头地区传统养殖品系和两个新品系(SW-81和HR-5)进行研究,发现新品系的遗传多样性较低。因此,应加大种质保护和选育范围,防止种质退化。在坛紫菜育苗生产过程中,建议采用多个品系联合应用,以适应多变的海洋环境。
参考文献
[1]王旭雷,马颖超,鲁晓萍,等.法紫菜生物多样性及其栽培生物学基础[J].海洋科学,2017,41(2):125-135.
[2]农业农村部渔业渔政管理局,全国水产技术推广总站,中国水产学会.2021中国渔业统计年鉴[M].北京:中国农业出版社,2022:50.
[3]刘颖,张鹏,王铁杆,等.浙南坛紫菜实验品系与传统品系遗传多样性的AFLP分析[J].浙江农业科学,2020,61(6):1157-1161.
[4]LI Y Y,SHEN S D,HE L H,et al.Sequence analysis of the ITS region and 5.8S rDNA of Porphyra haitanensis[J].Chinese Journal of Oceanology and Limnology,2009,27(3):493-501.
[5]CHEN C S,XIE C T,JI D H,et al.Molecular divergence and application of the ITS-5.8S rDNA and RUBISCO spacer in Porphyra haitanensis Chang et Zheng (Bangiales,Rhodophyta)[J].Aquaculture International,2010,18(6):1045-1060.
[6]XU P,YANG L E,ZHU J Y,et al.Analysis of hybridization strains of Porphyra based on rbcL gene sequences[J].Journal of Applied Phycology,2011,23(2):235-241.
[7]YOW Y Y,LIM P E,PHANG S M.Genetic diversity of Gracilaria changii(Gracilariaceae,Rhodophyta)from west coast,Peninsular Malaysia based on mitochondrial CO I gene analysis[J].Journal of Applied Phycology,2011,23(2):219-226.
[8]曹原,汪文俊,梁洲瑞,等.“黃优1号”条斑紫菜新品系遗传背景和营养成分分析[J].广西科学,2016,23(2):131-137.
[9]LIBRADO P,ROZAS J.DnaSP v5:a software for comprehensive analysis of DNA polymorphism data[J].Bioinformatics,2009,25(11):1451-1452.
[10]NEI M,MARUYAMA T,CHAKRABORTY R.The bottleneck effect and genetic variability in populations[J].Evolution,1975,29(1):1-10.
[11]TAMURA K,STECHER G,PETERSON D,et al.MEGA6:molecular evolutionary genetics analysis version 6.0[J].Molecular Biology and Evolution,2013,30(12):2725-2729.
[12]KIMURA M.A simple method for estimating evolutionary rates of base substitutions through comparative studies of nucleotide sequences[J].Journal of Molecular Evolution,1980,16(2):111-120.
[13]LEIGH J W,BRYANT D.Popart:full-feature software for haplotype network construction[J].Methods in Ecology and Evolution,2015,6(9):1110-1116.
[14]TAJIMA F.Statistical method for testing the neutral mutation hypothesis by DNA polymorphism[J].Genetics,1989,123(3):585-595.
[15]EXCOFFIER L,LISCHER H E L.Arlequin suite ver 3.5:a new series of programs to perform population genetics analyses under Linux and Windows[J].Molecular Ecology Resources,2010,10(3):564-567.
[16]张静,王绪敏,刘涛,等.藻类线粒体DNA研究进展[J].高技术通讯,2010,20(2):214-220.
[17]张鹏,蔡一凡,王铁杆,等.浙江沿海不同地理群体铜藻Sargassum horneri的AFLP分析[J].浙江农业学报,2015,27(9):1586-1592.
[18]施立明.遗传多样性及其保存[J].生物科學信息,1990,2(4):158-164.
[19]GRANT W S,BOWEN B W.Shallow population histories in deep evolutionary lineages of marine fishes:insights from sardines and anchovies and lessons for conservation[J].Journal of Heredity,1998,89(5):415-426.
[20]吴晓雯,王铁杆,刘颖,等.我国东南沿海坛紫菜遗传多样性研究[J].海洋学研究,2020,38(4):58-64.
[21]朱建一,严兴洪,丁兰平,等.中国紫菜原色图集[M].北京:中国农业出版社,2016:136.
[22]苏纪兰,袁业立.中国近海水文[M].北京:海洋出版社,2005:367.
Abstract: In order to explore the genetic differences between the traditional and new strains(SW-81,HR-5) of Neoporphyra haitanensis in Dongtou district,Wenzhou,the genetic diversity of 60 individuals in these three strains was studied using CO I and rbcL gene fragments.After comparative analysis,the 398 bp CO I gene sequence and 488 bp rbcL gene sequence were obtained with an isologous sequence,the joint analysis length was 886 bp,and the content of T,C,A and G in all obtained sequences was 35.100%,14.220%,34.320% and 16.360%,respectively.There were 6 mutation sites,including 5 single mutation sites and 1 simple information site;The nucleotide diversity of the three strains was 0.000 30±0.001,and the haplotype diversity was 0.249±0.074.60 individuals defined 6 haplotypes,and Hap3 was the core haplotype,accounting for 88.600% of all individuals,respectively;The haplotype-constructed NJ phylogenetic tree showed that there was gene communication in each strain,and genetic differentiation had not yet occurred.The results showed that the genetic diversity of the new strain of N. haitanensis was low,so the scope of germplasm protection and breeding should be improved to prevent germplasm degradation.It is recommended to use multiple strains in the production process of seaweed seedlings to adapt to the changing marine environment.
Key words: Neoporphyra haitanensis; new strain; CO I gene; rbcL gene; genetic diversity
作者简介:吴晓雯(1993—),女,助理研究员,研究方向为海洋生物多样性。E-mail:xwen0106@126.com
通信作者:张鹏(1982—),男,高级工程师,主要从事海洋生物生理生态研究。E-mail:zhangpeng20011918@163.com
项目资助:浙江省农业(水产新品种选育)新品种选育重大科技专项(2021C02069-9);温州市科研项目(S2020008);农业农村部藻类产 业技术体系(CARS-50)。

