基于全基因组重测序的藏猪脂质沉积相关基因鉴定
张彦 熊和丽 张斌 相德才 刘韶娜 赵智勇

张彦(1986-),博士,现为云南生猪产业前沿技术顶尖团队主要成员,近期重点开展地方猪杂交组合筛选及功能基因挖掘、地方猪性别分选精子差异蛋白筛查等研究工作。主持国家重点研发计划项目子课题“肉用山羊种畜场疫病防控技术示范基地建设”、云南省青年科学基金项目“云南地方猪性别分选精子差异表达蛋白鉴定与分析”,作为主要成员参与国家自然科学基金项目“膜穿透性海藻糖在猪卵母细胞玻璃化冷冻中的保护作用及机制研究”、云南省重大科技专项“云南地方家畜(猪、羊)种质资源开发平台建设及成果转化”等省部级以上科研项目5项;在《Journal of Proteomics》《中国畜牧杂志》《南方农业学报》等国内外期刊发表学术论文10余篇;获授权发明专利4项、实用新型专利10项;参与制定地方标准4项。
摘要:【目的】从全基因组水平解析藏猪与大白猪间的遗传差异,并鉴定出与藏猪脂质沉积相关的基因,为藏猪优质肉质特征的利用及新品种培育提供理论参考。【方法】选用5份自测云南迪庆藏猪的全基因组重测序数据,以及NCBI数据库中来自西藏、四川、甘肃地区34份藏猪和具有低脂肪沉积特征的14份大白猪全基因组重测序数据,获得全基因组SNP后以VCFtools进行选择信号检测,采用VCFtools分别计算候选SNP在藏猪和大白猪中的等位基因频率,再利用自编Python脚本及所有SNP注释文件进行注释以确定候选基因,最后运用DAVID对候选基因进行GO功能注释和KEGG信号通路富集分析,进一步鉴定与脂质沉积相关的功能基因。【结果】经全基因组重测序及与参考基因组比对、过滤后得到19374832个高质量的SNPs,根据受选择区域条件筛选出370778个候选SNPs,其中符合在藏猪中等位基因频率>0.5而在大白猪中等位基因频率<0.5条件的SNPs有152877个,共注释到1486个候选基因。GO功能注释分析结果显示,1486个候选基因注释到17个GO功能簇(Cluster1~Cluster17),其中Cluster11和Cluster12与脂肪生成相关,涉及与脂滴转运、结合过程相关的基因NME4、C4BPA、AP2M1、CD36、FABP5、PMP2、PDZD8、OSBPL6、APOD和OSBPL1A,与脂肪酸合成、延伸和代谢过程相关的基因TECR、ELOVL7、HACD2,以及与脂肪形成相关的细胞外基质基因(CCN3、EGFL6、KAZALD1、ASPN、ADAMTS18、ECM2、COL18A1、COL3A1和COL4A5);通过文献分析还发现8个基因(APOOL、PLAG1、PLAUR、SUCLG2、CPE、AKR1B1、SLC4A4和CHPT1)在藏猪与大白猪皮下脂肪或肌肉组织呈差异表达,且与脂肪形成相关。【结论】藏猪与大白猪间的脂肪沉积差异涉及脂肪形成的多个过程和多个基因,细胞外基质基因也可能与藏猪脂肪形成相关,且这些基因在藏猪中均具有较高的等位基因频率。
关键词: 藏猪;脂质沉积;选择信号;等位基因频率;全基因组重测序
中图分类号:S828.89 文獻标志码: A 文章编号:2095-1191(2023)02-0325-11
Abstract:【Objective】Exploring the genetic differences between Tibetan pigs (TIB) and Large white pigs(LW) at the whole-genome level, and identifying genes related to lipid deposition in TIB, to provide informative insights into the genetic utilization of its high meat quality traits. 【Method】 Whole genome re-sequencing data of 5 Yunnan Diqing TIB and whole-genome re-sequencing data of 34 TIB from Tibet, Sichuan and Gansu, and 14 LW with low fat characteristics in NCBI database were selected, which were totally 53 whole genome re-sequencing data for followed analysis.Whole genome SNPs were obtained and selection sweep analysis was performed using VCFtools. Allele frequencies for candidate SNPs in TIB and LW populations were calculated using VCFtools, and candidate SNPs were annotated using in house Pythonscript and all annotation files. Then, DAVID was used for GO functional annotation and KEGG pathway enrichment analysis to further identify genes related to lipid deposition. 【Result】By mapping whole genome sequence to refere-nce genome, 19374832 high quality SNPs were obtained. The selection region contained 370778 candidate SNPs, among which 152877 SNPs met the condition of allele frequency>0.5 in TIB and <0.5 in LW and were annotated into 1486 genes. The GO analysis showed that 1486 candidate genes were enriched into 17 GO functional clusters (Cluster 1-Cluster 17), among which Cluster 11 and Cluster 12 were related to lipid synthesis. The two clusters included genes involved in lipid droplet transport, binding processes (NME4, C4BPA, AP2M1, CD36, FABP5, PMP2, PDZD8, OSBPL6, APOD and OSBPL1A), fatty acid synthesis, elongation, and metabolism (TECR, ELOVL7, and HACD2), as well as extracellular matrix genes (CCN3, EGFL6, KAZALD1, ASPN, ADAMTS18, ECM2, COL18A1, COL3A1 and COL4A5). In addition,by surveying literature, eight genes (APOOL, PLAG1, PLAUR, SUCLG2, CPE, AKR1B1, SLC4A4 and CHPT1) were found to have differential expression in subcutaneous adipose tissue or muscle tissue between TIB and LW, and related to lipid formation. 【Conclusion】This study reveals that the difference in lipid deposition between TIB and LW involves multiple processes and genes related to lipid formation, extracellular matrix genes might also involved in fat depostition of TIB,and these genes have a relatively high allele frequencies in TIB.
Key words: Tibetan pigs; lipid deposition; selective signal; allele frequency; whole genome re-sequencing
Foundation items:Yunnan Major Science and Technology Project(202102AE090039);General Project of Yunnan Fundamental Research Plan(202101AT070061)
0 引言
【研究意义】藏猪是世界上少有能适应高原海拔气候的猪种,具有耐寒、耐低氧、抗逆性强、耐粗饲等特点,且肉质鲜美,素有藏香猪的美誉,是培育新品种的独特优质遗传素材(李明洲等,2008;裴利君等,2021;杨烨城等,2021)。目前,国内主要利用藏猪与瘦肉型猪种(大白、长白、杜洛克等)杂交以培育适应当地且具有优质肉质性状的高效猪种(龚建军等,2007),但由于肌内脂肪不易活体测量,导致其肉质很难通过性状测定进行选育。因此,解析藏猪脂质沉积的遗传机理及鉴定脂肪沉积相关基因,可为分子标记辅助育种提供理论参考,对推进高效优质新品种培育具有重要意义。【前人研究进展】至今,已有较多学者以藏猪和具有低脂肪沉积特征的大白猪(瘦肉型)为研究对象,通过转录组及蛋白组学揭示藏猪肌内脂肪及皮下脂肪沉积的调控作用机制,为藏猪脂肪沉积机制的解析提供了大量候选基因(Wang et al.,2015;Shang et al.,2019)。Wang等(2015)对180日龄藏猪—滇南小耳猪与大白—长白的背最长肌mRNA和miRNA进行差异表达分析,结果发现FASN、SCD、ADORA1、miR-4332、miR-182、miR-92b-3p、miR-let-7a和miR-let-7e等是调节藏猪及滇南小耳猪肌内脂肪沉积的关键基因或miRNA;而蛋白组学分析发现LPL、APOA1、APOC3、ACADM、FABP3、ACADVL、ACAA2、ACAT1、HADH和PECI等基因參与藏猪及滇南小耳猪的肌内脂肪沉积(Wang et al.,2017)。Tao等(2017)对藏猪肌内脂肪和皮下脂肪进行转录组分析,发现多种基因参与藏猪脂肪沉积调控,其中,部分基因同时参与肌内脂肪和皮下脂肪沉积,而部分基因分别是肌内脂肪或皮下脂肪沉积调控的关键基因。Shang等(2019)对180日龄藏猪和大白猪皮下脂肪的转录组测序分析发现,FGF2、PLAG1、ADIPOQ、IRX3、MIF、IL-34、ADAM8、HMOX1、Vav1和TLR8等基因与藏猪皮下脂肪沉积调控相关。Gong等(2022)通过转录组分析发现ACACA、SLC2A4和THRSP基因正调控藏猪皮下脂肪沉积,CHPT1基因则产生负调控作用。近年来,随着测序技术的快速发展,基因组测序成本不断降低,越来越多学者利用全基因组重测序从基因组水平进行动物重要经济性状候选基因定位,包括狗(Axelsson et al.,2013)、猪(Lü et al.,2016)、鸭(Zhou et al.,2018)、猴(Liu et al.,2020)、牛(Luo et al.,2020)、山羊(Zheng et al.,2020)等物种经济性状或适应性相关基因的定位。Zhou等(2018)对40羽绿头鸭、30羽本地鸭和30羽北京鸭进行全基因组重测序,结果发现Mitf 基因是北京鸭白羽形成的关键基因;李恒等(2022)通过全基因组重测序发现,PRLHR外显子G529A与DRD1外显子A281T突变可能是调控山羊多羔性状的关键遗传标记。【本研究切入点】目前,关于藏猪脂质沉积机制的研究主要从转录组或蛋白组学角度进行探究,为揭示藏猪脂质沉积调控机制提供了大量候选基因,但从基因组水平解析藏猪脂质沉积遗传机制的研究鲜见报道。【拟解决的关键问题】从全基因组水平解析藏猪与大白猪间的遗传差异,并鉴定出与藏猪脂质沉积相关的基因,为藏猪优质肉质特征的利用及新品种培育提供理论参考,最终有效促进藏猪种质资源的保护与开发利用。
1 材料与方法
1. 1 试验材料
试验材料为5份自测云南迪庆藏猪的全基因组重测序数据,以及NCBI数据库中来自西藏、四川、甘肃地区34份藏猪和具有低脂肪沉积特征的14份大白猪全基因组重测序数据,样本信息详见表1。
1. 2 试验方法
1. 2. 1 全基因组重测序及变异检测 提取5头云南迪庆藏猪血液DNA,琼脂糖凝胶电泳检测DNA完整性,NanoDrop检测DNA纯度(OD260/OD280),选择OD在1.8~2.0及含量在1 μg以上的DNA样品用于构建文库;检验合格的DNA样品通过破碎机随机打断成350 bp的片段,建库后采用Illumina NovaSeq 6000进行测序。获得的5份原始序列(Sequenced reads)与NCBI数据库中的48份全基因组重测序数据一起进入下游分析。利用Fastp_0.20.0进行过滤,去除带接头、含N(无法确定碱基信息的位点)比例大于5%及质量值Q≤10碱基数占整个测序片段20%以上的Reads,过滤后的测序数据通过BWA比对到猪参考基因组Sscrofa11.1,然后采用SAMtools对比对结果进行排序(Li et al.,2009),再以Picard标记重复序列(Zhou et al.,2018),使用GATK 4.0.4.0检测SNP(DePristo et al.,2011),并对检测到的SNP进行过滤,以获得高质量的SNP。过滤条件:(1)SNP的Reads支持数低于4;(2)SNP的质量值Q?20;(3)次等位基因频率(Minor allele frequency,MAF)<0.05;(4)缺失率>0.1。最后以SnpEff对获得的高质量SNP进行注释分析(Cingolani et al.,2012)。
1. 2. 2 选择信号检测 根据藏猪与大白猪基因组的等位基因频率分化及多样性差异检测藏猪受选择区域,利用VCFtools以50 kb的滑动窗口进行检测(Danecek et al.,2011),并计算遗传分化系数(FST)及核苷酸遗传多样性(π)。在定义受选择区域时,考虑到中国家猪与欧洲家猪起源于不同野猪,二者的遗传差异较大,遗传分化明显(Groenen et al.,2012),因此扩大受选择区域的阈值范围,将同时满足等位基因FST>0.5且π(LW/TIB)最高5%(Top 5%)和最低5%(Low 5%)的区域定义为受选择区域(Li et al.,2013),利用自编Python脚本提取出受选择区域中的所有SNP。
1. 2. 3 候选基因等位基因频率分析 藏猪具有较高的脂肪沉积能力,而大白猪脂质沉积力较低,即与脂质沉积相关的基因在藏猪群体中应具有较高的等位基因频率,因此进一步统计候选SNP在藏猪和大白猪中的等位基因频率。首先利用自编Python脚本从VCF文件中取出受選择区域的所有SNP,采用VCFtools分别计算候选SNP在藏猪和大白猪中的等位基因频率,然后以自编Python脚本过滤筛选在藏猪中等位基因频率>0.5且在大白猪中等位基因频率<0.5的SNP,获得的SNP利用自编Python脚本及所有SNP注释文件进行注释分析,SNP所在的基因即为候选基因。
1. 2. 4 藏猪脂质沉积相关基因鉴定 利用在线DAVID对候选基因进行GO功能注释及KEGG信号通路富集分析,富集到与脂肪合成及代谢相关通路的基因可能就是藏猪脂质沉积相关基因。
2 结果与分析
2. 1 全基因组重测序数据统计及变异检测结果
5份自测云南迪庆藏猪及NCBI数据库中34份藏猪和14份大白猪的全基因组重测序数据统计结果如表2所示,其中原始序列(Raw reads)约135亿条,平均比对率为99.12%,平均测序深度为9.77×。通过与参考基因组比对并过滤后得到19374832个高质量的SNPs。
2. 2 选择信号分析结果
利用VCFtools以50 kb的滑动窗口进行检测,结果共检测到46912个区间,其FST介于0~0.98,平均为0.32(图1-A),π(LW/TIB)介于0~39.19,平均为0.59(图1-B),其中389个区间的π为0,即这些区间SNP只存在于藏猪或大白猪中,并非在2个品种共有,说明这些SNP是相对于另一品种的特有SNP,因此在后续研究中将这些区间也定义为受选择区域。通过选取FST>0.5,且满足π(LW/TIB)最高5%(>1.18)及最低5%(<0.05)的区间,以及π为0的区间,共有1379个区间定义为受选择区间,包含370778个SNPs,即为候选SNP。
2. 3 候选SNP等位基因频率分析结果
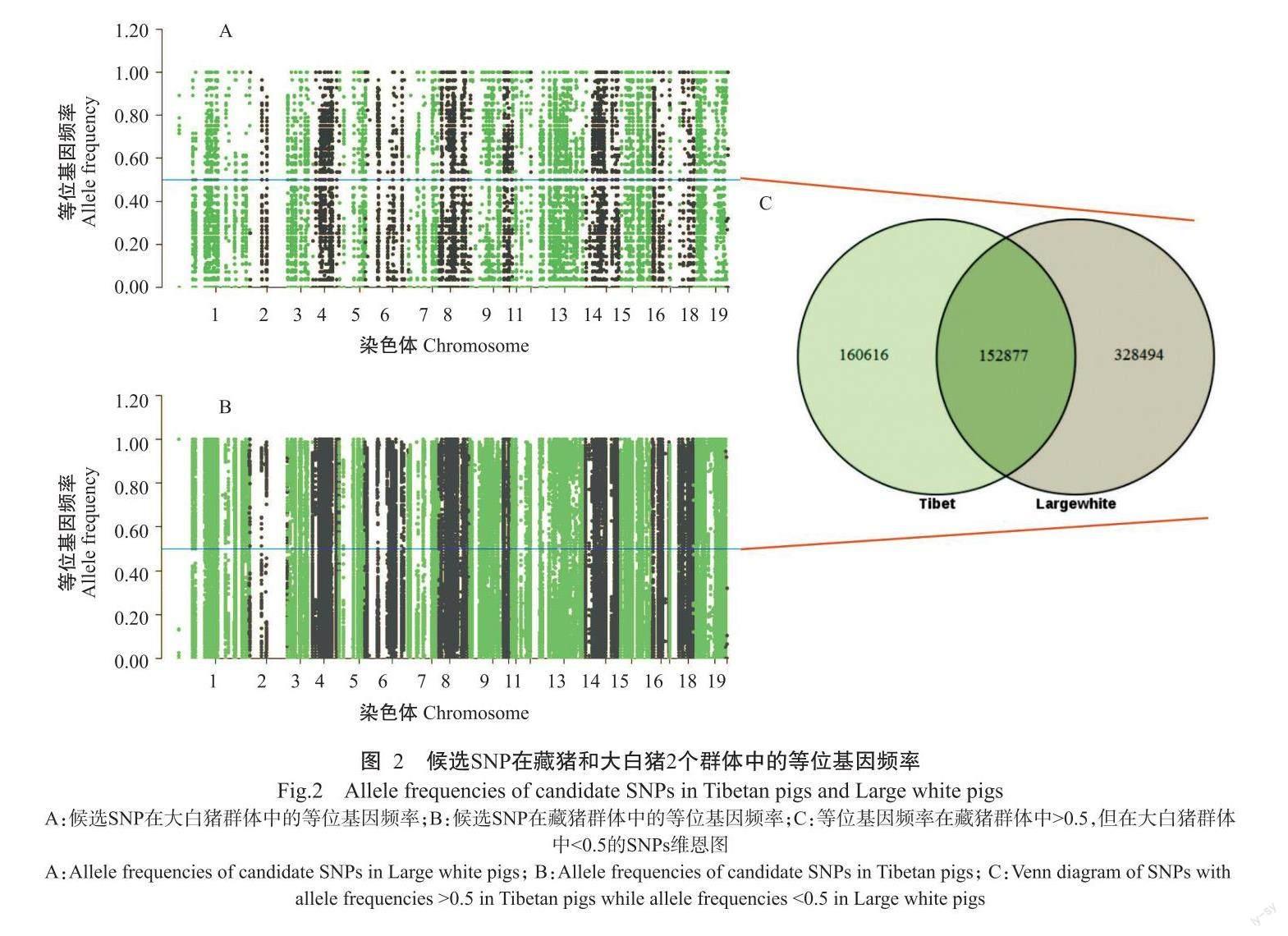
通过分析370778个SNPs在藏猪和大白猪2个群体中的等位基因频率,结果发现在藏猪群体中的等位基因频率介于0~1(图2-A),平均等位基因频率0.47,等位基因频率>0.5的SNPs有160016个;在大白猪中的等位基因频率也介于0~1(图2-B),平均等位基因频率0.12,等位基因频率>0.5的SNPs有42285个,等位基因频率<0.5的SNPs有328494个。说明通过选择压力分析筛选获得的候选区间包含大量分别在2个群体中具有低等位基因频率和高等位基因频率的SNPs,因此需要进一步筛选。本研究聚焦于在藏猪中等位基因频率>0.5,但在大白猪中等位基因频率<0.5的SNPs。经筛选,共有152877个SNPs符合条件(图2-C),其中73050个SNPs位于基因间区、73595个SNPs分布在内含子区,其他位置的SNPs有6234个;符合条件的SNPs共注释到1486个基因。
2. 4 藏猪脂肪沉积相关基因鉴定结果
为进一步筛选出藏猪脂肪沉积相关基因,利用DAVID对候选基因进行GO功能注释分析,结果(图3-A)发现有1486个候选基因注释到17个GO功能簇(Cluster1~Cluster17),其中Cluster11和Cluster12与脂肪生成相关。Cluster11包含3个GO功能集(图3-B),分别是BP_Lipid binding、MF_Lipid transport和MF_ Cholesterol binding,涉及10个基因(NME4、C4BPA、AP2M1、CD36、FABP5、PMP2、PDZD8、OSBPL6、APOD和OSBPL1A);Cluster12包含4个与脂肪细胞外基质相关的GO功能集(图3-C),分别是BP_Extracellular matrix organization、CC_Extracellular matrix、CC_Collagen trimer和MF_Extracellular matrix structural constituent,涉及14个基因(CCN3、EGFL6、KAZALD1、RXFP1、NYX、ASPN、ADAMTS18、ECM2、ADAMTS3、MMP16、ADAMTS17、COL18A1、COL3A1和COL4A5)。
同时利用DAVID对1486个候选基因进行KEGG信号通路富集分析,结果富集到10个KEGG通路富集簇(Cluster1~Cluster10),其中Cluster6与脂肪生成相关(图4)。Cluster6包含3条KEGG信号通路:Fatty acid elongation、Biosynthesis of unsaturated fatty acids和Fatty acid metabolism,涉及3个基因(TECR、ELOVL7和HACD2)。GO功能簇和KEGG富集簇中与脂肪形成相关的基因所包含的SNPs,在藏猪群体中具有中等及以上等位基因频率,而在大白猪中的等位基因频率均较低或为0(表3)。
通过查阅文献发现,1486个候选基因中有28个是Tao等(2017)研究报道180日龄藏猪与大白猪皮下脂肪组织的差异调控基因,44个是Shang等(2019)研究报道180日龄藏猪与大白猪皮下脂肪组织的差异调控基因。其中,有19个基因在上述2个研究中均被报道,分别是APOOL、ARMCX1、ASPN、BEX1、CD48、COL4A5、CPE、CRISPLD1、FSTL5、HCLS1、KCNQ1、LRRIQ3、LTBP4、PCDH11X、PDE1C、PLAG1、PLAUR、PURG和SUCLG2基因,而APOOL、ASPN、PLAG1、PLAUR、SUCLG2、COL4A5和CPE等7个基因已知与脂肪形成相关(表4)。在1486个候选基因中,有7个是Tao等(2017)研究报道180日龄藏猪与大白猪背最长肌的差异调控基因,分别是APOOL、AKR1B1、ITGB1BP2、RAP1GDS1、SLC26A7、SLC4A4和SNCA,其中APOOL、AKR1B1和SLC4A4基因与脂肪形成相关。另外,候选基因CHPT1在Gong等(2022)的研究中是180日龄藏猪与大白猪皮下脂肪组织的差异表达基因。
3 讨论
脂肪形成是脂肪细胞形成、脂肪合成与脂肪水解相平衡的过程,包括脂肪前体细胞在细胞外因子作用下启动脂肪前体细胞分化为成熟脂肪细胞,成熟脂肪细胞内脂肪的形成,以及甘油三酯的合成与分解,因此脂肪形成涉及一系列级联反应:脂肪细胞分化与增殖、脂肪酸合成、脂肪链延长及脂肪酸转运等。目前,已有大量研究证实这一系列级联反应中有众多基因参与,如参与脂肪前体细胞分化过程的基因PPARγ(Peroxisome proliferator-activated receptor gamma)、CEBPβ(CCAAT/enhancer-binding protein)、CEBPδ、CEBPα、SREBP(Sterol regulatory element-binding proteins)、LPL(Lipoprotein lipase)、FABP(Fatty acid-binding proteins)及GLUT4(Glucose transporter type 4)等,与脂肪从头合成相关的基因GLUT4(Glucose transporter type 4)、ACLY(ATP-citrate lyase)、FATP(Fatty acid transport protein-1)、FASN(Fatty acid synthase)及ACACA(Acetyl-CoA carboxylase-α)等(Malgwi et al.,2022)。本研究从基因组水平比较藏猪与大白猪间的基因组SNP,结果鉴定出1486个是藏猪与大白猪间存在遗传差异的基因,通过GO功能注释及KEGG信号通路富集分析,发现藏猪与大白猪间的脂肪沉积差异涉及多个过程的多个基因,主要包括与脂滴转运、结合过程相关的基因NME4、C4BPA、AP2M1、CD36、FABP5、PMP2、PDZD8、OSBPL6、APOD和OSBPL1A,以及与脂肪酸合成、延伸及代谢过程相关的基因TECR、ELOVL7和HACD2。但在已报道的研究中这些基因在180日齡藏猪与大白猪皮下脂肪组织并未表现出差异表达,因此是否在其他生长阶段或脂肪组织中发生转录且翻译为功能蛋白,并最终调控藏猪脂肪沉积还有待进一步探究。
细胞外基质对细胞起支撑和固定作用,同时对促进细胞间交流发挥重要调节作用,其成分主要包括葡糖氨基葡聚糖、糖复合物、蛋白聚糖、胶原蛋白及层黏连蛋白(Divoux and Clément,2011)。在体外培养过程中,细胞外基质成分对原血管基质组分分化为脂肪前体细胞的过程有重要影响,层黏连蛋白与整合蛋白在脂肪前体细胞发育过程中对脂肪细胞形态起关键作用,其中,层黏连蛋白能促进脂肪前体细胞吸附(Hausman,2012),而整合蛋白avb3有利于脂肪形成过程中的血管生成(Crandall et al.,2000)。Hausman等(2014)通过采用乙基-3,4-二氢-苯甲酸异丙酯(EDHB)抑制剂抑制胶原蛋白合成,证实了细胞外基质胶原蛋白在脂肪前体细胞分化过程中的重要性,EDHB抑制胶原蛋白合成后能有效降低牛肌内脂肪前体细胞、猪脂肪前体细胞克隆细胞系及其他脂肪前体细胞系的脂肪细胞分化。本研究共鉴定到14个细胞外基质相关基因在藏猪与大白猪间存在遗传差异,且已有研究证实部分基因与脂肪形成相关,如ASPN基因能促进脂肪细胞的生成。3个胶原蛋白类基因均与脂肪形成相关,其中,COL18A1基因支持脂肪前体细胞分化,并维持脂肪细胞的分化状态及白色脂肪组织大小和脂质的储存与清除(Aikio et al.,2014),缺失特定的COL18A1基因异构体会影响小鼠脂肪细胞分化(Pet?ist? et al.,2020);COL3A1基因是脂肪细胞形成的重要调控基因(Al Hasan et al.,2021);COL4A5基因则表达成脂肪细胞基底膜成分(Ninomiya et al.,1995)。敲除基质金属蛋白酶基因ADAMTS18后,小鼠脂肪比重增加,且内脏脂肪细胞增大(Zhu et al.,2018);与细胞外基质相互作用的CCN3基因是一种脂肪细胞因子,其缺失能抑制肥胖(Martinerie et al.,2016);EGFL6基因能促进脂肪前体细胞增殖,与脂肪细胞体积增大极显著相关(Landgraf et al.,2022);KAZALD1基因是胰岛素生长因子结合蛋白(IGFBP)超家族成员,而IGFBP是以自分泌或旁分泌形式调节脂肪前体细胞形成前的血管基质细胞增殖(Hausman et al.,2014)。在14个细胞外基质相关基因中,ASPN和COL4A5基因已被证实在180日龄藏猪皮下脂肪组织中呈差异表达(Tao et al.,2017;Shang et al.,2019),其他基因在180日龄藏猪与大白猪皮下脂肪组织中并未表现出差异表达,其是否最终翻译成功能蛋白调控藏猪脂肪沉积还有待进一步验证。
Poklukar等(2020)通过综述近年来27项通过转录组解析脂肪型猪与瘦肉型猪脂肪沉积差异相关的关键基因研究,发现不同脂肪型猪的脂肪沉积关键基因存在差异,可能与采样日龄、试验猪营养供给、脂肪组织部位等差异有关,由此推测不同脂肪型猪的脂肪沉积具有特异调控机制。Tao等(2017)同时比较7个180日龄中国地方猪种与大白猪皮下脂肪和背最长肌的转录表达谱,发现中国地方猪种与大白猪间的差异表达基因只有极少部分在7个中国地方猪种间同时表达,也表明不同地方猪种的脂肪沉积可能存在特异调控机制。本研究鉴定到10个已证实在180日龄藏猪与大白猪皮下脂肪组织差异表达的基因,其中,COL4A5基因是Tao等(2017)研究报道成华猪、藏猪、内江猪、青峪猪、乌金猪、丫杈猪与大白猪皮下脂肪组织的差异表达基因,APOOL基因是藏猪、内江猪、雅南猪与大白猪皮下脂肪组织的差异表达基因,同时又是藏猪、成华猪、青峪猪与大白猪肌内脂肪的差异表达基因,SLC4A4基因是藏猪、成华猪、雅南猪与大白猪肌内脂肪的差异表达基因,CPE基因是藏猪、内江猪与大白猪皮下脂肪组织的差异表达基因,而ASPN、PLAG1、SUCLG2、PLAUR、AKR1B1和CHPT1基因是藏猪与大白猪皮下脂肪和肌内脂肪组织的差异表达基因,故推测这些基因是藏猪脂肪沉积的特异调控基因。
4 结论
藏猪与大白猪间的遗传差异基因涉及脂肪形成的多个过程,包括脂滴转运、结合过程(NME4、C4BPA、AP2M1、CD36、FABP5、PMP2、PDZD8、OSBPL6、APOD和OSBPL1A基因),脂肪酸合成、延伸及代謝过程(TECR、ELOVL7和HACD2基因),以及与脂肪形成相关的细胞外基质基因(CCN3、EGFL6、KAZALD1、ASPN、ADAMTS18、ECM2、COL18A1、COL3A1和COL4A5基因);APOOL、PLAG1、PLAUR、SUCLG2、CPE、AKR1B1、SLC4A4及CHPT1等8个基因在180日龄藏猪与大白猪脂肪组织中差异表达。藏猪与大白猪间的脂肪沉积差异涉及脂肪形成的多个过程和多个基因,细胞外基质基因也可能与藏猪脂肪形成相关,且这些基因在藏猪中均具有较高的等位基因频率。
参考文献:
龚建军,何志平,李正确,吕学斌,应三成,陈晓军. 2007. 藏猪及不同杂交组合肥育与胴体性能研究[J]. 西南农业学报,20(5):1109-1112. [Gong J J,He Z P,Li Z Q,Lü X B,Ying S C,Chen X J. 2007. Investigation on fattening and carcass traits in Tibtan pigs and its combinations[J]. Southwest China Jounal of Agricultrual Sciences,20(5):1109-1112.] doi:10.3969/j.issn.1001-4829.2007.05.052.
李恒,字向东,王会,熊燕,吕明杰,刘宇,蒋旭东. 2022. 基于全基因组重测序的山羊产羔数性状关键调控基因的筛选[J]. 中国农业科学,55(23):4753-4768. [Li H,Zi X D,Wang H,Xiong Y,Lü M J,Liu Y,Jiang X D. 2022. Screening of key regulatory genes for litter size trait based on whole genome re-sequencing in goats (Capra hircus)[J]. Scientia Agricultura Sinica,55(23):4753-4768.] doi:10.3864/j.issn.0578-1752.2022.23.015.
李明洲,李学伟,帅素容,朱砺,刘海峰,王金勇,蒋忠荣,滕晓坤. 2008. 雌性藏猪不同生长发育阶段肌肉生长和脂肪沉积相关基因的表达模式[J]. 动物学报,54(3):442-452.[Li M Z,Li X W,Shuai S R,Zhu L,Liu H F,Wang J Y,Jiang Z R,Teng X K. 2008. Expression patterns of genes associated with muscle growth and adipose deposition during growth and development of female Tibetan pigs[J]. Acta Zoologica Sinica,54(3):442-452.]
裴利君,杨巧丽,王鹏飞,滚双宝. 2021. 合作猪夏冬季的肠道菌群结构[J]. 甘肃农业大学学报,56(4):8-15. [Pei L J,Yang Q L,Wang P F,Gun S B. 2021. Study on the structure of intestinal microflora in Hezuo pigs in summer and winter[J]. Journal of Gansu Agricultural University,56(4):8-15.] doi:10.13432/j.cnki.jgsau.2021.04.002.
杨烨城,黄秋艳,辛海云,李宝红,杜宗亮,孟繁明,朱向星,李剑豪,王塑天,唐冬生. 2022. 藏猪和杜藏猪生长、肉质性状及脂肪沉积相关基因的表达差异分析[J]. 南方农业学报,53(4):926-934. [Yang Y C,Huang Q Y,Xin H Y,Li B H,Du Z L,Meng F M,Zhu X X,Li J H,Wang S T,Tang D S. 2022. Analysis of differences in growth,meat quality traits and expression of genes related to fat deposition between Tibetan and Duzang pigs[J]. Journal of Southern Agriculture,53(4):926-934.] doi:10.3969/j.issn.2095-1191.2022.04.005.
Ai H H,Fang X D,Yang B,Huang Z Y,Chen H,Mao L K,Zhang F,Zhang L,Cui L L,He W M,Yang J,Yao X M,Zhou L S,Han L J,Li J,Sun S L,Xie X H,Lai B X,Su Y,Lu Y,Yang H,Huang T,Deng W J,Nielsen R,Ren J,Huang L S. 2015. Adaptation and possible ancient interspecies introgression in pigs identified by whole-genome sequencing[J]. Nature Genetics,47(3):217-225. doi:10. 1038/ng.3199.
Aikio M,Elamaa H,Vicente D,Izzi V,Kaur I,Seppinen L,Speedy H E,Kaminska D,Kuusisto S,Sormunen R,Heljasvaara R,Jones E L,Muilu M,Jauhiainen M,Pihlajamaki J,Savolainen M J,Shoulders C C,Pihlajaniemi T. 2014. Specific collagen XVIII isoforms promote adipose tissue accrual via mechanisms determining adipocyte number and affect fat deposition[J]. Proceedings of National Academy of Sciences of the United States of America,111(30):E3043-E3052. doi:10.1073/pnas.1405879111.
Al Hasan M,Martin P E,Shu X H,Patterson S,Bartholomew C. 2021. Type III collagen is required for adipogenesis and actin stress fibre formation in 3T3-L1 preadipocytes[J]. Biomolecules,11(2):156. doi:10.3390/biom11020
156.
Axelsson E,Ratnakumar A,Arendt M L,Maqbool K,Webster MT,Perloski M,Liberg O,Arnemo J M,Hedhammar ?,Lindblad-Toh K. 2013. The genomic signature of dog domestication reveals adaptationto a starch-rich diet[J]. Nature,495(7441):360-364. doi:10.1038/nature11837.
Cingolani P,Platts A,Wang L L,Coon M,Nguyen T,Wang L,Land S J,Lu X Y,Ruden D M. 2012. A program for annotating and predicting the effects of single nucleotide polymorphisms,SnpEff:SNPs in the genome of Droso-phila melanogaster strain w1118;iso-2;iso-3[J]. Fly (Austin),6(2):80-92. doi:10.4161/fly.19695.
Crandall D L,Busler D E,McHendry-Rinde B,Groeling T M,Kral J G. 2000. Autocrine regulation of human preadipocyte migration by plasminogen activator inhibitor-1[J]. The Journal of Clinical Endocrinology and Metabolism,85(7):2609-2614. doi:10.1210/jcem.85.7.6678.
Danecek P,Auton A,Abecasis G,Albers C A,Banks E,DePristo M A,Handsaker R E,Lunter G,Marth G T,Sherry S T,McVean G,Durbin R. 2011. The variant call format and VCFtools[J]. Binformatics,27(15):2156-2158. doi:10.1093/bioinformatics/btr330.
DePristo M A,Banks E,Poplin R,Garimella K V,Maguire J R,Hartl C,Philippakis A A,Angel G D,Rivas M A,Hanna M,McKenna A,Fennell T J,Kernytsky A M,Siva-chenko A Y,Cibulskis K,Gabriel S B,Altshuler D,Daly M J. 2011. A framework for variation discovery and genotyping using next-generation DNA sequencing data[J]. Nature Genetics,43(5):491-498. doi:10.1038/ng.806.
Divoux A,Clément K. 2011. Architecture and the extracellular matrix:The still unappreciated components of the adipose tissue[J]. Obesity Reviews,12(5):e494-e503. doi:10.1111/j.1467-789X.2010.00811.x.
Gong X L,Zheng M,Zhang J,Ye Y R,Duan M Q,Chamba Y Z,Wang Z B,Shang P. 2022. Transcriptomics-based study of differentially expressed genes related to fat deposition in Tibetan and Yorkshire pigs[J]. Froniers in Veteri-nary Science,9:919904. doi:10.3389/fvets.2022.919904.
Groenen M A M,Archibald A L,Uenishi H,Tuggle C K,Takeuchi Y,Rothschild M F,Rogel-Gaillard C,Park C,Milan D,Megens H J,Li S T,Larkin D M,Kim H,Frantz L A F,Caccamo M,Ahn H,Aken B L,Anselmo A,Anthon C,Auvil L,Badaoui B,Beattie C W,Bendixen C,Berman D,Blecha F,Blomberg J,Bolund L,Bosse M,Botti S,Zhan B J,Bystrom M,Capitanu B,Carvalho-Silva D,Chardon P,Chen C L,Cheng R,Choi S H,Chow W,Clark R C,Clee C,Crooijmans R P M A,Dawson H D,Dehais P,de Sapio F,Dibbits B,Drou N,Du Z Q,Eversole K,Fadista J,Fairley S,Faraut T,Faulkner G J,Fowler K E,Fredholm M,Fritz E,Gilbert J G R,Giuffra E,Gorodkin J,Griffin D K,Harrow J L,Hayward A,Howe K,Hu Z L,Humphray S J,Hunt T,Hornshoj H,Jeon J T,Jern P,Jones M,Jurka J,Kanamori H,Kapetanovic R,Kim J,Kim J H,Kim K W,Kim TH,Larson G,Lee K,Lee K T,Leggett R,Lewin H A,Li Y R,Liu W S,Loveland J E,Lu Y,Lunney J K,Ma J,Madsen O,Mann K,Matthews L,McLaren S,Morozumi T,Murtaugh M P,Narayan J,Nguyen D T,Ni P X,Oh S J,Onteru S,Panitz F,Park E W,Park H S,Pascal G,Paudel Y,Perez-Enciso M,Ramirez-Gonzalez R,Reecy J M,Rodriguez-Zas S,Rohrer G A,Rund L,Sang Y M,Schachtschneider K,Schraiber J G,Schwartz J,Scobie L,Scott C,Searle S,Servin B,Southey B R,Sperber G,Stadler P,Sweedler J V,Tafer H,Thomsen B,Wali R,Wang J,Wang J,White S,Xu X,Yerle M,Zhang G J,Zhang J G,Zhang J,Zhao S H,Rogers J,Churcher C,Schook L B. 2012. Analyses of pig genomes provide insight into porcine demography and evolution[J]. Nature,491(7424):393-398. doi:10. 1038/nature11622.
Hausman G J,Basu U,Wei S,Hausman D B,Dodson M V. 2014. Preadipocyte and adipose tissue differentiation in meat animals:Influence of species and anatomical location[J]. Annual Review of Animal Biosciences,2:323-351. doi:10.1146/annurev-animal-022513-114211.
Hausman G J. 2012. Meat science and muscle biology symposium:The influence of extracellular matrix on intramuscular and extramuscular adipogenesis[J]. Journal of Animal Science,90(3):942-949. doi:10.2527/jas.2011-4616.
Kanno Y,Matsuno H,Kawashita E,Okada K,Suga H,Ueshima S,Matsuo O. 2010. Urokinase-type plasminogen activator receptor is associated with the development of adipose tissue[J]. Thrombosis Haemostasis, 104(6):1124-1132. doi:10.1160/TH10-02-0101.
Konieczna A,Szczepańska A,Sawiuk K,W?grzyn G,?y?eń R. 2015. Effects of partial silencing of genes coding for enzymes involved in glycolysis and tricarboxylic acid cycle on the enterance of human fibroblasts to the S phase[J]. BMC Cell Biology,16:16. doi:10.1186/s12860-015-0062-8.
Labrecque J,Michaud A,Gauthier M F,Pelletier M,Julien F,Bouvet-Bouchard L,Tchernof A. 2019. Interleukin-1β and prostaglandin-synthesizing enzymes as modulators of human omental and subcutaneous adipose tissue function[J]. Prostaglandins Leukot Essent Fatty Acids,141:9-16.doi:10.1016/j.plefa.2018.11.015.
Landgraf K,Kühnapfel A,Schlanstein M,Biemann R,Isermann B,Kempf E,Kirsten H,Scholz M,K?rner A. 2022. Transcriptome analyses of adipose tissue samples identify EGFL6 as a candidate gene involved in obesity-related adipose tissue dysfunction in children[J]. International Journal of Molecular Sciences,23(8):4349. doi:10.3390/ijms23084349.
Li H,Handsaker B,Wysoker A,Fennell T,Ruan J,Homer N,Marth G,Abecasis G,Durbin R. 2009. The sequence alignment/map format and SAMtools[J]. Binformatics,25(16):2078-2079. doi:10.1093/bioinformatics/btp352.
Li M Z,Tian S L,Jin L,Zhou G Y,Li Y,Zhang Y,Wang T,Yeung C K L,Chen L,Ma J D,Zhang J B,Jiang A A,Li J,Zhou C W,Zhang J,Liu Y K,Sun X Q,Zhao H W,Niu Z X,Lou P E,Xian L J,Shen X Y,Liu S Q,Zhang S H,Zhang M W,Zhu L,Shuai S R,Bai L,Tang G Q,Liu H F,Jiang Y Z,Mai M M,Xiao J,Wang X,Zhou Q,Wang Z Y,Stothard P,Xue M,Gao X L,Luo Z G,Gu Y R,Zhu H M,Hu X X,Zhao Y F,Plastow G S,Wang J Y,Jiang Z,Li K,Li N,Li X W,Li R Q. 2013. Genomic analyses identify distinct patterns of selection in domesticated pigs and Tibetan wild boars[J]. Nature Genetics,45(12):1431-1438. doi:10.1038/ng.2811.
Li S J,Khan R,Raza S H A,Hong J Y,Mei C G,Kaster N,Cheng G,Zhao C P,Schreurs N M,Zan L S. 2020. Function and characterization of the promoter region of perilipin 1 (PLIN1):Roles of E2F1,PLAG1,C/EBPbeta,and SMAD3 in bovine adipocytes[J]. Genomics,112(3):2400-2409. doi:10.1016/j.ygeno.2020.01.012.
Liu Z J,Zhang L Y,Yan Z Z,Ren Z J,Han F M,Tan X X,Xiang Z Y,Dong F,Yang Z M,Liu G J,Wang Z M,Zhang J L,Que T C,Tang C H,Li Y F,Wang S,Wu J Y,Li L G,Huang C M,Roos C,Li M. 2020. Genomic mechanisms of physiological and morphological adaptations of limestone langurs to karst habitats[J]. Molecular Biology and Evolution,37(4):952-968. doi:10.1093/molbev/msz301.
Lü M D,Han X M,Ma Y F,Irwin D M,Gao Y,Deng J K,Adeola A C,Xie H B,Zhang Y P. 2016. Genetic variationsassociated with six-white-point coat pigmentation in Diannan small-ear pigs[J]. Scientific Reports,6(1):27534.doi:10.1038/srep27534.
Luo X E,Zhou Y,Zhang B,Zhang Y,Wang X B,Feng T,Li Z P,Cui K Q,Wang Z Q,Luo C,Li H,Deng Y F,Lu F H,Han J L,Miao Y W,Mao H M,Yi X X,Ai C,Wu S G,Li A L,Wu Z C,Zhuo Z J,da Giang D,Mitra B,Vahidi M F,Mansoor S,Al-Bayatti S A,Sari E M,Gorkhali N A,Prastowo S,Shafique L,Ye G Y,Qian Q,Chen B S,Shi D S,Ruan J,Liu Q Y. 2020. Understanding divergent domestication traits from the whole-genome sequencing of swamp-and river-buffalo populations[J]. National Scien-ce Review,7(3):686-701. doi:10.1093/nsr/nwaa024.
Malgwi I H,Halas V,Grunvald P,Schiavon S,Jocsák I. 2022. Genes related to fat metabolism in pigs and intramuscular fat content of Pork:A focus on nutrigenetics and nutrigenomics[J]. Animals (Basel),12(2):150. doi:10.3390/ani12020150.
Martinerie C,Garcia M,Do T T H,Antoine B,Moldes M,Dorothee G,Kazazian C,Auclair M,Buyse M,Ledent T,Marchal P O,Fesatidou M,Beisseiche A,Koseki H,Hi-raoka S,Chadjichristos C E,Blondeau B,Denis R G,Luquet S,Fève B. 2016. NOV/CCN3:A new adipocytokine involved in obesity-associated insulin resistance[J]. Diabetes,65(9):2502-2515. doi:10.2337/db15-0617.
Moon Y,Moon R,Roh H,Chang S,Lee S,Park H. 2020. HIF-1α-dependent induction of carboxypeptidase A4 and carboxypeptidase E in hypoxic human adipose-derived stem cells[J]. Molecules and Cells,43(11):945-952. doi:10.14348/molcells.2020.0100.
Ninomiya Y,Kagawa M,Iyama K,Naito I,Kishiro Y,Seyer J M,Sugimoto M,Oohashi T,Sado Y. 1995. Differential expression of two basement membrane collagen genes,COL4A6 and COL4A5,demonstrated by immunofluorescence staining using peptide-specific monoclonal antibo-dies[J]. Journal of Cell Biology,130(5):1219-1229. doi:10.1083/jcb.130.5.1219.
Pet?ist? T,Vicente D,M?kel? K A,Finnil? M A,Miinalainen I,Koivunen J,Izzi V,Aikio M,Karppinen S M,Devarajan R,Thevenot J,Herzig K H,Heljasvaara R,Pihlajaniemi T. 2020. Lack of collagen XVIII leads to lipodystrophy and perturbs hepatic glucose and lipid homeostasis[J]. The Journal of Physiology,598(16):3373-3393. doi:10.1113/JP279559.
Petersen C,Nielsen M D,Andersen E S,Basse A L,Isidor M S,Markussen L K,Viuff B M,Lambert I H,Hansen J B,Pedersen S F. 2017. MCT1 and MCT4 expression and lactate flux activity increase during white and brown adipogenesis and impact adipocyte metabolism[J]. Scientific Reports,7(1):13101. doi:10.1038/s41598-017-13298-z.
Poklukar K,Candek-Potokar M,Batorek L N,Tomazin U,Skrlep M. 2020. Lipid deposition and metabolism in local and modern pig breeds:A review[J]. Animals (Basel),10(3):424. doi:10.3390/ani10030424.
Sakashita H,Yamada S,Kinoshita M,Kajikawa T,Iwayama T,Murakami S. 2021. Mice lacking PLAP-1/asporin counteracts high fat diet-induced metabolic disorder and alveolar bone loss by controlling adipose tissue expansion[J]. Scitific Reports,11(1):4970. doi:10.1038/s41598-021-84512-2.
Shang P,Li W T,Liu G,Zhang J,Li M R,Wu L C,Wang K J,Chamba Y Z. 2019. Identification of lncRNAs and genes responsible for fatness and fatty acid composition traits between the Tibetan and Yorkshire pigs[J]. International Journal of Genomics,2019:5070975. doi:10.1155/2019/5070975.
Tao X,Liang Y,Yang X M,Pang J H,Zhong Z J,Chen X H,Yang Y K,Zeng K,Kang R M,Lei Y F,Ying S C,Gong J J,Gu Y R,Lv X B. 2017. Transcriptomic profiling in muscle and adipose tissue identifies genes related to growth and lipid deposition[J]. PLoS One,9(12):e184120. doi:10.1371/journal.pone.0184120.
Wang Z X,Li Q G,Chamba Y Z,Zhang B,Shang P,Zhang H,Wu C X. 2015. Identification of genes related to growth and lipid deposition from transcriptome profiles of pig muscle tissue[J]. PLoS One,10(10):e141138. doi:10.1371/journal.pone.0141138.
Wang Z X,Shang P,Li Q G,Wang L Y,Chamba Y Z,Zhang B,Zhang H,Wu C X. 2017. iTRAQ-based proteomic analysis reveals key proteins affecting muscle growth and lipid deposition in pigs[J]. Scientific Reports,7:46717. doi:10.1038/srep46717.
Weber TA,Koob S,Heide H,Wittig I,Head B,van der Bliek A,Brandt U,Mittelbronn M,Reichert A S. 2013. APOOL is a cardiolipin-binding constituent of the Mitofilin/MINOS protein complex determining cristae morphology in mammalian mitochondria[J]. PLoS One,8(5):e63683. doi:10.1371/journal.pone.0063683.
Zheng Z Q,Wang X H,Li M,Li Y J,Yang Z R,Wang X L,Pan X Y,Gong M,Zhang Y,Guo Y W,Wang Y,Liu J,Cai Y D,Chen Q M,Okpeku M,Colli L,Cai D W,Wang K,Huang S S,Sonstegard TS,Esmailizadeh A,Zhang W G,Zhang T T,Xu Y B,Xu N Y,Yang Y,Han J L,Chen L,Lesur J,Daly K G,Bradley D G,Heller R,Zhang G J,Wang W,Chen Y L,Jiang Y. 2020. The origin of domestication genes in goats[J]. Science Advances,6(21):z5216. doi:10.1126/sciadv.aaz5216.
Zhou Z K,Li M,Cheng H,Fan W L,Yuan Z R,Gao Q,Xu Y X,Guo Z B,Zhang Y S,Hu J,Liu H H,Liu D P,Chen W H,Zheng Z Q,Jiang Y,Wen Z G,Liu Y M,Chen H,Xie M,Zhang Q,Huang W,Wang W,Hou S S,Jiang Y. 2018. An intercross population study reveals genes associated with body size and plumage color in ducks[J]. Nature Communications,9(1):2648. doi:10.1038/s41467-018-04868-4.
Zhu R,Cheng M T,Lu T T,Yang N,Ye S,Pan Y H,Hong T,Dang S Y,Zhang W. 2018. A disintegrin and metalloproteinase with thrombospondin Motifs 18 deficiency leads to visceral adiposity and associated metabolic syndrome in mice[J]. The American Journal of Pathology,188(2):461-473. doi:10.1016/j.ajpath.2017.10.020.
(責任编辑 兰宗宝)

