微生物中自抗性基因导向的基因组挖掘及其在天然产物发现中的应用
谢丽媛,姜逢霖,胡友财
(中国医学科学院&北京协和医学院药物研究所,天然药物活性物质与功能国家重点实验室,北京 100050)
微生物来源的天然产物由于具有结构多样、活性显著等特点,一直以来是药物先导化合物的重要来源。以活性为导向的微生物天然产物研究模式帮助研究人员获得了大量具有药物开发潜力的化合物,曾经引领了新药尤其是抗生素研发的黄金时代。然而,自然界中存在的微生物分布极其广泛,产生的天然产物更是数量巨大,仅采用传统的研究模式已无法实现对活性天然产物的高效挖掘。已知化合物的重复发现、实验室环境下微生物部分代谢产物的缺失等问题亟需开发新技术、新方法来解决[1-2]。
随着基因组测序成本的大幅降低以及生物信息学技术的飞速发展,基因组导向的天然产物发现策略吸引了越来越多的关注。利用生物信息学手段对微生物的基因组数据库进行挖掘,可以高效快速地锁定具有研究价值的生物合成基因簇。基于基因簇中各个基因的功能,可以对天然产物的结构信息进行预测,从而助力研究人员实现目标天然产物的针对性分离。若基因簇在原始产生菌中不表达或表达量低,则可通过引入强启动子、敲除转录抑制因子、过表达转录激活因子、异源表达或报告基因指导下的突变株筛选(reporter-guided mutant selection,RGMS)等策略对该基因簇进行激活,以获得其表达产物[3-5]。
目前基因组挖掘的主要手段之一是基于核心酶进行挖掘。由于催化同一类型化合物骨架形成的核心酶具有序列保守性,可通过同源比对寻找编码核心酶的基因,继而对其上下游基因进行功能分析,获得完整的生物合成基因簇[6-8]。有研究人员针对天然产物药效团的生物合成基因进行基因挖掘,以此发现了许多结构新颖的活性天然产物[9]。
基因组挖掘的另一有效手段是基于自抗性基因进行挖掘,这是发现活性天然产物的另一高效策略。微生物产生的次级代谢产物通过抑制竞争者体内行使基本代谢功能的管家酶活性来杀死竞争者,或限制其生长。当这些代谢产物的产生菌体内也存在同源的管家酶时,为避免自身受到伤害,其天然产物生物合成基因簇内进化出自抗性基因,通过多种自抗性机制来发挥解毒作用。特别地,当簇内的自抗性基因编码了管家酶的同源抗性变体时,该基因还可为天然产物的靶点发现提供线索,这也搭起了天然产物、生物合成基因簇和靶点之间的桥梁[10]。
本文旨在综述微生物中常见的自抗性机制以及基于自抗性基因进行基因组挖掘的策略和应用,并对近年发展起来的相关生物信息学工具进行简要介绍。
1 常见的自抗性机制
微生物为了生存,会合成有毒的次级代谢产物来杀死其竞争者或抑制其生长。为了不被自身代谢产物影响,同时利用它们创造竞争优势,产生菌必须进化出某种自抗性机制,以实现“杀敌不伤己”的效果。常见的自抗性机制包括外排泵、化合物封闭、前药、化学修饰、靶点修饰、DNA 修复和管家酶抗性变体(即自抗酶)等[11](见图1)。此外,某些微生物体内还存在多种自抗性机制的协同作用,从而实现更加有效的自我保护。
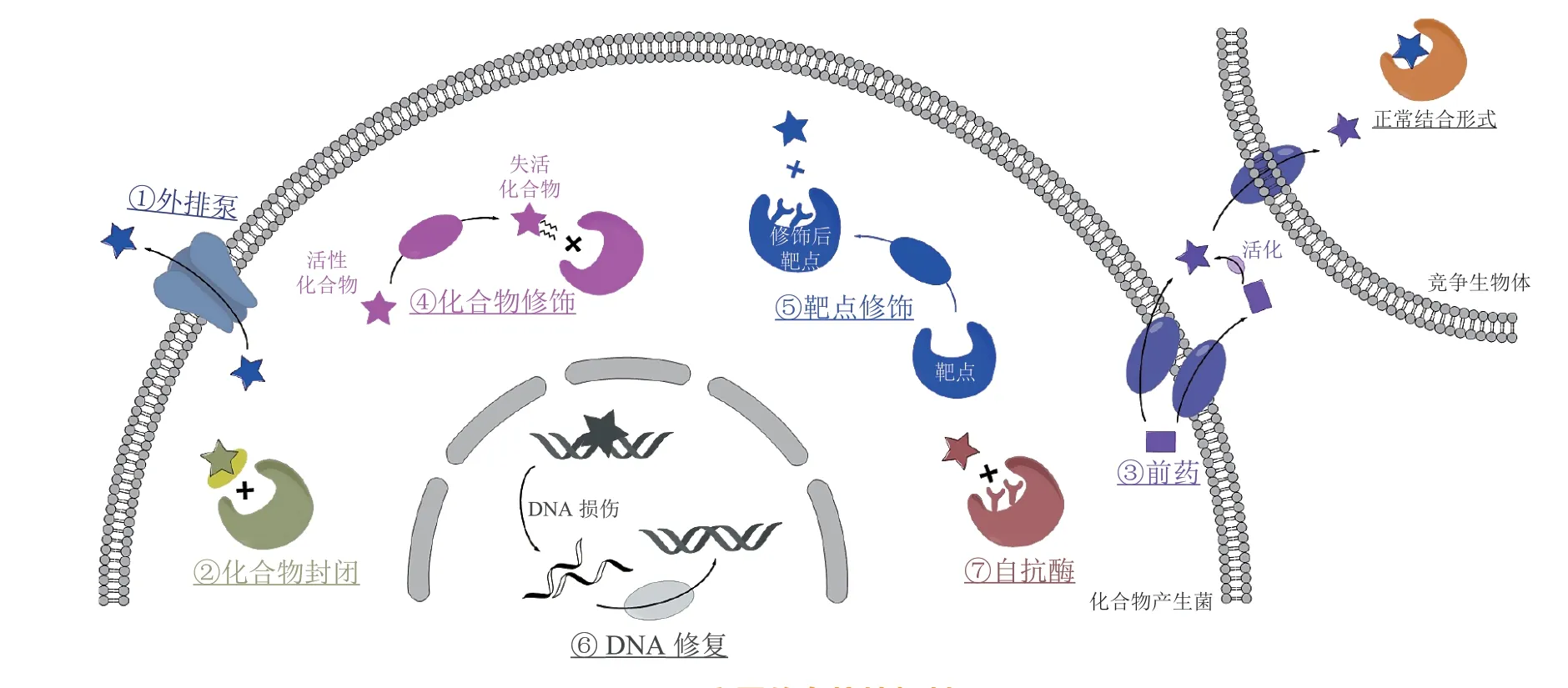
图1 主要的自抗性机制Figure 1 Main mechanisms of self-resistance
1.1 外排泵
外排泵(efflux pumps)可将微生物产生的次级代谢产物排出胞外,从而避免毒性化合物与胞内靶点蛋白相互作用。外排泵主要分为5 类:ATP 结合盒(ATP binding cassette,ABC)家族、主要易化超家族(major facilitator superfamily,MFS)、小多药耐药(small multidrug resistance,SMR)家族、多药与毒物外排(multidrug and toxic microbial extrusion,MATE)家族和耐药结节细胞分化(resistance-nodulation-cell division,RND)家族,其中最常见的是ABC 和MFS 家族。编码外排泵的基因在许多天然产物的生物合成基因簇中均有报道,例如柔红霉素(daunorubicin)生物合成基因簇中的drrA和drrB[12-13],土霉素(oxytetracycline)生物合成基因簇中的otrB[14]以及myxin 生物合成基因簇中的lexABC[15]等,它们与生物合成基因共同表达和调控,准确高效地发挥了自抗性作用。除此之外,部分编码外排泵的基因并不与生物合成基因簇毗邻。例如,基因efrT不在efrotomycin B1 的生物合成基因簇内,但其编码的转运蛋白在efrotomycin B1 的异源宿主体内起到了增强菌株自抗性、提高化合物产量的作用[16]。
1.2 化合物封闭
通过编码化合物结合蛋白,将化合物封闭在蛋白空腔内而不能与靶点蛋白结合的策略,称为化合物封闭。例如,乳酸链球菌素(nisin)可与合成细胞壁的前体lipid Ⅱ结合,插入细菌细胞膜并形成孔洞,造成细菌死亡。为了避免自身的细胞膜受到影响,乳酸链球菌素的产生菌表达了脂蛋白NisI。NisI 不会对乳酸链球菌素进行修饰或降解,而仅仅是将其可逆地封闭起来,使其不能与细胞膜上的靶点结合[17]。在蒽醌骈合的烯二炔类化合物tiancimycin(TNM)的产生菌体内也存在类似的封闭策略,自抗性基因编码的蛋白TnmS1,TnmS2 和TnmS3 形成β-桶状结构,将tiancimycin 封闭在蛋白空腔中,保护产生菌不被烯二炔类化合物极强的毒性作用所影响[18]。同样地,DNA 促旋酶抑制剂closthioamide 产生菌中编码的聚硫酰胺结合蛋白CtaZ[19]、抗肿瘤化合物zorbamycin(ZBM)产生菌中编码的ZBM 结合蛋白ZbmA[20]以及抗结核化合物capreomycin IIB 产生菌中存在的磷酸转移酶Cph[21]也可通过化合物封闭的策略实现自我保护。
1.3 前药
部分微生物在胞内仅产生低毒或无毒的药物前体,称为前药。有些前药在排出胞外后,就会在分泌蛋白或膜蛋白的催化下转化为活性形式,如5-O-磷酸化安普霉素被分泌蛋白AprZ 去磷酸化形成活性终产物安普霉素(apramycin)[22],precolibactin 的N-酰基-D-天冬酰胺部分被周质中的内膜蛋白ClbP 水解形成colibactin[23]等;有些化合物则在产生菌体内一直保持前药状态,直至被摄取后才会转化为活性形式,如microcin C 只有被敏感细胞摄取后,其结构中的N-末端甲酰基和肽基部分才会被去除,形成活性产物[24]。
1.4 化合物修饰
为了降低胞内积累的天然产物对自身产生的毒性,微生物还会合成相应的酶对毒性产物进行化学修饰,如乙酰化、磷酸化、糖基化、氧化还原等,使其转化为低毒产物。例如,bleomycin(BLM)和tallysomycin(TLM)等博来霉素家族化合物需要与金属离子结合形成螯合物,继而在氧气存在时被活化,才能发挥其诱导DNA降解和抗肿瘤的作用[25-27]。为了保证自身不受到伤害,该类化合物的产生菌合成了N-乙酰转移酶,将乙酰基转移到参与金属螯合的其中1 个氨基上,抑制了金属螯合物与氧气的相互作用,避免了活化产物的形成[28-29]。同样地,乙酰转移酶AurG 可将真菌毒素aurovertin E 转化为酰基化的低毒产物aurovertin B[30],磷酸转移酶Cph 和Mur28 可分别使capreomycin IIA 和muraymycin 发生磷酸化失活[21,31]。
1.5 靶点修饰
活性天然产物可以特异性地识别并结合靶点,进而干扰机体的正常生命活动。当靶点的特异性结合位点被修饰后,药物与靶点结合的亲和力会显著降低,从而起到自我保护的效果。例如,dityromycin 可通过结合核糖体蛋白S12 来抑制核糖体移位,从而影响蛋白质的合成。在dityromycin 的产生菌Streptomycessp.中,核糖体蛋白S12 上高度保守的抗生素结合位点发生2 个氨基酸的替换,降低了其与dityromycin 的亲和力,避免了dityromycin对自身的伤害[32]。同样,holomycin 生物合成基因簇中的hom12编码了1 个RNA 甲基转移酶,使得靶点位置的RNA 被甲基化,避免了holomycin 对产生菌体内RNA 合成的抑制作用[33]。糖肽类抗生素的自抗性机制也属于典型的靶点修饰。糖肽类抗生素通过与肽聚糖前体脂质Ⅱ的D-丙氨酰-D-丙氨酸(D-alanyl-D-alanine,D-Ala-D-Ala)末端结合抑制细胞壁的合成。为了避免对自身生命活动的影响,糖肽类抗生素的产生菌对靶点进行了不同方式的修饰,如将肽聚糖前体脂质Ⅱ的D-Ala-D-Ala 末端替换为D-丙氨酰-D-乳酸(D-alanyl-D-lactate,D-Ala-D-Lac)[34-38]或去除末端的D-Ala 残基[39-41]等。
1.6 DNA 修复
抗肿瘤活性化合物能够造成DNA 损伤,影响功能基因的正常转录和翻译,进而导致细胞死亡。为了保证生命活动不受影响,微生物进化出了DNA修复机制。其中,碱基切除修复(base excision repair,BER)机制主要用于消除小的、非螺旋扭曲的碱基损伤,如烷基化、氧化和脱氨基等。例如,yatakemycin(YTM)诱导DNA 发生烷基化,其生物合成基因簇中编码的YtkR2 可启动BER 机制对DNA 进行修复[42]。核苷酸切除修复(nucleotide excision repair,NER)机制则用于去除体积大的、螺旋不稳定的损伤,例如daunorubicin 生物合成基因簇中编码的DrrC 可通过NER 机制将daunorubicin从非共价嵌合部位去除,恢复DNA的正常转录[43-44]。
1.7 管家酶抗性变体(自抗酶)
如前所述,若微生物次级代谢产物靶向的管家酶在产生菌体内保守存在,则该代谢产物必然对产生菌也有毒性作用。因此微生物会在某些代谢产物的基因簇内编码1 个管家酶的抗性变体,即自抗酶。自抗酶与管家酶的氨基酸序列高度相似,具有相同的催化活性;同时自抗酶中存在部分氨基酸突变使得其对代谢产物的敏感性降低,在管家酶被抑制时可以代偿管家酶行使相应的功能[10]。例如,降脂药物洛伐他汀(lovastatin,1,见图2)是通过抑制3-羟基-3-甲基戊二酸单酰辅酶A 还原酶(3-hydroxy-3-methyl glutaryl coenzyme A reductase,HMGR)活性发挥降脂作用,其生物合成基因簇中的lvrA可编码HMGR 的抗性变体,从而发挥自抗作用[45-46];霉酚酸(mycophenolic acid,2,见图2)可通过抑制肌苷-5'-单磷酸脱氢酶(inosine-5'-monophosphate dehydrogenase,IMPDH)的活性来发挥免疫抑制作用,其生物合成基因簇中编码的IMPDH 抗性变体实现了产生菌对霉酚酸的自抗作用[47];烟曲霉素(fumagillin,3,见图2)是一种有效的抗血管生成药物,可通过抑制甲硫氨酸氨肽酶-2(methionyl aminopeptidase 2,MetAP-2)来发挥其药理作用,为了实现自我保护,烟曲霉素的基因簇中编码了MetAP-2 的抗性变体,可在管家酶活性被抑制时行使代偿功能[48]。此外,cladosporin(4,见图2)生物合成基因簇中编码的赖氨酰-tRNA 合成酶同源蛋白Cla4[49]、heptelidic acid(5,见图2)簇中编码的甘油醛-3-磷酸脱氢酶同工酶HepG[50-51]以及 agrocin 84(6,见图2)簇中编码的亮氨酸tRNA 合成酶抗性变体AgnB2[52-53]等也采用了这种策略来抵抗自身代谢产物对产生菌的影响。
1.8 多种自抗性机制协同作用
值得关注的是,许多微生物体内对代谢产物的自抗性并不是通过单一机制实现的,而是利用了多种自抗性机制的协同作用[54]。例如,在萘啶霉素(naphthyridinomycin)[55-56]和A26771B[57]的生物合成过程中,产生菌均同时采用了前药机制和化合物修饰机制,保护自身不被代谢产物的毒性作用所影响。在萘啶霉素的生物合成过程中,低毒前药分泌至胞外后,才会被膜蛋白NapG 和分泌蛋白NapU 活化,转化为活性产物萘啶霉素[55],NapW则在胞内通过化合物修饰将毒性中间体及进入胞内的萘啶霉素转化为低毒形式[56]。A26771B 生物合成基因簇中编码的分泌蛋白BerkD 可将排出胞外的低毒前药berkeleylactone E 转化为活性化合物A26771B。当A26771B 进入胞内时,短链还原酶/脱氢酶BerkC 还可将A26771B 的C-4 位羰基还原为羟基来降低其毒性[57]。同样地,在最小霉素(minimycin)的生物合成过程中也存在多种自抗性机制的协同作用。最小霉素生物合成基因簇中编码的MinCN 可将毒性中间体最小霉素单磷酸的磷酸基团脱去,从而缓解其毒性作用;同时,尿嘧啶磷酸核糖转移酶MinD 可催化形成大量尿苷单磷酸,与胞内残留的最小霉素单磷酸竞争靶点,从而降低最小霉素单磷酸与靶点的结合能力,最终实现对宿主的自我保护[58]。
2 基于自抗性基因的基因组挖掘在天然产物研究中的应用
自抗性基因将天然产物的生物合成基因簇与其生物活性联系起来,为基因组挖掘提供了新策略[10,59]。一方面,自抗性基因通常与天然产物生物合成基因簇共定位,可作为基因簇的“定位标签”,用于指导天然产物生物合成基因簇的发现;另一方面,自抗性基因还可作为“活性标签”,指示基因簇产物的生物活性,或用于对基因组数据库进行广泛挖掘,探索特定活性天然产物的生物合成基因簇。
2.1 活性天然产物生物合成基因簇的发现
在自抗性基因指导基因簇发现的早期研究阶段,研究人员基于自抗功能来寻找目标基因簇,即利用代谢产物对产生菌的基因组文库进行抗性筛选,能够赋予基因文库受体菌抗性的片段必然包含自抗性基因,而基因组中与自抗性基因毗邻的生物合成基因簇最有可能参与该代谢产物的生物合成。借助这一策略,杀稻瘟菌素(blasticidin S,BS)(7,见图3)和andrimid(8,见图3)等化合物的生物合成基因簇相继被鉴定出来。BS 是来源于Streptomyces griseochromogenes的肽基核苷类抗生素,广泛应用于植物病原真菌Pyricularia oryzae的防治[60]。Cone 等[61]利用BS 对S. Griseochromogenes的基因组文库进行抗性筛选,以获得的抗性DNA 片段作为探针成功鉴定到BS 的完整生物合成基因簇bls。该基因簇包含19 个开放阅读框,抗性DNA 探针中包含的blsJ编码了1 个ABC 家族的转运蛋白,可将毒性产物转运出去以避免对自身的伤害[62]。除此之外,簇中blsK编码的氨酰tRNA 转移酶通过催化低毒药物前体亮氨酰脱甲基杀稻瘟菌素(leucyldemethylblasticidin S,LDBS)和亮氨酰杀稻瘟菌素(leucylblasticidin S,LBS)的形成也参与了对BS 的自抗[63-64]。采用类似的策略对成团泛菌(Pantoea agglomerans)的基因组文库进行筛选,Jin 等[65]获得了乙酰辅酶A 羧化酶(acetyl CoA carboxylase,ACC)抑制剂andrimid 的完整生物合成基因簇adm。其中admQ编码的MFS 家族转运蛋白负责将胞内积累的andrimid 转运出去,admT编码的自抗酶与管家酶乙酰辅酶A 羧化酶β 亚基AccD 的氨基酸序列高度相似,但203 位的亮氨酸突变为甲硫氨酸使得AdmT 对andrimid 的抗性显著增加[65-67]。

图3 自抗酶基因指导下鉴定生物合成基因簇的天然产物示例Figure 3 Examples of natural products identifying biosynthetic gene clusters guided by self-resistant enzyme genes
随着对自抗酶的认识不断深入,研究人员开始将编码自抗酶的基因作为基因簇筛选的条件之一。当化合物的作用靶点(即管家酶)已知时,可根据序列相似性寻找基因组中的抗性拷贝,继而获得完整的生物合成基因簇。霉酚酸(2,见图2)是由青霉属真菌产生的免疫抑制剂,通过抑制IMPDH 的活性来发挥其药理作用[68]。利用产生菌Penicillium brevicompactum基因组中克隆得到的IMPDH 序列片段作为探针对基因组文库进行筛选,Regueira 等[69]获得了2 组编码IMPDH 的DNA 片段。由于原始IMPDH 基因周围总是存在1 个编码rasGTPase 激活蛋白的基因,作者利用排除法成功鉴定到霉酚酸的生物合成基因簇mpa。异源表达实验结果表明,簇内mpaF编码的IMPDH 对霉酚酸具有显著的自抗作用[47]。Coumermycin A1(9,见图3)是由Streptomyces rishiriensis产生的香豆素类化合物,具有抑制DNA 促旋酶活性的作用。Wang 等[70]同时以核心基因(dTDP-葡萄糖4,6-脱水酶基因)和抗性促旋酶基因gyrBr作为探针对产生菌的基因组文库进行筛选,成功定位到coumermycin A1 的生物合成基因簇cum。
由于基因组测序成本大幅降低,仅通过生物信息学手段对基因组数据进行分析,而不必借助繁杂的实验操作就可对基因簇进行初步定位。烟曲霉素(3,见图2)是由萜类化合物fumagillol 和聚酮decatetraenedioic acid 酯化形成的MetAP-2 抑制剂[71]。Lin 等[48]对烟曲霉素产生菌Aspergillusfumigatus的基因组进行筛选,发现fma基因簇中同时存在高度还原型聚酮合酶(highly-reducing polyketide synthase,HR-PKS)基因af370和MetAP-2基因af410,最有可能参与烟曲霉素的生物合成。基因敲除和体外生化实验结果表明,fma基因簇中聚酮合酶(polyketide synthase,PKS)和酰基转移酶负责聚酮部分的生物合成,而簇中编码的新型萜环化酶fma-TC 能够催化法尼基焦磷酸(farnesyl pyrophosphate,FPP)转化为烟曲霉素的中间体β-顺式-佛手柑油烯。Kato 等[72]利用潮霉素B 对Fusariumsp. RK97-94 进行诱导,在代谢产物中分离得到3-羟基-3-甲基戊二酸单酰辅酶A 合成酶(3-hydroxy-3-methyl glutaryl coenzyme A synthase,HMGS)抑制剂1233A(10,见图3)[73]。结合转录组数据和基因组数据,作者发现在潮霉素诱导下转录水平升高的7 个基因簇中,3 号基因簇同时存在负责骨架形成的PKS 基因和HMGS 基因,于是将该基因簇确定为候选基因簇。基因敲除实验结果表明,簇中各基因均与HMGS 抑制剂1233A 的生物合成相关,这也说明自抗酶在基因簇鉴定过程中可作为重要的筛选条件[72]。
当天然产物产生菌的基因组尚未测序或基因组数据难以获得时,还可利用抗性基因对公开数据库进行挖掘,在新的产生菌中获得目标基因簇。例如,restricticin(11,见图3)和lanomycin(12,见图3)能够通过与血红素铁配位发挥抑制甾醇14α-去甲基化酶(sterol 14α-demethylase,CYP51)活性的作用,但由于已知产生菌Penicillium restrictum和Pycnidophora dispersea的基因组尚未测序,这2 个化合物的生物合成途径迟迟未能得到解析。Liu 等[74]采用自抗酶导向的基因组挖掘策略,利用其课题组开发的算法对公共数据库进行挖掘,发现了5 个同时编码CYP51 和相关生物合成酶的基因簇。其中rstn簇参与了restricticin 的生物合成,簇中各基因的功能也通过异源表达和体外生化反应得到了阐明。另外,在Curvularia lunata和Pyrenophora dematioidea(TTI-1096)中均保守存在的基因簇则参与了lanomycin 的生物合成。研究人员利用单菌株多次级代谢产物(one strain many compounds,OSMAC)策略对P. dematioidea中该基因簇进行激活,发现利用Wheat1 培养基进行发酵时,在发酵产物中检测到了lanomycin 的产生。
值得注意的是,有些基因簇中编码的管家酶同源蛋白未必行使自抗功能,而是负责化合物的一步生物合成。例如,SB-203207(13,见图4A)和SB-203208(14,见图4A)是Streptomycessp.NCIMB 40513 产生的异亮氨酸tRNA 合成酶抑制剂。利用自抗性基因指导的基因组挖掘策略,Hu等[75]鉴定了化合物13 和14 的生物合成基因簇sbz(见图4A)。但对簇中各基因功能进行研究时,作者发现sbz簇中编码的异亮氨酸合成酶SbzA 催化tRNAIle氨酰化的效率远不如基因组中的管家酶Ssp_IleRS,且SbzA 可以在Ile-tRNAIle存在时将异亮氨酸转移到中间体altemicidin 上,即参与了化合物的生物合成(见图4B)。提示当生物合成酶与管家酶的催化功能存在相似性时,可能会对自抗性基因导向的基因组挖掘造成信息误导,需要仔细甄别。

图4 未行使自抗功能的自抗酶基因举例Figure 4 Examples of self-resistant enzyme genes without self-protection function
2.2 自抗性基因导向的天然产物活性研究
由于自抗性基因具有“活性标签”的特点,研究人员逐渐意识到,天然产物的生物合成基因簇除了能够阐明生物合成途径,还为天然产物的生物活性研究提供了线索。例如,rumbrins 类化合物是含氯多烯吡咯类化合物,具有抗癌、抗脂质过氧化和细胞保护作用等多种药理活性[76-77]。笔者课题组[78]在研究rumbrin(15,见图5)的生物合成过程中,发现了1 个潜在的自抗酶RumB。簇中基因rumB在rumbrin 的生物合成中保持转录,但并不参与其生物合成。生物信息分析表明,RumB 与人源硫酯酶Ⅱ(human thioesterase II,hTE)具有较高的同源性,hTE 是人类免疫缺陷病毒(human immunodeficiency virus,HIV)辅助蛋白Nef 在宿主中的分子伴侣,在HIV 感染的早期阶段发挥了重要作用。因此笔者课题组推测rumbrins 类化合物可能通过靶向hTE 来发挥抗HIV 活性。体外活性评价实验证明,大多数rumbrins 对HIV 复制的早期阶段具有明显的抑制作用(纳摩尔级),并且化合物12E-rumbrin 的抑制率与阳性药相当,这与生物信息分析的结果一致。同时,笔者课题组在对同源基因簇进行分析时,发现参与malbranpyrroles 生物合成的mas簇中缺少rumB的同源基因。相应地,malbranpyrroles 展示出极弱的抗HIV 活性。以上结果表明自抗酶对化合物靶点发现具有启示作用,为基于自抗性基因发现先导化合物及其作用靶点提供了新的思路。

图5 自抗性基因指导下阐明天然产物靶点的示例Figure 5 Natural products targets elucidated under the guidance of self-resistant enzyme genes
在浅灰霉素类化合物(griselimycins)作用靶点的发现过程中,自抗酶基因也发挥了不可或缺的作用。浅灰霉素(griselimycin,GM,16,见图5)由Streptomycessp. DSM-40835 产生,具有抗多药耐药结核分枝杆菌活性。Kling 等[79]鉴定了GM 的生物合成基因簇,并在簇中发现了dnaN的同源基因griR,dnaN负责编码管家酶DNA 聚合酶的滑动钳部分,在DNA 复制过程中发挥重要作用。由于griR基因的引入可使原本对GM 敏感的异源宿主产生抗性,且表面等离子共振(surface plasmon resonance,SPR)结果显示GriR 与GM 的亲和力远低于DnaN,因此作者推测GriR 为基因簇中编码的自抗酶,而DnaN 为浅灰霉素类化合物的靶点。Obafluorin(17, 见图5) 是由Pseudomonas fluorescensATCC 39502 产生的β-内酯类化合物,对革兰阳性菌和阴性菌均有抑制作用。Scott 等[80]发现在obafluorin 的生物合成基因簇oba中存在1 个编码苏氨酰-tRNA 合成酶的基因obaO,当obaO被敲除时,敲除菌株对obafluorin 的敏感性显著增加,且导入obaO的大肠埃希菌显示出了对obafluorin的抗性。因此,作者推测obafluorin 的靶点为苏氨酰-tRNA 合成酶,而ObaO 为基因簇中的自抗酶。体外生化实验验证了这一推测。当存在1 μmol · L-1的obafluorin 时,管家酶苏氨酰-tRNA 合成酶可被obafluorin 完全抑制,而在最大浓度的obafluorin 中自抗酶ObaO 依然保持部分活性。同样地,Xie 等[81]在研究harzianic acid(18,见图5)的靶点时也采用了自抗性基因导向的策略。由于Trichoderma afroharzianumt-22(Tht22)体内参与harzianic acid生物合成的基因簇中编码了1 个乙酰羟酸合成酶(acetohydroxyacid synthase,AHAS,在该真菌中命名为ThAHAS),作者推测harzianic acid 的作用靶点为AHAS。结合生物信息分析、体外生化反应和对ThAHAS 的晶体结构研究,作者发现harzianic acid 不仅可抑制原始的AHAS,对P188 发生突变的抗性AHAS 也产生了明显的抑制作用。
2.3 自抗性基因导向的新化合物发现
除了为天然产物的生物活性研究提供线索之外,自抗性基因还可用于指导海量基因组数据中特定活性化合物生物合成基因簇的发现,从而获得新的活性化合物。Tang 等[82]基于自抗酶基因的特点开发出一套完整的基因组挖掘流程,并利用这一流程对盐孢菌属细菌基因组进行了广泛的挖掘。利用同源基因聚类、直系同源组功能鉴定等方法,作者在参与脂质转运和代谢的直系同源组中发现了管家酶FabB/F 的同源蛋白Salin8269。Salin8269 位于tlm基因簇内,当对tlm簇进行异源表达时,作者在代谢产物中分离到具有Ⅱ型脂肪酸合成酶(fatty acid synthase type II,FASII)抑制活性的thiotetronic acids 类化合物(19 ~ 22,见图6),其中包括已知的thiolactomycin(TLM)。同时,作者利用同源比对方法,在Streptomyces afghaniensis中找到了类似的基因簇ttm,并在其异源表达产物中分离到一系列新的TLM 同系物(23 ~ 26,见图6)。Thiotetronic acids 类化合物的发现证实了基于自抗酶进行基因组挖掘这一策略的可行性。

图6 以自抗性基因为导向挖掘的天然产物示例Figure 6 Discovery of natural products guided by self-resistant gene
自抗性基因导向的基因组挖掘在新型除草剂的开发中也发挥了重要作用。支链氨基酸(branchedchain amino acid,BCAA)的生物合成对植物的生长至关重要,由于该途径在动物中并不存在,因此是除草剂开发的理想靶点。BCAA 的生物合成过程中有3 个酶参与,包括乙酰乳酸合酶(acetolactate synthase,ALS)、乙酰羟基酸异构还原酶(ketolacid reductoisomerase,KARI)和二羟基酸脱水酶(dihydroxyacid dehydratase,DHAD)。Zhang 等[83]以ALS 为探针对459 个真菌测序基因组进行筛选,发现了4 个含有ALS 编码基因的生物合成基因簇,其中Aspergillus candidus基因组中的cfo簇参与了氯黄酮(27,见图6)的生物合成。氯黄酮可有效抑制拟南芥种子萌发和病原菌生长,且基因敲除和异源表达实验证实,簇中ALS 编码基因cfoL参与了A. candidus对氯黄酮的自抗作用。Yan 等[84]则在已测序的真菌基因组中对与生物合成核心基因共定位的DHAD 基因进行挖掘,发现在多个真菌基因组中保守存在的ast簇编码了1 个DHAD 同源蛋白AstD。作者利用酿酒酵母对ast簇进行异源表达,并分离得到基因簇产物aspterric acid(28,见图6)。Aspterric acid 表现出对多种植物的生长和根系发育的抑制作用[84-85],这使其有望发展成为新一代广谱除草剂。体外生化反应和异源表达实验结果表明,aspterric acid 可以竞争性地抑制管家酶DHAD 的活性,而ast簇中编码的同源蛋白AstD 在aspterric acid 的浓度达到溶解度极限(8 mmol · L-1)的情况下,依然可以行使正常的催化功能。同时,当拟南芥中导入密码子优化的astD基因时,转基因植株表现出对aspterric acid 的抗性。提示astD基因可用于抗aspterric acid 作物的培育,这也使得aspterric acid的除草剂开发更具前景。
Elfamycins 是一系列具有链延长因子Tu(elongation factor Tu,EF-Tu)抑制活性的抗生素[86],由kirromycin,enacyloxin IIa,pulvomycin 和GE2270A 这4 类结构不同的化合物组成。Kirromycin通过阻断核糖体上EF-Tu·GDP(guanosine diphosphate,二磷酸鸟苷)的构象变化而发挥抑制作用[87],链霉菌中针对kirromycin 的抗性突变主要发生在EF-Tu 的375 位丙氨酸(A375T)上。Yarlagadda 等[88]基于抗性EF-Tu(A375T,EF-TuKirR)基因对链霉菌基因组进行挖掘,发现3个含有潜在elfamycins 生物合成基因簇的菌株,其中Streptomyce cattleya已知可以产生elfamycin L-681217。此外,作者在筛选到的S. sulphureusATCC 27468 代谢产物中分离得到phenelfamycin A(29,见图6)和phenelfamycin B(30,见图6),其中phenelfamycin B 可通过干扰EF-Tu 抑制蛋白质合成,具有抗耐药淋病奈瑟菌的活性。
ClpP 蛋白酶是ATP 依赖的蛋白水解酶,在原核及真核生物的线粒体中广泛存在。Culp 等[89]以ClpP 为探针进行基因组挖掘,筛选到10 条含有clpP基因的生物合成基因簇,并对其中同时包含双模块非核糖体肽合成酶(nonribosomal peptide synthase,NRPS)基因和Ⅰ型PKS 基因的cac基因簇进行了深入研究。由于cac基因簇异源表达产物可对ClpP 进行共价修饰(ClpP 相对分子质量增加约328 000),作者认为cac簇必然参与了共价ClpP 抑制剂的生物合成,并基于以上结果分离得到cac簇的真实产物clipibicyclene(31,见图6)。Clipibicyclene 可显著抑制管家酶ClpP 的活性,且cac簇中编码的ClpP 同源蛋白(Cac16 和Cac17)参与了产生菌对clipibicyclene 的自抗作用。
有些自抗性基因虽然并非编码管家酶抗性变体的基因,但由于在特定靶点抑制剂的生物合成基因簇中广泛存在,也可用于指导基因组挖掘。例如,拓扑异构酶是抗肿瘤药物和抗生素作用的重要靶点,而五肽重复蛋白(pentapeptide repeat protein,PRP)提供了微生物对拓扑异构酶抑制剂的抗性[90]。Panter 等[91]以PRP 为探针,对黏细菌基因组进行自抗性基因导向的基因组挖掘,在PyxidicoccusfallaxAn d48 中发现了新型拓扑异构酶抑制剂pyxidicycline A(32, 见图6) 和pyxidicycline B(33,见图6)。Li 等[92]则利用普遍存在的四环素类化合物自抗性基因(tetR/marR和邻近转运蛋白基因如tetA)以及核心酶基因(PKS 基因和链延长因子基因)对国家生物技术信息中心(National Center of Biotechnology Information,NCBI)中已注释的20 000 余个基因组进行筛选,通过对候选基因簇中链延长因子的进化树分析,发现了30 个可能参与四环素类化合物生物合成的基因簇。经实验验证,其中的hai基因簇为高度糖基化的新颖四环素类化合物hainancycline(34,见图6)的生物合成基因簇。
2.4 生物信息学工具的开发
随着基因组数据的爆炸式增长,对基因组数据进行手动挖掘已难以满足科研人员的需求。因此许多生物信息学工具涌现出来,这为研究者基于自抗性基因进行基因组挖掘提供了有力帮助。
AntiSMASH(antibiotics and secondary metabolites analysis shell,https://antismash.secondarymetabolites.org)是广泛应用于细菌和真菌生物合成基因簇分析的生物信息学平台。2019 年推出的antiSMASH 5.0使用耐药蛋白数据库Resfams 的隐马尔可夫模型(profile Hidden Markov Models,pHMMs)对基因簇中潜在的自抗性基因进行注释,为自抗性基因导向的基因组挖掘提供了条件[93]。ClusterTools(https://github.com/emzodls/clusterArch)是基于BLAST(basic local alignment search tool)和pHMMs 构建的基因组挖掘软件。用户以核心酶或存在特定结构域组合的蛋白为筛选条件,寻找感兴趣的基因簇。除了对参与生物合成的基因进行筛选之外,clusterTools 还可将参与基因调控和自抗的基因作为筛选条件,因而可用于自抗性基因导向的基因组挖掘[94]。
Alanjary 等[95]开发了搜索引擎Antibiotic Resistant Target Seeker(ARTS,https://arts.ziemertlab.com)。该网站基于3 个标准对自抗性基因进行筛选:是否存在管家基因的重复拷贝;是否定位于生物合成基因簇内;是否存在水平基因转移现象。筛选结果最终汇总到交互式输出表中,用户可根据需要进行筛选和排序,非常便捷。最初版本的ARTS 只能应用于放线菌中,2020 年更新的ARTS 2.0[96]则可对所有细菌基因组和宏基因组数据进行分析。此外,ARTS 2.0还可对不同基因组中的相似基因簇及簇中的自抗性基因进行比较。由于ARTS 只能充当网络服务器,每次只能执行单项分析任务,且需要一定的时间进行数据处理,开发者们又推出了ARTS-DB 数据库(https://arts-db.ziemertlab.com/)[97]。ARTS-DB 提供了70 000 余个基因组和宏基因组预先计算的ARTS 结果,用户可根据ARTS 筛选的基本标准(基因拷贝、临近基因簇和水平基因转移)对数据库进行检索。不仅如此,ARTS-DB 数据库中还提供了其他含有相关信息数据库的链接,便于用户对筛选结果进行进一步分析。
Stahlecker 等[98]开发了基于系统发育对目标基因周围片段(neighborhoods of gene of interest,NGIs)进行可视化分析的工具——SYN-View(https://bitbucket.org/jstahlecker/syn-view/)。由于在亲缘关系接近的菌株中管家基因的NGIs 往往保守存在,而自抗性基因的NGIs 之间存在明显差异,因此SYN-View 的可视化结果可作为ARTS 等分析工具的补充,对ARTS 筛选得到的自抗性基因进行进一步筛选和确认。
Vandova 等[99]开发了与ARTS 类似的算法(https://github.com/GerganaVandova/TargetMining)。该算法除了采用基因的重复拷贝以及自抗性基因与生物合成基因簇之间的距离作为筛选标准之外,还增加了对自抗酶与核心酶之间共进化关系的分析,对核心基因与自抗性基因之间距离的要求也更严格。作者利用阳性对照组验证了算法的可行性,并对NCBI 数据库中的PKS 基因簇进行了筛选和排序,筛选结果可为新型抗生素的研发提供参考。
Kjærbølling 等[100]开发的真菌自抗性基因导向的基因组挖掘(fungal resistance gene-directed genome mining,FRIGG)管线也是基于自抗酶基因的特点进行基因挖掘的:自抗酶基因位于基因簇内,并且是基因簇内管家基因的同源拷贝。该管线包含5 步筛选过程,对存在自抗性基因的基因簇进行筛选并过滤掉可能存在的假阳性结果。利用主成分分析(principal-component analysis,PCA)、进化树分析、功能注释和BLASTp(对蛋白质的氨基酸序列进行同源性比对,寻找同源蛋白或对蛋白质进行功能预测)对筛选得到的候选基因簇进行进一步分析,可使筛选更高效。利用这一筛选策略,作者在51 株曲霉菌和青霉菌的基因组中鉴定到72 个潜在的自抗性基因,其中包含已经得到功能表征的自抗性基因inpE(参与产生菌对fellutamide B 的自抗性机制),证实了该工具的有效性。值得注意的是,该管线更适用于对亲缘关系较为接近的真菌基因组进行分析,若数据库中包含亲缘关系较远的真菌,则需要对筛选参数进行适当调整。
3 总结与展望
自抗性是保证产生天然产物的微生物“杀敌不伤己”的重要机制,也为后基因组时代的基因组挖掘提供了新的策略和思路。本文归纳了微生物常见的自抗性机制,对自抗性基因导向的基因组挖掘策略及其应用进行了分类介绍,同时还对已开发的基于自抗性基因进行基因组挖掘的生物信息学工具进行了汇总。尽管基于自抗性基因进行基因组挖掘的策略卓有成效,但仍存在一些亟待解决的问题。例如,自抗性基因可能存在于基因组中的其他位置而非基因簇内[101];部分自抗性机制与天然产物的生物合成途径融合,自抗性基因编码的酶既具有抗性,同时又作为天然产物的生物合成途径酶而存在[102];某些生物合成基因簇中的管家基因拷贝并未编码抗性酶,而是参与了化合物生物合成[75]等。随着对天然产物生物合成以及自抗性机制的深入研究,研究人员有望利用人工智能、机器学习等手段设计出更加合理高效的生物信息学工具和数据库,更精准地预测生物合成基因簇、基因簇产物及天然产物的靶点,从而实现对微生物中天然产物资源更好的开发和利用。

