人体微生物天然产物研究进展
张雨薇 ,刘煜峰 ,陈义华 *
(1. 中国科学院微生物研究所微生物资源前期开发国家重点实验室,北京 100101;2. 中国科学院大学生命科学学院,北京 100049;3. 中国科学院大学存济医学院,北京 100049)
上世纪40 年代以来,土壤、海洋等环境中的微生物所产生的活性天然产物成为药物的重要来源之一[1]。在2 代测序技术发展起来以前,人们已经认识到人体中存在着大量的微生物,并通过传统手段分离获得一些它们所产生的活性化合物,从而拉开了人体微生物天然产物研究的序幕。近年来,人类微生物组计划(Human Microbiome Project,HMP)[2]、人类肠道宏基因组计划(Metagenomics of the Human Intestinal Tract,MetaHIT)[3]等组学研究的开展,更新了研究人员对人体内微生物数量和多样性的了解,也拓展了其对人体微生物天然产物的结构类型和丰富程度的认识。人体微生物作为活性天然产物的潜在重要来源的概念正逐渐被更多的研究者认可[4-8]。
与环境来源的微生物相比,人体微生物经历了与人体环境的长期共同进化过程[9],适应了人体和人体微生物群落构成的复杂而独特的内部生态系统。这决定了人体微生物产生的小分子化合物天然靶向人体或人体微生物组来行使信号传递、免疫调节、种间竞争等功能的活性特征[5]。与人体健康之间的天然联系暗示着人体微生物天然产物具有良好的成药性。另外,有些病原菌所产生的天然产物与其致病性密切相关,对这类天然产物的研究将有助于发展预防和治疗相关疾病的新型诊疗手段[4,10]。基于相关认知,近年来人体微生物天然产物研究备受重视,得到了快速发展。本文将以发现人体微生物活性天然产物的策略为脉络,介绍近年来报道的代表性天然产物的发现方法、结构特点及其活性特征,以期为进一步优化人体微生物活性天然产物的挖掘方法、促进相关领域的研究提供思路。
1 基于传统策略发现人体微生物天然产物
如前所述,在2 代基因测序技术发展起来以前,研究者们利用传统的天然产物发现策略已获得了一些人体微生物产生的活性化合物。这一策略的实现依赖于对菌株的分离和培养,之后在活性指导下借助高效液相色谱法(high performance liquid chromatography,HPLC)、 质谱(mass spectrum,MS)等手段对菌株代谢物进行分析,并利用液相色谱-质谱法(liquid chromatography/mass spectrometry,LC/MS)、串联质谱法(tandem mass spectrometry,MS/MS)、核磁共振(nuclear magnetic resonance,NMR)等经典的化学手段对目标化合物进行结构表征(见图1)。利用传统的天然产物发现策略在人体微生物中获得的活性天然产物大多为核糖体合成和翻译后修饰多肽类(ribosomally synthesized and post-translationally modified peptides,RiPPs) 化合物。它们通常抑制亲缘关系较近的物种,具有较窄的抗菌谱,在协助产生菌占据和维持生态位方面发挥作用。在以往的很多研究中对这类化合物都进行了详细的介绍[5,7],图1 简单列举了几种代表性化合物。从表皮葡萄球菌(Staphylococcus epidermidis)中分离的表皮抗菌肽(epidermin)能够有效抑制与表皮葡萄球菌竞争皮肤生态位的痤疮丙酸杆菌(Propionibacterium acnes)[11];由唾液链球菌(Streptococcus salivarius)产生的salivaricin B 对唾液链球菌、化脓链球菌(Streptococcus pyogenes)、轻型链球菌(Streptococcus mitis)、藤黄微球菌(Micrococcus luteus)等常见人体微生物具有抑制活性[12];大肠埃希菌(Escherichia coli)能够产生多种相对分子质量较小的小菌素(microcins),其中MccC7 具有腺苷酸化修饰的C 端,对肺炎克雷伯菌(Klebsiella pneumoniae)、沙门菌属(Salmonella)、耶尔森菌属(Yersinia)等微生物都具有抑制活性[13]。
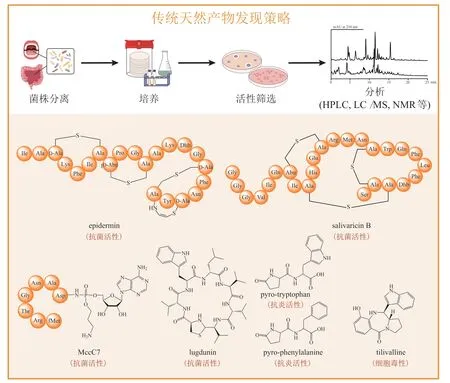
图1 基于传统策略获得人体微生物活性天然产物Figure 1 Obtaining bioactive natural products from human microbiota based on traditional strategies
除此之外,利用传统的天然产物发现策略也从人体微生物中发现了少数其他类型的化合物。与其他生境中的生物一样,人体微生物之间也存在着激烈的营养和生态位竞争,它们在长期的共存中发展出了能够相互抑制的“分子武器”。在人体的皮肤或上呼吸道这类营养物质贫乏的环境中,人体微生物之间的竞争可能尤为激烈[14-15]。基于这一生态学假设,Zipperer 等[16]从一系列鼻腔共生菌中筛选到了可以抑制同一生态位的条件致病菌——金黄色葡萄球菌(Staphylococcus aureus)生长的路邓葡萄球菌(Staphylococcus lugdunensis)IVK28。随后,通过非靶向转座子诱变,作者以抑制金黄色葡萄球菌活性为指示,对突变株与野生型菌株的代谢谱进行比对分析后,分离得到了具有良好抗菌活性的非核糖体多肽(non-ribosomal peptides,NRPs)类化合物lugdunin(见图1)。此外,lugdunin 还显示出对耐甲氧西林金黄色葡萄球菌 (methicillin-resistantS.aureus,MRSA)、耐万古霉素肠球菌(vancomycinresistantEnterococcus,VRE)和单核细胞增生李斯特菌(Listeria monocytogenes)的良好抑制活性,并在小鼠金黄色葡萄球菌皮肤感染模型中起到了抑制感染的作用。后续研究中,Bitschar 等[17]还发现lugdunin 能够与人体产生的抗菌肽DCD-1L 以及LL-37 协同作用,发挥对MRSA 的杀菌作用,并能够增强人原代角质形成细胞的先天免疫反应。这些结果显示出lugdunin 潜在的应用价值,也展现了生态学观念的引入在人体微生物活性天然产物挖掘过程中的重要性。
类似地,研究者们利用活性导向的天然产物发现方法对一些人体益生菌的产物进行了表征。多项研究表明,益生菌植物乳杆菌(Lactobacillus plantarum)能够减少体内炎症[18],其发酵液在体外试验中也显示出对核因子κB(nuclear factorkappa B,NF-κB)信号通路相应的调节功能[19]。通过主成分分析(principal component analysis,PCA)探究植物乳杆菌不同生长阶段代谢产物的差异,Zvanych 等[20]从中鉴定出了4 个具有独特焦谷氨酸环结构的pyro-dipeptides 类化合物。其中,pyro-phenylalanine 和pyro-tryptophan(见图1)在小鼠腹腔注射实验中能够显著减少脾脏产生的γ 干扰素(interferon-gamma,IFN-γ),显示出这类化合物潜在的抗炎能力。
除了挖掘对人类健康有益的人体微生物天然产物,研究者们还利用传统的天然产物发现策略研究了人体病原菌的毒力因子,其中最典型的例子就是tilivalline(见图1)的发现。2014 年,Schneditz 等[21]以对人上皮细胞的细胞毒作用为指示,利用转座子诱变,从引发抗生素相关出血性结肠炎(antibioticassociated hemorrhagic colitis,AAHC)的病原菌产酸克雷伯菌(Klebsiella oxytoca)中鉴定出该菌产生的细胞毒分子tilivalline,通过体外试验和动物模型验证了tilivalline 对肠道黏膜和屏障功能的破坏,揭示了产酸克雷伯菌引发结肠炎的可能机制之一,并提供了潜在的诊断标志物和治疗策略。可见,研究人体病原菌产生的天然产物也能够促进病原菌相关疾病预防和治疗手段的发展。
利用传统策略挖掘人体微生物产生的活性天然产物具有不依赖于序列预测、能够以目标活性或结构特征为导向等优势。然而,该方法在应用过程中也存在不少限制因素,例如:对可培养菌株的依赖性,以及难以避免对已知化合物的重复发现。令人鼓舞的是,培养组学(culturomics)的应用和发展使得越来越多的人体微生物能够被培养[22-23],该方法首次在多种人体粪便样品中应用就将可培养的人体肠道细菌数量从原先的688 种增加到了1 057种[24];此外,基于LC/MS,MS/MS 和NMR 数据的去重复方法的进步也赋予了传统天然产物发现方法新的活力[25],这一策略依然能够在未来人体微生物活性天然产物发现中提供有力的帮助。
2 基于基因组挖掘方法获得人体微生物天然产物
自HMP 等计划开展以来,对于人体微生物领域的持续研究积累了大量的组学数据,这让研究人员越来越清晰地认识到人体中蕴藏有丰富的微生物和微生物代谢产物资源[26-29]。另外,基因组文库高通量筛选方法的优化、(宏)基因组数据中生物合成基因簇(biosynthetic gene clusters,BGCs)识别算法和分析工具的不断更新,以及针对不同种类微生物遗传操作方法和异源表达系统的建立,有力地推动了通过基因组挖掘来获取人体微生物天然产物的方法进步。相关研究根据出发点不同可以分为功能导向和序列导向2 种策略(见图2)。
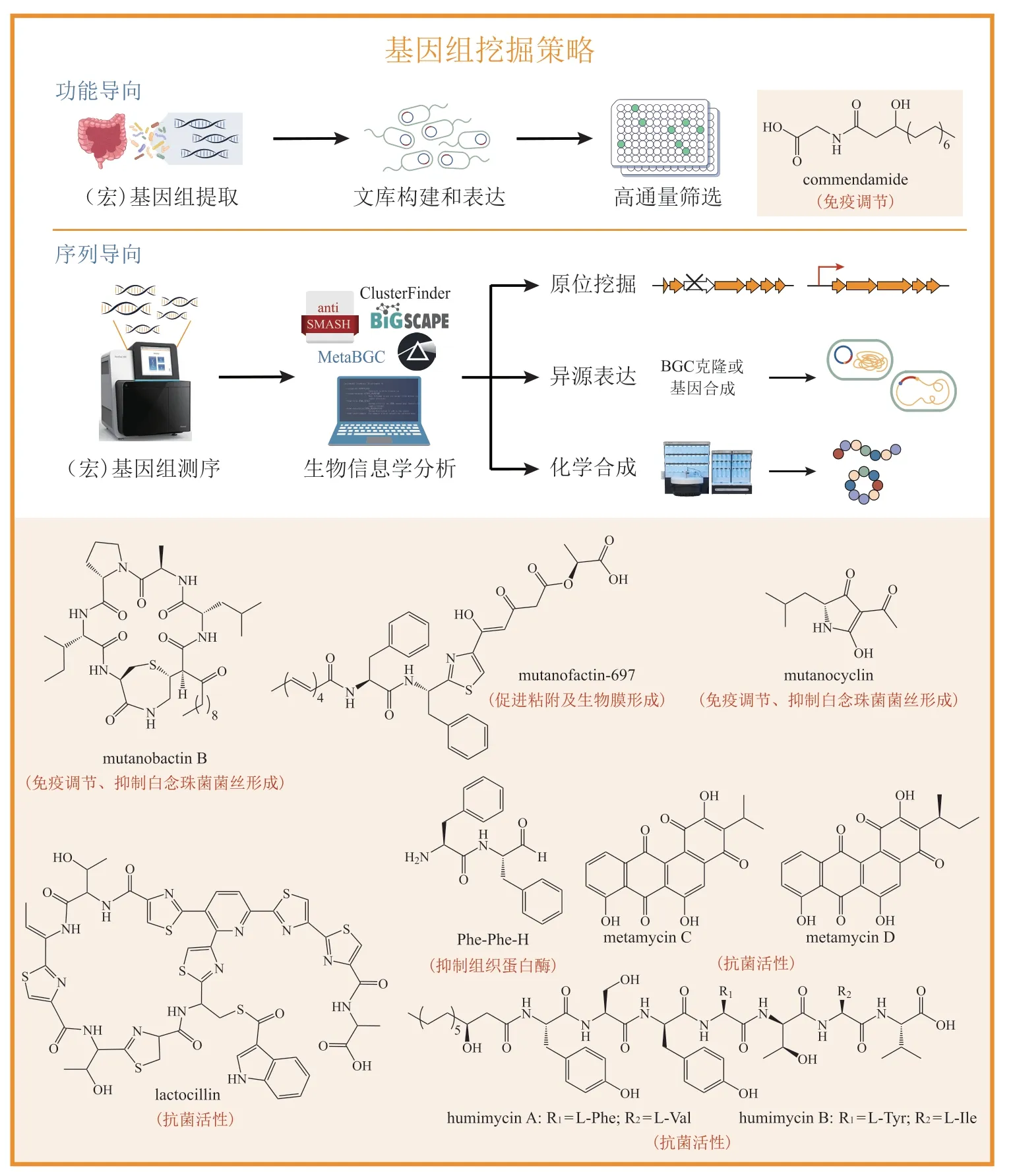
图2 基于基因组挖掘方法获得人体微生物活性天然产物Figure 2 Obtaining bioactive natural products from human microbiota by genome mining
2.1 功能导向的人体微生物基因组挖掘
功能导向的人体微生物基因组挖掘以功能宏基因组学相关的工作为代表,该方法首先提取特定环境微生物组的脱氧核糖核酸(deoxyribonucleic acid,DNA)来构建宏基因组文库,随后针对该文库产生的特定活性的化合物进行高通量筛选,获得具有目标活性的化合物后,再从对应的克隆中鉴定出负责产生活性天然产物的基因序列。Cohen等[30]利用人体粪便样本提取的DNA 构建了约含有75 000 个克隆的宏基因组文库,随后利用细胞报告系统来筛选具有调节NF-κB 活性的克隆,再通过转座子诱变确认产生相关化合物的基因。这一过程鉴定出26个独特的人体共生细菌效应基因(commensal bacteria effector genes)。其中的1 个效应基因家族Cbeg12被注释为N-酰基转移酶,能够催化产生新颖的N-酰基酰胺类天然产物commendamide(见图2)。在随后针对242 种G 蛋白偶联受体(G proteincoupled receptors,GPCRs)的活性筛选中,发现commendamide 可以特异性地激活调节细胞增殖和免疫的关键受体GPCR132/G2A。此外,与Cbeg12高度同源的基因存在于一些人体共生拟杆菌中,在普通拟杆菌(Bacteroides vulgatus)的发酵液中也能够直接检测到commendamide,提示人体共生拟杆菌可能会产生commendamide,并通过GPCR132/G2A 参与对宿主免疫的影响。Commendamide 的结构与一些人体内源性代谢物类似,它的发现和功能鉴定显示出人体微生物具有通过模拟宿主内源性信号分子来实现与宿主相互作用的能力。在此基础上,Cohen 等[31]在后续工作中系统地分析和研究了HMP 数据中的N-酰基转移酶,并发现了人体微生物产生的更多类型的N-酰基酰胺以及它们对其他GPCRs 的影响。近年来,Piscotta 等[32]利用功能宏基因组学对14 个健康婴儿的粪便样本进行研究,不仅在针对NF-κB 调节活性的筛选中再次验证了commendamide 的产生及其功能,还鉴定出一些具有诱导细胞自噬或调节氧化还原电位功能的效应基因。可见,功能宏基因组学在建立人体微生物组序列和功能的关联方面具有突出优势。
2.2 序列导向的人体微生物基因组挖掘
(宏)基因组研究伴随着DNA 测序技术的快速进步,为人体微生物天然产物挖掘提供了大量可用的序列信息,同时也对序列和数据的分析提出了更高的要求。负责天然产物产生的基因通常共定位于基因组邻近区域形成BGC,而且一般来说具有某些特征序列。基于这一特性,研究者们开发了多种预测和评估BGC 的方法。表1 展示了目前常用的部分BGC 预测工具。序列导向的人体微生物基因组挖掘研究基于对(宏)基因组序列的深入分析,通过针对性地选取目标BGC 来进行研究。已开展的工作中研究的BGCs 有的具有高度的新颖性或突出的代表性;有的来源于高丰度物种或与人体健康、疾病有密切关联的物种;有的则能够被其他组学数据映射,具有潜在的生理学意义。对已经开展的研究进行归纳,根据所采取研究策略的不同可以分为以下3 类。

表 1 常用的生物合成基因簇预测工具Table 1 Commonly used biosynthetic gene cluster prediction tools
2.2.1 在原始菌株中进行原位挖掘人体肠道和口腔是微生物丰度最高的2 个部位。与肠道微生物天然产物一样,口腔微生物所蕴含的活性天然产物资源也引发了研究人员广泛的兴趣。在已经开展的口腔微生物天然产物研究中,针对变形链球菌(Streptococcus mutans)的研究具有一定的代表性。变形链球菌在人类口腔中普遍存在且长期被认为与龋齿相关[52]。Ajdić 等[53]在详细分析S. mutansUA159 的基因组后,从中鉴定出了TnSmu2 基因组岛。随后对这一区域进行比较分析,发现不同变形链球菌中该基因组岛可能包含了不同类型的BGCs[54]。通过敲除S. mutansUA159 该区域中编码聚酮合酶-非核糖体肽合成酶(polyketide synthasenonribosomal peptide synthetase,PKS-NRPS)杂合酶的mub基因,研究者们鉴定出了具有特殊的C—C成键大环和含氮、硫七元杂环结构的mutanobactin A[55],并在随后的几项研究中发现了一系列类似物mutanobactin B-J[56-57]。笔者课题组通过研究发现mutanobactins 类化合物在生物合成过程中通过还原酶结构域释放带有醛基末端的线性脂肽,再历经3 步非酶催化反应产生mutanobactins,而这一过程中自发的C—S 键形成和C—C 键断裂极大丰富了mutanobactins 的结构多样性[58]。此外,在mutanobactins 的研究过程中,还利用基于MS/MS的算法iSNAP(informatic search algorithm for natural products)鉴定了该基因簇的中间产物mutanamide[57]。与口腔中变形链球菌需要应对氧胁迫以及该菌与白念珠菌(Candida albicans)密切关联等事实相符,mub的存在有利于变形链球菌的抗氧化能力,部分mutanobactins 类化合物也表现出对白念珠菌菌丝形成和生物膜形成的有效抑制。此外,在利用脂多糖刺激的RAW264.7 巨噬细胞中,mutanobactin B(见图2)能够显著上调促炎细胞因子白细胞介素-6(interleukin-6,IL-6)和IL-12 的表达,并下调单核细胞趋化蛋白-1(monocyte chemotactic protein-1,MCP-1)、粒细胞集落刺激因子(granulocyte colony-stimulating factor,G-CSF)和肿瘤坏死因子-α(tumor necrosis factor-α,TNF-α)的表达。
最近,Li 等[59]通过对S. mutansNMT4863 中编码PKS-NRPS 杂合酶的muf进行敲除,发现了一系列mutanofactins 类化合物(见图2)。Mutanofactins能够非特异性结合变形链球菌细胞并改变细菌表面的物理化学性质(增加细菌疏水性),还能直接与胞外DNA 结合并促进胞外DNA 介导的细胞聚集作用,从而以剂量依赖的方式促进细菌细胞的自聚集、细菌间的粘附,以及随后的生物膜形成。这一特征可能有助于mutanofactins 产生菌在牙菌斑生物膜形成过程中与周围的变形链球菌或其他微生物结合,促进其在口腔中的定植,并影响生物膜的组成和结构。
此外,人体其他部位的微生物同样具有产生活性天然产物的巨大潜能。2014 年,Cimermancic等[39]开发了基于隐马尔可夫模型(Hidden Markov Model,HMM)的概率算法ClusterFinder,扩大了所能预测的BGC 类型和范围。Donia 等[60]利用ClusterFinder,从2 430 个HMP 参考基因组中预测出14 000 余个BGCs,其中,3 118 个出现在健康人体微生物组中,且超过一半BGCs 分布于肠道和口腔中;基于编码硫肽类核糖体肽BGC 在HMP 数据中体现出的广泛性,以及已报道硫肽类化合物的良好活性,作者选取了其中1 个与抗生素高硫青霉素(thiocillin)的BGC 相似的bgc66进行了研究。通过比较1 株分离自阴道的含有bgc66的格氏乳杆菌(Lactobacillus gasseri)JV-V03 的野生型与基因簇插入突变株的代谢谱,获得了新颖的天然产物lactocillin(见图2)。活性测试发现lactocillin 可以抑制革兰阳性菌生长,对病原菌金黄色葡萄球菌和粪肠球菌(Enterococcus faecalis)都表现出了纳摩尔水平的抗菌活性。
若含有目标BGC 的菌株易于培养且能够进行遗传操作,可以针对BGC 中的关键基因,构建突变株,并通过代谢谱比对,直接建立BGC 与化合物的关联。这种原位挖掘的方式会较大程度地反映出天然产物产生的真实状态。然而,在实际研究中,很多人体微生物难以分离和培养,有些虽然可以培养但无法遗传操作,往往需要通过原位挖掘以外的策略来获取它们产生的天然产物。
2.2.2 通过异源表达进行挖掘得益于培养组学等方法在人体微生物研究中的应用,实验室条件下可培养的人体微生物数量持续增加。然而,一些低丰度物种的分离依然很困难,针对不同微生物建立稳定的培养和遗传操作方法也需要投入大量时间和精力。即便是在可培养,并已成功实现遗传操作的种属内,不同菌株之间的可操作性差异也阻碍了相关方法的广泛应用。因此,利用异源表达宿主来进行人体微生物天然产物研究得到了广泛的重视。
Guo 等[61]结合以往研究以及HMP 公布的最新数据鉴定了47 个未知功能的NRPS 类BGCs,这些BGCs 存在于超过90%的HMP 粪便样品中,且几乎完全来源于肠道微生物中的厚壁菌门(Firmicutes)梭菌纲(Clostridia)。由于梭菌的遗传操作较为困难,作者利用2 种常用的异源表达宿主大肠埃希菌和枯草芽孢杆菌(Bacillus subtilis)选取了14 个序列和结构域排布具有代表性的BGCs进行异源表达。通过直接克隆或经密码子优化后合成相关BGCs,将其转入大肠埃希菌或枯草芽孢杆菌中,分别置于强启动子T7 或hyper-Pspac下进行表达,成功获得了32 个pyrazinones 和dihydropyrazinones 类化合物。作者根据这些化合物的结构推测其环化由非酶促机制产生,而未发生环化的二肽醛(dipeptide aldehydes)形式前体可能是细菌的真正产物。结合肽醛类化合物在以往研究中表现出的蛋白酶抑制活性,作者通过体外研究发现其中一些二肽醛确实具有类似的活性,如:Phe-Phe-H 对于组织蛋白酶表现出显著的选择性(见图2)。考虑到组织蛋白酶在免疫监视中的作用,以及编码产生二肽醛的BGCs在肠道中广泛存在,这一研究结果提示人体微生物可能通过产生具有组织蛋白酶抑制活性的天然产物来影响宿主免疫,从而促进自身定植。
大肠埃希菌和枯草芽孢杆菌等常用异源表达宿主在表达部分厌氧微生物的BGCs 时表现出一定的局限性。基于此,笔者课题组以兼性厌氧菌S.mutansUA159 作为异源表达宿主,利用其天然感受态建立了适用于低G+C 含量的厌氧菌的大片段克隆技术——基于自然感受态的大片段克隆技术(natural competence based large DNA fragment cloning,NabLC)[62]。NabLC克隆过程中不需要引入中间载体,能够避免由于载体不稳定或不兼容引起的克隆失败问题。利用该技术,课题组对不同来源的低G+C 含量的厌氧菌BGCs 成功实现了克隆和表达,包括来源于表皮葡萄球菌的pyrazinones 类化合物的BGC和S. mutans35 中编码PKS-NRPS 杂合酶的BGC,后者能够产生tetramic acids 类化合物mutanocyclin(见图2)。活性测试发现mutanocyclin 具有抑制免疫细胞浸润的活性,并表现出抑制白念珠菌菌丝形成的能力[63]。随后,Tang 等[64]通过对S. mutansB04Sm5 直接进行遗传操作也检测到了mutanocyclin 的产生,还发现了muc基因簇可以产生reutericyclins 类化合物。其中,reutericyclin A 也曾经从罗伊乳杆菌(Limosilactobacillus reuteri)中分离得到[65]。Reutericyclins 具有广谱抗革兰阳性菌活性,不仅对乳杆菌、枯草芽孢杆菌、金黄色葡萄球菌等菌种具有抗菌作用,还能够抑制血链球菌(Streptococcus sanguinis)、戈登链球菌(Streptococcus gordonii)、轻型链球菌等与变形链球菌竞争同一生态位的口腔微生物。据推测,reutericyclins 是mutanocyclin 生物合成过程中的中间代谢产物,能够被mucF编码的脱酰基酶转化为mutanocyclin,从而在不同情况下为变形链球菌提供更多生存优势。
已开发的大部分BGC 预测工具一般适用于基因组信息较为完整的物种,而在分析一些从临床人体微生物样本获得的微量、碎片化测序结果时可能会遗失很多新颖BGC 的信息;此外,最近的几项宏基因组研究揭示了人体微生物组中还有很多尚未被测序的罕见微生物,它们的BGCs 依然有待挖掘。为了突破这一限制,Sugimoto 等[45]开发了MetaBGC 算法,该算法包含Build,Identify,Quantify 和Cluster 4 个模块。 其中,Build 模块基于轮廓隐马尔可夫模型(profile Hidden Markov Models,pHMMs)开发了分段pHMMs(segmented pHMMs,spHMMs)算法,能够直接从读段(read)水平(约100 bp)的人体微生物宏基因组数据中识别BGCs,在一定程度上避免了对低丰度序列的遗漏。此外,MetaBGC 还具有较高的运算效率,便于同时分析大量宏基因组样本。利用该方法,研究者针对HMP 以及MetaHIT 的2 544 个宏基因组样本进行了分析,从中鉴定出13 个完整的Ⅱ型PKS BGCs,并通过将公开的转录组数据映射到已鉴定的BGCs 中,证明其中至少6 个BGCs 能够在人体内表达。随后,作者利用异源表达的方法对1 个口腔来源的BGC(bgc3)和1 个肠道来源的BGC(bgc6)进行了研究。他们依据链霉菌属的密码子偏好性优化后合成了bgc3,并整合至白色链霉菌(Streptomyces albus)J1074 中进行表达,最终鉴定得到了metamycins A-D;另外,还从Blautia wexleraeDSM 19850 中克隆了bgc6,将其置于大肠埃希菌-枯草芽孢杆菌穿梭载体中的强启动子下游,整合至基因组平均G+C 含量与Blautia wexlerae(41.5%)接近的枯草芽孢杆菌(Bacillus subtilis)168sfp+(43.5%)中进行表达,成功获得了蒽环类聚酮化合物wexrubicin。活性测试发现wexrubicin,metamycin A 和metamycin B 对测试细胞和菌株无明显毒性,metamycin C 和metamycin D(见图2)对一些革兰阳性菌,尤其是链球菌属、奇异菌属(Atopobium)、放线菌属(Actinomyces)、罗斯菌属(Rothia)以及棒状杆菌属(Corynebacterium)的口腔分离株具有良好的抑制活性,提示bgc3有利于宿主菌竞争口腔生态位,这一结果也和转录组分析显示的bgc3在人龈上菌斑样本中早期生物膜形成期间表达的现象一致。
目前,利用已有的序列分析程序预测BGCs,再根据目标BGCs 特性选择合适的异源宿主进行表达,已经成功实现了对多种不同来源、不同类型的BGCs 的表征。随着更多BGC 克隆方法的建立和完善、DNA 合成成本的下降以及可作为异源表达宿主的菌株列表的扩充,异源表达策略有望在人体微生物活性天然产物的挖掘中发挥更重要的作用。
2.2.3 化学合成手段助力挖掘不管是通过原位激活,还是通过异源表达来获取目标天然产物,都需要较长的研究周期,可否获得目标化合物具有不确定性。随着基于序列信息预测天然产物结构的准确性越来越高,在结构预测基础上直接通过有机合成来制备目标化合物,减少了微生物培养、BGC 表达、产物分离纯化等过程中可能存在的限制和不确定性。这一方法在挖掘不含修饰或具有简单修饰的RiPPs 类以及NRPs 类化合物时得到了成功应用。
例如,Chu 等[66]发展了将BGC 预测与化学合成相结合的方法来研究人体微生物中的天然产物,利用该方法获得的分子称为合成-生物信息学天然产物(synthetic-bioinformatic natural products,syn-BNPs)[47]。作者利用抗生素和次级代谢产物分析工具(antibiotics and secondary metabolite analysis shell,antiSMASH)对HMP 和人类口腔微生物组数据库(human oral microbiome database,HOMD)中的基因组进行预测,并选取其中的NRPS 类BGCs 进行后续研究。考虑到产物少于5 个氨基酸的非核糖体肽通常被高度修饰,经过长度筛选后鉴定出57 个预测产物多于或等于5 个氨基酸的NRPS 类BGCs,排除其中不完整、包含PKS 模块或杂环化结构域的BGCs 后,利用NRPSPredictor2 等3 种NRPS 预测算法对25 个BGCs 的产物进行了预测。根据预测结果,作者设计了30 个潜在的syn-BNPs。对于可能发生N-酰化的syn-BNPs,在合成时利用非核糖体肽中常见的β-羟基肉豆蔻酸进行修饰,经过2 轮固相多肽合成后成功获得了其中25 个syn-BNPs。随后,作者利用1 组常见人体共生菌和致病菌对syn-BNPs 进行了抗菌活性筛选,发现来源于马红球菌(Rhodococcus equi)的humimycin A 和红串红球菌(Rhodococcus erythropolis)的humimycin B(见图2)对于厚壁菌门、放线菌门(Actinomycetes)的测试菌株具有抗菌活性,尤其对金黄色葡萄球菌(包括一些耐甲氧西林金黄色葡萄球菌的临床分离株)和肺炎链球菌(Streptococcus pneumoniae)表现出突出的抗菌活性。此外,作者还尝试通过LC-MS检测探究了红球菌属菌株的发酵液中是否能够产生humimycins,但未观察到此类化合物的产生,暗示着在实验室条件下该BGC 可能不表达,也印证了syn-BNPs 方法具有的潜在优势。
3 人工智能辅助人体微生物活性天然产物的发现
近年来,人体微生物相关的组学数据增长迅速,对研究者们的挖掘效率和准确性提出了更为严格的要求。引入人工智能算法极大地提升了研究人员对海量数据进行分析的能力,不仅在BGCs 预测方面突破了以往生物信息学分析工具在预测新颖BGCs,尤其是RiPPs 合成酶类BGCs 时的局限,在化合物的活性和作用靶点预测、减少重复发现等方面也表现出了巨大的应用潜力[67]。
BGCs 预测方面,表1 中所列的antiSMASH和RODEO 等工具包及时更新,在以往主要基于BLAST 和HMM 进行BGCs 预测的基础上,引入了二元分类器支持向量机(support vector machine,SVM)等人工智能算法。基于HMM的ClusterFinder 在以往工作中已经体现出识别新型BGCs 的强大功能,但该方法在准确性上存在一定不足。基于自然语言处理(natural language processing,NLP)和深度学习(deep learning)策略的DeepBGC[47]在一定程度上弥补了ClusterFinder在准确性方面的缺陷,在针对同一验证集的分析结果中,该方法相较于ClusterFinder 显示出更高的识别精度和准确率。最近,Liu 等[48]又发布了在此基础上改进的e-DeepBGC(extension of DeepBGC),在原有深度学习的基础上引入了新的数据增强(data augmentation)步骤,以识别更多类型的BGCs。RiPPs 合成酶类BGCs 序列的高度多样性,以及相关BGCs 基因的分散分布性,很长时间以来都限制了该类BGCs 的准确预测和新型基因簇的发现。近几年来,一些针对RiPPs 合成酶类BGCs 特点开发的工具,如:NeuRiPP[46]和DeepRiPP[25],通过在预测过程中使用神经网络和自然语言处理在很大程度上突破了传统预测方法的局限。
最近,笔者课题组与合作者基于attention、长短期记忆(long short-term memory,LSTM)以及基于transformer 的双向编码器表征(bidirectional encoder representations from transformers,BERT)3 种自然语言处理神经网络模型构建了用于抗菌肽(antimicrobial peptides,AMPs)挖掘的方法[68](见图3),预测精确度相比以往的预测工具大幅提高,达到91.31%。通过对4 409 个人体微生物代表性基因组进行挖掘,笔者课题组与合作者鉴定了2×107余个候选AMPs。随后,经过与宏蛋白组学数据的交叉验证将候选AMPs 数量精简至2 349 个。考虑到具有抗菌活性的AMPs 和其能够抑制的细菌之间存在潜在的负相关性,进一步使用15 个独立队列的宏基因组数据构建了AMP-微生物相关网络,将候选AMPs 的列表优化到241 个。最后,通过多肽合成的方法成功获得了216 个AMPs,并进行了抗菌活性测试,阳性率达到83.8%(181/216)。这些AMPs 与以往报道的AMPs 序列一致性均低于40%,说明基于自然语言学习的方法可以有效发现新型AMPs。对活性突出的11 种AMPs 进一步测试后显示10 种AMPs 对多株临床分离的多重耐药细菌表现出良好抗菌活性。笔者课题组与合作者选择了3 种在溶血性测试和细胞毒性测试中表现良好的AMPs 用于治疗感染肺炎克雷伯菌的小鼠模型,观察到了显著的疗效。进一步对综合活性最好的抗菌肽c_AMP1043 的细菌耐药性进行了测试,结果在30 天针对大肠埃希菌的抗性诱导实验后,未观察到明显耐药性的产生。

图3 人工智能辅助的人体微生物活性天然产物挖掘和分析Figure 3 AI-assisted mining and analysis of bioactive natural products from human microbiota
这一工作更新了从序列信息中挖掘活性天然产物发现的逻辑,证明在AMPs 等类型的化合物发现过程中,人工智能方法的引入可以避开BGCs 预测等步骤,直接发现具有特定生物活性的分子,大大简化了活性化合物的获取过程。
4 结语与展望
人体内蕴藏着数量庞大的微生物群体,它们之间的相互作用及其对人体产生的影响与人类的健康息息相关。人体微生物所产生的活性天然产物是人体环境长期筛选的结果,对相关微生物适应人体环境发挥着重要的作用。相应地,这就决定了活性天然产物靶向人体或人体微生物组来行使生理功能的活性特征。目前,人们已经发展了多种获取人体微生物天然产物的研究策略,成功地获得了一些化合物。对这些化合物的活性研究发现其往往具有免疫调节和抑制其他人体微生物的作用,但由于人体环境的复杂性和缺乏研究模型的限制,对其真实生理功能的理解进展缓慢。
除了产生次级代谢产物之外,人体微生物通过代谢食物或人体分泌物产生的活性分子也能够从多方面影响人体健康。例如:1)肠道微生物以膳食纤维或宿主分泌的黏蛋白为底物进行发酵产生的短链脂肪酸(short-chain fatty acids,SCFAs)能够激活GPCRs 或游离脂肪酸受体(free fatty acid receptors,FFARs),从而对维持肠道屏障、肠道运动、激素分泌等过程产生影响,并与非酒精性脂肪肝等多种代谢疾病相关;2)肠道微生物代谢氨基酸产生5-羟色胺、吲哚等衍生物,能够调节宿主情绪、睡眠以及免疫反应,并与神经系统炎症和疾病的发展相关;3)人体微生物代谢膳食胆碱可产生三甲胺,其转化生成的三甲胺N-氧化物(trimethylamineN-oxide,TMAO)是心血管疾病和其他慢性疾病的潜在风险因子等[69]。最近,Tintelnot 等[70]发现肠道微生物产生的色氨酸代谢物吲哚-3-乙酸(indole-3-acetic acid,3-IAA)水平与胰腺导管腺癌(pancreatic ductal adenocarcinoma,PDAC)的化疗疗效相关,这一活性源于中性粒细胞衍生的髓过氧化物酶能够氧化3-IAA,与化疗联用时会引起癌细胞中活性氧的积累和自噬下调,从而损害癌细胞的代谢适应性。相比次级代谢产物,目前针对人体微生物产生的初级代谢产物所使用的检测、分析方法和研究流程都更为成熟和完备,因此,进一步解析这些活性分子在错综复杂的人体环境中具体的作用靶点和通路无疑将极大推动对人体微生物活性化合物的理解和应用。
从改善健康的视角来看,人体微生物天然产物的研究蕴藏了巨大的潜力。一方面,很多来源于人体微生物的天然产物有望成为新型的抗菌化合物或免疫调节分子;另一方面,对相关天然产物进行研究能深入地了解它们的作用机制及在人体中产生的影响,从而在健康促进和疾病预防中采取更有效、更有针对性的预防或治疗措施。近些年来,人体微生物研究领域的技术手段和计算工具得到了飞速发展。然而,在高效整合已有的组学数据、寻找适用于更多微生物及不同类型BGCs 的宿主菌或遗传操作工具、探究微生物天然产物在宿主中发挥的真实作用等方面还有巨大的进步空间。相信随着这些问题的改善和解决,人体微生物活性天然产物的发现和利用将会更为高效和精准。

