希尔斯山羊草1Ss S 和3Ss S 染色体特异dCAPS 标记的建立
倪佳俊,韩冉,徐文竞,王开,祁广,曾小雪,訾妍,李豪圣,刘建军,汪晓璐,刘成
(1. 鲁东大学农学院,山东烟台 264025;2. 山东省农业科学院作物研究所/小麦玉米国家工程研究中心/农业农村部黄淮北部小麦生物学与遗传育种重点实验室/山东省小麦技术创新中心,山东济南 250100)
小麦(Triticum aestivum, 2n =6x =42,AABBDD)是世界上最重要的粮食作物之一[1]。近年来,中国小麦种植面积稳定在2400 万公顷左右,占全球小麦种植面积的10.9%[2]。 然而,由于近几十年来少数优异亲本的广泛使用以及新病原菌的变异,导致我国小麦生产面临着遗传基础狭窄及抗病性丧失的双重问题[3]。 小麦近缘种属中蕴藏着丰富的优异性状,如抗病、抗逆和抗虫等特性,为小麦的遗传改良提供了珍贵的基因资源[4,5]。 山羊草属(Aegilops)包含20 多个物种,广泛分布于世界各地,是小麦抗病基因的重要来源[1]。 希尔斯山羊草(Ae.searsiiFeldman &Kislevex Hammer,2n =2x =14,SsSs)主要分布于以色列、约旦、叙利亚西南部和黎巴嫩东南部的亚地中海地区[6],高抗小麦叶锈病和白粉病[7]、秆锈病[6]以及禾谷类二叉蚜虫[8],对干旱和盐胁迫也有较好的抗性[9]。
分子标记可以用于鉴定和追踪小麦背景中近缘物种染色体。 目前,应用最为普遍的基于聚合酶链式反应(polymerase chain reaction,PCR)的分子标记主要有简单重复序列(simple sequence repeats,SSR)标记、竞争性等位基因特异性PCR(kompetitive allele specific PCR,KASP)和限制性内切酶多态性(cleavable amplified polymorphic sequences,CAPS)标记等。 CAPS 标记是在序列信息及单核苷酸多态性(single nucleotide polymorphism,SNP)位点已知的条件下,用限制性内切酶对PCR 产物进行酶切而产生长度多态性的目标片段。 由于多数SNP 不在限制性内切酶位点上,不能直接转化为CAPS 标记,人们常通过PCR 在SNP 位点的侧翼引入突变碱基,将其改造为限制性内切酶位点,进而产生长度多态性的目标片段,该技术被称为衍生限制性内切酶多态性标记(dCAPS)[10],具有共显性、位点特异性、操作简单、成本低等特点,目前已成功用于作物分类和辅助育种等研究[11-13]。
为了获得更多的小麦-希尔斯山羊草染色体易位系用于定位希尔斯山羊草优异性状并将其用于小麦遗传育种,本试验室从美国引进了中国春-希尔斯山羊草1Ss~7Ss附加系,并将这套附加系分别与小麦进行杂交,获得了一大批杂交后代材料。 然而,分子标记的缺乏极大地限制了这批杂交后代材料的有效筛选与鉴定。 因此,本研究基于希尔斯山羊草重测序的SNP 分析,开发了希尔斯山羊草新的dCAPS 分子标记,为小麦背景中希尔斯山羊草染色质的检测提供新的手段。
1 材料与方法
1.1 试验材料
试验材料如表1 所示。 中国春(Chinese Spring,CS)由电子科技大学杨足君教授提供。 序号2~23 的材料由美国堪萨斯州立大学的小麦遗传和基因组资源中心的John Raupp 先生提供。序号24 ~31 的材料是山东省农业科学院作物研究所小麦遗传育种研究团队自育品种(系)。

表1 试验材料
1.2 样品检测及文库构建
(1)利用DNA 提取试剂盒(QIAGEN,德国)提取希尔斯山羊草叶片的基因组DNA;(2)琼脂糖凝胶电泳分析DNA 降解程度以及是否有RNA污染;(3)紫外分光光度计检测DNA 的纯度(OD260/OD280比值);(4)Qubit 对DNA 浓度进行精确定量。 选取OD 值在1.8 ~2.0 之间、含量在1.5 μg 以上的DNA 样品用于建库。
检验合格的DNA 样品用Covaris 破碎机随机打断成长度为350 bp 的片段,采用TruSeq 文库构建试剂盒,经末端修复、加ployA 尾、加测序接头、纯化、PCR 扩增等步骤完成整个文库构建。 构建好的文库通过Illumina HiSeq 进行测序。
1.3 数据分析
对测序得到的原始数据进行质控得到过滤后数据:(1)去除带接头的双端读数;(2)当单端测序读数中含有的N 含量超过该条读数长度比例的10%时,去除此对双端读数;(3)当单端测序读数中含有的低质量(Q≤5)碱基数超过该条读数长度比例的50%时,去除此对双端读数。 有效测序数据通过BWA 软件[14](参数为mem-t4-k32-M)比对到小麦参考基因组(ftp:/ /ftp.ensemblgenomes.org/pub/plants/release - 46/fasta/triticum_aestivum/dna/Triticum_ aestivum. IWGSC. dna. toplevel.fa.gz),比对结果经软件SAMTOOLS[15]去除重复(参数为rmdup)。
1.4 SNP 检测及注释
利用软件SAMTOOLS[15](参数为mpileupm2-F0.002-d1000)进行个体SNP 检测。 为了降低SNP 检测的错误率,选用如下标准进行过滤:(1)SNP 的读数支持数不低于4;(2)SNP 的质量值(MQ)不低于20。 利用软件ANNOVAR 对SNP检测结果进行注释。
1.5 dCAPS 标记开发
利用dCAPS Finder 2.0(http:/ /helix.wustl.edu/dcaps/)软件设计差异碱基序列的酶切位点及上游或下游引物,再利用Primer Premier 5.0 设计序列另一端的下游或上游引物。
1.6 DNA 提取
供试材料基因组总DNA 用快捷型植物基因组DNA 提取试剂盒[天根生化科技(北京)有限公司]提取。 取约100 mg 叶片放入2 mL 离心管(提前加入直径2 mm 的钢珠)中,液氮中速冻,然后用SCIENTZ-192 组织研磨机在30 Hz 频率下将叶片打碎至粉末,加入400 μL 缓冲液FP1 和6 μL TNase,涡漩振荡1 min,室温放置10 min。 然后加入130 μL 缓冲液FP2,轻轻混匀,涡旋振荡1 min。 12000 r/min 离心5 min,将上清转移至新的离心管中。 向上清液中加入70%体积的异丙醇,充分混匀,12000 r/min 离心2 min,弃上清,保留沉淀。 加入500 μL 70%乙醇洗涤DNA,12000 r/min 离心2 min,弃上清,再次用500 μL 70%乙醇洗涤一次,步骤与第一次相同。 晾干后加入TE 溶解DNA 备用。
1.7 PCR 扩增及酶切
根据引物合成推荐的退火温度进行PCR 扩增。 PCR 反应体系(15 μL):DNA(25 ng/μL)1.0 μL,2×PCR mix 7.5 μL,上、下游引物(10 μmol/L)各1 μL,用无菌双蒸馏水补充反应体系至15 μL。PCR 反应扩增程序为:94℃预变性3 min;94℃变性30 s,56℃退火30 s,72℃延伸1 min,35 个循环;最后72℃延伸10 min。
PCR 扩增结束后,采用相应的内切酶(Thermo Scientific 公司提供)进行酶切。 酶切反应体系(5 μL):内切酶0.2 μL,快速酶切缓冲液1 μL,无菌双蒸馏水补充反应体系至5 μL。 37℃酶切15 min。 酶切产物在1.5%的琼脂糖凝胶中电泳,电泳后凝胶在紫外凝胶成像仪GDS-Gel Dol 2000下扫描照相。
2 结果与分析
2.1 基因组重测序数据质量评估及基因组比对情况
本次测序共产生原始数据149.713 G,过滤后的数据149.389 G,各样本的原始数据为149713.013 M,测序质量高(Q20≥96.38%、Q30≥91.08%),GC 含量47.27%。 综上,所有样本的数据量足够,测序质量合格,GC 分布正常,建库测序成功。
参考基因组大小为14547 261565 bp,所有样本的比对率为97.26%,对参考基因组(排除N区)的平均覆盖深度为29.82×,1×覆盖度(至少有一个碱基的覆盖)在43.43%以上。 比对结果正常,可用于后续的变异检测及相关分析。
2.2 SNP 位点检测及注释
检测到的SNP 位点总数为8390 257。 基因上游1 kb 区变异位点数量为148038,基因下游1 kb 区位点数量为145070,同属两个基因上下游1 kb 区位点数量为6558,基因间区位点数量为7075 127,获得终止密码子位点数量为1190,丧失终止密码子位点数量为335,外显子区同义变异位点数量为237253,非同义变异位点数量为161321,内含子区位点数量为513874,剪接位点数量为653,转换位点数量为5346 489,颠换位点数量为3043 768,全基因组杂合率(杂合SNP 个数)为0.131。
2.3 dCAPS 标记开发及鉴定
依据整理的SNP 序列信息,结合dCAPS 软件,设计了150 对dCAPS 标记引物,最终筛选出2个dCAPS 标记用于检测中国春-希尔斯山羊草附加系。 标记信息如表2 所示。
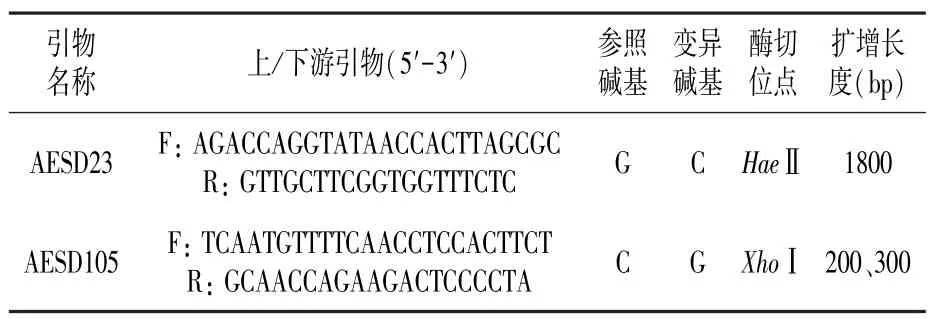
表2 dCAPS 标记信息
PCR 扩增及电泳检测结果发现,这2 对引物能在中国春-希尔斯山羊草附加系中扩增出不同的多态性条带。 AESD23 在中国春-希尔斯山羊草1Ss附加系中扩增出1800 bp 左右的多态性条带;AESD105 在中国春-希尔斯山羊草3Ss附加系中扩增出200 bp 和300 bp 左右的多态性条带(图1)。
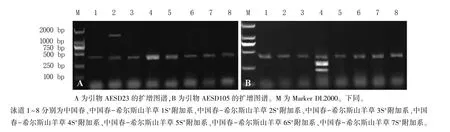
图1 2 对dCAPS 引物在中国春-希尔斯山羊草附加系中扩增产物的电泳检测图谱
为了验证这2 对引物可以用于检测希尔斯山羊草杂交后代,我们又以济南17、济麦20、济麦22、济麦23、济麦4075 和济麦6206 为材料进行PCR 扩增。 结果发现,这2 对引物可以在含有希尔斯山羊草染色体的附加系中扩增出多态性条带,而在中国春等六倍体普通小麦中没有扩增出多态性条带(图2)。

图2 2 对dCAPS 引物在中国春等普通小麦中扩增产物的电泳检测图谱
对这两个引物分别在中国春与中国春-希尔斯山羊草1Ss附加系杂交F2后代和3Ss附加系杂交F2后代上随机点样,发现AESD23 只在中国春-希尔斯山羊草1Ss附加系上扩增出多态性条带,而AESD105 只在中国春-希尔斯山羊草3Ss附加系上扩增出多态性条带(图3)。

图3 2 对dCAPS 引物在附加系杂交后代中的扩增电泳检测图谱
另外我们分别用AESD23 和AESD105 在中国春-希尔斯山羊草的1SsS 和1SsL 端体附加系、3SsS 和3SsL 端体附加系中扩增,发现这两对引物分别在中国春-希尔斯山羊草1SsS 端体附加系和3SsS 端体附加系中扩增出多态性条带(图4),说明这两对引物分别定位在1SsS 染色体和3SsS 染色体上。

图4 2 对dCAPS 引物在端体附加系中的扩增电泳检测图谱
3 讨论与结论
3.1 希尔斯山羊草基因组重测序结果的重要性
目前,许多研究报道了在小麦近缘物种中利用基因组测序开发分子标记,能够更精确地鉴定抗病基因,从而为将小麦近缘物种的优异基因导入小麦提供有利保障。 Xu 等[16]利用粗山羊草基因组测序信息,比较了SNP 标记和SSR 标记对绘制QTL 图谱的差异,分析了小麦染色体3A、3B 和3D 上21 个镰刀菌抗性QTLs。 Bansal 等[17]通过基因组测序将小伞山羊草渐渗系抗叶锈病和条锈病基因定位在5DS 末端染色体。 Liu 等[18]利用RNA 测序技术开发高大山羊草4S 特异分子标记,通过诱导同源重组从4S 染色体上转移Bgt抗病基因。 Li 等[19]通过对5 种山羊草(二角山羊草、高大山羊草、希尔斯山羊草、沙融山羊草和拟斯卑尔脱山羊草)染色体水平的基因组序列进行组装,分析了小麦B 基因组的进化关系,为分析小麦-山羊草进化和遗传研究提供了新思路。 希尔斯山羊草作为小麦近缘种属,具有抗病、抗虫和抗逆等优异性状[6-9],但关于其基因组重测序的研究甚少。 本研究对希尔斯山羊草基因组进行重测序,结果显示测序质量好,数据可靠,后续可用于开发标记和用于比较基因组分析。
3.2 开发的标记可用于检测杂交后代
Sun 等[20]通过对希尔斯山羊草高分子量谷蛋白的分析,开发了可以检测小麦背景中1Ss染色体的生化标记。 Garg 等[21]对中国春-希尔斯山羊草1Ss~7Ss附加系进行分析,发现希尔斯山羊草1Ss染色体上含有新型高分子量谷蛋白亚基,对希尔斯山羊草1Ss染色体上的高分子量谷蛋白亚基基因进行了克隆并建立了分子标记。Liu 等[6]以中国春、希尔斯山羊草、中国春-希尔斯山羊草3Ss附加系及端体系为材料,筛选小麦EST-STS 引物,获得了希尔斯山羊草3Ss染色体特异标记,用这批标记鉴定了希尔斯山羊草3Ss染色体的整臂易位系,结合抗病性鉴定将抗秆锈病基因定位在3SsS 染色体上。 Gong 等[22]以中国春、绵阳11、绵阳15、希尔斯山羊草、小麦-希尔斯山羊草1Ss~7Ss附加系、小麦-希尔斯山羊草染色体端体系为材料,筛选EST-STS、EST-SSR、COS、PLUG 和SSR 引物,建立了希尔斯山羊草1SsS、1SsL、2SsS 和2SsL 染色体臂特异标记,并对相应杂交后代群体材料进行了扩增验证,为小麦背景中希尔斯山羊草染色体的检测提供了方法。 Liu等[23]以中国春、希尔斯山羊草、中国春-希尔斯山羊草2Ss附加系及端体系为材料,筛选小麦ESTSTS 引物,获得了希尔斯山羊草2Ss染色体特异标记,用这些标记鉴定了涉及希尔斯山羊草2Ss染色体小片段易位系,结合抗病性鉴定将抗白粉病基因Pm57 定位在2SsL 染色体上。 但是目前对希尔斯山羊草dCAPS 标记的研究缺乏。
本研究通过对希尔斯山羊草基因组重测序SNP 的分析,开发了2 个dCAPS 标记,分别位于希尔斯山羊草1Ss和3Ss染色体上,并且都位于短臂上。 Garg[21]和Wang[4]等将显著提高小麦品质及籽粒铁和锌元素含量的基因都定位在希尔斯山羊草1Ss染色体上,Liu 等[6]将抗小麦秆锈病基因Sr51定位在希尔斯山羊草3Ss染色体上,因此希尔斯山羊草1Ss和3Ss上可能携带丰富的与小麦品质和抗病相关的基因。 我们开发的新的dCAPS 标记可作为其特异标记检测希尔斯山羊草的杂交后代,为小麦近缘物种标记开发提供新的研究思路,也为挖掘小麦抗病和优质基因提供有利保障。
3.3 开发标记的效率
Liu 等[6]分别从170 个小麦SSR 引物和144个STS-PCR 引物中筛选到2 个和18 个希尔斯山羊草3Ss染色体特异标记,开发标记效率为1.17%和12.50%。 Liu 等[23]又分别从96 个STS-PCR和95 个全长cDNA(FlcDNA)引物中筛选到希尔斯山羊草2Ss染色体特异标记1 个和3 个(长臂和短臂各2 个),开发标记效率为1.04%和3.16%。Gong 等[22]从476 个EST 或SSR 引物中共筛选到17 个希尔斯山羊草1Ss或2Ss染色体标记,开发标记的效率为3.57%。 可见,上述研究报道的希尔斯山羊草标记开发效率在1.04%~12.50%之间。本研究从150 对dCAPS 引物中筛选出2 个希尔斯山羊草特异标记,开发标记的效率为1.33%。开发标记的效率不是特别高的原因可能是基因组重测序中存在大量的重复序列,产生许多错误的重叠,造成拼接产生的序列过短,因此获得的SNP位点相对减少。 但我们开发的标记仍可以作为筛选希尔斯山羊草染色体的特异标记,用于鉴定小麦-希尔斯山羊草后代材料。

