潮滩湿地硝化螺菌的代谢潜力和环境适应机制
陈国浩, 毛铁墙, 董宏坡, 欧亚飞, 张家伟
(华东师范大学 河口海岸学国家重点实验室, 上海 200241)
0 引言
硝化过程是氮素转化的重要环节, 包括氨氧化作用和亚硝酸盐氧化作用, 主要是由微生物主导[1-2].氨氧化古菌(ammonia-oxidizing archaea, AOA)和氨氧化细菌(ammonia-oxidizing bacteria, AOB)主要负责将氨氧化为亚硝酸盐[3-4], 而亚硝酸盐氧化细菌(nitrite-oxidizing bacteria, NOB)主要负责将亚硝酸盐氧化为硝酸盐[5]. 尽管最近发现了能够直接将氨氧化为硝酸盐的细菌[6], 即全程氨氧化细菌(complete ammonia oxidizer, Comammox), 但至今仍未在海洋环境中发现Comammox的基因组序列[7],故NOB对于亚硝酸盐的氧化仍为硝酸盐的主要来源之一[8-9].
硝化螺菌(Nitrospira)是NOB中多样性最为丰富的属, 至今已发现至少有6个世系(lineages)[10],然而, 仅5个lineages的亚硝酸盐氧化还原酶(nitrite oxidoreductase, NXR)基因序列[11]和3个lineages的基因组被揭示. 特别地, lineage Ⅲ的培养代表和基因组至今未被发现, 这意味着Nitrospira仍然存在较多未知的类群. 其中, Comammox在进化上属于Nitrospiralineage Ⅱ, 是近年来氮循环中的一个重大发现, 该细菌可以独立地完成两步硝化过程[6].Nitrospira广泛分布在土壤、淡水、热泉、地下水、人工系统和海洋中[10-12]. 根据调查和生理实验,Nitrospira的分布和多样性受到盐度、温度和底物浓度等因素的影响[13-15].
近年来, 发现Nitrospira不仅存在全球分布的特征, 还有着多样的代谢功能和生存方式. 有的Nitrospira编码着脲酶和氰酸酶基因[16-17], 能将这些有机氮素降解为氨, 为氨氧化微生物提供能源. 特别地, 部分ComammoxNitrospira能直接利用尿素和氰酸盐作为能源[6,18-19], 拓宽了对Nitrospira利用氮素的认识. 另外,Nitrospira还可利用氢气、甲酸盐和硫化物作为电子供体驱动生长[16,19-20]. 这些功能潜力对氮循环和Nitrospira自身生长均至关重要. 然而, 目前从潮滩湿地获得的Nitrospira基因组及培养样品较为缺乏, 对其在潮滩湿地中的功能认识非常有限. 因此, 利用一些新技术获取更多基因组对Nitrospira进行研究很必要.
潮滩湿地是陆地和海洋的交界地带, 其在全球的生物地球化学循环中发挥着重要作用. 大量富含氮的生活污水和农业肥料通过径流作用在潮滩湿地富集, 经过一系列的转化作用后, 部分流入海洋.根据已有的报道,Nitrospira在人工污水处理系统中发挥着关键的作用, 具有较高的功能多样性[18-19,21].天然的潮滩湿地, 包括红树林湿地、盐沼湿地和河口等, 是庞大而重要的污水处理系统, 含有丰富多样的Nitrospira物种[22-25]. 为了进一步认识Nitrospira在潮滩湿地中的功能, 本研究选取了中国沿海5个跨不同纬度的潮滩湿地, 从沉积物样品中恢复了14个Nitrospira基因组. 通过对系统发育、代谢通路和环境适应等方面的分析, 深入研究了这些基因组, 以期为理解Nitrospira在潮滩湿地中的重要生态功能提供更多的科学依据.
1 材料与方法
1.1 研究站位和采样
本研究从中国沿海的3个潮滩湿地获得12个沉积物样品, 包括辽河口盐沼湿地(LH01和LH02)、崇明岛东滩湿地(CM01和CM02)、三亚青梅港红树林自然保护区(QM). 使用重力柱采集器,在崇明岛东滩湿地和三亚青梅港红树林自然保护区获得3个1 m深的沉积物柱, 且每个沉积柱样品被平均分割成3 ~ 4份; 在辽河口盐沼湿地获得两个表层(0 ~ 5 cm)沉积物样品(LH01和LH02). 所有样品采集完后立即装进密封的无菌样品盒, 并放入装有干冰的储物箱中运送至实验室, 将样品储藏在 –80℃冰箱中用于测序和理化分析. 另外, 两个潮滩湿地的宏基因组样品数据(表1)从开放的公共数据库美国国家生物技术信息中心(the national center for biotechnology information, NCBI, https://www.ncbi.nlm.nih.gov)中获得.
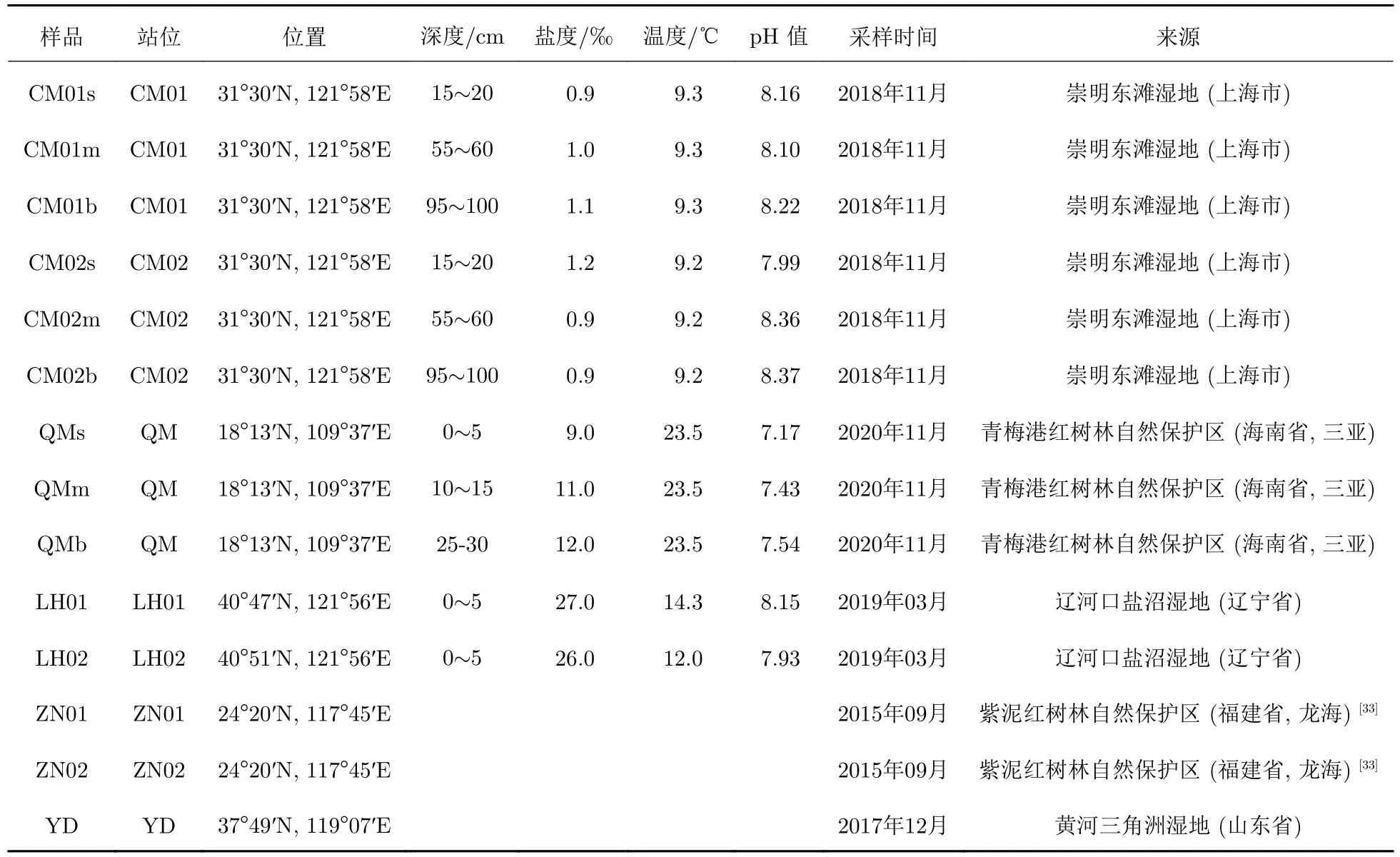
表1 样品的物理和化学性质Tab. 1 Physical and chemical properties of the samples
1.2 DNA提取和测序
使用DNA提取试剂盒 (MoBio Laboratories, Carlsbad, CA, USA), 从5 ~ 10 g沉积物中提取基因组DNA, 使用Qubit 2.0和Nanodrop One (Thermo Fisher Scientific, Waltham, USA)检测DNA提取质量. 检测合格的 DNA 样品被用于文库构建和测序. 宏基因组测序数据由Illumina HiSeq 2500仪器生成, 每个样品大约获得60 G双端原始测序序列数据.
1.3 宏基因组组装和基因组重构
本研究共获得15个宏基因组数据(表1), 包括从NCBI下载的3个数据 (YD、ZN01 和 ZN02) ,其中, YD由2个较小的宏基因组混合组成. 宏基因组拼接与分箱步骤包括: 先使用Trimmomatic软件[26]对原始序列进行质量裁剪, 再使用MEGAHIT软件[27]进行组装, 参数为(--k-min 27, --k-max 127,--k-step 10), 然后将拼接得到的序列使用MetaBAT软件[28]进行分箱, 最后使用CheckM软件[29]将分箱得到的基因组进行完整度和污染度的质量分析. 多样的基因组组装参数通过QUAST[30]进行评估.基因组的分类使用GTDB-TK工具[31]获得, 仅属于Nitrospirae门的基因组被关注. 序列分类通过CAT软件 (https://github.com/dutilh/CAT/releases/tag/v5.2.3)识别, 分类成其他门的序列被移除;同时, 不一致的序列被CheckM软件[29]基于单拷贝基因进行识别和手动移除. 最后, 对候选的基因组基于GC (guanine and cytosine)含量、覆盖度和四核苷酸频率使用RefineM[32]进行检查和校正, 完整度大于70%和污染度小于5%的基因组会被进一步分析.
1.4 系统发育分析和基因组注释
为确定重构基因组的系统发育地位, 本研究从NCBI数据库下载了Nitrospirae门的基因组. 通过使用GTDB-TK软件[31]对细菌的120个标记蛋白进行串联, 并使用IQ-TREE软件[34]构建最大似然树, 利用iTOL[35]对进化树进行可视化. 为进一步识别基因组的进化地位, 使用Barrnap软件(https://github.com/tseemann/barrnap)预测基因组的16S rRNA基因. 同时, 从NCBI数据库中下载了其他的16S rRNA基因, 使用MAFFT软件[36]对这些序列进行比对, 并使用BMGE软件[37]进行切边处理. 最后, 使用IQ-TREE软件[34]构建最大似然树和利用iTOL[35]对进化树进行可视化.
为探究氰酸酯酶基因的进化地位, 从NCBI中下载已知的NOB和AOA的氰酸酶基因的氨基酸序列及相似的部分序列, 并利用HMMER3[38]识别公共数据库及本研究获得的Nitrospira属基因组中的氰酸酶基因. 比对所有这些氨基酸序列, 对所得结果使用IQ-TREE软件[35]进行最大似然树的构建.
使用Prodigal软件[39]对基因组的开放阅读框(open reading frame, ORF)进行预测, 并将核酸序列翻译为蛋白序列. 然后, 使用BlastP工具[40]比对COG和NCBI 非冗余蛋白数据库, 进行蛋白功能的注释(E-value 阈值 < 1 × 10–5). 同时, 通过在线的KASS[41]工具进行注释.
2 结果与讨论
2.1 Nitrospira基因组的恢复
从5个跨越温带和热带的潮滩湿地沉积物中, 使用宏基因组测序的方法拼接与组装得到14个较高质量(完整度>70%、污染度<5%)的Nitrospira基因组 (metagenome-aassembled genomes, MAGs)(表2). 依据大于95%的种间平均核酸相似度 (average nucleotide identity, ANI)阈值[42], 这些MAGs可被划分为10个新物种. 同时, 基因组系统发育树(图1)显示, 这些MAGs位于3个不同lineages中, 包括lineage Ⅱ、lineage Ⅳ和一个未被定义的系统发育分支. 其中, 3个MAGs与clade A comammoxNitrospira聚在一起. 依据已有的研究, lineage Ⅱ和lineage Ⅳ分别来自两个完全不同的生态环境系统, 分别为陆地和海洋[10], 这意味着潮滩湿地的Nitrospira有更高的物种多样性. 有趣的是,基因组进化树显示, QMm6和QMb135这两个Nitrospira物种形成独立的一簇(图1), 且不属于任何已知的世系, 可能为新的Nitrospira物种.
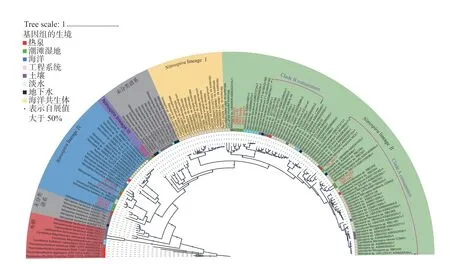
图1 基于120个保守串联蛋白构建的Nitrospira基因组最大似然树Fig. 1 Nitrospira genomic maximum likelihood tree based on 120 conserved proteins
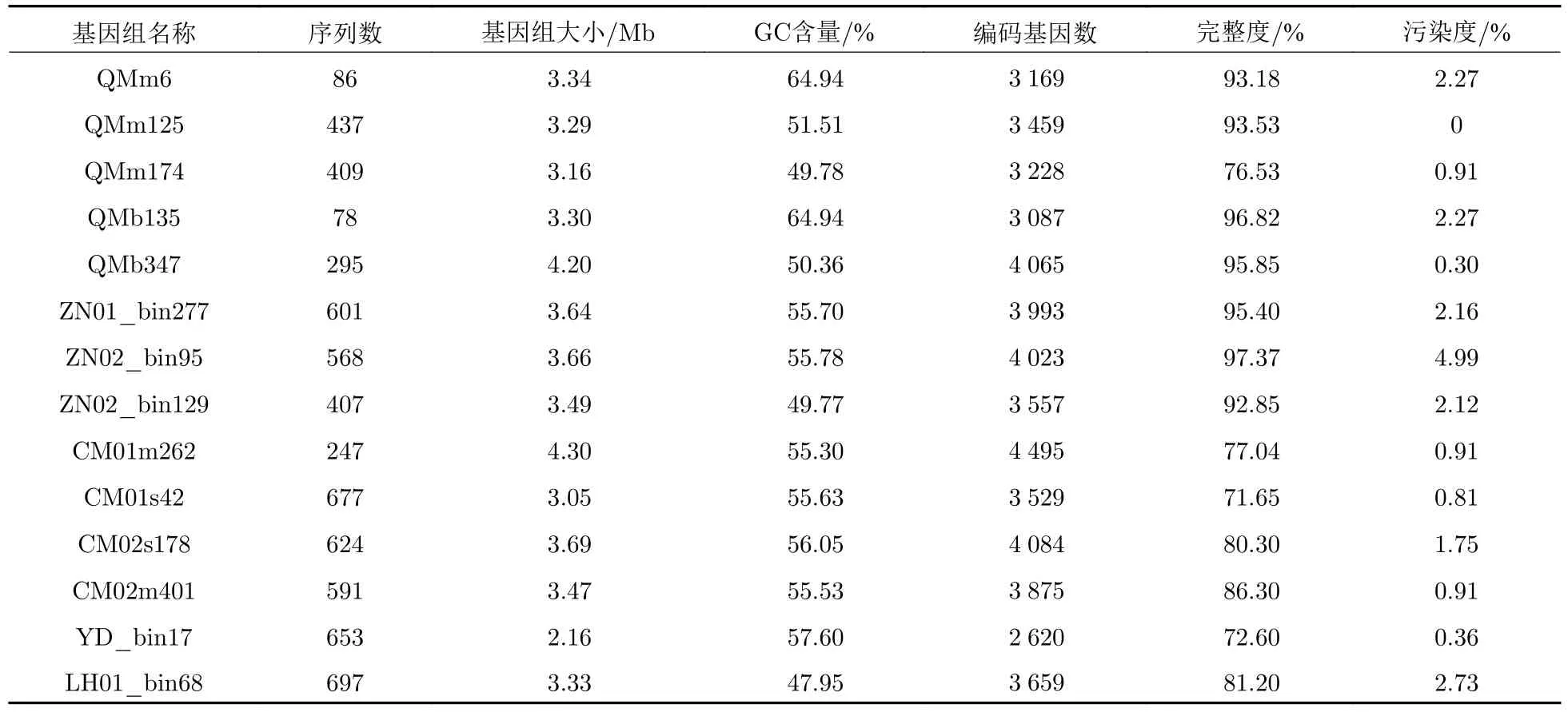
表2 从潮滩湿地宏基因组组装中提取的14个硝化螺旋菌的基因组特征Tab. 2 Genomic features of 14 Nitrospira MAGs recovered from metagenomic assembly in tidal flat wetlands
为了进一步揭示由QMm6和QMb135聚类成的这一未知的系统发育分支, 基于Nitrospira16S rRNA 基因构建系统发育树(图2). 本研究发现16S rRNA 基因树与Nitrospira基因组树有着近乎一致的拓扑结构, 且QMm6和QMb135这两个未曾被揭示的基因组所含的16S rRNA基因序列与前人报道的lineage Ⅲ中的16S rRNA序列聚类成一簇, 这意味着这两个MAGs隶属于此前被划分的lineage Ⅲ[12]. 这一独特的lineage从发现至今已超过20年, 但只有相应的16S rRNA基因被揭示, 而该分支的培养样品和基因组均未被获得[11-12], 因此, 其生理功能和代谢潜力仍有着很大的未知. 本研究首次发现的这两个隶属于lineage Ⅲ的MAGs有着较高的恢复质量(完整度分别为95.91%和96.82%),在中国三亚青梅港红树林沉积物中获得. 同时, 对16S rRNA序列的分析发现, 该lineage的成员分布在深海沉积物[43-44]、陆地喀斯特岩洞[45-46]和盐碱土环境中 (图2). 而本研究在红树林沉积物中首次发现了lineage Ⅲ, 扩大了它们的生境分布范围.

图2 基于Nitrospira 16S rRNA序列构建的最大似然树Fig. 2 Maximum likelihood tree based on Nitrospira 16S rRNA sequences
2.2 代谢潜力分析
本研究重构了14个NitrospiraMAGs的代谢途径(图3), 结果表明, 这些MAGs有着典型的Nitrospira所具备的中心碳代谢和能量传递系统, 如还原型三羧酸循环、糖酵解/糖质新生及磷酸戊糖途径等. 尽管少部分MAGs缺失亚硝酸盐氧化还原酶alpha亚基和亚硝酸盐氧化还原酶beta亚基, 但有多个拷贝的亚硝酸盐氧化还原酶gamma亚基存在于这些基因组中. 在3个Comammox的基因组中, 仅CM01m262含有氨单加氧酶gamma亚基和CM01s42含有羟胺氧化还原酶(hydroxylamine oxidoreductase, HAO), 但它们均含有区别于严格亚硝酸盐氧化Nitrospira的Rh类型氨转运体[18,47],意味着这3个MAGs为典型的ComammoxNitrospira. 这些MAGs除具有进行亚硝酸盐氧化或全程氨氧化的作用外, 还具有更多的代谢潜力.
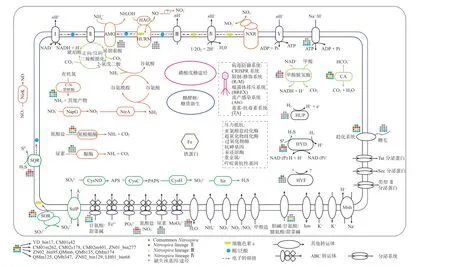
图3 重构14个Nitrospira基因组的代谢途径Fig. 3 Metabolic pathways of 14 Nitrospira MAGs
近年来, 研究者们发现Nitrospira可将尿素或氰酸盐降解为铵盐, 通过“互喂作用”为氨氧化微生物和Comammox 提供底物[16-18,48]. 尿素的裂解已经在一些富集培养的Comammox菌株(Ca.Nitrospira nitrosa,Ca.Nitrospira nitrificans和Ca.Nitrospira inopinata) 中被观察到[6,47]. 在本研究中, 脲酶基因在两个Comammox MAGs(CM01m262 和 YD_bin17)中被识别(图3), 其中YD_bin17含有部分相应的ABC转运系统, 这意味着尿素也可以被这些来自潮滩湿地的Comammox所利用. 这些Comammox主要存在于盐度较低的CM01和YD站位中. 最近的研究表明, 有机肥的使用和工厂产生了大量的尿素, 这些尿素通过径流进入水生生态系统中, 并在河口湿地中积累[45], 导致河口湿地富营养化[49]和水体缺氧[50], 而本文获得的这些Comammox可以直接转化尿素并减缓尿素的积累. 尽管尿素的利用已经在一些富集培养的典型Nitrospira中被观察到[16,51], 然而, 本研究获得的所有11个典型的Nitrospira基因组均没有包含脲酶基因, 这意味着典型的Nitrospira与氨氧化微生物通过降解尿素进行互惠生长的过程在潮滩湿地中可能并不普遍. 研究表明, 尿素可自发地裂解为氰酸盐, 广泛存在于环境中并参与氮循环[52-53]. 本研究获得的6个NitrospiraMAGs中含有氰酸酯酶基因(图3), 其中3个来自CM02和QM站位的NitrospiraMAGs编码着相应的氰酸盐转运体, 表明这些Nitrospira可分解潮滩湿地环境中的氰酸盐为铵盐, 并为硝化微生物提供能源, 对氮素循环发挥着重要的作用.本研究中的3个Comammox MAGs均不含有氰酸酯酶基因, 这些基因仅在污水处理系统的几个ComammoxNitrospira成员中被发现[18-19]. 令人意外的是, 从lineage Ⅲ两个基因组中发现的氰酸酯酶与此前在其他Nitrospira成员中发现的有着不同的进化关系(图4(a)), 它们之间有着较低的氨基酸相似度(<40%); 前人发现的Nitrospira中的氰酸酯酶与AOA 菌株Nitrososphaera gargensis的氰酸酯酶有着更近的发育关系(图4(b)), 而本研究获得的这些序列与Nitrospinae门和厌氧氨氧化细菌(anaerobic ammonium oxidation, anammox)的氰酸酯酶有着更近的进化关系(图4(c)). 在AOA 菌株Nitrososphaera gargensis中, 氰酸酯酶可分解氰酸盐产生氨作为单一能源[17]; 而海洋中的anammox和Nitrospina细菌也表现出降解氰酸盐的潜力, 可以使用氰酸盐作为氮源, 或基于对氨氧化微生物的“互喂” 机制作为间接能源[54-56]. 在潮滩湿地MAGs中发现的这些氰酸酯酶氨基酸序列均含有保守的Arg96、Glu99、Ser122活性位点残基[57](图5), 这意味着Nitrospira成员中发现的这两种有着不同亲缘关系的氰酸酯酶均具有裂解氰酸盐的潜力. 这些新发育分支的氰酸酯酶氨基酸序列与Nitrospinasp.(MBM14886.1)有着较高的氨基酸相似度(75% ~ 82%), 意味着它们之间可能发生了水平基因转移事件. 在Nitrospira中新的氰酸酯酶进化分支的这一发现, 拓展了研究人员对Nitrospira成员中氰酸酯酶的认知, 表明该酶在Nitrospira中的分布可能更为多样化. 这两个进化分支的氰酸酯酶基因广泛存在于Nitrospira不同的lineage基因组中, 而这些不同lineage的Nitrospira广泛分布于全球的各种生态系统, 包括开阔大洋和陆地生态系统, 意味着氰酸盐在全球广泛被Nitrospira作为有机氮降解的底物, 并为氨氧化微生物提供能源.

图4 氰酸酯酶氨基酸序列的系统发育分析Fig. 4 Phylogenetic analysis of cyanase amino acid sequences

图5 氰酸酯酶氨基酸位点的比对Fig. 5 Alignment of cyanase amino acid sites
特别地, 这些NitrospiraMAGs可能在参与有机氮转化方面具有更大的功能潜力. 本研究中获得的9个NitrospiraMAGs中注释有“nitrilase/cyanide hydratase and apolipoprotein N-acyltransferase”或“carbon-nitrogen hydrolase family protein”, 它们属于氮-碳裂解酶或腈水解酶超家族蛋白, 所有这些蛋白质似乎都参与了有机氮化合物的还原和氨的产生[58-59](图3). 另外, 本文还注意到多个站位的NitrospiraMAGs含有将一些氨基化合物降解产生氨的酶, 包括烟酰胺酶、酰胺酶和胺氧化酶, 这意味着Nitrospira可能有更多未被揭示的与氨氧化微生物耦合生长的机制, 这些酶可能对潮滩湿地有机氮的去除发挥着关键的作用.
在本研究中,NitrospiraMAGs含有多种潜在的替代能源, 暗示着Nitrospira在潮滩湿地环境中有更广泛的生存策略. 甲酸已被发现作为一些Nitrospiralineage Ⅰ、Ⅱ和Ⅵ菌株的电子供体和碳源[16,21,60].仅4个来自CM01、ZN02和QM站位的NitrospiraMAGs含有甲酸脱氢酶, 意味着这些Nitrospira可能有着利用甲酸盐作为生长能源的潜力. 尽管NitrospiraMAGs被识别出含有多种有机物利用途径,但大多数有机物被验证并不能维持Nitrospira的持续生长[15,61-62]. 有研究表明,Nitrospira在混合营养中能更好地生长[12,60-61], 意味着其可能利用部分有机物作为碳源, 这些过程可能对潮滩湿地中有机物的去除有着重要作用. 10个NitrospiraMAGs含有周质的亚硫酸盐脱氢酶(sulfite oxidoreductase,SOR)和4个lineage Ⅱ MAGs 含有硫化氢氧化还原酶(sulfide/quinone oxidoreductase, SQR) (图3),这两种酶可催化亚硫酸盐(SO3–)或硫化氢 (H2S)的氧化, 并产生电子[63-65]; 尽管这两种酶也存在于Nitrococcus和Nitrotoga中[66-67], 但仍缺乏相关的生理实验证实NOB能够利用这两种酶催化相应硫化物作为生长能源. 另外,NitrospiraMAGs含有不同类型的氢气氧化酶, 其中11个MAGs编码着Group 3b镍铁硫还原氢化酶(group 3b [Ni-Fe] sulfur-reducing hydrogenase, HYD), 该酶能将氢气氧化并产生NADPH, 同时能消耗NADPH将硫还原[55,66-68]. 3个Comammox MAGs和来自CM02站位的CM02s178 (图3) 中含有假定的Group 4氢化酶 (putative group 4 hydrogenase, HYF). 但该酶的大亚基缺失两对在镍铁类型氢化酶中均存在的镍铁中心的半胱氨酸配体[69], 这一特征在其他典型Nitrospira中也被揭示[16,70], 暗示着该酶发挥着未知的功能. 来自QM站位的QMb347和QMm174中含有Group 2a镍铁氢化酶(group 2a [Ni-Fe] hydrogenase, HUP), 该酶在Nitrospira moscoviensis菌株中被证实能够催化氢气氧化, 从而获得能量用于碳的固定和生长[16].
2.3 Nitrospira在潮滩湿地环境的适应
许多研究表明, 潮滩湿地是重金属和污染物积累的区域[71-73], 并且有着较大范围的盐度和底物浓度的变化[74]. 本研究获得的Nitrospira基因组编码着次氯酸盐歧化酶、砷酸盐还原酶和汞还原酶, 这或许能消除细胞对来自潮滩中的毒性[75-77]. 我们还发现, 这些基因组编码着超过氧化物歧化酶和过氧化物酶,这些酶能抵抗高浓度的活性氧(reactive oxygen species, ROS)[78]. 大部分的Nitrospira还编码着碳酸酐酶(carbonic anhydrase, CA), 可以将进入细胞的转化为CO2[79], 为还原型的三羧酸循环提供碳源. 根据报道, 大部分培养的Nitrospira含有鞭毛[51], 而我们获得的14个基因组中仅QM、ZN02和LH01站位中的3个编码着较完整的鞭毛系统, 这意味着潮滩湿地中的Nitrospira可能有着独立的生态位.
由于潮滩湿地有着较大的盐度差, 故Nitrospira可能需要有特殊的代谢途径来适应盐度变化较大的潮滩环境. 有研究表明,Nitrospira编码着调节渗透压的重要物质转运通道, 包括水通道 (aquaporin Z, AqpZ)、机械离子感应通道 (mechanosensitive ion channel, MSC)和甜菜碱ABC转运蛋白等[80-82].值得注意的是, 本研究获得的MAGs中, 仅隶属于lineage Ⅳ和lineage Ⅲ的MAGs编码有H+/Na+逆向转运体和甘氨酸/甜菜碱ABC转运体, 这些基因组主要来自盐度更高(9‰ ~ 27‰)的QM和LH站位. 水通道蛋白仅在隶属于lineage Ⅱ的3个MAGs中被发现(图3), 这些基因组来自盐度更低(<5‰)的CM02和ZN01站位. 有研究表明, 许多生活在低营养海洋环境中的细菌通常存在一些有机渗透物(甜菜碱、脯氨酸和甘露糖醇等)的ABC转运体[83], 使得这些细菌能够更好地适应高盐度的海洋环境[84]. 这意味着在潮滩湿地中, 不同盐度站位的Nitrospira成员有着不同的渗透压调控机制, 高盐度站位的Nitrospira可通过编码有机渗透物的ATP转运体来适应高盐度的胁迫.
令人惊讶的是, 本研究获得的这些NitrospiraMAGs的抗病毒系统有着较高的多样性(图3), 大部分MAGs编码CRISPR (clustered regularly interspaced short palindromic repeats) 相关蛋白. 然而,由于一个完整的CRISPR防御系统至少含有一个核心的cas基因[85], 而本研究获得的MAGs中仅QMb347和QMb135含有核心的cas基因, 故而暗示着其他MAGs可能不能利用CRISPR系统进行病毒的防御. 但有研究表明, 细菌的限制-修饰 (restriction-modification, R-M) 病毒免疫系统广泛存在于原核生物中, 能攻击进入细胞的外来DNA[86]. 本研究获得的NitrospiraMAGs中注释有Type Ⅰ -ⅢR-M系统的关键基因 (Restriction endonuclease和DNA methylase), 意味着该免疫系统可能在这些NitrospraMAGs中发挥着重要的病毒防御作用. 此外, 毒素抗毒素系统(toxin-antitoxin, TA) 和流产感染系统(abortive infection system, Abi)的相关基因也普遍存在于这些MAGs中, 这两种免疫系统均采用自杀方式进行病毒的抵抗[87-88]. 另外, QMm125基因组中包含噬菌体排斥系统 (bacteriophage exclusion, BREX) 的大部分基因, 也可能发挥着相应的病毒防御功能[89]. 尽管噬菌体让大量细菌感染致死并影响着微生物群落结构, 但同时也是驱动基因进化和生物遗传多样性的重要动力[90]. 特别地,NitrospiraMAGs中发现含有大量噬菌体组装和侵染相关基因, 这表明噬菌体介导的水平基因转移(horizontal gene transfer, HGT) 可能广泛发生在Nitrospira群体中[91-92], 并推动Nitrospira基因组趋于多样化和适应全球广泛的生态环境.
本研究中从潮滩湿地恢复的NitrospiraMAGs分布在较宽的纬度范围 (18.22°N ~ 40.78°N), 这些长期栖息在不同纬度位置的Nitrospira可能受到不同温度的胁迫. 研究表明, 极端嗜热蛋白需要更多的带正电荷的残基及通过牺牲极性氨基酸残基来保持蛋白质的稳定性[93]. 因此, 带电氨基酸和极性氨基酸的百分比差异(difference between the proportions of charged versus polar (non-charged) amino acids, CvP-bias)常被用来研究微生物的热适应能力[94-95]. 我们计算了14个Nitrospira基因组的CvPbias值, 将每个站位基因组的CvP-bias平均值与纬度位置进行线性回归. 结果显示, CvP-bias平均值与纬度之间有一定负相关性, 但并不显著 (y= – 5.708x+ 46.58,R2= 0.2038,p= 0.4454; 图6(a)).最小和最大的平均基因组CvP-bias值分别对应最高纬度和最低纬度. 这意味着, 在有更高温度的较低纬度位置,NitrospiraMAGs可能有更高的CvP-bias值, 即需要使用更多的带电氨基酸以适应更高的温度. 但这一结果并不显著, 这可能与基因组和站位数量有关. 为进一步验证纬度及温度与Nitrospira基因组CvP-bias值关系的显著性, 所有含纬度信息(1°N ~ 82°N)的99个Nitrospira基因组的CvP-bias值被计算. 为克服不同生境差异带来的偏差, 取每10° 纬度间的基因组CvP-bias值的平均值, 且热泉基因组被假定为赤道样品(0°), 并与纬度位置进行线性回归. 线性拟合结果显示, 基因组的CvP-bias与纬度呈现显著的负相关性 (y= – 67.99x+ 227.7,R2= 0.7739,p< 0.005; 图6(b)).这表明, 生活在较低纬度和热环境的Nitrospira在一定程度上拥有更多带电氨基酸来维持蛋白质的热稳定性, 而在高纬度位置的Nitrospira普遍有更低的CvP-bias值.

图6 纬度与平均CvP-bias值的线性关系Fig. 6 Linear relationship between latitude and CvP-bias
3 结论
本研究获得了不同纬度位置潮滩湿地的宏基因组样品, 并分析了14个从中恢复的NitrospiraMAGs的系统发育、功能潜力和潮滩环境适应机制. 结果显示, 这些NitrospiraMAGs有着丰富的物种多样性和生态功能潜力. 本研究首次获得了两个隶属于Nitrospiralineage Ⅲ的基因组, 暗示潮滩湿地含有未被揭示的类群. 代谢通路分析发现, 这些NitrospiraMAGs有多种可选择能源利用途径、压力防御方式和病毒抵抗机制. Comammox 和典型的Nitrospira在尿素和氰酸酶的利用上存在明显差异. 此外, 更多含氮有机化合物可能被这些Nitrospira作为硝化作用的能源, 这对潮滩湿地有机氮素的去除起着重要作用. 同时, 这些Nitrospira来自不同的纬度位置, 研究表明了它们在不同的站位应对盐度、温度和含氮化合物等展现出不同的适应策略. 这些研究结果加深了对Nitrospira在代谢潜力和环境适应机制上的认识, 为深入理解潮滩湿地的氮循环过程提供了重要的依据。

