基于多组学分析血红素加氧酶1在肿瘤预后中的作用
田义恒,陈玉清
(1.江西科技师范大学 药学院,江西 南昌 330038;2.河南省人民医院 血液科,河南 郑州 450000)
血红素加氧酶1(heme oxygenase 1,HMOX1)也称HO-1,是血红素分解代谢过程中的限速酶,在全身组织广泛存在,其可代谢血红素产生一氧化碳、胆绿素和亚铁,HMOX1及其酶产物在维持细胞稳态和调节重要的生物过程中发挥重要作用,包括氧化应激、炎症、细胞凋亡、细胞增殖、纤维化和血管生成[1]。HMOX1与多种癌细胞增殖密切相关,在多种肿瘤和组织中高表达,与肿瘤生长、浸润、耐药有关[2]。正常情况下,HMOX1可以保护机体,但对肿瘤组织来说,HMOX1可以增强肿瘤的抗杀伤能力,抑制肿瘤细胞死亡[3]。使用公共数据库肿瘤基因组图谱(The Cancer Genome Atlas,TCGA)和基因表达综合(Gene Expression Omnibus,GEO)数据库对HMOX1从基因表达、生存状态、基因改变、蛋白磷酸化、免疫浸润以及细胞的相关途径进行基因表达、预后生存、免疫浸润以及相关蛋白互作分析,有助于从临床肿瘤样本的角度理解HMOX1在肿瘤发生中的作用。
1 资料与方法
1.1 利用TIMER2.0和UALCAN分析HMOX1在各癌症中的表达在肿瘤免疫数据库TIMER2.0(http://timer.cistrome.org/)“Gene-DE模块”中输入HMOX1,观察不同肿瘤或特定肿瘤亚型下HMOX1在肿瘤与相邻正常组织中的表达差异[4];对于某些无正常组织或者正常组织非常有限的肿瘤,使用GEPIA2(http://gepia.cancer-pku.cn/)数据库,选择Expression DIY的“Box plot板块”,设置P值为0.01,Log2FC为1,以及“匹配TCGA正常和GTEx数据”[5]。获取这些肿瘤组织与基因型和基因表达量关联(Genotype Tissue Expression,GTEx)数据库中相应正常组织表达差异的箱线图。UALCAN(http://ualcan.path.uab.edu/)是用于分析癌症组学数据的交互式网络资源,通过使用临床蛋白质组学肿瘤分析联盟(Clinical Proteomic Tumor Analysis Consortium,CPTAC)数据集进行蛋白表达分析[6]。选择乳腺癌、卵巢癌、肾细胞癌、肺腺癌、子宫内膜癌、结肠癌、头颈部鳞状细胞癌、胰腺癌、多形胶质母细胞瘤、肝细胞癌等,分析HMOX1在10种原发肿瘤组织和正常组织蛋白中的表达情况。
1.2 利用基因表达谱在线分析平台(gene expression profiling interactive analysis,GEPIA)2数据库分析HMOX1在各癌症中的生存预后使用GEPIA2中“Survival Map”模块,获取所有TCGA数据库中表达HMOX1的肿瘤的总生存期(overall survival,OS)和无病生存期(disease-free survival,DFS)显著性图,设定临界值-高、临界值-低分别为50%、50%,初步探索HMOX1表达与肿瘤患者OS和DFS的关系。
1.3 利用肿瘤免疫浸润评估资源(Tumor Immune Estimation Resource,TIMER)2.0数据库分析HMOX1在各癌症中的免疫浸润使用TIMER2.0数据库中“Immune”模块,输入HMOX1并选择癌症相关成纤维细胞,研究TCGA不同癌症类型中HMOX1表达与癌症免疫浸润的关系。应用TIDE、MCPCOUNTER、EPIC、EXCELL算法进行免疫浸润评估。
1.4 利用STRING进行HMOX1相关蛋白互作分析利用STRING网站(https://cn.string-db.org/),输入蛋白名称HMOX1,物种选择“Homo sapiens”,得到HMOX1的蛋白互作用网络[7];使用GEPIA2网站获取与HMOX1表达相关的前100个基因,并选取5个基因,使用GEPIA2工具的“Correlation Analysis”模块分析这5个基因与HMOX1在各癌症中表达的相关性,并利用TIMER2.0数据库的“Gene_Corr”模块将这 5个靶基因在各个癌症中的表达以热图形式展示出来。此外,将前100个基因和蛋白互作的基因结合起来,并进行京都基因与基因组百科全书(kyoto Encyclopedia of Genes and Genomes,KEGG)通路分析。将基因列表上传至DAVID,选择“OFFICIAL_GENE_SYMBOL”和“Homo sapiens”,获取功能注释图的数据,使用OmicShare(https://www.omicshare.com/)平台绘制气泡图使KEGG富集通路可视化,使用微生信(http://www.bioinformatics.com.cn/)进行基因本体(Gene Ontology,GO)富集分析,使生物学过程(biological process,BP)、细胞成分(cell components ,CC)和分子功能(molecular function,MF)数据可视化。
2 结果
2.1 基因表达分析HMOX1在TCGA数据库多种癌症中的表达:HMOX1在膀胱尿路上皮癌(bladder cancer,BLCA)、乳腺癌(breast carcinoma,BRCA)、食道癌(esophageal cancer,ESCA)、多形胶质母细胞瘤(glioblastoma multiforme,GBM)、肾透明细胞癌(clear cell carcinoma of the kidney,KIRC)、肾乳头状细胞癌(papillary cell carcinoma of the kidney,KIRP)、甲状腺癌(thyroid cancer,THCA)和子宫内膜癌(endometrial cancer,UCEC)等癌症组织中的表达均高于相应正常组织(P<0.05)(图1A)。
使用GEPIA2数据库将GTEx数据集的正常组织作为对照,进一步评估HMOX1在正常组织和各癌症组织中的表达。HMOX1在GBM、KIRC、急性髓系细胞白血病(acute myeloid leukemia,AML)、脑低级别胶质瘤(lower grade glioma,LGG)、胃癌(stomach cancer,STAD)、睾丸生殖细胞瘤(testicular cancer,TGCT)和子宫癌肉瘤(uterine carcinosarcoma,UCS)等癌症组织中的表达高于相应正常组织,并且HMOX1在GBM和KIRC中呈高表达(P<0.05),与TIMER2.0分析结果一致(图1B)。
运用UALCAN平台中CPTAC数据集提供的蛋白质组数据评估HMOX1在肿瘤组织中的表达,结果显示与正常组织相比,HMOX1在肾细胞癌(renal cell cancer,RCC)、UCEC、头颈鳞状细胞癌(head and neck squamous cell carcinoma,HNSC)、胰腺癌(pancreatic cancer,PAAD)和GBM中的总蛋白表达高于正常组织(图1C)。
2.2 生存分析使用GEPIA2网站,结合TCGA和GEO数据库进行生存预后分析,结果显示HMOX1高表达与LGG(P=0.025)和葡萄膜黑色素瘤(uveal melanoma,UVM)(P<0.05)的OS预后不良相关;DFS分析结果显示,高表达HMOX1与前列腺癌(prostate cancer,PRAD)(P=0.013)、皮肤黑色素瘤(skin cutaneous melanoma,SKCM)(P=0.001 1)和肺腺癌(lung adenocarcinoma,LUAD)(P=0.048)预后不良相关。此外,HMOX1低表达的KIRC(P=0.000 62)与OS预后不良相关,低表达HMOX1与胆管癌(bile duct cancer,CHOL)(P=0.007 9)和KIRC(P=0.001 1)的DFS预后不良相关。见图2。
2.3 免疫浸润分析应用TIDE、MCPCOUNTER、EPIC、EXCELL算法探索不同癌症免疫浸润水平与HMOX1表达的相关性,发现HMOX1表达与BLCA、结肠癌(colon cancer,COAD)、HNSC、LGG、PAAD、PRAD、STAD、TGCT、UCS的癌症相关纤维细胞浸润水平呈正相关(P<0.05)。见图3。
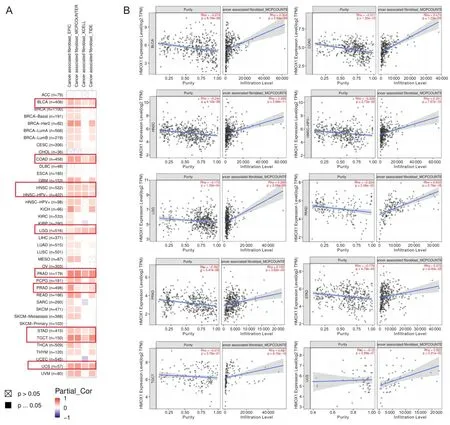
A为HMOX1表达与BLCA、COAD、HNSC、LGG、PAAD、PRAD、STAD、TGCT、UCS的关系;B为HMOX1表达癌症相关纤维细胞浸润水平呈正相关(P<0.05)。
2.4 蛋白互作分析为进一步研究HMOX1在肿瘤发生和发展中的分子机制,筛选出HMOX1相互作用蛋白以及与HMOX1蛋白表达的相关基因,并对HMOX1表达相关基因进行富集分析。使用STRING数据库获取10个HMOX1结合蛋白,使用GEPIA2工具获取HMOX1表达相关前100个基因,结果显示HMOX1与LIPA(r=0.41)、DAB2(r=0.52)、ABI3(r=0.39)、CD4(r=0.32)和CD163(r=0.40)基因表达均呈正相关。相应热图数据显示,HMOX1与上述5个基因在大部分癌症中存在正相关。将两组数据合并,通过DAVID网站进行KEGG和GO富集分析。KEGG数据表明,HMOX1在癌症中发病机制可能与破骨细胞分化、卟啉和叶绿素代谢、金黄色葡萄球菌感染、TNF信号通路有关。GO富集分析进一步表明,这些基因与炎症反应、细胞黏附有关,并且大多数基因编码定位在质膜上,促使三磷酸鸟苷和蛋白结合。见图4。

A为使用STRING数据库获取10个HMOX1结合蛋白;B为热图,数据显示HMOX1与LIPA、DAB2、ABI3、CD4和CD163基因在大部分癌症中呈正相关;C显示HMOX1在癌症中发病机制可能与破骨细胞分化、卟啉和叶绿素代谢、金黄色葡萄球菌感染、TNF信号通路有关;D为HMOX1与LIPA(r=0.41)、DAB2(r=0.52)、ABI3(r=0.39)、CD4(r=0.32)和CD163(r=0.40)基因表达均呈正相关。
3 讨论
肿瘤浸润性免疫细胞在肿瘤微环境中与肿瘤起始、发展和转移密切相关[8-9]。研究报道成纤维细胞促进肿瘤细胞生长、侵袭及免疫抑制,而且基因组稳定不易耐药,有望成为肿瘤免疫治疗的新靶标[10]。HMOX1是一种催化血红素在体内氧化降解的限速酶,广泛存在于人体各器官中,其酶活性降低可以导致氧化应激降低、炎症反应减弱和细胞凋亡率降低[11]。另外,HMOX1是血管内皮生长因子的有效诱导剂,而血管内皮生长因子是引起肿瘤血管新生的一种重要活化因子,可以促进内皮细胞增殖,从而使血管通透性增加,使细胞外基质对血管形成的诱导性生物学作用发生改变,并且高表达的HMOX1可以刺激癌细胞浸润,导致血管生成、肿瘤转移速度加快、血管通透性增加。在很多癌症中,HMOX1被认为是一种重要的生存因子,研究表明HMOX1的过度表达可促使肿瘤细胞增殖、抵抗氧化应激、血管生成及病灶转移[12],例如抑制卵巢癌细胞中HMOX1,可以抑制癌细胞增殖[13]。虽然有报道表明了肿瘤与HMOX1之间有联系,但是HMOX1在不同肿瘤中的发病机制是否起到关键作用,通过怎样的分子机制起作用还有待回答。
因此,本研究使用TCGA和GEO数据库分析了数据库中不同肿瘤类型,发现HMOX1在大多数肿瘤中高表达。研究表明HMOX1在膀胱癌、卵巢癌、食管鳞状上皮细胞癌中的表达均高于相应的正常组织,发挥促进肿瘤增殖和迁移的作用[9,14-15]。但是预后生存分析表明HMOX1表达的高低在不同肿瘤中的预后有一定的差异性。DFS分析结果显示,高表达HMOX1与PRAD、SKCM和LUAD预后不良相关。低表达KIRC与OS预后不良相关,低表达HMOX1与CHOL和KIRC的DFS预后不良相关。
肿瘤微环境主要由免疫细胞、肿瘤相关成纤维细胞、炎症因子、细胞外基质以及癌细胞自身等共同组成,癌细胞的存活、生长、侵袭、迁移都受到周围肿瘤微环境的影响。癌症相关成纤维细胞作为肿瘤微环境中的主要成分之一,被认为是影响肿瘤微环境免疫调节的主要参与者,可通过多种途径促进肿瘤的免疫逃逸[16],并且癌症相关成纤维细胞在肿瘤的发展中发挥着多种作用,例如可以通过分泌促进癌细胞增殖、免疫排斥的生长因子和细胞外基质蛋白发挥促进肿瘤细胞增殖、转移和侵袭作用,促进肿瘤耐药,调节免疫细胞活性并抑制抗肿瘤免疫应答[17]。在原发性和转移性肿瘤中发现的癌症相关成纤维细胞是高度通用的、可塑的和有弹性的细胞,它们通过与肿瘤微环境中其他细胞类型的复杂相互作用积极参与癌症进展[18]。本研究主要分析了HMOX1表达与癌症相关成纤维细胞浸润水平之间的相关性,发现HMOX1表达与BLCA、COAD、HNSC、HPV-HNSC、LGG、PAAD、PRAD、STAD、TGCT、UCS的癌症相关纤维细胞浸润值呈正相关。
本研究对HMOX1靶向结合蛋白和HMOX1蛋白表达相关基因进行了一系列富集分析,结果显示HMOX1在癌症中发病机制可能和肿瘤坏死因子(tumor necrosis factor,TNF)信号通路、炎症反应、细胞黏附有关。有研究表明TNF信号与塑造肿瘤微环境有关,并且在癌症发展过程中,TNF有一定的抗癌作用,其途径有诱导细胞凋亡、影响肿瘤血管系统和增强宿主免疫力3个方面。但是TNF对肿瘤的作用具备双向性,既有抗瘤作用也具有促进肿瘤作用,其浓度非常关键,高浓度TNF-α可选择性破坏肿瘤血管系统发挥抗肿瘤效应,低浓度的TNF通过激活NF-κB信号通路,发挥抑制细胞凋亡和促进肿瘤增殖作用[19]。通过建立小鼠结肠癌肝转移模型,研究TNF与HO-1与结肠癌发展之间关系,发现增加TNF水平,降低HO-1水平可以抑制肿瘤生长[20]。
综上所述,本研究通过对HMOX1进行了全面的泛癌分析,发现HMOX1表达与多种人类癌症的临床预后、免疫浸润等存在相关性,有助于从临床肿瘤样本的角度阐述HMOX1在肿瘤发生中的作用,为临床诊断和治疗提供依据与帮助。

