基于微卫星标记的中华鲟亲子关系判别及案例分析
赵娜 常剑波 陶江平 孙行


摘要:利用野外和人工繁殖中华鲟(Acipenser sinensis)均有的产卵洄游入海特性,通过在长江口水域捕获滞留的中华鲟幼鱼并区分人工繁殖放流个体和野外个体,可以评估其人工繁殖放流个体占比情况。基于孟德尔遗传模式,在应用前期开发中华鲟特异性四倍体微卫星标记的基础上,构建了基于四倍体微卫星位点遗传模式的亲子鉴定方法,并用全同胞家系、半同胞家系和非亲缘关系群体样本验证方法的有效性,将此方法用于1999年度中华鲟人工繁殖效果评估案例中,计算人工繁殖个体在长江口中华鲟幼鲟群体中的比例。结果显示,3个全同胞家系个体间的平均遗传距离分别为0.43、0.44和0.44,半同胞家系的遗传距离居中(0.57),长江口中华鲟幼鲟群体间的平均遗传距离为0.74。人工繁殖效果评估显示,在2000年度长江口幼鱼群体中,人工繁殖个体的比例较低(0~3.8%)。研究表明,当年度中华鲟补充群体主要来源于自然繁殖,需要继续加大对中华鲟野生群体的保护力度,增加人工繁殖群体的放流规模。
关键词: 中华鲟;亲子鉴定;人工繁殖;自然群体;微卫星DNA
中图分类号:Q503 文献标志码:A 文章编号:1674-3075(2023)05-0092-08
中华鲟属于大型溯河产卵洄游鱼类,其生活史复杂(Wu,1963;Ke & Tian,1988;Chang & Cao,1999);对长江群体而言,其生活史大多在中国黄海和东海大陆架海域度过(Wu,1963;Zhang,1988)。每年春夏季,即将成熟的繁殖群体进入长江口并溯河而上,在长江度过14~17个月,繁殖后返回大海(Zhang,1988;危起伟,2003;Zhuang et al,2009)。秋季在葛洲坝坝下江段自然繁殖的中华鲟受精卵,在产卵场孵化后随水流向长江下游漂移(庄平,1999;Zhuang et al,2002);幼鲟一般于次年4月中旬到达或接近长江口浒浦江段,5月中下旬开始出现在长江口(危起伟,2003)。
1981年实现长江截流的葛洲坝一期工程阻断了中华鲟繁殖洄游通道,随后天然种群数量急剧减少,1983年长江中华鲟人工繁殖放流工作开展。危起伟(2003)采用微型线码标志(Coded Wire Tag,CWT)和外挂银制标志牌的方法研究幼鲟迁移路线,发现人工放流的中华鲟2月龄幼鲟具有与自然种群相似的洄游特性,最早在第二年3月到达长江口。由于野外和人工繁殖中华鲟幼鲟有这种相似的归海洄游特性,在长江口捕获的滞留幼鲟中,包含了野外和人工繁殖放流个体,因此为评价人工繁殖放流个体占比提供了途径。前期已有学者分别应用荧光染色、微卫星和人工神经网络结合以及CWT方法进行人工繁殖放流效果评估(常剑波,1999;Zhu et al,2002;危起伟,2003)。水工程生态研究所实验室通过多年研究,积累了大量自行开发的中华鲟特异性微卫星DNA标记(Zhu et al,2005),并对其多倍体模式进行了深入分析(Zhao et al,2015)。本研究拟采用四倍体遗传模式的中华鲟特异性微卫星标记,构建其在亲子关系判别中的统计分析方法,并应用本方法鉴定和区分长江口幼鲟自然繁殖放流个体,评估当年度长江口中华鲟人工繁殖放流个体占比。
1 材料和方法
1.1 材料选取
1.1.1 人工繁殖家系样本 本研究中的样本来自水利部中国科学院水工程生态研究所样本库。为检验本研究统计方法的准确性,将2001年长江水产研究所通过人工催产方法获得的3个人工繁殖家系作为检验样本,其中A和B是同父异母的半同胞家系(表1)。研究中分别用Af、Bf和Cf代表3个家系的母本,ABm代表A、B家系的同一父本,Cm为C家系父本。家系子代个体分别用A1~A27,B1~B26和C1~C43编号。亲本取鳍条或肌肉样本保存在无水乙醇中。子代样本用70%乙醇溶液浸泡,置于4℃冰箱中备用。
1.1.2 人工繁殖和放流亲本 1999年度中华鲟人工繁殖放流工作由葛洲坝中华鲟研究所和长江水产研究所共同承担。当年一对亲本1999-YL-103(♂)×1999-YR-402(♀)配对繁殖成功;1999年12月28日,中华鲟研究所成功放流这对亲本繁殖的子代30 000尾。
1.1.3 长江口幼鲟群体样本 2000年5-7月,在长江口崇明岛东旺沙和团结沙地区,通过插网方式和收集渔民误捕共采集到幼鲟90尾,以Juv表示幼鲟个体。每尾幼鲟剪取2~4 cm2的尾鳍或胸鳍作为样本,浸泡于无水乙醇中保存,活体重新放回长江口,已死亡的个体用乙醇浸泡后带回实验室保存。样本编号为Juv01~Juv90。
1.2 微卫星引物来源
水工程生态研究所实验室自行开发的中华鲟微卫星特异性引物As-073、As-074和As-102扩增图谱清晰、稳定,并具有较强的个体识别能力(Zhao et al,2005;2015)。微卫星位点特征(Zhu et al,2005)见表2。
1.3 PCR方法
使用DNeasy 组织试剂盒 (Qiagen,Valencia,CA,USA) 提取DNA样本。PCR 10 μL反应体系包括正反向引物各1 μM,dNTP 0.2 mM,0.4 U Taq DNA聚合酶和10~15 ng模板DNA。反应条件如下:94℃预变性5 min,45个循环,包括94℃变性40 s,56℃或58℃退火40 s,72℃延伸50 s;72℃最后延伸10 min。以H2O为模板设置PCR阴性对照。采样阴性对照和PCR阴性对照,扩增结果均为阴性。扩增产物上样于10%聚丙烯酰胺凝胶(19:1),于200 V电压下电泳3.5 h,溴化乙锭染色20 min,用Gene Genius凝胶成像系统(SynGene,英国)扫描记录。通过GeneTool凝胶分析软件(SynGene)比对pBR322 DNA/Msp Ⅰ Markers并确定片段大小。根据微卫星片段长度进行基因分型。
1.4 数据分析
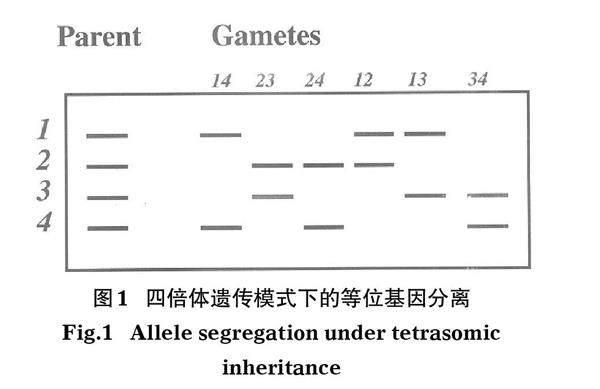
研究中应用的微卫星位点均在中华鲟上呈现四倍体孟德尔遗传模式(Zhao & Chang,2006;Zhao et al,2015),即每个亲本在1个位点上携带4个等位基因,形成配子时,都会有任意2个等位基因随机组合并遗传给子代(图1)。因此,可以通过计算长江口子代个体与葛洲坝放流亲本之间的遗传距离来判断是否存在亲子关系,从而评估人工繁殖放流个体占比。
首先,明确所有研究个体的基因型。如在1个微卫星位点检测到6个等位基因,分别是220 bp(等位基因1)、224 bp(等位基因2)、228 bp(等位基因3)、232 bp(等位基因4)、236 bp(等位基因5)、240 bp(等位基因6)长度的片段,可以用片段长度(220/224/232/240)表示1个个体的基因型,也可以用基因编号(1/2/4/6)表示,本研究采用基因编号型。如同二倍体中的纯合子,四倍体中也有携带相同等位基因的情况出现,称之为“剂量”,可以通过测序图谱的峰值或者电泳凝胶的亮度判断(如图2中个体4和个体5)。在这种情况下,用(1/1/2/3)表示基因型。如果有无效等位基因的存在,可能会出现(1/2/3)情况。
第二步,根据Lynch(1990)公式计算个体之间的遗传距离:
Dxy = 1- [2NxyNx+Ny] ①
式中:Dxy表示个体x和个体y之间的遗传距离;Nxy表示个体x和y在一个微卫星位点上共有的等位基因数目;Nx和Ny分别表示个体x、y在这个位点上所拥有的所有等位基因数目。本研究中使用的3个微卫星位点是四倍体位点,因此N应该是4。在双亲基因型完全相同的情况下,子代与亲本的遗传距离可能是0(D = 1- [2×44+4] )。在没有无效等位基因和双亲不共享任一等位基因的情况下,子代与亲本的遗传距离应该是0.5(D = 1- [2×24+4])。但也存在1个无效等位基因的情况下,N会是3(更多无效等位基因的情况不考虑)。因此,若子代与成体存在亲子关系,则至少会共有1个(有无效等位基因存在)或2个(正常情况)等位基因,其遗传距离可能会是0.714 (D = 1- [2×13+4])或者0.429 (D = 1- [2×23+4])。所以,只有当子代个体同繁殖父本、母本之间距离都在这个范围内时,才可以判定其是真实子代。
为检验方法的准确性,首先用引物As-073对3个家系样本进行验证;另外,分析全同胞、半同胞和无亲缘关系3种情况下个体之间的遗传距离;最后,分析长江口幼鲟和葛洲坝放流亲本之间的遗传距离,判断是否存在亲子关系,评估人工繁殖的子代在长江口幼鲟群体中的占比。
2 结果与分析
2.1 已知亲缘关系的家系子代与亲本的遗传距离
在As-073位点上3个已知亲缘关系的家系中个体之间的遗传距离见表3。家系A中,绝大多数子代个体与亲本的遗传距离是0.5,由于父本和母本的基因型分别为2/3/11/11 和2/5/6/8,共享一个等位基因2,因此有部分子代与亲本的遗传距离是0.25。在家系B中,同样是双亲(2/3/11/11 ×1/2/10/12)共享等位基因2,因此也有部分子代与亲本的遗传距离表现为0.25。在家系C中,双亲的基因型分别为2/3/5/9 和4/4/7/8,不共享任何等位基因,子代个体从双亲中遗传任意2个等位基因的组合;在这些组合中,没有等位基因相同的可能性,因此所有子代都一致性地表现出与亲本的遗传距离为0.5;子代C16和C41个体DNA样品提取不成功,因此没有获得遗传学数据;除5个个体(C9、C12、C20、C36和C42)与C家系双亲遗传距离为0.5外,同时与A家系亲本之间的遗传距离也为0.5,这是因为家系A双亲的基因型为2/3/11/11和2/5/6/8,家系C双亲的基因型为2/3/5/9和4/4/7/8,2个家系的双亲之间共有等位基因2、3、5、8;以家系C9子代为例,具有基因型2/3/4/8,那么其与Af、ABm、Cm和Cf任一亲本都共有2个等位基因,因此遗传距离均为0.5。
在As-073位点上,3个家系的遗传图谱没有揭示无效等位基因的存在。因此,如果疑似亲本是真实的,子代与疑似双亲之间的遗传距离都应在0~0.5,单是与一方亲本的遗传距离不在这个范围内都将被排除。照此标准判断,A家系的27个子代都准确地定位于双亲Af×ABm中,准确率为100%;B家系的26个子代也全部归属于双亲Bf×ABm中;C家系43个子代中的38个个体判断为亲本Cm×Cf 的后代,准确率为88.4%。对于其余5个个体(C9、C12、C20、C36、C42),若要正确判断是双亲Cm×Cf 的后代,还是双亲Af×ABm的后代,需要使用更多的微卫星位点。
2.2 不同亲缘关系的个体间遗传距离
全同胞家系、半同胞家系以及无亲缘关系的个体间遗传距离分布见图3。其中,A、B、C为3个全同胞家系,其内部同胞个体间平均距离分别为0.43、0.44、0.44,平均值为0.437;A和B是同父异母的半同胞家系,个体间平均距离为0.57;A和C家系个体间平均距离为0.73,B和C家系间平均距离为0.83;长江口随机采样的中华鲟幼鲟个体间平均距离为0.74。
2.3 长江口幼鲟群体人工繁殖放流个体占比
2000年长江口采集的90尾幼鲟中,只有52尾在As-073、As-074和As-102全部3个位点得到清晰并可准确计带的指纹图谱,因此只对这些幼鲟的遗传学数据进行分析。2000年长江口捕获的幼鲟个体与1999年参与人工繁殖放流亲本之间的遗传距离见表4。1999年成功参与人工繁殖的一对亲鲟用♀、♂表示。♀在As-073和As-074位点上都只扩增出3个亮度相同的条带,因此存在1个无效等位基因;在随后的距离分析中,疑似子代与♀、♂之间遗传距离在0~0.714都应该判断为其子代个体。
单个位点的分析结果显示,在As-073位点上,有13个个体与♀、♂遗传距离在0~0.714,疑似子代比例为25%;As-074位点上,62%的个体为疑似子代;As-102位点上,疑似子代的比例最小,仅21%。
2个位点的联合使用结果显示,As-073/As-074组合排他能力最弱,判断8个个体为疑似子代,比例为15.4%;As-073/As-102组合的排他能力最强,判断2个为疑似子代,比例为3.8%;As-074/As-102组合的排他能力居中,判断疑似子代为6个,比例为11.5%。
3个位点联合使用后,只有Juv40个体与♀、♂遗传距离满足判别条件,因此在52个长江口幼鲟中,只有1尾被判断为人工繁殖放流的子代个体,仅为1.9%。随着更多微卫星位点的使用,Juv 40个体与♀、♂之间的亲子关系也可能会被排除,因此2000年捕获的长江口幼鲟群体属人工繁殖放流的个体仅占0~1.9%。
3 讨论
3.1 微卫星标记用于亲子分析的技术缺陷
微卫星标记由于高度多态性,在亲子鉴定中有很强的个体识别能力 (Hansen et al,2001;Wilson & Ferguson,2002;Baumsteiger et al,2008);但标记本身也存在技术缺陷 (Van Oosterhout et al,2004),如无效等位基因、连锁不平衡、突变或者计带错误等原因,都会造成子代基因型与真实亲本不相符,从而降低亲子鉴定的准确性 (Wilson & Ferguson,2002; Neff et al,2000a; 2000b; Gold et al,2011)。无效等位基因的存在可以导致哈迪-温伯格平衡偏离,因此可以通过群体研究检测出无效等位基因存在与否。在亲子分析中,位点物理连锁或者连锁不平衡所造成的结果不同 (Waples,2006; Waples & Do,2010)。在使用存在连锁不平衡的2个位点时,由于位点之间存在的非随机联合,减少了有效的变化水平,因此降低了标记的排除能力和亲子鉴定结果的准确性。然而,在实际研究中,由于应用的标记位点较少,多数研究者都假设这些位点不存在物理连锁或连锁不平衡;另外,由于对基因突变机制的认识不足,识别突变问题最好的途径是比较亲本和子代的遗传图谱 (Neff et al,2000a; 2000b)。
3.2 实际应用中微卫星标记的选择
本研究中,位点As-102具有最高的个体识别能力(排除能力强),As-073居中,As-074的识别能力最小。亲子鉴定的准确性依赖于使用的统计方法,同时也受到标记本身的影响。使用的多态性位点数目越多,鉴定结果越接近真实情况。但在实际应用中,考虑实验成本和实验时间等因素(Jone & Ardren,2003),使用高效标记位点可以达到事半功倍的效果。如使用排他能力最强的As-073/As-102组合,排除了52个子代中的50个个体;使用排他能力最弱的As-073/As-074组合,只能排除44个个体。因此,检验微卫星位点的个体识别能力也是亲子鉴定实践中的重要一环。
3.3 平均遗传距离揭示的亲缘关系
四倍体生物微卫星位点遗传模式复杂 (Jones & Ardren,2003;Jones et al,2010),特别是当双亲之间共享等位基因或者存在无效等位基因时,同胞个体之间的遗传距离变化很大。通过计算两两个体之间的平均遗传距离,可以粗略揭示其亲缘关系。本次研究中,家系A、B、C尽管样本量有差异,但3个全同胞家系的平均遗传距离很接近(0.43、0.44、0.44);A和C家系的亲本共享4个等位基因,因此个体之间的平均遗传距离要小于不共享任一等位基因的B和C家系(0.73<0.83)。A和B家系属于半同胞家系,因此遗传距离居中(0.57),这也从侧面验证了本研究数据处理方法的可行性;此分析方法可用于鱼类人工增殖放流效果评价、鱼类个体间亲缘关系判别、集群鱼类的分布与扩散模式分析以及鱼类养殖中的遗传学管理等 (Berejikian et al,2001; Hansen et al,2001; Blouin,2003; Suski et al,2003; Hessenauer et al,2012; Hessenauer et al,2014)。
3.4 当年度长江口人工繁殖放流个体占比
危起伟(2003)采用CWT和外挂银制标志牌的方法对人工放流的部分幼鲟进行标记放流和回捕试验,发现人工放流对长江口幼鲟的补充作用不明显,连续4年(1998-2002)各标志放流约2万尾对长江口幼鲟群体的贡献率为0~0.45%;常剑波(1999)根据荧光标记放流,估算1996-1999年全长8~10 cm人工放流个体占长江口幼鲟的数量分别为1.71%、1.89% 和3.03%;Zhu等(2002)应用中华鲟微卫星数据按照二倍体显性标记(只记录图谱条带有无,不考虑剂量)的分析方法评价人工繁殖放流效果,发现1999年度放流的中华鲟人工繁殖个体在长江口幼鲟群体中占5%~10%,2000年度人工放流个体占13.3%。本研究结果评估1999年度中华鲟人工繁殖放流个体对长江口中华鲟幼鱼群体的贡献率为0~1.9%。以上结果均表明,各研究年度内中华鲟补充群体主要来源于自然繁殖,珍稀特有物种的恢复十分艰难,需要继续加大对中华鲟野生群体的保护力度,增加人工繁殖群体的放流规模。
参考文献
常剑波,1999. 长江中华鲟繁殖群体结构特征和数量变动趋势研究[D]. 武汉:中国科学院水生生物研究所.
危起伟,2003. 中华鲟繁殖行为生态学与资源评估[D]. 武汉:中国科学院水生生物研究所.
庄平,1999. 鲟科鱼类个体发育行为学及其在进化与实践上的意义[D]. 武汉:中国科学院水生生物研究所.
Baumsteiger J, Hand D M, Olson D E, et al, 2008. Use of Parentage Analysis to Determine Reproductive Success of Hatchery-Origin Spring Chinook Salmon Outplanted into Shitike Creek, Oregon[J]. North American Journal of Fisheries Management, 28:1472-1485.
Berejikian B A, Tezak E P, Schroder S L, 2001. Reproductive Behavior and Breeding Success of Captively Reared Chinook Salmon[J]. North American Journal of Fisheries Management, 21:255-260.
Blouin M S, 2003. DNA-based methods for pedigree reconstruction and kinship analysis in natural populations[J]. Trends in Ecology and Evolution, 18:503-511.
Chang J B, Cao W X, 1999. Histroy and prospect of conservation on Chinese sturgeon in the Yangtze River[J]. Acta Hydrobiol, 23:713-720.
Gold J R, Saillant E,Cummings N J,et al,2011. Genetic Divergence and Effective Size among Lane Snapper in US Waters of the Western Atlantic Ocean[J]. North American Journal of Fisheries Management, 31:209-223.
Hansen M M,Kenchington E,Nielsen E E,2001. Assigning individual fish to populations using microsatellite DNA markers[J]. Fish and Fisheries, 2:93-112.
Hessenauer J M,Bremigan M T,Scribner K T,2012. Genetic pedigree reconstruction facilitates lake wide estimates of age-0 largemouth bass dispersal[J]. Transactions of the American Fishery Society, 141:1672-1681.
Hessenauer J M,Bremigan M T,Scribner K T,2014. Genetic pedigree reconstruction detects bias in largemouth bass nest sampling procedures[J]. North American Journal of Fisheries Management, 34:175-183.
Jones A G,Ardren W R,2003. Methods of parentage analysis in natural populations[J]. Molecular Ecology,12:2511-2523.
Jones A G,Small C M,Paczolt K A,et al, 2010. A practical guide to methods of parentage analysis[J]. Molecular Ecology Resource, 10:6-30.
Ke X T,Tian Y P,1988. Nature spawning of Chinese sturgeon[C]// In:Shi,BN. (Ed.) The biology of sturgeons in Yangtze River and their artificial propagation[M]. Chengdu: Sichuan Scientific and Technical Publishing House.
Lynch M,1990. The similarity index and DNA fingerprinting[J]. Molecular Biology and Evolution, 7:478-484.
Neff B D,Repka J,Gross M R,2000a. Parentage analysis with incomplete sampling of candidate parents and offspring[J]. Molecular Ecology, 9:515-528.
Neff B D,Repka J,Gross M R,2000b. Statistical confidence in parentage analysis with incomplete sampling: how many loci and offspring are needed[J]. Molecular Ecology, 9:529-539.
Suski C D,Svec J H,Ludden J B,et al, 2003. The Effect of Catch-and-Release Angling on the Parental Care Behavior of Male Small mouth Bass[J]. Transactions of the American Fisheries Society, 132:210-218.
Van Oosterhout C, Hutchinson W, Will D, et al, 2004. Micro-checker: software for identifying and correcting genotyping errors in microsatellite data[J]. Molecular Ecology Notes, 4:535-538.
Waples R S, 2006. A bias correction for estimates of effective population size based on linkage disequilibrium at unlinked gene loci[J]. Conservation Genetics, 7:167-184.
Waples R S,Do C,2010. Linkage disequilibrium estimates of contemporary Ne using highly variable genetic markers: a largely untapped resource for applied conservation and evolution[J]. Evolutionary Applications, 3:244-262.
Wilson A J,Ferguson M M,2002. Molecular pedigree analysis in natural populations of fishes: approaches, application, and practical consideration[J]. Canadian Journal of Fisheries and Aquatic Sciences, 59:1696-1707.
Wu X W,1963. Economic Animals of China (Freshwater Fishes)[M]. Beijing:Science Press.
Zhang M K,1988. Migration and distribution of Chinese sturgeon[C]//In: Shi BN(Ed), The biology of sturgeons in Yangtze River and Their Artifical propagation[M]. Chengdu: Sichuan Scientific and Technical Publishing House: 85-89.
Zhao N,Ai W,Shao Z,et al,2005. Microsatellites assessment of Chinese sturgeon (Acipenser sinensis Gray) genetic variability[J]. Journal of Applied Ichthyology, 21:7-13.
Zhao N,Chang J B,2006. Microsatellite loci inheritance in the Chinese sturgeon Acipenser sinensis with an analysis of expected gametes ratios in polyploidy organisms[J]. Journal of Applied Ichthyology, 22:89-96.
Zhao N,Qiao Y,Zhu B,et al,2015. Identification ability of tetraploid microsatellite loci in parentage analysis[J]. Journal of Applied Ichthyology,31:614-619.
Zhu B,Zhou L F,Cao H,et al,2002. Analysis of genetic variation in the Chinese sturgeon, Acipenser sinensis: estimating the contribution of artificially produced larvae in a wild population[J]. Journal of Applied Ichthyology,18:301-306.
Zhu B,Liao X,Shao Z,et al,2005. Isolation and characterization of microsatellites in Chinese sturgeon, Acipenser sinensis[J]. Molecular Ecology Notes, 5:888-892.
Zhuang P,Kynard B,Zhang L Z,et al,2002. Ontogenetic behavior and migration of Chinese sturgeon, Acipenser sinensis[J]. Environmental biology of fish, 65:83-97.
Zhuang P,Liu J,Wang Y,et al,2009. The Yangtze estuary nature reserve for Chinese sturgeon: Scientific studies and management[M]. Beijing: China Ocean Press.
(责任编辑 万月华)
Parentage Identification and Case Analysis of Chinese Sturgeon
(Acipenser sinensis) Populations Based on Microsatellite Markers
ZHAO Na1, CHANG Jian‐bo2, TAO Jiang‐ping1, SUN Hang1
(1. Key Laboratory of Ecological Impacts of Hydraulic-Projects and Restoration of Aquatic Ecosystem
of Ministry of Water Resources, Institute of Hydroecology, Ministry of Water Resources and
Chinese Academy of Sciences, Wuhan 430079, P.R. China;
2. State Key Laboratory of Water Resources and Hydropower Engineering Science,
Wuhan University,Wuhan 430072, P.R. China)
Abstract:Artificially propagated juvenile Chinese sturgeons have the same migration pattern of arriving at the estuary of the Yangtze River. This provides a way to evaluate the effect of artificial breeding and release of Chinese sturgeon by distinguishing between artificially bred and wild individuals collected in the estuary of Yangtze River. Based on Mendelian inheritance and previously developed tetraploid microsatellite markers for Chinese sturgeon, a paternity test method using a tetraploid microsatellite locus inheritance model was constructed and verified with specimens of full-sibling families, half-sibling families and unrelated populations. This method was then applied to determine the percentage of released artificial individuals among the wild Chinese sturgeons in 90 samples collected in the Yangtze River estuary from May to July of 2000. The results were used to evaluate the effectiveness of the artificial bred Chinese sturgeon released on December 28, 1999. The average genetic distance of the three full-sib families calculated by the paternity testing was 0.43, 0.44 and 0.44. The genetic distance of the half-sib family was 0.57. The distance among the juveniles collected in the estuary was 0.74 and the proportion of artificially propagated individuals among juveniles collected in 2000 was in the range of 0-3.8%. Most of the juveniles in the estuary were therefore from natural reproduction. It is necessary to continue to strengthen the protection of the wild population of Chinese sturgeon and increase the scale of the release of artificial breeding population.
Key words:Acipenser sinensis; paternity testing; artificial breeding; natural population; microsatellite DNA
收稿日期:2020-06-08 修回日期:2023-06-12
基金项目:国家重点基础研究发展计划项目(2015CB150701)。
作者简介:赵娜,1978年,女,博士,研究员,主要从事分子生态学研究。E-mail:zhaona@mail.ihe.ac.cn

