基于环境DNA宏条形码的鄱阳湖真核浮游植物多样性研究
郭婷 付智豪 周春花 陈金萍 欧阳珊 吴小平


摘要:探索鄱阳湖真核浮游植物多样性,可为环境DNA监测水生态系统的应用及标准化提供基础资料。于2019年4月在鄱阳湖北部通江水道湖区、中部鄱阳湖湖区和南部人工养殖湖泊区共设18个采样点,采集鄱阳湖环境水样,针对真核浮游植物18S rDNA 基因的V9区域进行PCR扩增,高通量测序并结合生物信息学技术分析鄱阳湖浮游植物的群落组成。结果表明,基于环境DNA宏条形码技术鉴定到浮游植物10门24纲54目101科166属,其中绿藻门和硅藻门种类较为丰富。中部区域的浮游植物群落多样性和均匀度较高,且鄱阳湖浮游植物整体丰富度较高。非度量多维尺度分析(NMDS)表明,中部与北部区域(P=0.004)、南部与北部区域(P=0.011)之间群落结构差异显著。冗余分析表明,叶绿素a、pH、流速对各区域的浮游植物群落影响较显著。环境DNA宏条形码作为一种新兴的生物多样性监测手段,可快速检测鄱阳湖浮游植物生物多样性及其空间分布,为鄱阳湖生物多样性监测以及生态系统健康评估提供新的技术手段。
关键词:环境DNA宏条形码;浮游植物;多样性;群落结构;鄱阳湖
中图分类号:Q145 文献标志码:A 文章编号:1674-3075(2023)05-0067-09
浮游生物是水生生态系统的重要参与者之一,能够维持水体环境中食物网的结构和功能(Rubin & Leff, 2007),浮游植物是水生态系统的主要初级生产者(Reynolds, 1984),其群落和分布特征可以反映水体的环境变化和营养状态(Reynolds et al, 1993;Chen et al, 2003;Wu et al, 2011)。目前,对于浮游植物的传统定量调查研究都是通过采集水样固定后沉淀,然后使用显微镜对其进行形态分类并计数(胡鸿钧和魏印心, 2006;Soares et al, 2011)。传统的监测方法在形态鉴定方面存在一定困难,需掌握专业的分类学知识。浮游生物种类繁多且个体微小,导致难以实现大量样本镜检,容易产生人工误差(Xiao et al,2014;Bucklin et al,2016);此外,传统方法较难估计浮游植物的生物多样性,监测通常仅限于某些群体(Eiler et al,2013;Visco et al, 2015)。
近年来,快速发展的环境DNA (eDNA)技术提供了新的解决方案,可以更容易评估生态系统的生物多样性。从最简单的意义上讲,环境 DNA 是从任何类型的环境样本(土壤、水和空气等)中提取DNA,无需分离特定生物体(Thomsen & Willerslev, 2015)。环境DNA宏条形码(eDNA metabarcoding)技术通过对环境DNA序列分析即可检测物种的存在,无需干扰或者观察实际生物体(Bohmann et al,2014;Thomsen & Willerslev,2015),可用于描述具有高质量参考库的环境DNA的分类组成,提供了一种高效率、非侵入性的生物多样性调查方法,同时也提供了克服基于形态分类法生物评估局限性的机会(Yang & Zhang,2019)。但该方法也存在丰度较低的物种被过滤、采样流程等过程缺乏统一标准等问题(Bush et al, 2019)。Bombin等(2020)使用环境DNA宏条形码技术,利用部分LSU rDNA和23S rDNA质体分子标记,阐述了墨西哥北部湾滨海的藻类多样性。
鄱阳湖是长江流域最大的通江湖泊,对维持区域生态平衡具有重要意义(Li et al, 2019)。尽管人类活动和非生物因素对鄱阳湖的生态系统造成了较为严重的影响,但其作为中国最大的淡水湖泊,为本地经济发展提供了丰富的资源。因此,持续和长期的水质及生物监测对于保护这一生态系统至关重要(Wu et al,2013)。鄱阳湖浮游植物群落结构的动态变化及驱动机制成已成为研究热点(Chen et al,2013),但鲜见利用环境DNA宏条形技术的相关研究。本研究将环境DNA宏条形码技术应用于鄱阳湖浮游植物群落调查,旨在揭示鄱阳湖浮游植物的组成及空间分布,为鄱阳湖水体环境变化监测提供一种新的技术手段,这种方法可长期稳定监测生态系统变化并有机会识别一些响应外部干扰的早期信号。
1 材料与方法
1.1 样品采集
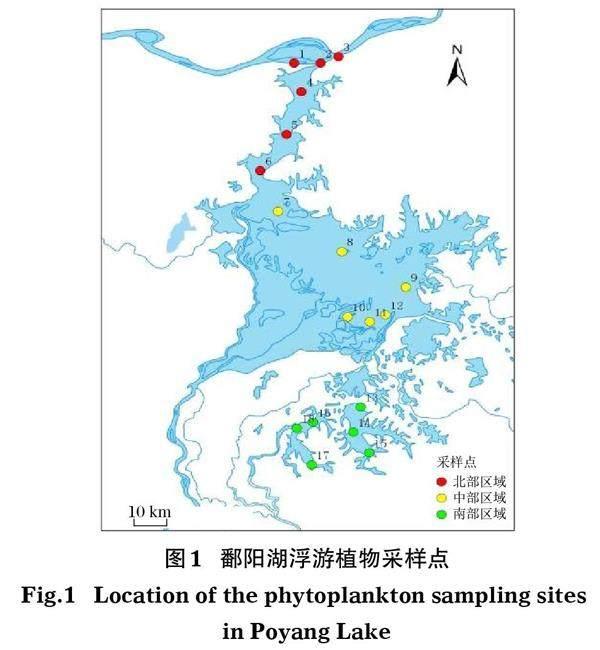
根据各样点的地理位置,将鄱阳湖划分为3个采样区域,共设18个采样点(图1),即北部区域(1~6)主要是通江水道湖区,中部区域(7~12)为鄱阳湖湖区,南部区域(13~18)为人工养殖湖泊,即青岚湖和军山湖2个湖汊。于2019年4月进行水样采集,每个采样点使用容量为1 L的有机玻璃采水器采集样品,每个样点重复3次。样品保存在已灭菌并可密封的广口瓶中,置于冰盒上尽快送回实验室过滤。每个样品使用0.45 μm的混合纤维素滤膜 (天津津腾)和真空蠕动泵(Rocker400, 中国台湾)进行抽滤。为了防止在抽滤过程中的DNA污染,在每次抽滤样品的同时,过滤同体积的超纯水作为空白对照。在样品抽滤完成之后,将富集DNA的滤膜分别装入在2 mL离心管中,-20℃保存直至提取。
1.2 DNA提取
获取的样本采用DNeasy Blood & Tissue Kit试剂盒(DNeasy plant kit, QIAGEN, 德国)进行滤膜DNA的提取,最后用60 [μ]L Elution Buffer洗脱DNA,保存于-20℃冰箱。为确保操作过程无污染,提取1张未使用的滤膜作为空白对照。使用Qubit (Thermo Fisher Scientific,中国)测量DNA浓度。
1.3 PCR扩增
利用通用引物针对真核浮游植物18S rDNA 基因的 V9 区域(长度约130 bp)进行PCR扩增(Amral-Zettler et al, 2009),并设置阴性对照,确保测定过程中未受到DNA污染。引物前带有8个碱基的barcode。25 μL反应体系包括正反向引物(10 μM)各1 μL,5×reaction buffer 5 μL, 5×GC buffer 5 μL, dNTP(2.5 mM) 2 μL, DNA 模板2 μL, ddH2O 8.75 μL, Q5 DNA Polymerase 0.25 μL。反应条件如下:98℃预变性2 min,30个循环包括98℃变性15 s、55℃退火30 s、72℃延伸30 s,72℃最后延伸5 min。
每个样品重复扩增3份,将同一样品的PCR产物混合通过2%的凝胶电泳进行检测,PCR阴性对照和空白对照的样本均未出现扩增条带,表明在采样及实验操作过程中均未受污染。
1.4 高通量测序
将上述获取的DNA样本送至上海派森诺生物科技有限公司进行高通量测序,采用Illumina Miseq平台对样本DNA片段进行双端(Paired-end)测序。通过QIIME软件(Quantitative Insights Into Microbial Ecology, v1.8.0,http://qiime.org/)调用USEARCH(v5.2.236, http://www.drive5.com/usearch/)检查并剔除嵌合体序列;之后调用UCLUST序列比对工具(Edgar, 2010)对前述获得的序列按97%的序列相似度进行归并和OTU划分,并选取每个OTU中丰度最高的序列作为该OTU的代表;最后,利用QIIME软件获取每个OTU所对应的分类学信息。18S rRDA的参考数据库为 Protist Ribosomal Reference (PR2) (Guillou et al, 2013)。
1.5 数据分析
物种分类信息使用NCBI中的taxonomy数据库进行校对。
测定每个取样点的水深(WD),使用校准后的多参数水质检测仪(YSI,美国)分别测量并记录浊度(Turb)、水温(WT)、盐度(Sal)、溶解氧(DO)和pH,使用叶绿素计(HL-168C06,中国)测量叶绿素浓度(Chl-a),使用流速仪(FP111, Global Water,精度为0.1 m/s)测量流速(V)。每个样点另外再采集500 mL水样保存在样品瓶中带回实验室,紫外分光光度法分析测定总氮(TN)和总磷(TP)含量。将每个采样点的水体理化因子求平均得到每个区域的水体理化因子值。
本文采用物种分类关系图表示部分浮游植物的物种组成。利用Chao 1指数(Chao1 index)反映群落丰富度,香农指数(Shannon index, H['])、辛普森指数(Gini-Simpson index, GS)反映群落多样性程度,Pielou均匀度指数(Pielou evenness index, J['])度量群落中相对物种丰富度。基于Bray-Curtis距离矩阵进行非度量多维尺度分析(NMDS),通过相似性分析(ANOSIM)判别样本组间差异。采用降趋对应分析(DCA)完成对模型的选择,对浮游植物群落和环境因子的关系进行RDA分析。利用相关性Heatmap图直观观察各个群落与环境因子之间正负相关性程度。采用物种优势度(Y)表示浮游植物类群某一OTU所占的优势程度。各指数计算公式如下:
式中:ni为第i种OTU的个数,N为所有OTU总个数,fi为第i种OTU在各采样点出现的频率。本文将优势度Y ≥ 0.02的OTU确定为优势种(白海锋等,2021)。
以上数据在R语言、软件SPSS 26.0.0.0、软件Canoco 5 (赖江山, 2013)、图图云平台(https://www.cloudtutu.com)、生科云平台(https://www.bioincloud.tech)以及基因云平台完成(https://www.genescloud.cn)完成。
2 结果
2.1 水环境因子
鄱阳湖水体理化因子的空间分布见表1。3个区域的水温、溶解氧、总氮和总磷差异不明显。水温在20.17~22.12℃,溶解氧在7.26~8.66 mg/L,总氮在1.46~1.90 mg/L,总磷在0.12~0.16 mg/L。各区域的水深、浊度、盐度、流速、pH和叶绿素a的差异较大,北部平均水深达到了9.17 m,流速达到了0.45 m/s,pH、总氮、总磷也比其他2个区域更高,中部的平均浊度最大(31.95),南部最小(8.82)。叶绿素a含量中部最高(12.89 [μ]g/L)。中部和南部的水体pH呈弱碱性,北部水体呈弱酸性。
2.2 藻类物种组成及空间分布
根据区域将环境DNA宏条形码测序得到的OTU进行注释并分类,共检测到2 135个OTU,隶属于10门24纲54目101科166属188种(种未显示) (表2)。其中,硅藻门(345个OTU)和绿藻门(679个OTU)的种类最丰富,而定鞭藻门(8个OTU)未注释到属,褐藻门(12个OTU)仅注释到科。因此物种分类关系图只显示这4个门,且因未注释到属的OTU相对较多,均以未注释到显示。硅藻门中的圆筛藻纲相对丰度最高,绿藻门中共球藻纲溪菜目的溪菜科相对丰度最高,同时也存在一些OTU未能准确鉴定到目或其他分类单元(图2)。从OTU的分布区域来看,南部的OTU数目比中部和北部的多。硅藻门的OTU主要分布在北部,绿藻门的OTU主要分布在南部,甲藻门、裸藻门的OTU主要分布在中部,金藻门、隐藻门的OTU则主要分布在南部。
在鄱阳湖检测到的浮游植物群落组成序列相对丰度见图3。北部硅藻门的相对丰度最高,其次是绿藻门,均为优势类群;与北部不同的是,中部和南部区域绿藻门的相对丰对最高,其次是隐藻门,也均为优势类群。在整个调查区域内,褐藻门和定鞭藻门的相对丰度均较低。
2.3 优势OTU的确定及其空间分布
在2 135个OTU中,共有6个被确定为优势OTU (表3)。其中,隐藻门2个,绿藻门2个,硅藻门1个,裸藻门1个。中部和南部OTU2的优势度最大(0.05和0.11),属于隐藻门隐藻纲隐藻目,在南部和中部占绝对优势;北部OTU8的优势度最大(0.05),属于硅藻门圆筛藻纲海链藻目。OTU4的优势度为0.08,对应绿藻门共球藻纲溪菜目,但仅在南部出现;OTU48的优势度为0.02,对应裸藻门裸藻纲裸藻目,是北部特有的优势OTU,也是裸藻门唯一的优势OTU。
2.4 多样性指数
通过计算香农指数(H['])、辛普森指数(GS)、Pielou均匀度(J['])和Chao 1指数,对鄱阳湖浮游植物的群落进行多样性分析,并绘制多样性指数差异检验箱线图(图4)。在3个分组的样本间,H[']、GS和J[']的变化趋势大致相似。H[']均值为3.18(1.27~4.01),GS均值为0.87(0.59~0.96),J[']均值为0.50,最大值是0.62;中部的H[']、GS和J[']均较南部和北部的高,说明中部的浮游植物群落的多样性和均匀度较高。Chao 1指数的平均值为726.31(385.22~1129.51),其值较大,说明鄱阳湖浮游植物群落丰富度较高。但3组样本的各多样性指数之间没有显著差异性(P>0.05)。
2.5 NMDS分析
对北部、中部和南部3组样本的群落结构组成进行了NMDS分析。Stress值为0.0922,表明结果较好;R值大于0,说明各组之间的距离大于组内距离;整体P值小于0.01,说明分组样本间的浮游植物群落结构组成具有极显著性差异(图5)。南部和北部的部分样点距离较近,但大部分样点之间的距离较远,因此南、北部之间的群落结构组成具有一定的差异;进一步的ANOSIM分析表明,南部与中部之间的群落结构组成无显著差异(R=0.263, P=0.06);中部与北部的群落结构组成具有极显著差异(R=0.343, P=0.004);南部与北部的浮游植物群落结构组成具有显著差异(R=0.444, P=0.011)。
2.6 环境因子关联性
基于各门的序列数进行DCA分析,Lengths of gradient的最大值小于3,因此选择RDA分析环境因子的关联性。各环境因子在排序结果中的相关性指数(r)以及显著性检验值(P)见表4。
由表4可见,环境因子中Chl-a和pH极显著影响排序结果(P<0.01)。流速(V)显著影响排序结果(P<0.05)。因此,在进行RDA分析前,去除对浮游植物群落影响不显著的环境因子,仅使用显著影响因子进行RDA分析。RDA分析中(图6),Permutation test的P<0.01,即环境因子对浮游植物群落结构影响显著,可以看出北部、中部和南部3组样本间表现出了较高的离散性,北部区域样本之间的离散性较南部和中部的低。大部分北部样本与流速之间的射线夹角为锐角,说明北部区域的大部分样本与流速为正相关关系;南部区域的大部分样本与pH为正相关关系。从RDA图6还可以看出,Chl-a与pH也为正相关关系,而Chl-a和pH与V为负相关关系。
浮游植物各门与环境因子之间的相关性Heatmap分析见图7。可以看出,黄藻门、褐藻门和红藻门与Chl-a为极显著正相关关系(P<0.01),硅藻门和裸藻门与pH为极显著负相关关系(P<0.01),硅藻门与流速为极显著正相关关系(P<0.01)。
3 讨论
3.1 基于环境DNA的鄱阳湖浮游植物多样性
本次调查表明,基于环境DNA宏条形技术的鄱阳湖区域春季浮游植物共有10门24纲54目101科166属188种,Jia等(2020)利用传统方法检测到8门77属133种。比较发现,利用环境DNA技术可以检测到更多的物种。本研究中,硅藻门和绿藻门在群落组成中占优势,这与以往利用传统方法监测鄱阳湖浮游植物的情况一致(Liu et al, 2015;Cao et al,2016;Liu et al, 2020)。
物种多样性被用来评价群落中种类组成的稳定程度及其数量分布均匀程度和群落组织结构特征,也是衡量一定区域内浮游植物资源丰富程度的客观指标之一(陈家长等, 2009);根据香农多样性指数大小的等级划分见表5(孟顺龙等,2016)。本研究中,北部湖区多样性丰富(2.6~3.5)的样点有5个,占北部湖区所有样点的83.3%,说明北部湖区浮游植物的多样性处于Ⅳ级,多样性丰富;中部湖区多样性非常丰富(>3.5)的样点有4个,占中部湖区所有样点的66.7%,说明中部湖区浮游植物的多样性处于Ⅴ级,多样性非常丰富;南部区域多样性丰富(2.6~3.5)的点只有2个,而多样性较好(1.6~2.5)的点有2个,均占调查样点的33.3%,说明南部区域浮游植物的多样性处于Ⅲ~Ⅳ级,多样性较好或丰富。由各区域多样性等级可以看出,北部和南部区域的多样性比中部丰富,推测是南部区域(青岚湖和军山湖)人工养殖大量经济鱼类,并且以浮游植物为饵料的鱼类较多,对浮游植物的消耗较大所致;而中部区域水动力学较为平稳,较为适合浮游植物的繁殖和生长。
3.2 环境因子对浮游植物群落结构的影响
通过环境DNA宏条形码技术对鄱阳湖真核浮游植物的研究发现,硅藻门、绿藻门和隐藻门的相对丰度均较高,OTU数目也较多。硅藻对水环境变化极为敏感,水温、流速等都会引起硅藻及其含量的变化(黄学辉等, 2018),硅藻门的相对丰度较高,硅藻门OTU数目也较多(图3,表2),本研究结果与黄兰贵等(2021)的结果一致。这是由于硅藻适宜生长温度在10~25℃,春季温度适宜硅藻生长(Admiraal, 1976)。硅藻门主要与流速呈极显著正相关关系(图7)。北部区域是鄱阳湖的通江口,流速较大(表1),对硅藻生长较为有利,这是硅藻门在北部相对丰度最高的原因;此外,常温条件下一些单细胞绿藻生长速度也较快,导致绿藻门的相对丰度较高(彭宁彦等, 2018);隐藻纲是隐藻门仅有的1纲,其分布广泛,常在水体中形成优势类群(胡鸿钧和魏印心, 2006)。
相关性热图中并未出现绿藻门和隐藻门(图7),这是因为绿藻门与水温关系为正相关(夏爽等, 2013),隐藻门的繁殖与生长主要受磷、铁和光照影响(黄学辉等, 2018);而在RDA分析中,水温和总磷与鄱阳湖整体浮游植物之间的关系并不显著。营养盐的富集有利于藻类的繁殖与生长(戴星照, 2016),但本研究中的营养盐对鄱阳湖整体浮游植物之间的关系并不显著,可能是春季鄱阳湖水位上涨稀释了营养盐浓度(吴召仕等, 2014)。
此外,本研究利用环境DNA宏条形技术检测到了红藻门在鄱阳湖的分布(表2),而在以往传统的研究中均未出现红藻门,这可能是红藻个体较大,利用传统方法定性采样时较难涉及所致(张军毅等,2021);同时,在本研究还检测到褐藻门,但其OTU较少且仅能注释到科水平,一方面可能是褐藻在淡水中种类极少(胡鸿钧和魏印心, 2006);另一方面也可能是数据库缺少序列的相关信息所致。
3.3 环境DNA技术用于浮游植物监测的优缺点
与传统调查方法相比,环境DNA宏条形技术可以检测到更多的物种,对传统调查方法得到的物种名录是一个补充,也可以用于评估浮游植物的生物多样性,还能减少对分类学专家的依赖(Bombin et al, 2020)。本研究也发现环境DNA宏条形技术存在有些OTU不能鉴定到种水平的问题,这是由于参考数据库中浮游植物序列信息不全所致。研究表明,数据库对环境DNA技术注释物种这一环节尤为重要,目前数据库都在不断更新和补充缺失物种。因此,在物种注释时更应选择对应关系较强、更新较频繁的数据库。
志谢:感谢南昌大学孙威威、王维开和刘雄军博士在样品采集方面提供的帮助。
参考文献
白海锋,王怡睿,宋进喜,等,2021. 渭河陕西段浮游植物群落结构时空变化与影响因子分析[J]. 环境科学学报, 41(8):3290-3301.
陈家长,孟顺龙,尤洋,等,2009. 太湖五里湖浮游植物群落结构特征分析[J]. 生态环境学报, 18(4):1358-1367.
陈康,孟子豪,李学梅,等,2022. 鄱阳湖流域柘林水库秋季浮游植物群落结构及其构建过程驱动机制[J]. 湖泊科学, 34(2):433-444.
戴星照,2016. 鄱阳湖科学考察[M]. 上海:上海科学技术出版社.
胡鸿钧,魏印心,2006. 中国淡水藻类[M]. 北京:科学出版社.
黄兰贵,殷环环,张航,等,2021. 鄱阳湖出口浮游植物群落与环境因子分析[J]. 水利水电快报, 42(8):77-82.
黄学辉,陈传红,袁轶君,等,2018. 鄱阳湖浮游植物群落季节性变化分析[J]. 江西化工, (2):180-184.
赖江山,2013. 生态学多元数据排序分析软件Canoco5介绍[J]. 生物多样性, 21(6):765-768.
孟顺龙,肖代,陈小丽,等,2016. 丰、枯水期里下河腹地典型水体浮游动物群落结构与水质评价[J]. 水生态学杂志, 37(6):76-83.
彭宁彦,戴国飞,张伟,等,2018. 鄱阳湖不同湖区营养盐状态及藻类种群对比[J]. 湖泊科学, 30(5):1295-1308.
吴召仕,张路,刘宝贵,等,2014. 鄱阳湖丰水期水体中叶绿素a含量空间分布及其与环境因子的关系[J]. 湿地科学, 12(3):286-292.
夏爽,张琪,刘国祥,等,2013. 人工试验湖泊浮游藻类群落的生态学研究[J]. 水生生物学报, 37(4):640-647.
张军毅,孙蓓丽,朱冰川,等,2021. 基于分子标记的藻类鉴定研究进展[J]. 湖泊科学, 33(6):1-20.
Admiraal W, 1976. Influence of light and temperature on the growth rate of estuarine benthic diatoms in culture[J]. Marine Biology, 39(1):1-9.
Amral-Zettler L A, Mccliment E A, Ducklow H W, et al, 2009. A method for studying protistan diversity using massively parallel sequencing of V9 hypervariable regions of small-subunit ribosomal RNA genes[J]. PloS One, 4(12):e6372.
Bohmann K, Evans A, Gilbert M T, et al, 2014. Environmental DNA for wildlife biology and biodiversity monitoring[J]. Trends in Ecology & Evolution, 29(6):358-367.
Bombin S, Wysor B, Lopez-Bautista J M, 2020. Assessment of littoral algal diversity from the northern Gulf of Mexico using environmental DNA metabarcoding[J]. Journal of Phycology, 57(1):269-278.
Bucklin A, Lindeque P K, Rodriguez-Ezpeleta N, et al, 2016. Metabarcoding of marine zooplankton: prospects, progress and pitfalls[J]. Journal of Plankton Research, 38(3):393-400.
Bush A, Compson Z G, Monk W A, et al, 2019. Studying Ecosystems With DNA Metabarcoding: Lessons From Biomonitoring of Aquatic Macroinvertebrates[J]. Frontiers in Ecology and Evolution,DOI: 10.3389/fevo.2019.00434
Cao J, Chu Z S, Du Y, et al, 2016. Phytoplankton dynamics and their relationship with environmental variables of Lake Poyang[J]. Hydrology Research, 47(S1):249-260.
Chen Y W, Qin B Q, Teubner K, et al, 2003. Long-term dynamics of phytoplankton assemblages: microcystis-domination in Lake Taihu, a large shallow lake in China[J]. Journal of Plankton Research, 25(4):445-453.
Eiler A, Drakare S, Bertilsson S, et al, 2013. Unveiling distribution patterns of freshwater phytoplankton by a next generation sequencing based approach[J]. PLoS One, 8(1):e53516.
Guillou L, Bachar D, Audic S, et al, 2013. The protist ribosomal reference database (PR2): a catalog of unicellular eukaryote small sub-unit rRNA sequences with curated taxonomy[J]. Nucleic Acids Research, 41(D1):597-604.
Jia J J, Gao Y, Zhou F, et al, 2020. Identifying the main drivers of change of phytoplankton community structure and gross primary productivity in a river-lake system[J]. Journal of Hydrology, 583(C):124633.
Liu X, Qian K M, Chen Y W, 2015. Effects of water level fluctuations on phytoplankton in a Changjiang River floodplain lake (Poyang Lake): Implications for dam operations[J]. Journal of Great Lakes Research, 41(3):770-779.
Li K, Liu X J, Zhou Y, et al, 2019. Temporal and spatial changes in macrozoobenthos diversity in Poyang Lake Basin, China[J]. Ecology and Evolution, 9(11):1-13.
Liu X J, Lu Q F, Zhou Y, et al, 2020. Community characteristics of phytoplankton and management implications in Poyang Lake Basin[J]. Limnology, 21(2):207-218.
Reynolds C S, 1984. Phytoplankton periodicity-the interactions of form, function and environmental variability[J]. Freshwater Biology, 14(2):111-142.
Reynolds C S, Padisák J, Sommer U, 1993. Intermediate disturbance in the ecology of phytoplankton and the maintenance of species diversity: a synthesis[J]. Hydrobiologia, 249(1/3):183-188.
Rubin M A, Leff L G, 2007. Nutrients and other abiotic factors affecting bacterial communities in an Ohio River (USA)[J]. Microbial Ecology, 54(2):374-383.
Soares M C S, Lobo L M, Vidal L O, et al, 2011. Light microscopy in aquatic ecology: methods for plankton communities studies[J]. Methods in Molecular Biology, 689:215-227.
Thomsen P F, Willerslev E, 2015. Environmental DNA—an emerging tool in conservation for monitoring past and present biodiversity[J]. Biological Conservation, 183(1):4-18.
Visco A J, Apothoz-Perret-Gentil L, Cordonier A, et al, 2015. Environmental monitoring: inferring diatom index from next-generation sequencing data[J]. Environmental Science & Technology, 49(13):7597-7605.
Wu N C, Schmalz B, Fohrer N, 2011. Distribution of phytoplankton in a German lowland river in relation to environmental factors[J]. Journal of Plankton Research, 33(5):807-820.
Wu Z S, Cai Y J, Liu X, et al, 2013. Temporal and spatial variability of phytoplankton in Lake Poyang: The largest freshwater lake in China[J]. Journal of Great Lakes Research, 39(3):476-483.
Xiao X, Sogge H, Lagesen K, et al, 2014. Use of high throughput sequencing and light microscopy show contrasting results in a study of phytoplankton occurrence in a freshwater environment[J]. PLoS One, 9(8):e106510.
Yang J H, Zhang X W, 2019. eDNA metabarcoding in zooplankton improves the ecological status assessment of aquatic ecosystems[J]. Environment International, 134:105230.
(责任编辑 万月华)
Diversity of Eukaryotic Phytoplankton in Poyang Lake based
on Environmental DNA Metabarcoding
GUO Ting1, FU Zhi‐hao1, ZHOU Chun‐hua1, CHEN Jin‐ping1, OUYANG Shan1,2, WU Xiao‐ping1,2
(1. College of Life Science, Nanchang University, Nanchang 330031, P.R. China;
2. Ministry of Education, Key Laboratory of Environment and Resource Utilization of Poyang Lake,
Nanchang University, Nanchang 330031, P.R. China)
Abstract:In this study, the new technology of environmental DNA (eDNA) metabarcoding was used to explore the phytoplankton community in Poyang Lake, focusing on species composition, spatial distribution, and community diversity. We aimed to provide basic data for the application and standardization of eDNA for monitoring aquatic ecosystems. Poyang Lake was divided into three regions and a total of 18 sampling sites were selected, including the channel connecting north Poyang Lake to the Yangtze River (Site 1-6), central Poyang Lake (Site 7-12) and the aquaculture area of south Poyang Lake (Site 13-18). Water samples were collected in triplicate at each sampling site during April of 2019. The V9 region of the 18S rDNA gene in eukaryotic phytoplankton was amplified and high-throughput sequencing and bioinformatics were used to analyze the community composition of phytoplankton in Poyang Lake. A total of 2 135 Operational Taxonomic Units (OTUs) of phytoplankton were obtained, consisting of 188 phytoplankton species from 166 genera, 101 families, 54 orders, 24 classes and 10 phyla. Chlorophyta and Bacillariophyta had high species richness. The phytoplankton community richness in Poyang Lake was generally high, and community diversity and evenness were higher in the central area of Poyang Lake. Non-metric multidimensional scale (NMDS) analysis shows that there were significant differences in the phytoplankton community structure between central and northern Poyang Lake (P=0.004), and between the southern and northern areas (P=0.011). Redundancy analysis (RDA) shows that chlorophyll-a, pH, and flow velocity greatly effected on the phytoplankton community in all areas of the lake. In conclusion, eDNA metabarcoding was used to rapidly assess phytoplankton biodiversity and spatial distribution in Poyang Lake and it provides a new and effective tool for monitoring the biodiversity and ecosystem health of Poyang Lake.
Key words:environmental DNA metabarcoding; phytoplankton; diversity; community structure; Poyang Lake
收稿日期:2021-12-11 修回日期:2023-04-11
基金项目:江西省大学生创新创业训练计划重点项目(202210403062);国家重点研发计划“蓝色粮仓”重点专项(2018YFD0900801)。
作者简介:郭婷,1995年,女,硕士研究生,研究方向为生物多样性。E-mail: Guotingsx@163.com
通信作者:周春花,1979年生,女,博士,副教授,主要从事生物多样性研究。E-mail: zhouchunhuajx@hotmail.com

