基于黄褐棉导入系群体定位抗黄萎病QTL
常鑫燚,李轩照,唐秉晖,潘振远,吴元龙,沈超,努日曼古丽·艾尼,林忠旭,4,尤春源,4*,聂新辉*
(1.石河子大学农学院/ 新疆生产建设兵团绿洲生态农业重点实验室,新疆 石河子 832003;2.石河子农业科学院棉花研究所,新疆 石河子 832011;3.广东石油化工学院生物与食品工程学院,广东 茂名 525000;4.华中农业大学植物科学技术学院/ 作物遗传改良国家重点实验室,武汉 430070)
棉花作为我国重要的经济作物及战略物资来源,在国民经济中具有举足轻重的地位。黄萎病(Verticillium wilt)被称为棉花的“癌症”,是直接影响棉花产量和品质的重要因素,对棉花生长发育造成不可逆的伤害。棉花黄萎病属于土传真菌病害,黑白轮枝菌(V.albo-atrum)和大丽轮枝菌(V.dahilae)是其致病菌[1-2],主要危害棉花维管束及叶片等组织(器官),致使棉花叶片黄化、萎蔫、脱落及植株死亡[3],严重阻碍棉花营养物质运输及光合作用的进行。在棉花生产中,其防治非常困难。因此,加强对棉花黄萎病抗性调控机理的研究[4]和选育抗病新品种[5]是防治棉花黄萎病最有效的措施。数年来,众多科研工作者对棉花抗黄萎病调控机理进行研究[6-7],发掘与黄萎病抗性相关的数量性状位点(quantitative trait loci,QTL)[8-9],并通过遗传转化及基因功能分析[10-11]等方法进行验证。
陆地棉(Gossypium hirsutum)是全球种植面积最大的棉花栽培种,其产量占世界棉花总产量95%以上[12]。但其遗传背景较为狭窄,通过种内杂交改良性状非常困难[13]。黄褐棉(G.mustelinum)原产于巴西,与陆地棉遗传背景差异大,具有丰富的抗病基因[14],其野生棉种质资源对提高陆地棉纤维品质、拓宽遗传背景具有重大价值[15-16]。
近年来,随着高通量测序技术不断发展完善,被广泛应用于大规模群体基因型鉴定和QTL 定位,大大提高了发掘调控优良性状基因或标记的效率。利用特异性位点扩增片段测序技术(specific-locus amplified fragment sequencing,SLAF-seq),能够在全基因组范围内鉴定和开发特异性的单核苷酸多态性(single nucleotide polymorphism,SNP)标记[17]。SNP 标记已被广泛应用于亲缘关系及遗传变异分析、全基因组关联分析和构建遗传图谱,具有高效、精确等特点[18-19]。
国内外许多研究报道是关于利用分子标记技术和不同群体检测到大量与棉花黄萎病抗性相关的QTL。赵君等[20]以苏VR018 为母本、泗棉3 号为父本构建F2群体,并检测到4 个与棉花黄萎病抗性相关的QTL,分别位于A05、D05、D05和D06 染色体上。邓琳等[21]构建了以陆地棉PD94042 为轮回亲本、黄褐棉为供体亲本的导入系群体,检测到10 个与黄萎病抗性相关的QTL,解释14.0%~39.3%的表型变异。Rashid[22]利用300 份有中棉所36 背景的陆海渐渗系材料进行QTL 定位,检测到60 个与黄萎病抗性相关的QTL,解释3.8%~11.9%的表型变异。鲁宁宁[23]以抗、感黄萎病棉花品种构建的重组自交系(recombinant inbred line,RIL)和F2群体为研究材料,在RIL 群体中检测到8 个与黄萎病抗性相关的QTL,在F2群体检测到6 个QTL 与黄萎病抗性相关。李廷刚等[24-25]以299 份陆地棉材料构建关联群体,检测到17 个与黄萎病抗性相关的SNP 位点,且A10 染色体上的SNP(A10_99672586)位点在3 个环境内均被检测到。杨芮[26]利用海陆渐渗系BMI9626 和中棉所36 构建F2、F2:3、F2:4群体,检测到10 个与黄萎病抗性相关的QTL,可解释3.32%~10.00%的表型变异,其中7 个QTL 在多个环境下均被检测到。
黄褐棉导入系群体 (以下简称为BC5S5群体)的轮回亲本陆地棉B0011(以下简称为B0011)有产量高、综合性状优良等特性[27],是华杂棉H318 的亲本之一[28]。供体亲本黄褐棉具有纤维品质优良、抗黄萎病等特性[14]。因此,本研究在多年多点环境下鉴定BC5S5群体的黄萎病抗性,并结合基因型数据进行QTL 定位,以期挖掘到与黄萎病抗性相关的稳定QTL,为抗黄萎病分子标记辅助选择育种提供参考。


以黄褐棉为供体亲本,与轮回亲本B0011 杂交产生F1,并与B0011 连续回交5 次,回交过程选取具有代表性株系与轮回亲本杂交,并进行标记鉴定,获得71 个BC5株系。然后通过单子传法将71 个株系连续自交5 代,最后获得有71 个株系的黄褐棉导入系群体(BC5S5群体)并作为试验材料[16]。回交和自交过程利用分子标记法进行鉴定,以获得遗传稳定的导入系材料。

以71 个株系和亲本 (陆地棉B0011、黄褐棉)幼嫩叶片为材料,利用液氮速冻,在-70 ℃条件下贮藏。随后使用Plant Genomic DNA Kit(天根生化,北京,中国)提取基因组DNA,利用Nano-Drop 2000C 超微量分光光度计(赛默飞,美国)检测提取基因组DNA 的浓度和纯度,OD260/OD280在1.8~2.0 视为符合质量要求,用1%(质量分数)琼脂糖凝胶进行电泳进一步检测提取基因组DNA 的纯度和完整性,满足建库要求后,送至百迈客生物科技有限公司用Illumina HiSeq-2500平台(因美纳,美国)进行高通量测序、标签分析和变异鉴定[29-30]。亲本测序深度为20 倍,子代测序深度为5 倍。

2018―2019 年分别在玛纳斯农业试验站病圃地(2018 年记为E1、2019 年记为E3)和库尔勒试验站病圃地(2018 年记为E2、2019 年记为E4)种植BC5S5群体和亲本B0011。采用随机区组设计,2 次重复,每小区种植2 行,行长5 m;种植模式:一膜四行,行距(28+50+28)cm+55 cm,株距9.5 cm。以新陆早36 为感病对照,以中植棉2号为抗病对照。
当感病对照病情指数 (disease index,DI)高于35 时开始进行抗病性鉴定。玛纳斯试验点分别于2018 年6 月28 日和2019 年7 月3 日、库尔勒试验点分别于2018 年7 月5 日和2019 年7月3 日进行抗病性鉴定,每个环境每隔10 d 进行1 次抗病性鉴定,共鉴定3 次。参照国家标准GB/T 22101.5-2009 《棉花抗病虫性评价技术规范 第5 部分:黄萎病》[31]的五级法进行棉花黄萎病抗性鉴定,并计算病情指数和相对病情指数(relative disease index,RDI)。

1.4.1表型分析。对4 个环境下的表型数据进行整理,用SPSS 24.0 进行描述统计分析(如均值、标准偏差、变异系数、峰度及偏度等)和方差分析(单因素一般线性模型)[32]。利用R 语言lme4 包(https://cran.r-project.org/web/packages/lme4/index.html)进行最佳线性无偏预测(best linear unbiased prediction,BLUP)评估及遗传力计算,用R语 言corrplot 包(https://cran.r-project.org/web/packages/corrplot/index.html)对BC5S5群体在4个环境下的RDI 均值进行皮尔逊(Pearson)相关分析。其中,方差分析、BLUP 值评估和相关分析选用3 次调查中变异系数最大的1 次抗病性鉴定结果。
1.4.2QTL 定位。本研究选用4 个环境中RDI变异系数最大的1 次抗病性鉴定结果作为该环境的表型值进行QTL 定位。QTL 定位参照沈超等[16]和Wang 等[33]的定位方法。利用2 839 个SNP标记[16],结合QTL IciMapping 4.1(https://isbreeding.caas.cn/)软件[33]CSL 功能基于逐步回归加性效应模型(RSTER-LRT- 加性效应值)检测与黄萎病抗性相关QTL,设对数优势比(logarithm of the odd score,LOD)阈值≥2.5。根据软件数据格式要求,将与受体亲本B0011 相同的基因型记为“0”,与供体亲本黄褐棉相同的基因型记为“2”。参照McCouch 等的规则命名QTL[34],根据BLUP 值检测的QTL 后面加上“B”作区分,在2个或2 个以上环境同时被检测到的QTL 被认为是稳定QTL[35],并利用R 语言LinkageMapView包(https://cran.r-project.org/web/packages/Linkage-MapView/)绘制与黄萎病抗性相关QTL 的染色体分布图。
在CottonQTLdb 数 据 库(http://www.cottonqtldb.org)中检索并下载抗黄萎病相关的QTL[36],并利用QTL 标记在Cottongen 数据库(https://www.cottongen.org/find/markers)中确定相应QTL的物理位置[37]。物理位置相同或物理位置包含本研究检测到的QTL 视为对已鉴定QTL 的重复鉴定,反之,则视为新检测到的QTL。


对4 个环境下BC5S5群体RDI 进行描述统计分析,结果见表1。BC5S5群体平均RDI 为13.62~43.66,变异系数为21.45%~67.93%,遗传力为83.89%~95.17%。其中,E1 的发病相对较轻,RDI 为1.04~60.61,平均为13.62~27.92;E3的发病最重,RDI 为14.94~85.77,平均为39.49~43.66(表1 和附图1)。其中,E1_1、E2_1、E3_1 和E4_1 是相应环境中离散程度较高、变异系数最大的调查结果。基于这4 次调查数据的BLUP值的频率分布,发现BC5S5群体RDI 呈现相对连续的正态分布,且分离较大,峰度和偏度满足正态分布要求,符合数量性状分布特性(图1)。因此,本研究后续的方差分析、相关分析及QTL 分析均基于E1_1、E2_1、E3_1 和E4_1。
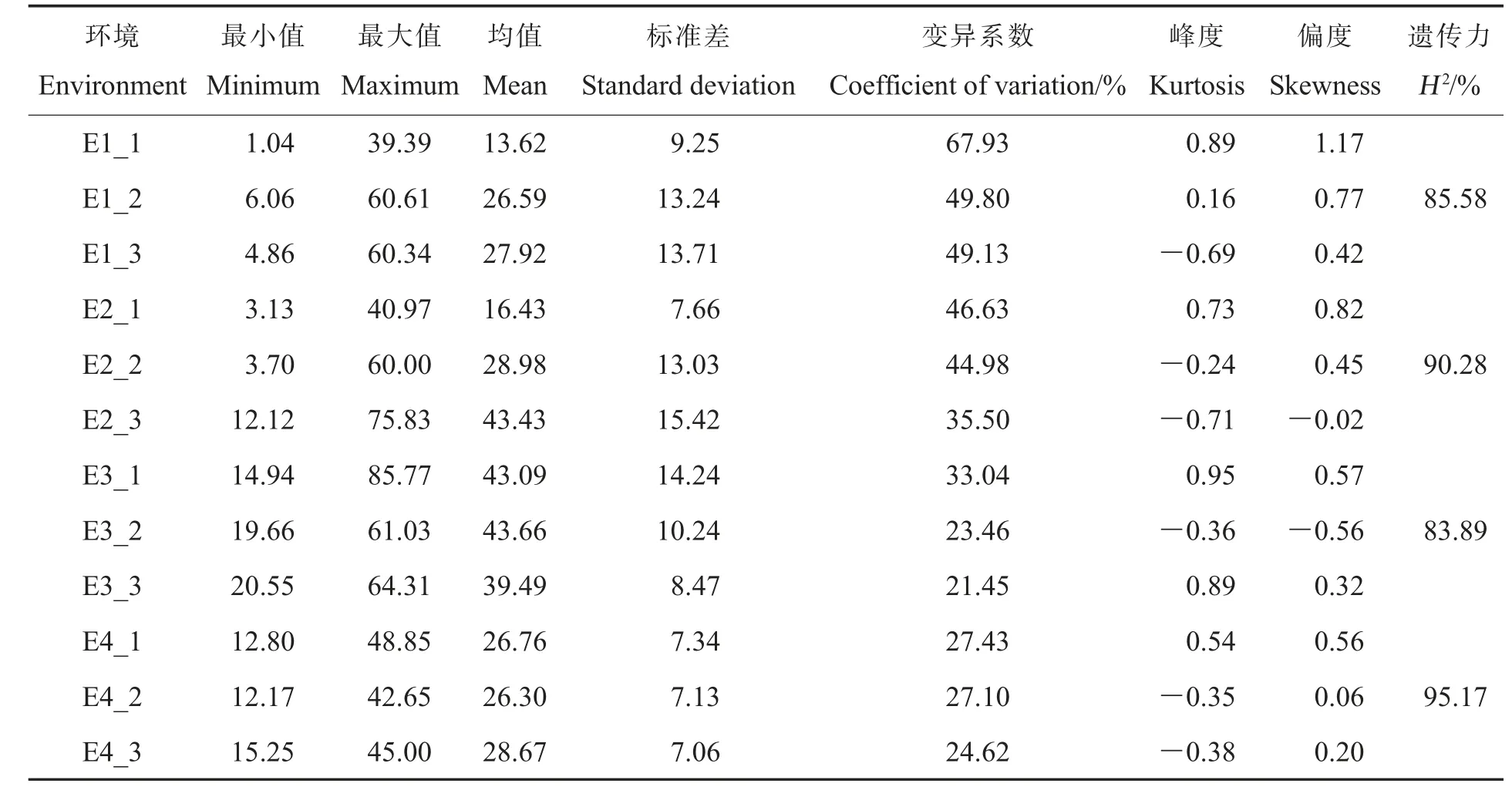
表1 BC5S5 群体相对病情指数描述统计Table 1 Relative disease index description statistics of the BC5S5 population
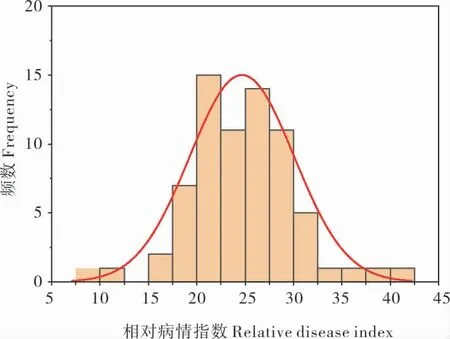
图1 BC5S5 群体相对病情指数在多环境中BLUP的频率分布Fig.1 BLUP frequency distribution of relative disease index of BC5S5 population in multi-environments
为了解棉花黄萎病的发生是否受到基因型和环境的影响,对2.1 选择的E1_1、E2_1、E3_1和E4_1 的RDI 进行方差分析,结果(表2)表明基因型、环境以及基因型与环境的互作对RDI 的影响均在0.001 水平极显著。表明棉花黄萎病的发生程度不仅受基因型和环境的影响,也易受基因型与环境互作的影响。
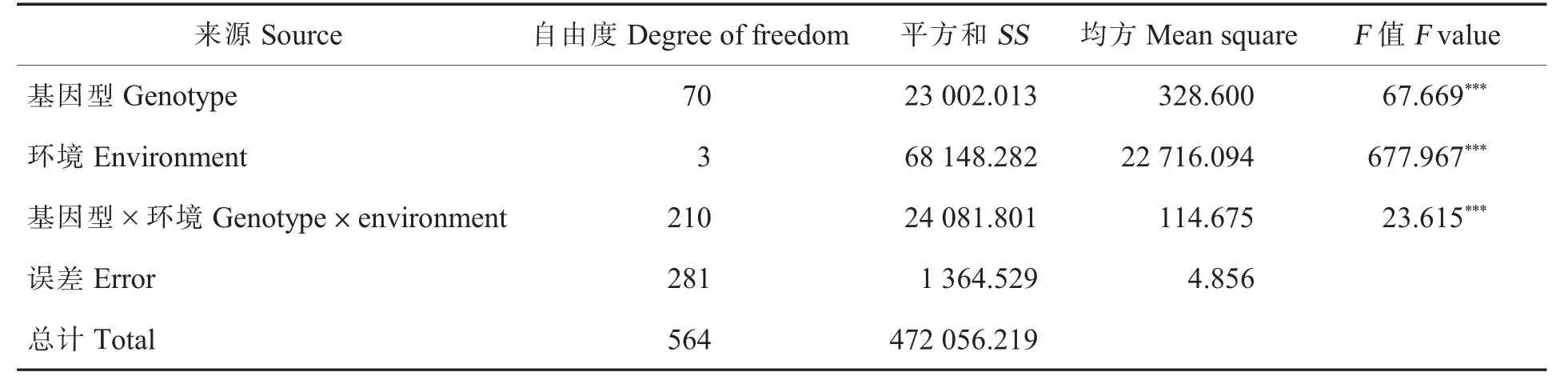
表2 相对病情指数不同环境间的方差分析Table 2 ANOVA analysis of relative disease index in different environments

对在相应环境内变异系数最大的4 个环境进行BC5S5群体RDI 相关分析,结果(表3)表明:4 个环境间RDI 均存在显著或极显著相关关系,相关系数为0.29~0.65,其中,E1 与E3 的相关系数为0.65,存在较强的相关性;E2 与E3 的相关系数为0.29,相关性较弱。表明不同环境间RDI具有较大差异。
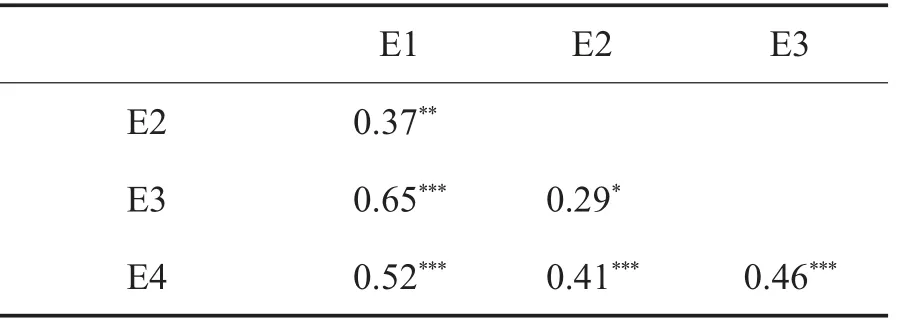
表3 不同环境下BC5S5 群体相对病情指数相关分析Table 3 Correlation analysis of relative disease index of BC5S5 population in different environments

2.4.1黄萎病抗性相关QTL。利用QTL IciMapping 4.1 软件,共检测到15 个与黄萎病抗性相关的QTL,表型变异解释率(phenotypic variance explained,PVE)为4.21%~26.77%,LOD 值为2.69~12.44,加性效应值为-13.29~14.59。在At 亚基因组共检测到10 个QTL,其中A02 和A07 染色体上各检测到2 个QTL;在Dt 亚基因组共检测到5 个QTL,分别在D03、D04、D06、D10 和D12染色体上。其中,qVW-A01-1(E1 和E3 环境)、qVW-A02-2(E1、E2 和E3 环境)、qVW-A07-2(E3和E4 环境)在2 个及以上环境中被检测到,PVE分别为15.56%~16.56%、11.95%~24.62%和13.22%~16.73%(表4 和附图2),表明这些QTL比较稳定、可能与棉花黄萎病抗性有较强的连锁性。
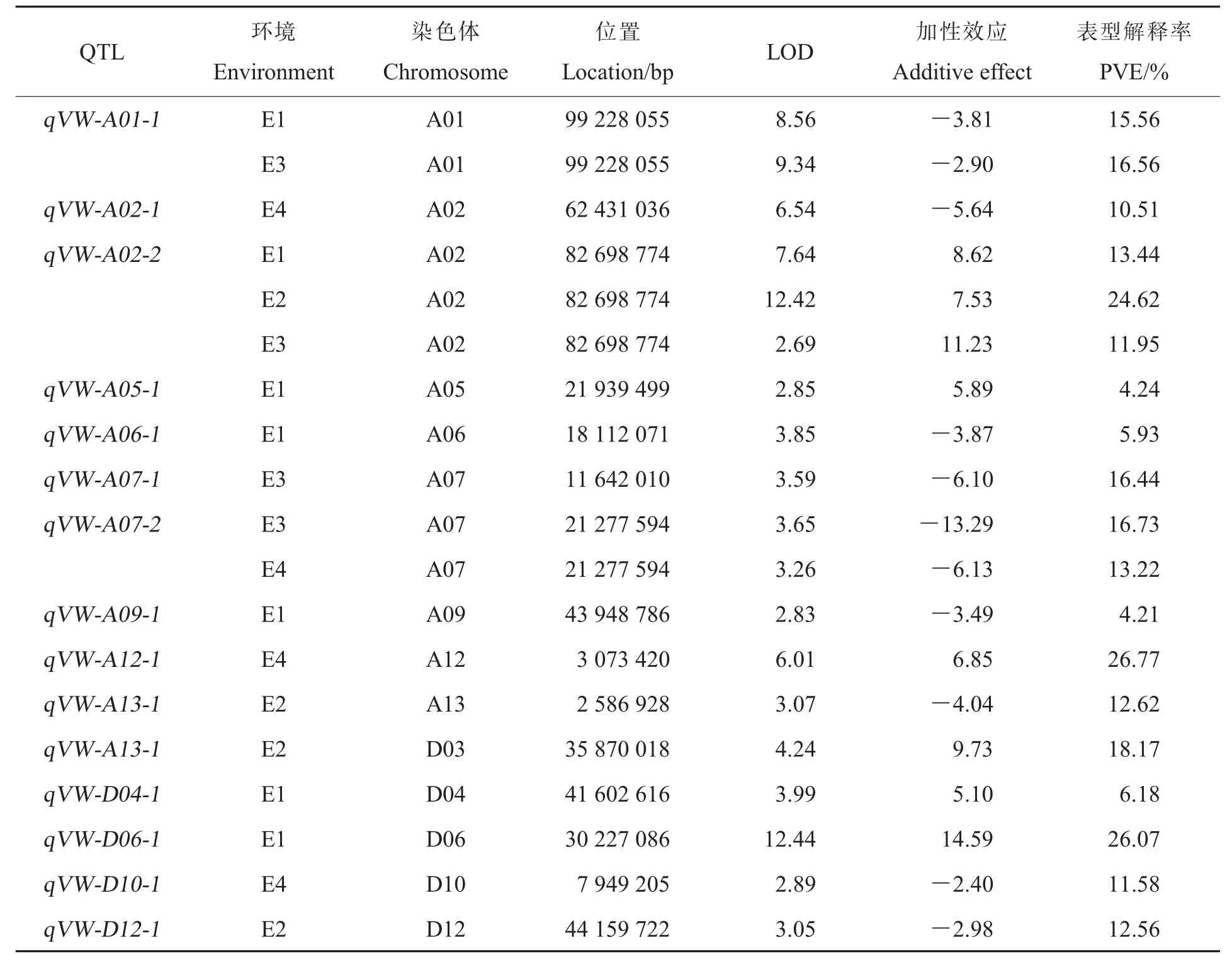
表4 黄萎病抗性相关的QTL 信息Table 4 Information of QTL related to Verticillium wilt resistance
2.4.2基于BLUP 值的黄萎病抗性相关QTL。基于BC5S5群体在4 个环境中RDI 的BLUP 值,共检测到5 个与黄萎病抗性相关的QTL (表5),PVE 为6.10%~23.67%,LOD 值为3.43~10.42,加性效应值为-3.94~5.66,分布在A01、A02、A10、D02 和D06 染色体上。其中qVW-A01-1B、qVW-A02-1B和qVW-D06-1B的PVE 分 别 为23.67%、17.90%和12.66%,分别与前文鉴定的qVW-A01-1、qVW-A02-2和qVW-D06-1的 物 理位置一致(表5 和附图3)。
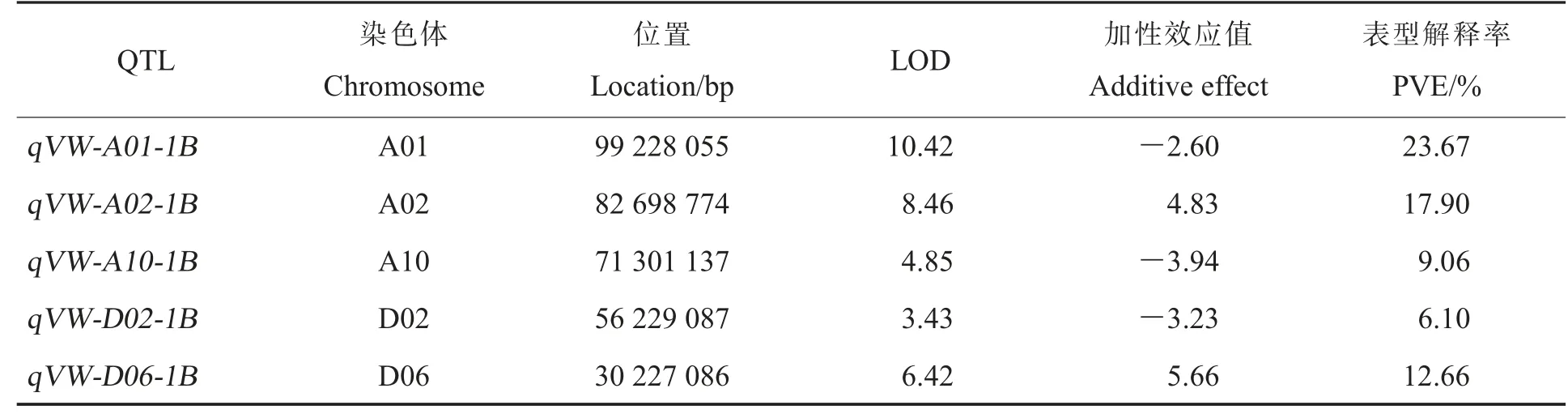
表5 基于BLUP 值鉴定的黄萎病抗性相关的QTLTable 5 QTL related to Verticillium wilt resistance based on BLUP value


作图群体不仅是QTL 分析的基础,更是构建遗传连锁图谱的根本。目前,常用于QTL 定位的群体有F2、F2:3、回交一代(backcross generation,BC1)、加倍单倍体(doubled haploid, DH)和RIL等分离群体[38],但这些群体遗传背景较为复杂,且棉花黄萎病是多基因控制的数量性状,易受环境及基因的共同影响,导致QTL 的精确定位相对困难。然而,导入系群体经过不断与轮回亲本回交,遗传背景仅含有供体亲本1 个或几个染色体片段[39],使导入系群体成为QTL 分析的理想材料。因此,亲本选择及作图群体类型是影响QTL分析的关键因素。黄褐棉具有纤维品质优良及抗病性强等优良特点[14],本研究以黄褐棉为供体亲本构建有71 个株系的BC5S5群体可用于检测棉花黄萎病抗性相关的QTL,为检测稳定QTL 位点、分子标记辅助选择育种及候选基因功能鉴定奠定基础。

我国棉花黄萎病菌的分布及棉花黄萎病抗性调控机理十分复杂。张桂寅等[40-41]利用1 个海岛棉和15 个陆地棉品种为分离寄主对大丽轮枝菌进行分析,共分离出Teyu1、Han7860、Han6208等67 个菌系;致病力鉴定表明从同一田块不同棉花品种上分离的黄萎病菌是由致病力不同的多种菌系构成,不同品种上分离的菌系致病力存在差异;寄主品种的抗性与分离的黄萎病菌系的致病力无关,且不同基因型品种可能会影响不同致病力菌落的消长。本研究将包含71 个株系的BC5S5群体种植于病圃中,并进行多年多点的黄萎病抗性鉴定,方差分析显示,基因型和环境对棉花黄萎病发生程度的影响均达极显著水平,同时基因型与环境互作对棉花黄萎病发生程度的影响也极显著。相关分析显示,4 个环境下群体的RDI 间存在显著或极显著正相关关系,且不同环境间RDI 存在较大差异。因此,棉花黄萎病发生易受基因型及种植环境的影响。

研究表明,并非所有的有利等位基因均来源于高值亲本[42]。孙颖[43]以黄褐棉为供体亲本,中棉所35 为轮回亲本构建有564 个单株的BC3F2群体,连锁分析共检测到121 个QTL 与棉花产量和纤维品质性状相关,其中,46 个QTL 有利等位基因来源于黄褐棉。陈奇等[44]基于黄褐棉导入系进行棉花衣分QTL 定位,共检测到21 个QTL,其主效QTLqLP-3增效基因均来源于黄褐棉。邓林等[21]基于黄褐棉导入系定位棉花抗黄萎病相关的QTL,共检测到10 个QTL,其中4 个QTL 增效基因来源于黄褐棉。
本研究中,在4 个环境下共检测到15 个与棉花黄萎病抗性相关的QTL,加性效应分析结果(表4)显示:有6 个QTL 为正向加性效应,且加性效应为5.10~14.59,表明有利等位基因来源于供体亲本黄褐棉,其中本研究检测到的稳定QTL:qVW-A02-2的加性效应为7.53~11.23;有9 个QTL 有利等位基因来源于B0011,加性效应为-13.29~-2.40,其中qVW-A01-1和qVWA07-2为本研究稳定检测的QTL。利用BLUP 值进行棉花抗黄萎病相关QTL 分析,共检测到5个QTL(表5),其中2 个QTL 的有利等位基因来源于黄褐棉,3 个QTL 的有利等位基因来源于B0011。此结果证实了有利等位基因并非都只来源于高值亲本。
关于棉花抗黄萎病相关QTL 定位已有较多报道,但大多是基于简单重复序列 (simple sequence repeat,SSR)标记进行,不能提供较为精确的物理位置。棉花全基因组测序的完成和高通量分子标记(如SNP)的开发应用,使得棉花黄萎病抗性相关基因定位更精确。
本研究共检测到3 个稳定QTL(qVW-A01-1、qVWA02-2和qVW-A07-2),且qVW-A01-1、qVW-A02-2在黄萎病抗性相关QTL 定位及基于BLUP 值的黄萎病抗性相关QTL 定位中均被检测到。通过与前人鉴定的QTL 进行比较,本研究检测到的部分位点与前人鉴定到的与棉花黄萎病抗性相关QTL 的物理位置相近或一致。Palanga等[45]利用有169 个株系的RIL 群体进行黄萎病抗性相关QTL 定位,共检测到119 个与黄萎病抗性相关的QTL,其中qDInc-C1-1(标记i23690Gh和i25127Gh)和qDInc-C1-2(标记i35470Gh 和i40498Gh)在陆地棉TM-1 参考基因组的物理位置分别为A01 染色体98 274 676~106 996 201 bp和99 181 202~102 158 017 bp,与本研究检测到的来源于黄褐棉的增效基因qVW-A01-1(99 228 055 bp)位置相近;qDInc-C10-3(标记i43465Gh和i44312Gh)物理位置为A10 染色体70 467 989~71 371 332 bp,与本研究检测到的qVW-A10-1(71 301 137 bp)相近;qDI-C12-2(标记:i45732Gh和i49103Gh)物理位置为A12 染色体3 042 213~5 012 230 bp,与本研究检测到的qVW-A12-1(3 073 420 bp)相近。因此,本研究所检测到的黄萎病抗性相关QTL 准确可靠。

本研究对黄褐棉导入系BC5S5群体进行黄萎病抗性鉴定及相关QTL 定位,共检测到15 个QTL,解释4.21%~26.77%的表型变异,其中qVW-A01-1、qVW-A02-2和qVW-A07-2能 在2个及以上环境中被检测到;基于BLUP 值检测到5 个QTL,解释6.10%~23.67%的表型变异,其中3 个是重复鉴定的QTL;其中qVW-A01-1在前人研究中已有相关报道。本研究发现的稳定QTL可为棉花抗黄萎病分子标记辅助选择育种等提供参考。
附图:详见本刊网站https://journal.cricaas.com.cn
附图1 不同环境下BC5S5群体相对病情指数分布
Fig.S1 The relative disease index distribution of BC5S5population in different environments
附图2 与棉花黄萎病抗性相关的QTL
Fig.S2 QTL related to resistance to Verticillium wilt disease in cotton
附图3 与棉花黄萎病抗性相关的QTL(BLUP 值)
Fig.S3 QTL related to resistance to Verticillium wilt disease in cotton(BLUP value)

