基于eDNA 技术的珠海外伶仃海洋牧场细菌多样性研究
臧能玮,朱爱意,陈丕茂,袁华荣,张红会,魏文迪,3
(1.浙江海洋大学国家海洋设施养殖工程技术研究中心,浙江 舟山 316022;2.中国水产科学院南海水产科学研究所,广东 广州 510300;3.上海海洋大学海洋科学学院,上海 201306)
【研究意义】海洋微生物是海洋初级生产力的代表,约占全球海洋初级生产力的一半[1],是海洋生态系统中生物要素的重要组成,在物质循环和能量流动中发挥着重要作用。海洋微生物个体或群落变化可以客观地反映水体变化[2],在生态监测实践中广泛应用[3]。海洋微生物还在环境污染的生物修复、全球气候变化调节等方面具有举足轻重的作用。研究珠海外伶仃海洋牧场微生物多样性,对于加强海洋牧场管理和维护具有重要意义。【前人研究进展】环境DNA(environmental DNA,eDNA)是采用非直接接触的方式提取环境中有机体遗留的DNA,即直接从环境样品 (土壤、沉积物、水等)获得遗传物质,是一种有效、无创且易于标准化的取样方法[4]。1990 年宏基因组编码(DNA metabarcoding)由Giovannoni 等[5]首次提出。1998 年Handelsman 等[6]提取了湖底沉积微生物的DNA 片段。2000 年Rondon 等[7]提出“eDNA 技术”概念,直到2005 年Martellini等[8]从水样中提取 DNA 寻找地表水污染源,开启了该技术运用于水生生态系统的篇章。2008 年Ficetola 等[9]利用eDNA 技术追踪入侵物种美国牛蛙,使eDNA 技术首次应用在水生生物的监测上[10]。在2010 年前后,有研究者逐渐将eDNA技术引入海洋调查中。结合海水中各种复杂的化学和物理变化过程,构建eDNA 研究相关计算模型,将eDNA 技术发挥更大的作用。目前,eDNA与DNA 技术相结合,已成为生物多样性监测有力的新型工具。近年来,eDNA 的应用经历了巨大的发展,已广泛应用于群落生态学、古环境研究、生物监测、生物保护学和入侵生态学等各个领域。珠海外伶仃国家级海洋牧场位于珠江口万山群岛中北部,海域面积9.83 km2,于2018 年入选第四批国家级海洋牧场示范区[11]。目前对珠海外伶仃海洋牧场的调查研究主要集中在生物资源种类组成[12]、数量分布[13]、时空变化[14]等方面,而有关微生物研究只涉及到吖啶橙染色直接镜检计数法[15],尚未见到采用eDNA 技术进行检测研究的报道。【本研究切入点】为解决珠海外伶仃海洋牧场微生物检测方法的单一性,提高检测的准确性,运用eDNA 技术检测珠海外伶仃海洋牧场的微生物多样性,并与传统检测方法进行对比验证,为珠海外伶仃海洋牧场的微生物检测提供一条新的途径。【拟解决的关键问题】基于eDNA 技术的检测结果,分析珠海外伶仃海洋牧场的微生物群落组成、生物多样性等特征,以期为珠海外伶仃海洋牧场生态系统管理维护提供参考。
1 材料与方法
1 站位点设置
为了解微生物多样性,本研究在广东省珠海外伶仃海域国家级海洋牧场示范区设立了S1~S9(22°05'25.107″~22°07'55.272″N、113°59'50.170″~114°03'35.238″E)9 个站位点,站位点位置分布见图1。

图1 9 个站位点位置Fig.1 Location of 9 stations
1.2 采样及样品预处理
为防止样品间出现交叉污染,在采样前对采样器皿进行消毒,以10%次氯酸钠溶液[16-17]浸泡10 min,蒸馏水冲洗3 遍,采样人员佩戴手套并及时更换。本研究于2021 年9 月13 日在9 个站位点进行样品采集,每个站位点设6 个平行试验,共采集54份水样。每份水样采集2 L表层水(在水面以下约2 m 处采集)[16-17],用0.45 μm 孔径的混合纤维滤膜过滤[18-19],加入5 mL 95%无水乙醇灭活,将过滤后的滤膜放置于无菌吸水纸,对折,使滤膜干燥,同一站位点的样品袋折叠放入2 mL 的离心管,贴上标签纸,最后放入液氮内保存。
1.3 DNA 提取
DNA 提取采用DNA Clean &Concentrator-25 试剂盒(购于北京天漠科技开发有限公司),参考Martin-Laurent[20]的方法:洗涤后的样品加入400 μL Cell 裂解液,旋涡振荡1 min,65℃水浴30 min,每5 min 振荡1 次,冷却到室温。以12 000 r/min、4℃离心10 min,取上清,加入等体积的酚/氯仿/异戊醇(25 ∶24 ∶1)混合溶液,上下颠倒、混匀;继续以12 000 r/min、4℃离心10 min 后取上清,加入等体积的氯仿/异戊醇(24 ∶1)混合溶液,上下颠倒、混匀;再以12 000 r/min、4℃离心10 min,取上清于新离心管中,加入0.6 倍体积的异丙醇,室温静置1 h 后,12 000 r/min、4℃离心10 min,弃上清,用70%冰乙醇溶液洗涤沉淀,倒置,于无菌干燥沉淀中加入100 μL 含20 μg/mL RNaesA 的无菌TE buffer溶解DNA 沉淀,于30℃水浴30 min 后取出置于-20℃冰箱保存。
1.4 PCR 扩增及高通量测序
将提取好的DNA 样品寄到生工生物工程(上海)股份有限公司,使用引物MF-F:5'-GTCGGTAAAACTCGTGCCAGC-3'进行PCR 扩增[21],使用Cutadapt 软件[22-23]去除Read1 3'端测序引物接头:AGATCGGAAGAGCACACGTCTG AACTCCAGTCA 和Read2 3'端测序引物接头:AG ATCGGAAGAGCGTCGTGTAGGGAAAGAGTGT,使用Prinseq 软件[24-25]切除Reads 尾部质量值20以下的碱基,设置10 bp 的窗口,若窗口内的平均质量值低于20,则从窗口开始截去后端碱基,过滤质控后的含N 序列和短序列,最终过滤掉低复杂度的序列(图2)。

图2 原始序列数据(A)和有效序列数据(B)长度分布Fig.2 Length distributions of raw sequence data (A) and valid sequence data (B)
1.5 数据分析
1.5.1 微生物种类鉴定 序列比对使用 QIIME 软件中的UCLUST[26],在默认参数中将序列与Silva、NCBI、FGR 数据库的模板序列相比对,利用高通量测序技术,将经过处理的序列进行OTU 聚类,然后对OTU 进行注释,完成物种分类。
1.5.2 多样性指数分析(1)Chao 指数[27]: 估计微生物群落中含OTU 数目的指数,计算公式如下:
式中,Schao是估计的OTU 数,Sobs是实际观测到的OTU 数,n1是只含有1 条序列的OTU 数目,n2是只含有2 条序列的OTU 数目 。
(2)Shannon 多样性指数[28]: 用于估算样品中微生物多样性指数之一。Shannon 值越大,说明群落多样性越高。计算公式如下:
式中,Sobs是实际观测到的OTU 数,ni是第i个OTU 包含的序列数,N是所有个体数目,此处为序列总数。
(3)Simpson 指数: 用于估算样品中微生物多样性指数之一,Simpson 指数值越大,说明群落多样性越低。计算公式如下:
式中,Sobs是实际观测到的OTU 数,ni是第i个OTU 包含的序列数,N是所有个体数目,此处为序列总数。
(4)物种优势度(Y):表示微生物物种所占的优势程度,计算公式为:
式中,ni是第i个OTU 包含的序列数,N是所有个体数目,fi为第i个OTU 在各采样点出现的频率[31]。本文将Y> 0.02 的微生物确定为优势种。
2 结果与分析
2.1 测序所得OTU
测序所得有效数据见表1,9 个站位点的样品共产生779 706 个OTU,平均长度255 bp,其中S9 站位点样品的OTU 最多、为95 914 个,S1 站位点的OTU 最少、为77 448 个。测序所得有效数据每个样品平均获得96 141 条原始序列,经过拼接和质量过滤[32-33],每个样品平均得到86 634 条序列,高质量数据占90.1%。

表1 样本有效序列数据统计Table 1 Statistics of valid sequence data of samples
2.2 珠海外伶仃海洋牧场微生物α 多样性
本次高通量测序有效数据与OUT 数量关系如图3,各个样本的稀释性曲线趋于平缓。其中S1-2、S4-1、S8-2 等3 个位点的稀释曲线趋于平缓前的斜率明显比其他曲线更高,表明3 个位点的OTU 数目少于其他位点,即这3 个位点检测出的物种数量少于其他位点。由表2 可知,S1 站位点无论是Shannon 指数还是Chao 指数均最低、分别为2.89 和508.49,S9 站位点最高、分别是3.37和660.37,表明S1 站位点的微生物多样性最低,S9 站位点的微生物多样性最高。

表2 α 多样性指数统计Table 2 Statistics of α diversity index

图3 α 指数稀释性曲线Fig.3 α index dilution curve
Pan/Core OTU 用于描述随着样本量加大物种总量和核心物种量变化的情况。Pan OTU 为泛OTU,是所有样本包含的OTU 总和;Core OTU 为核心OTU,是所有样本共有的OTU 数目。Pan/Core 曲线反映了持续抽样下新OTU 出现的速率。利用物种累积曲线可以判断样品量是否充分,如图4 所示,随着样品量的加大,Pan/Core 曲线趋于平缓,表示此环境中的物种并不会随样品量的增加而显著增多,证明本研究样品准备较好,准确性较高[34]。

图4 Pan/Core 曲线Fig.4 Pan/Core curve
2.3 珠海外伶仃海洋牧场微生物β 多样性
通过PCA 分析不同样本群落组成可以反映样本间的差异和距离,PCA 运用方差分解,将多组数据的差异反映在二维坐标图上,表示物种OTU在主成分上的贡献度,如样本物种组成越相似,反映在PCA 图中的距离越近。图5 显示,样本分散程度较大,距离较远,说明各站位点之间的微生物物种差异较大。

图5 各站位点样本的PCA 图Fig.5 PCA figure of samples from various staions
2.4 珠海外伶仃海洋牧场微生物物种
利用eDNA 技术从珠海外伶仃海洋牧场9 个站位点的样本中检测到779 706 个OTU,进行注释后得到的微生物物种共30 种,(表3)隶属于5 门7 纲14 目19 科26 属,分为变形菌门(Proteobacteria)、拟杆菌门(Bacteroidetes)、放线菌门(Actinobacteria)、浮霉菌门(Planctomycetes)、厚壁菌门(Firmicutes),其中放线菌门仅有两种微生物分别是放线杆菌(Actinobacterium)和微酸菌(Ilumatobacter),在拟杆菌门中只检测到鞘氨醇杆菌(Sphingobacterium)。
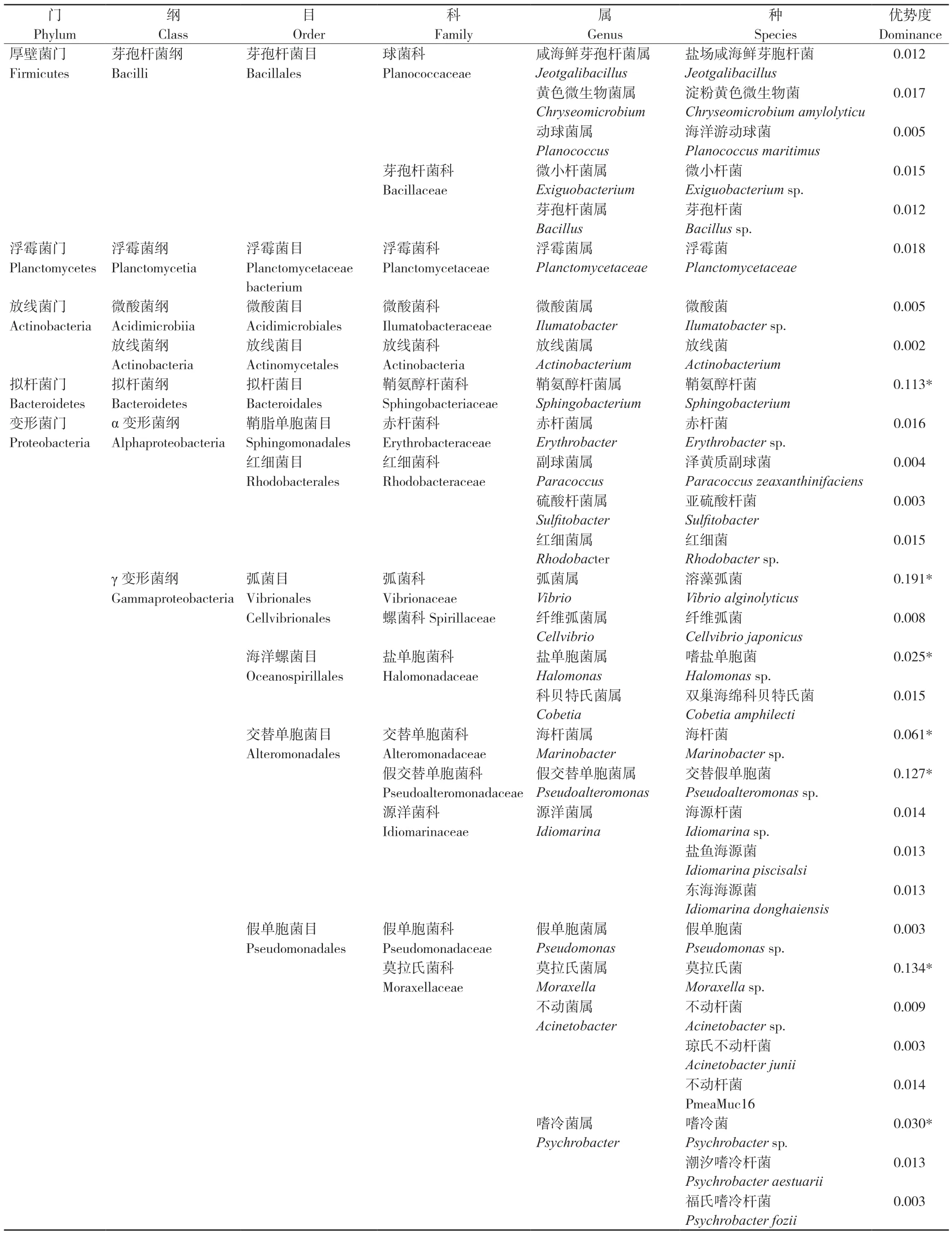
表3 9 个站位点的微生物物种统计Table 3 Microbial species statistics of 9 stations
2.5 珠海外伶仃海洋牧场微生物相对丰度
在珠海外伶仃海洋牧场各个站位点中变形菌门至少占61%以上,远高于其他门,是绝对优势门(图6)。放线菌门在各站位点分布最少,且在S1、S3、S8 中未被检测到,并且各站位点有少量检测到的序列未被标注。

图6 各站位点微生物相对丰度Fig.6 Relative abundance of microorganisms from various stations
基于eDNA 技术对珠海外伶仃海洋牧场9 个站位点的样本检测到的微生物物种分为5 门,其中放线菌门最少,只占总检测物种的1.06%,而变形菌门是绝对优势门,占总检测物种的75.06%,其中γ-变形菌纲(Gammaproteobacteria)最多,共5 目8 科11 属17 种;其余为α-变形菌纲(Alphaproteobacteria),共4 目4 科6 属6 种。此外,拟杆菌门占比11.35%,厚壁菌门占比5.28%,浮霉菌门占比1.91%。优势种共有7 种(表3),包含交替假单胞菌(Pseudoalteromonas)、海杆菌(Marinobacter)、莫拉克氏菌(Moraxella)、嗜冷菌(Psychrobacter)、溶藻弧菌(Vibrio alginolyticus)、嗜盐单胞菌(Halomonas)、鞘氨醇杆菌,其中溶藻弧菌优势度最高(0.191),为绝对优势种,其次是莫拉克氏菌和交替假单胞菌优势度分别达到0.14 和0.127,为主要优势种。
3 讨论
3.1 eDNA 方法的准确性
本研究中每个样品的高质量数据占90.1%。宋伦等[35]研究表明,DNA 高质量数据达到90%以上,则测得OTU 数据准确可靠。当各个样本的α 指数稀释性曲线趋于平缓,表明测序数据趋于饱和,测序深度已足够反映样品中的所有OTU 信息,也就可以认为测序深度已经基本覆盖到样品中所有物种[36]。且Pan/Core 曲线趋于平缓,表示此环境中的物种并不会随样品量的增加而显著增多。
深入研究海洋微生物群落组成、分布及其形成机制有助于监测海洋牧场生态系统的动态变化、预测人类活动、理解气候变化等自然现象对珠海外伶仃海洋牧场的影响。eDNA 技术与传统形态学方式相比,在微生物调查中更加省时省力、节约成本,并且对环境无损伤,对传统形态学方法监测结果有较高的还原性,并可检测到一些传统方法无法识别的稀有物种和隐匿物种,说明 eDNA 技术在生物多样性监测中具有重要应用潜力。
3.2 eDNA 技术应用于珠海外伶仃海洋牧场微生物检测的不足与建议
在珠海外伶仃海洋牧场的微生物多样性研究中,因受疫情影响,本研究只有2021 年秋季(9 月)的数据,尚未开展其他季度的调查研究,并且采集样品单一,无法较全面地掌握珠海外伶仃海洋牧场微生物群落的空间分布规律和时空变化,为提高数据结果的准确性、了解微生物群落在珠海外伶仃海洋牧场的变化趋势,应长期、定期地开展不同水深的采样研究。
本研究也揭示了eDNA 技术的一些不足之处,自然界中有大量未被发现、培养和测序的物种,其基因序列在数据库中不存在,导致数据库信息与自然界实际存在的物种不对等,无法将检测到的所有OTU 注释到种水平。同时,受引物的偏好性影响,有些物种无法准确定量地检测出。此外,季节、水温、流速等自然条件变化导致样品成分变化,从而影响物种的检测和相对丰度。因此,eDNA 技术仍需要与传统形态学鉴定方法紧密结合,才能构建可靠的多维生物监测系统。
4 结论
本研究运用eDNA 检测数据和海洋生物数据库,根据微生物特征条形码序列设计扩增引物,检测到珠海外伶仃海洋牧场30 种微生物。通过种类组成和优势种的对比分析,确定了变形菌门为绝对优势门,以及交替假单胞菌、海杆菌、莫拉克氏菌、嗜冷菌、溶藻弧菌、嗜盐单胞菌、鞘氨醇杆菌7 种优势菌,推进了广东省珠海外伶仃海域国家级海洋牧场示范区海洋生物多样性研究,为后续海洋牧场微生物方面的研究提供基础数据和科学参考。随着eDNA 技术的不断完善和发展,该方法不仅会用于大规模的生物监测,更有望融合到海洋牧场生物多样性监测的标准程序中。

