基于SLAF 标记的大豆遗传图谱构建及苗期耐盐性QTL 定位
陈奕博 杨万明 岳爱琴 王利祥 杜维俊 王敏
(1. 山西农业大学农学院,太谷 030801;2. 晋中学院生物科学与技术学院,榆次 030619)
土壤盐渍化是一个全球性的生态问题,会对作物产生严重的负面影响。全球盐渍土和碱性土的面积分别为3.97 亿hm2和4.34 亿hm2,并且有19.5%的灌溉地和2.1%的旱地受到盐胁迫的影响[1]。其中,我国盐碱土面积为0.991 3 亿hm2,占种植总面积的4.88%[2],并且呈现局部治理、整体恶化、面积增加的趋势。可盐渍地作为我国重要的后备耕地,未来可开垦为农业备用耕地。因此,对当代育种科学家而言,培育耐盐性强的作物,可保障我国粮食生产安全[3]。
大豆(Glycine max(L.)Merr.)是世界范围内重要的油料作物和粮食作物,原产于中国,是当今世界最重要的植物蛋白与植物油来源[4],近年来,我国对其需求量与进口量逐年增加。大豆通常被认为是一种盐敏感作物,当土壤盐度超过5 dS/m 时,大豆开始受到盐害,更高浓度的盐害下会出现减产,严重时会绝收[5]。盐浓度过高通常是由氯化钠引起的,它会引起2 种不同却相互关联的胁迫,渗透胁迫和离子胁迫[6]。在盐浓度升高时,渗透胁迫立即发生,并抑制水分吸收和细胞扩张。盐胁迫早期与干旱胁迫[7]相似,且对生长速率的影响大于离子毒性[8],盐胁迫发生一段时间后,离子胁迫的影响逐渐显现,有毒的离子在植物体内积累,增加了叶片死亡率,降低了光合作用。这些有害离子不仅直接抑制代谢,而且还通过减少有益营养物质[9]的摄取而破坏离子稳态。盐胁迫下植物表现出多种症状,包括生长抑制、衰老甚至死亡。盐胁迫是大豆生长发育过程中一种重要的非生物胁迫,会对其生长产生严重的负面影响。盐渍化土壤严重影响着大豆的正常生长,会造成大豆产量低下。但随着世界范围内盐渍化的不断加剧,且改良土壤具有比较高的成本,较难实现,因此,明确大豆耐盐机制,筛选耐盐基因,进而培育出耐盐性优良的大豆品种,是提高大豆产量的重要途径。
早期研究表明,大豆苗期耐盐性状是由单个显性基因决定,现有研究结果表明,大豆苗期耐盐性状是受多个不同基因控制的数量性状[10]。过去几十年来,通过正向遗传学方法获得大豆耐盐QTL 或基因,一方面结合构建高密度的分子图谱,获得与耐盐基因连锁的分子标记,寻找QTL 附近的分子标记去筛选获取最终的目的基因;另一方面利用不同的遗传群体、不同的定位指标及方法对大豆耐盐性状进行QTL 定位,运用分子标记辅助(marker assisted selection, MAS)育种技术对主效QTL 开展精细定位,从而挖掘出耐盐相关基因,为大豆耐盐育种提供基因资源[11]。Guan 等[12]在第3 染色体上进行精细定位,将其命名为GmSALT3,该基因定位于内质网,可以显著提高大豆耐盐性。Zhang 等[13]通过QTL定位确定GmCDF1为重要的候选基因,后续试验表明该基因是大豆萌发期耐盐性的负调控基因。通过反向遗传学方法鉴定的大豆耐盐基因也有很多,基因GmNHX1可将胞质Na+转运到液泡中[14],在大豆中过表达和拟南芥中异源表达都可以增强转基因株系的耐盐能力[15]。Zhao 等[16]在拟南芥中异源表达GmSOS1,可以降低Na+积累,并增加抗氧化酶活性,从而参与拟南芥耐盐性。Wei 等[17]研究表明,GmCLC1能够通过调控Cl-的积累增强植物耐盐性。Chen 等[18]发 现GmHKT1和GmHKT1;4均 可 以 调节Na+和K+的平衡,增强转基因烟草耐盐能力。当植物受盐胁迫时,相关转录因子也会参与,如一些MYB[19]、bZIP[20]、WRKY[21]、ERF[22]、NAC[23]等转录因子家族成员与大豆耐盐性相关。由于构建QTL 定位群体需要考虑多方面原因,如群体的遗传背景、定位时的环境变化、基因与基因互作、标记的准确性。因此,在这些因素变化后,所定位到的QTL 结果也可能会随之改变。但近年来,随着QTL精细定位研究工作不断深入,在了解QTL 的位置及效用机理后,将QTL 定位结果用于挖掘候选基因依然拥有可预见的前景。
前人主要利用温室和自然环境对大豆苗期耐盐性进行鉴定研究,定位的耐盐QTL 大多位于N 连锁群上,但是,由于鉴定环境的不同和遗传群体材料遗传背景的差异,鉴定的相关QTL 位点也存在巨大差异。
本研究以栽培大豆品种晋大53 为母本和野生大豆品种平南为父本杂交衍生的128 个株系的RIL 群体为材料,结合基于SLAF 标记构建的高密度遗传连锁图谱,再以大豆苗期盐处理后致死浓度(plant death concentration, PDC)作为耐盐指标,对连续2年大豆苗期耐盐性进行QTL 分析。发掘控制大豆苗期耐盐性的新QTL 位点,为大豆耐盐性的遗传改良提供参考,促进耐盐基因在大豆耐盐育种中的应用。
1 材料与方法
1.1 材料
所用RIL 群体由山西农业大学大豆种质创新与利用实验室提供,母本栽培大豆晋大53 和父本野生大豆平南。通过单粒传法获得由128 个株系组成F2:12重组自交系群体。
1.2 方法
1.2.1 遗传图谱的构建 应用北京百迈科生物科技有限公司自主研发的SLAF-seq(Specific-Locus Amplified Fragment Sequencing)技术[24]和High Map软件[25]对大豆128 份重组自交系群体开发高密度分子标签,根据大豆参考基因组大小、GC 含量,选择酶切方案。根据选定的酶切方案,分别对检测合格的各样品基因组DNA 进行酶切试验。处理后选取质检合格的酶切片段(SLAF 标签)进行测序。获得各个样品的reads,通过对reads 进行评估,使用reads 间聚类的方法,在亲本和子代中开发SLAF 标签。最后利用多态性的SLAF 标签,构建遗传图谱,并进行图谱评估。
1.2.2 大豆苗期耐盐性鉴定 2020-2021年连续2年在山西农业大学旱棚内进行试验,采用王聪等[26]方法进行,将大豆种子播种于35 穴的育苗盘中,蛭石与灭菌土等比例混合后作为基质,采用随机区组设计,重复3 次,重复内每个家系播1 穴,每穴定苗2 株。待大豆植株长到二叶一心期时,用130 mmol/L NaCl 溶液进行逆境处理(即开始处理时的初始浓度),每次每穴30 mL,每2 d 处理1 次,浇至320 mmol/L NaCl 时(即所有家系均死亡时的最大浓度),试验完成。
1.2.3 表型数据的调查和分析 待大豆长至二叶一心期开始胁迫处理,每2 d 统计一次,及下次胁迫处理前进行统计,记录每个家系死亡时NaCl 的浓度,直到该家系所有植株全部死亡,当每个家系植株死亡,NaCl 浓度不同时,取2 个植株死亡时浓度的均值,可计算出该家系的平均致死浓度,即为致死浓度(plant death concentration, PDC),然后以各个家系的致死浓度作为耐盐指标,致死浓度与耐盐等级对照如表1 所示,利用SPSS 数据处理系统对表型值进行统计分析。

表1 致死浓度与耐盐等级对照表Table 1 Comparison between plant death concentration and salt tolerance grade
1.2.4 QTL 定位分析 以复合区间作图法作图,以LOD=2.5 为阈值对QTL 进行定位分析。LOD 值≥5.0 即可认为该区间存在一个上位性QTL,并计算QTL 的加性效应、上位性效应及表型贡献率。QTL 命名方法后面的数字为染色体编号,参照McCouch 等[27]方法。PDC Ⅰ、PDC II、PDC Ⅲ、PDCA 分别表示致死浓度重复一、致死浓度重复二、致死浓度重复三和3 次致死浓度平均值。
2 结果
2.1 SLAF-seq文库构建
对参考物种基因组(Glycine maxCv. Williams 82.a2.v1)序列进行电子酶切预测,根据酶切方案选择原则,选择的酶为HaeⅢ,酶切片段长度为264-364 bp 的序列定义为SLAF 标签,预测可得到114 027 个SLAF 标签,位于重复序列区的SLAF 标签比例为7.11%,SLAF 标签在基因组各染色体上分布基本均匀,酶切方案可行。对测序数据进行,共获得260 187 738 reads(52.02 Gb)数据,测序平均Q30 为89.24%,平均GC 含量为41.04%,样本GC分布正常。综上所述,数据比对效率、酶切效率和质量均达到目的要求。
2.2 遗传连锁图谱的构建
在进行SLAF 标签文库构建时,共获得140 864个SLAF 标签,其中,多态性SLAF 标签有44 298 个,可以用于遗传图谱构建的标签有33 742 个,亲本有效多态性为23.95%。为保证遗传图谱质量,将多态性SLAF 标签进行过滤,终得到可用于作图的SLAF标签8 223 个。将筛选出的8 223 个SLAF 标签,及山西农业大学大豆种质创新与利用实验室提供的可以比对到参考基因组上的SSR 标记39 个共8 266 个,一起进行分群。通过与大豆参考基因组的定位将SLAF 标签分为20 个连锁群,以连锁群(每条染色体)为单位,获得连锁群内Marker 的线性排列,并估算相邻Marker 间的遗传距离,进行连锁图谱的构建。最终,得到一张包括20 个连锁群共7 945 个Marker的连锁遗传图谱(图1),该图谱总图距为3 148.46 cM,标记完整度为99.56%。各个连锁群Marker 数目、总图距、平均图距、最大Gap 和Gap<5 cM 的比例基本信息统计如表2 所示,其中,第18 染色体的标记数最多(740 个),第1 染色体的标记数最少(57个)。标记间平均距离最大值为3.30,最小值为0.25,分别位于第16 染色体上和第4 染色体上。最大Gap在第18 染色体上,值为27.09,最小在第17 染色体上,值为6.87。

表2 染色体标记信息Table 2 Chromosome marker information

图1 遗传图谱结果图Fig. 1 Genetic map results
2.3 大豆苗期耐盐性表型分析
为利用亲本平南和晋大53 进行大豆耐盐性的QTL 定位研究,对双亲连续2年不同环境PDC Ⅰ、PDC II、PDC Ⅲ和PDCA 等4 个耐盐指标差异进行分析,2年内,晋大53 的PDC Ⅰ、PDC Ⅲ、PDC II和PDCA 的4 个指标均显著高于平南(P<0.05)。
在2年不同环境下,RIL 群体中各株系的致死浓度(PDC)在160-320 mmol/L 都有分布,且呈连续分布规律,符合数量性状的特点,从亲本致死浓度在群体内的分布来看,存在双向超亲遗传规律,这也是数量遗传的典型特征。其中,2020PDC Ⅰ的变异系数最大,为35.1,2020PDCA 的变异系数最小,为22.1,正态分布的适合性检验结果表明,2年内致死浓度的偏度和峰度的绝对值均小于1。以上结果表明,耐盐性状是一种数量性状,RIL 群体耐盐相关性状数据适合QTL 定位(表3,图2)。
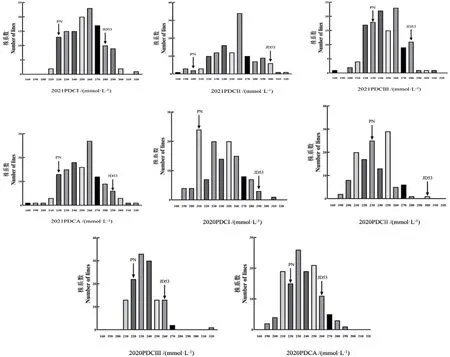
图2 RIL 群体耐盐性状频次分布Fig. 2 Frequency distribution of salt tolerance traits in RIL population

表3 RIL 群体中苗期表型统计Table 3 Phenotypic statistics of RIL population at seedling stage
2.4 RIL群体耐盐指标的相关性分析
通过对2020-2021年RIL 群体苗期耐盐性状进行相关性分析(表4)发现,2020PDC Ⅰ、2020PDC II、2020PDC Ⅲ、2020PDCA 各性状间多呈显著正相关,表明盐胁迫对大豆植株的影响是同向的。由相关性分析可知,2020PDC Ⅰ与2021PDC Ⅰ的相关系数为0.651,2020PDCA 与2021PDCA 的相关系数为0.415 5,各指标间呈正相关关系。综合2年结果,盐胁迫对于大豆表型耐盐性状影响是一致的,且2年各性状之间相关性较高,表明不同的自然环境对大豆苗期耐盐性状影响不大,说明128 个家系的耐盐指标可用于大豆苗期耐盐性的QTL 定位。

表4 耐盐性状的相关性分析Table 4 Correlation analysis of salt tolerance traits
2.5 大豆苗期耐盐性的QTL定位
通过对2年128 份RIL 群体耐盐性状相对值进行QTL 定位分析(图3),2年共检测到12 个相关性状的QTL 位点,分布于第2、3、5、7、9、11、13、15、18 和20 染色体上(表5)。2020年耐盐性状共定位到7 个QTL,分布在7 条染色体上,其中,第7 染色体上存在2 个位置相近的QTL 位点,性状的遗传变异解释率为24.58-31.84。2021年耐盐性状共定位到5 个QTL,分布于5 条染色体上,其中,第3 染色体上存在2 个位置相近的QTL 位点,性状的遗传变异解释率为31.22-31.22。通过比较2年各性状定位到的QTL,在第3 染色体上定位到多个QTL 位点,集中于58.81-108.22 cM 位置区间,在第7 染色体上定位到多个QTL 位点,集中于63.68-100.65 cM 位置区间,说明PDC 作为耐盐生理指标较准确,且在第3 染色体上可能存在耐盐相关的稳定QTL。对于QTL 加性效应分析发现,2020年qPDC15 为0.71,qPDC7-1 为1.28,qPDC2 为0.42,qPDC7-2 为0.97,2021年qPDC3-1 为0.58,qPDC9为1.20,qPDC20 为0.89,且皆为正值,说明这些位点增效基因来自父本平南,其他QTL 为负值,说明其他位点的增效基因主要来自母本晋大53(表5)。
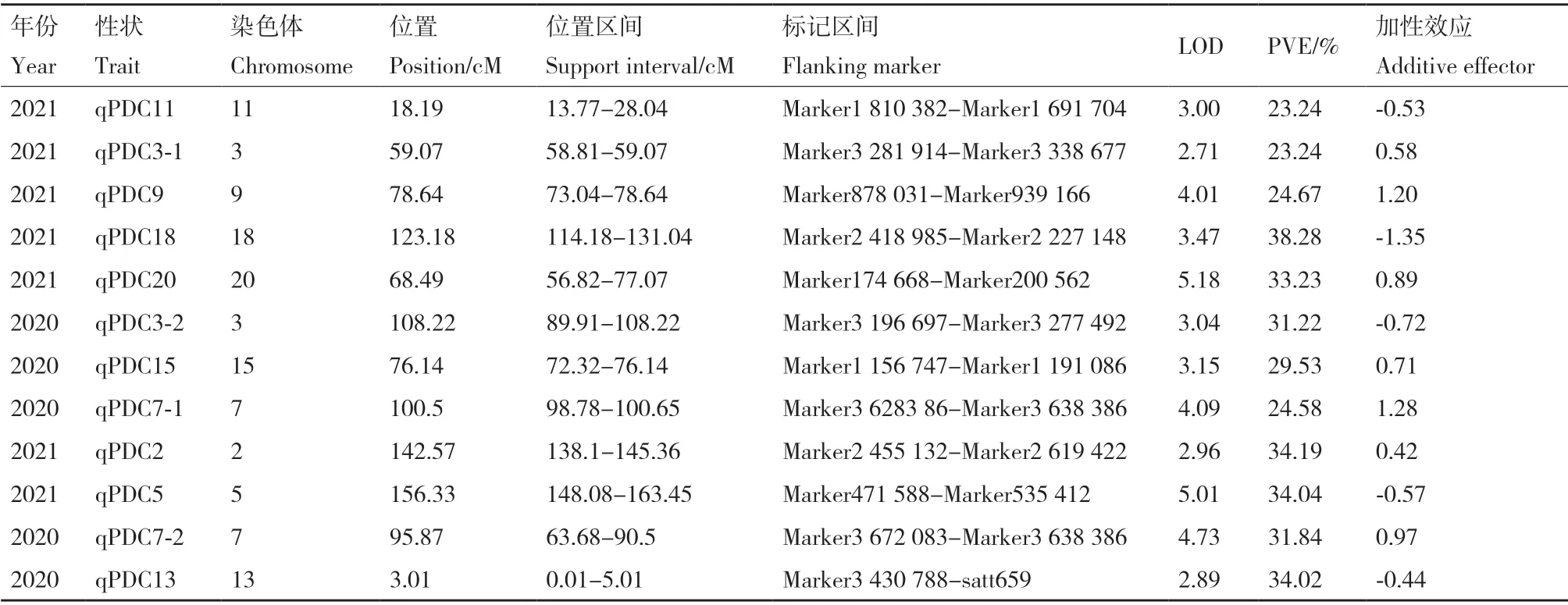
表5 大豆苗期耐盐性状QTL 分析Table 5 Analysis of QTL associated with salt tolerance in soybean seedling

图3 大豆苗期耐盐各性状的QTL 分布Fig. 3 QTL distribution of salt tolerance traits in soybean seedling stage
2.6 上位性QTL定位
检测到1 对与苗期耐盐相关的上位性位点(表6),该位点存在于第20 染色体的Marker174 668-Marker200 562,LOD 值为5.18,贡献率为33.32,上位性效应为0.89。

表6 大豆苗期的上位性位点Table 6 Epistatic loci of soybean at seedling stage
3 讨论
目前,在大豆耐盐QTL 定位和耐盐机制等方面取得很多进展[28]。近年来,大量研究使用大豆种内或种间重组自交系群体,在不同年份,不同地点,重复多次进行QTL 定位,这样定位到的结果比较可靠,前人普遍使用沙培或土培的方式进行耐盐性鉴定,调查的耐盐性状大多为叶片SPAD 值、耐盐系数、叶片受害面积等指标,这些鉴定方式和指标存在一定的合理之处,但也存在许多局限性,为了更好地反映大豆耐盐性,采用致死浓度作为耐盐指标进行调查。通过对2年耐盐性状的QTL 定位结果分析,各性状定位到的QTL 分布于不同染色体的不同位置上且2年的QTL 重叠度不高,这可能是由于环境的影响造成定位结果的不稳定。但是本研究在染色体上定位到的QTL 和前人研究结果较为一致,研究定位到的位置相近。
前人对苗期大豆耐盐QTL 定位研究较多[29]。Lee 等[29]利用S-100×Tokoy 为亲本的RILs 群体,以视觉耐盐等级(visual salt tolerance ratings, STR)作为盐处理后的耐盐指标,在N 和L 连锁群上定位 到6 个 耐 盐QTL。Hamwieh 等[30]以JWS156-1×Jackson 为亲本组成的F2群体,以叶片叶绿素含量SPAD 和STR 为指标在N 连锁群上定位到2 个QTL,都位于Satt339 分子标记附近。Chen 等[31]以南农1138-2×科丰1 号为亲本的RILs 群体,以STR、存活时间和存活率为指标定位出8 个耐盐QTL,分别位于7 个不同的连锁群上。Ha 等[32]使用野生大豆品种PI4-83463 与Hutcheson 杂交获得的106 个RILs 群体进行定位,其与耐盐相关的QTL基因定位在第3 染色体上Satt255 与BARC-038333-10036 之间。杨燕[33]以重组自交系群体NJRIKY(科丰1 号×南农1138-2)427 个家系为研究对象,结合构建的包含4 737 个重组bin(分子标记)的高密度大豆遗传图谱,以相对根长(relative root length,RRL)和相对干重(relative dry weight, RDW)为鉴定指标,一共检测到16 个大豆幼苗期耐盐 QTL 位点。陈华涛等[11]以植株盐处理后的存活时间(plant survival days, PSD)为指标,共检测到3 个耐盐QTL,它们分别位于B1、G 和K 三个连锁群上。据报道在大豆不同生育时期,大豆表现出不同的耐盐机制[34]。研究结果表明,第3 染色体上QTL,在大豆苗期耐盐相关性状定位[35]时,被多名研究人员重复检测到。其他报道也发现许多与大豆耐盐相关的新QTL。2008年,Chen 等[31]在大豆苗期耐盐QTL 定位中,通过田间和温室试验定位到8 个QTL分布在6 条染色体上,在大豆第3 染色体上和第18染色体上定位到了稳定的QTL 位点。Kan 等[36]利用由184 份F7:11组成的重组自交系,定位11 个大豆芽期耐盐相关QTL,同时在第8 染色体上和第18 染色体上定位到了耐盐显著相关的QTL。在2018年,Do 等[37]使用Williams 82(中度敏盐)和Fiskeby III(耐盐)构建的132 重组自交系群体进行苗期耐盐相关研究中,在第3 染色体和第13 染色体上都定位到了耐盐相关QTL。刘谢香[38]利用耐盐栽培大豆中黄39 与盐敏感野生大豆NY27-38 杂交后代衍生的142 个家系组建群体 ,定位到2 个大豆出苗期耐盐性QTL,分别位于第6 和第14 染色体。在实际生产种植上,大豆苗期是大豆生长发育的重要时期,对产量的影响较大。了解大豆苗期耐盐机制,挖掘耐盐基因可能成为今后大豆耐盐育种研究的一个重要方面。
4 结论
2020-2021年连续2年共定位到12 个QTL 位点。7 个QTL 位点具有加性效应,1 个QTL 位点具有上位性效应。有5 个与已报道的大豆耐盐相关QTL 物理位置相同,其余7 个QTL 则是本研究首次发现的新QTL 位点。在大豆第3 染色体58 cM 处与第18染色体123.18 cM 处定位到的苗期耐盐QTL 位点为大豆苗期耐盐主效QTL。

