真核翻译起始因子4γ2调控N-乙酰氨基半乳糖转移酶1 mRNA对胃癌细胞恶性表型的影响
李海洋, 张 淼, 陆 峰, 戴素华, 李芳芳, 鱼红亮, 后 博, 张 伟, 张明雷, 尹晓东
(江苏省滨海县人民医院, 1. 放疗科, 2. 消化内科, 3. 普外科, 4. 肿瘤科, 6. 血液科, 江苏 盐城, 224500; 5. 江苏省肿瘤医院 肿瘤科, 江苏 南京, 210009)
胃癌起源于胃黏膜上皮细胞,是消化道常见的侵袭性恶性肿瘤,发病率和病死率均很高[1]。研究[2]认为,胃癌可能是环境、饮食、幽门螺杆菌感染、遗传等多种因素共同作用的结果。目前,手术联合放疗、化疗是胃癌的主要治疗手段[3-4], 但胃癌早期发病隐匿,诊断较为困难,往往不能及时治疗,导致患者治疗效果差和预后不良[5]。因此,探究胃癌进展相关的分子靶标可为改善胃癌患者预后提供重要的理论依据。N-乙酰氨基半乳糖转移酶1(GALNT1)是N-乙酰氨基半乳糖转移酶(GALNT)家族的一员,其主要功能是参与O-糖基化合成[6]。异常的糖基化是多种人类肿瘤发生的标志,且与细胞多种生物学行为密切相关[7]。相关研究[8]报道, GALNT1在胃肠道肿瘤中主要充当致癌因子,可促进结直肠癌肿瘤生成及肝转移。然而, GALNT1是否在胃癌中异常表达并参与疾病进展尚未明确。真核翻译起始因子4γ2(EIF4G2)是真核翻译起始因子4γ(EIF4G)的一种亚型,其在大鼠胃黏膜中的高表达可诱导胃癌细胞体外增殖[9]。然而, EIF4G2表达对胃癌进展的影响及其可能的调控机制仍需深入探究。本研究探讨GALNT1对胃癌细胞生物学行为的具体影响,并分析GALNT1mRNA与EIF4G2的相互关系,现报告如下。
1 材料与方法
1.1 材料与设备
永生化人胃黏膜上皮细胞GES-1和人胃癌细胞HGC-27(北纳生物); 人胃癌细胞KE-39(上海酶研生物科技有限公司); 人胃癌细胞AGS、SNU-1、SNU-16(美国菌种保藏中心); RPMI-1640培养基、DMEM培养基(美国思拓凡公司); 胎牛血清(FBS)(浙江天杭生物科技股份有限公司); Lipofectamine®2000转染试剂、分光光度计、PierceTM磁性RNA-蛋白Pull-Down试剂盒(美国赛默飞世尔科技公司); sh-GALNT1-1、sh-GALNT1-2、shRNA-EIF4G2-1、shRNA-EIF4G2-2重组慢病毒和阴性对照sh-NC、shRNA-NC(广州市锐博生物科技有限公司); oe-EIF4G2和oe-NC质粒(北京义翘神州科技股份有限公司); RNAiso Plus试剂[宝日医生物技术(北京)有限公司]; ReverTraAce实时荧光定量聚合酶链反应(RT-qPCR)试剂盒[东洋纺(上海)生物科技有限公司]; FastStart Universal SYBR Master Mix、CCK-8试剂(德国罗氏诊断有限公司); GoTaq®实时定量PCR系统(美国普洛麦格公司); RIPA裂解液、二喹啉甲酸(BCA)蛋白浓度测定试剂盒(北京索莱宝科技有限公司); 兔源GALNT1、Bcl-2、Bax、cleaved-caspase3、caspase3、caspase9、MMP2、MMP9、GPADH抗体和辣根过氧化物酶(HRP)标记的羊抗兔免疫球蛋白G(IgG)(英国Abcam公司); 兔源EIF4G2、cleaved-caspase9抗体(美国Cell Signaling Technology公司); ECL发光液(北京酷来搏科技有限公司); TUNEL染色试剂盒(上海碧云天生物科技有限公司); 基质胶和Transwell小室(美国BD公司); 链霉亲和素磁珠、放线菌素D(美国MedChemExpress公司); Magna RNA结合蛋白免疫沉淀试剂盒(美国密理博公司); Image J软件(美国国立卫生研究院); 荧光显微镜(德国欧蒙医学诊断有限公司); 倒置显微镜(日本奥林巴斯)。
1.2 方法
1.2.1 生物信息学分析: 利用CCLE数据库(https://sites.broadinstitute.org/ccle)分析GALNT1、EIF4G2在胃癌细胞和正常细胞中的表达情况; 利用GEPIA数据库(http://gepia.cancer-pku.cn/)分析GALNT1、EIF4G2在胃癌组织和正常组织中的表达情况,并预测GALNT1、EIF4G2在胃癌组织中的相关性。
1.2.2 细胞培养: 永生化人胃黏膜上皮细胞GES-1和人胃癌细胞AGS、KE-39常规培养于DMEM培养基中; 人胃癌细胞SNU-1、HGC-27、SNU-16均用RPMI-1640培养基进行孵育。除了HGC-27细胞的培养基含有20%FBS以外,其余细胞的培养基含有10%FBS, 均置于37 ℃、5%CO2的孵育箱中。
1.2.3 细胞转染: 当接种于6孔板中的细胞(5×104个/孔)融合度达到60%~70%时,严格按照Lipofectamine®2000转染试剂使用说明将sh-GALNT1-1、sh-GALNT1-2、sh-NC、shRNA-EIF4G2-1、shRNA-EIF4G2-2、shRNA-NC、oe-EIF4G2和oe-NC瞬时转染至各组细胞中,分别设为sh-GALNT1-1组、sh-GALNT1-2组、sh-NC组、shRNA-EIF4G2-1组、shRNA-EIF4G2-2组、shRNA-NC组、oe-EIF4G2组和oe-NC组,另取未转染的细胞设为对照组; 在AGS细胞中分别共转染sh-GALNT1-1+oe-NC和sh-GALNT1-1+oe-EIF4G2, 并分别设为sh-GALNT1-1+oe-NC组和sh-GALNT1-1+oe-EIF4G2组, 48 h后转染完成,检测转染效率。
1.2.4 RT-qPCR检测GALNT1和EIF4G2表达: 使用RNAiso Plus试剂从各组细胞中提取总RNA。向每10 cm2的培养细胞中加入1 mL RNAiso Plus后匀浆,室温静置5 min, 加入RNAiso Plus试剂1/5体积量的氯仿,剧烈震荡混匀,室温静置5 min, 12 000 ×g 4 ℃离心15 min, 将上清液转移至新的离心管中,加入与上清液等体积的异丙醇,颠倒混匀,室温静置10 min, 12 000×g 4 ℃离心10 min, 弃上清,向沉淀中加入1 mL的75%乙醇清洗沉淀,弃上清保留沉淀,干燥,并加入适量的RNase-free水溶解沉淀。随后按照ReverTraAce RT-qPCR试剂盒说明书进行cDNA合成。以cDNA为模板,加入FastStart Universal SYBR Master Mix在GoTaq®实时定量PCR系统中进行PCR扩增反应。以GAPDH为内参,采用2-△△Ct法进行数据分析。
1.2.5 蛋白质印迹法(Western blot): 使用RIPA裂解液对蛋白进行提取,并用BCA试剂盒进行蛋白浓度检测。将行10%SDS-PAGE电泳的等量蛋白质转移至PVDF膜并在5%脱脂牛奶中进行室温封闭,加入一抗于4 ℃下进行孵育,并加入HRP标记的羊抗兔二抗于室温下进行孵育。加入ECL发光液对蛋白条带进行显影,用Image J软件记录灰度值。
1.2.6 CCK-8法检测细胞活性: 将转染后的AGS细胞种植于96孔板中进行培养(5×103个细胞/孔),分别于24、48、72 h后,加入体积分数为10%的CCK-8试剂。2 h后,使用分光光度计于450 nm处检测光密度值,并绘制增殖曲线。
1.2.7 克隆形成实验检测细胞克隆形成能力: 将转染后的AGS细胞接种于直径6 cm培养皿中,每皿1×104个细胞。2周后终止培养,以磷酸盐缓冲液(PBS)洗涤后用多聚甲醛固定细胞,室温下用0.5%结晶紫染色10 min。拍照保存并计算细胞克隆形成数目。
1.2.8 TUNEL染色法检测细胞凋亡: 将转染后的AGS细胞接种于96孔板中,经4%多聚甲醛和0.5% Triton X-100进行固定和透化处理后,添加TUNEL试剂,严格按照试剂盒说明书于37 ℃下反应45 min, 随后用DAPI对细胞核进行染色,于荧光显微镜下观察细胞凋亡情况。
1.2.9 划痕实验检测细胞迁移: 在6孔板中加入处于生长对数期的AGS细胞,细胞达到90%融合度时,使用200 μL无菌枪头划1条线, PBS洗涤3次后,加入无血清DMEM培养基。于倒置显微镜下测量0、48 h时的伤口宽度,计算细胞迁移数。
1.2.10 Transwell实验检测细胞侵袭能力: 将转染后的AGS细胞接种于含有基质胶的Transwell上室内,向Transwell下室加入500 μL含10%FBS的DMEM培养基。24 h后,用棉签擦去上室膜表层细胞,并分别用多聚甲醛和结晶紫进行固定和染色处理,最后使用倒置显微镜对侵袭细胞进行计数。
1.2.11 RNA结合蛋白免疫沉淀: 严格根据制造商的说明,使用Magna RNA结合蛋白免疫沉淀试剂盒进行RIP实验。将羊抗兔IgG或EIF4G2抗体包被的磁珠与制备好的细胞裂解物于4 ℃下孵育过夜,然后洗涤RNA-蛋白质复合物并与蛋白酶K缓冲液一起孵育,最后对RNA进行提取并纯化,利用RT-qPCR进行富集度分析。
1.2.12 RNA下拉实验: 严格根据说明书要求使用PierceTM磁性RNA-蛋白Pull-Down试剂盒进行RNA下拉实验。使用生物素标记的DNA探针与链霉亲和素磁珠在室温下孵育30 min, 并将细胞裂解物与链霉亲和素磁珠于4 ℃下孵育过夜。清洗磁珠,分离与磁珠结合的蛋白质,进行Western blot分析。
1.2.13 RNA稳定性检测: 将细胞接种于6孔板(6×105个细胞/孔)中, 37 ℃孵育过夜,然后用5 μg/mL放线菌素D对细胞进行处理(0、3、6、9、12 h),通过RT-qPCR检测mRNA稳定性。
1.3 统计学分析

2 结 果
2.1 GALNT1在胃癌组织和细胞中表达上调并与患者不良预后相关
CCLE数据库检测结果显示,GALNT1在胃癌细胞中表达上调,见图1A; GEPIA数据库检测结果显示,与正常组织比较,GALNT1在胃癌组织中表达上调,差异有统计学意义(P<0.05), 见图1B; GEPIA数据库检测结果显示,GALNT1高表达与胃癌患者低总体生存率相关,见图1C。RT-qPCR、Western blot检测结果显示,与永生化人胃黏膜上皮细胞GES-1比较,GALNT1mRNA、GALNT1蛋白在人胃癌细胞(HGC-27、AGS、SNU-1、SNU-16、KE-39)中的表达水平更高,差异有统计学意义(P<0.001), 见图1D、图1E。本研究选用GALNT1相对表达量最高的AGS细胞进行后续实验。

A: 基于CCLE数据库检测GALNT1在胃癌细胞中的表达; B: 基于GEPIA数据库检测GALNT1在胃癌组织、正常组织中的表达(不同组织比较,*P<0.05); C: 基于GEPIA数据库检测GALNT1表达与胃癌患者生存率的相关性; D、E: RT-qPCR检测GALNT1 mRNA、Western blot检测GALNT1蛋白在永生化人胃黏膜上皮细胞系和人胃癌细胞中的表达(与GES-1比较, ***P<0.001)。图1 GALNT1在胃癌组织和胃癌细胞中的表达及与患者不良预后的关系
2.2 干扰GALNT1对胃癌细胞增殖和凋亡的影响
为阐明GALNT1对胃癌细胞生物学行为的具体影响,本研究将sh-GALNT1-1、sh-GALNT1-2干扰质粒转染至AGS细胞并检测转染效率。结果显示,与sh-NC组比较, sh-GALNT1-1组、sh-GALNT1-2组GALNT1mRNA、GALNT1蛋白表达均降低,差异有统计学意义(P<0.001), 见图2A、图2B, 本研究后续选择转染效率较高的sh-GALNT1-1进行实验。CCK-8法检测结果显示,与sh-NC组比较, sh-GALNT1-1组中细胞活性降低,差异有统计学意义(P<0.001), 见图2C。克隆形成实验结果表明, sh-GALNT1-1组细胞克隆形成率低于sh-NC组,差异有统计学意义(P<0.001), 见图2D。TUNEL实验结果表明,与sh-NC组相较, sh-GALNT1-1组细胞凋亡率上升,差异有统计学意义(P<0.001), 见图2E。凋亡相关蛋白的Western blot测定结果表明, sh-GALNT1-1组中Bcl-2蛋白表达低于sh-NC组, Bax、cleaved-caspase3、cleaved-caspase9蛋白表达高于sh-NC组,差异有统计学意义(P<0.001), 2组caspase3、caspase9蛋白表达比较,差异无统计学意义(P>0.05), 见图2F。
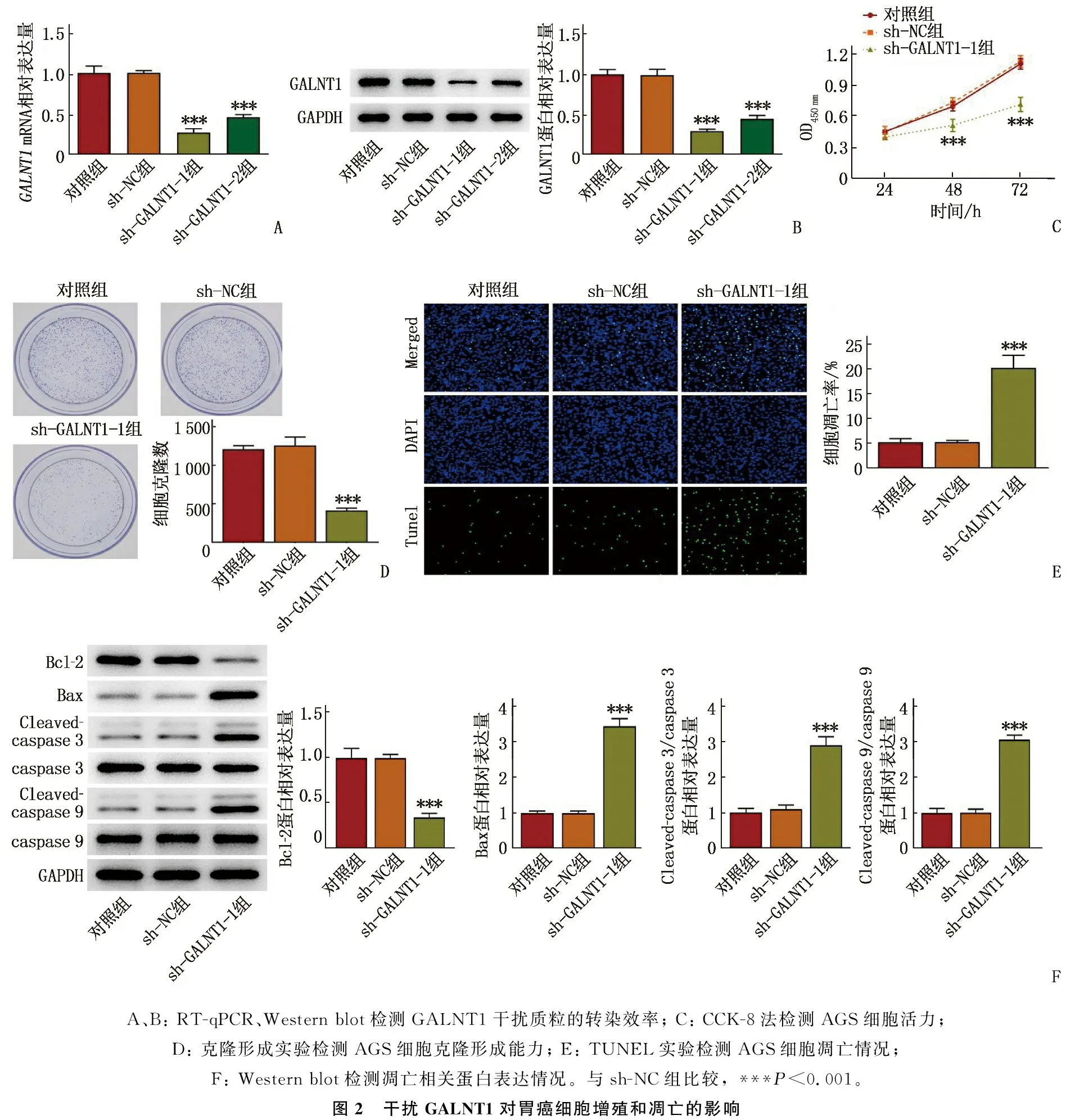
A、B: RT-qPCR、Western blot检测GALNT1干扰质粒的转染效率; C: CCK-8法检测AGS细胞活力; D: 克隆形成实验检测AGS细胞克隆形成能力; E: TUNEL实验检测AGS细胞凋亡情况; F: Western blot检测凋亡相关蛋白表达情况。与sh-NC组比较, ***P<0.001。图2 干扰GALNT1对胃癌细胞增殖和凋亡的影响
2.3 干扰GALNT1对胃癌细胞迁移和侵袭的影响
划痕实验显示,与sh-NC组比较, sh-GALNT1-1组划痕相对愈合率降低,迁移细胞数目减少,差异有统计学意义(P<0.001), 见图3A。Transwell实验显示,与sh-NC组比较, sh-GALNT1-1组侵袭细胞数目减少,差异有统计学意义(P<0.001), 见图3B。Western blot结果表明, sh-GALNT1-1组MMP2、MMP9蛋白表达水平低于sh-NC组,差异有统计学意义(P<0.001), 见图3C。
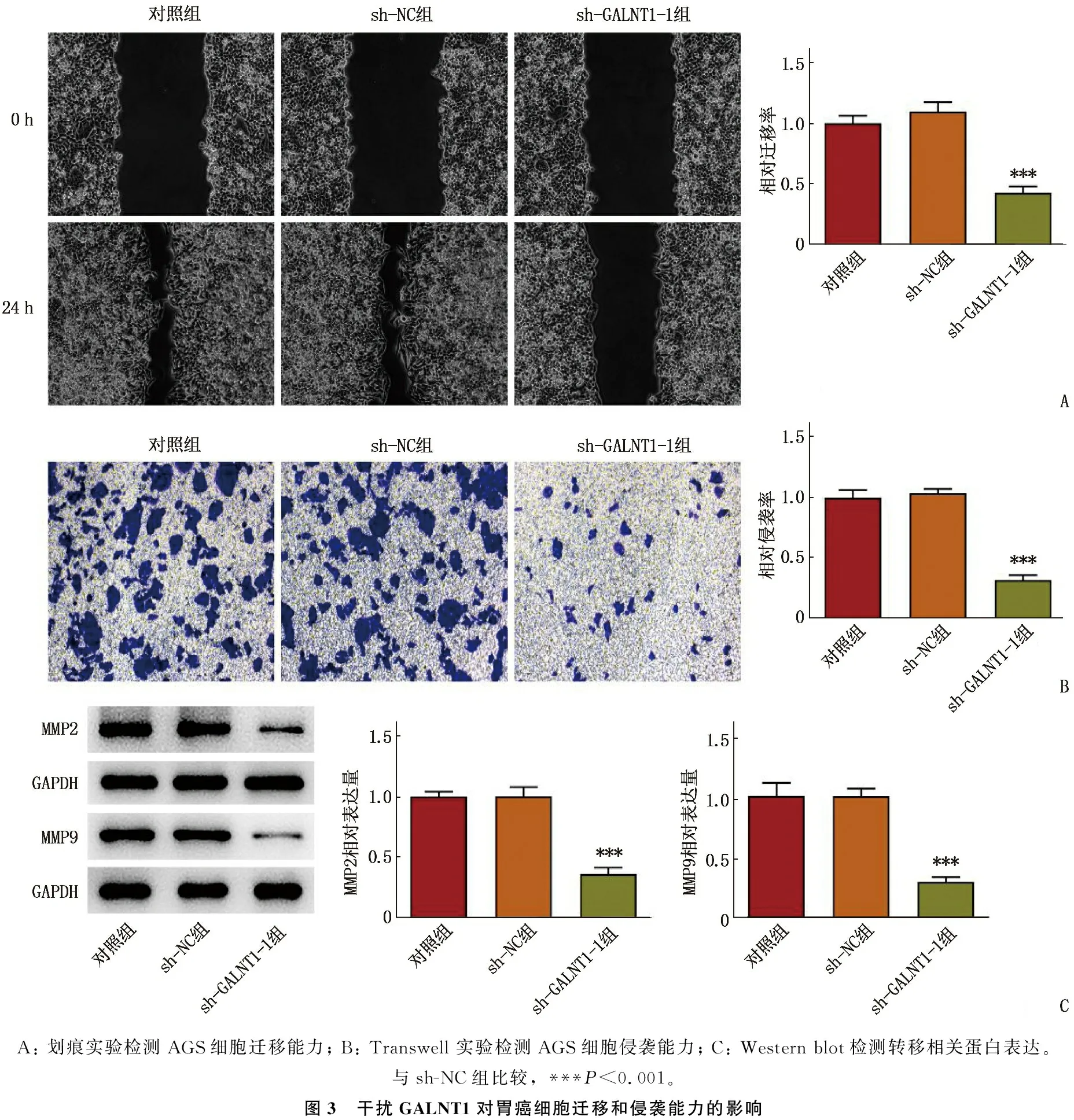
A: 划痕实验检测AGS细胞迁移能力; B: Transwell 实验检测AGS细胞侵袭能力; C: Western blot检测转移相关蛋白表达。与sh-NC组比较, ***P<0.001。图3 干扰GALNT1对胃癌细胞迁移和侵袭能力的影响
2.4 EIF4G2在胃癌组织和胃癌细胞中表达上调并与患者不良预后相关
CCLE数据库检测结果显示,EIF4G2在胃癌细胞中表达上调,见图4A; GEPIA数据库检测结果显示,与正常组织比较,EIF4G2在胃癌组织中表达上调,差异有统计学意义(P<0.05), 见图4B; GEPIA数据库检测结果显示,EIF4G2高表达与胃癌患者低总体生存率相关,见图4C。GEPIA预测结果发现,EIF4G2与GALNT1在胃癌组织中的表达呈正相关,见图4D。RT-qPCR、Western blot检测结果显示,与GES-1细胞比较,EIF4G2mRNA、EIF4G2蛋白在AGS细胞中的表达水平更高,差异有统计学意义(P<0.001), 见图4E、图4F。

A: 基于CCLE数据库检测EIF4G2在胃癌细胞中的表达; B: 基于GEPIA数据库检测EIF4G2在胃癌组织与正常组织中的表达(不同组织比较, *P<0.05); C: 基于GEPIA数据库检测EIF4G2表达与胃癌患者生存率的相关性; D: 基于GEPIA数据库检测GALNT1与EIF4G2在胃癌组织中表达的相关性; E、F: RT-qPCR检测EIF4G2 mRNA、Western blot检测EIF4G2蛋白在人胃黏膜上皮永生细胞系和胃癌细胞中的表达(与GES-1比较, ***P<0.001)。图4 EIF4G2在胃癌组织和胃癌细胞中的表达及与患者不良预后的关系
2.5 EIF4G2对GALNT1 mRNA稳定性的影响
为探究GALNT1mRNA与EIF4G2在胃癌组织中的结合关系,本研究利用RIP实验和RNA下拉实验进行验证,结果显示,相较于IgG中,GALNT1在EIF4G2抗体中的富集度升高,差异有统计学意义(P<0.001), 见图5A、图5B。为检测EIF4G2对GALNT1mRNA的调控关系,分别转染EIF4G2过表达和EIF4G2 shRNA干扰质粒, RT-qPCR和Western blot检测结果发现, oe-EIF4G2质粒转染导致EIF4G2mRNA和EIF4G2蛋白表达增加, shRNA-EIF4G2-1、shRNA-EIF4G2-2质粒转染导致EIF4G2mRNA和EIF4G2蛋白表达降低,差异有统计学意义(P<0.01或P<0.001), 见图5C、图5D, 后续选择干扰效率较高的shRNA-EIF4G2-2进行实验。RT-qPCR检测结果显示,与shRNA-NC组比较, shRNA-EIF4G2-2组GALNT1mRNA稳定性降低,差异有统计学意义(P<0.001), 见图5E。与oe-NC组比较, oe-EIF4G2组GALNT1mRNA和GALNT1蛋白表达水平升高,差异有统计学意义(P<0.01或P<0.001), 见图5F、图5G。

A、B: RIP实验、RNA下拉实验检测EIF4G2与GALNT1 mRNA间的结合关系(两者比较, ***P<0.001); C、D: RT-qPCR、Western blot检测EIF4G2过表达和干扰质粒的转染效率(与oe-NC组比较, ***P<0.001; 与shRNA-NC组比较, ##P<0.01, ###P<0.001); E: RT-qPCR检测GALNT1 mRNA的稳定性(与shRNA-NC组比较, ***P<0.001); F、G: RT-qPCR和Western blot检测过表达EIF4G2后的GALNT1 mRNA和GALNT1蛋白表达情况(与oe-NC组比较, **P<0.01, ***P<0.001)。图5 EIF4G2对GALNT1 mRNA稳定性的影响
2.6 过表达EIF4G2能够部分逆转GALNT1敲低对胃癌细胞增殖和凋亡的作用
CCK-8和克隆形成实验结果显示,与sh-NC组比较, sh-GALNT1-1组AGS细胞增殖能力减弱,差异有统计学意义(P<0.001); 与sh-GALNT1-1+oe-NC组比较, sh-GALNT1-1+oe-EIF4G2组AGS细胞增殖能力增强,差异有统计学意义(P<0.05或P<0.001), 见图6A、图6B。TUNEL实验结果表明, sh-GALNT1-1组细胞凋亡率高于sh-NC组,而sh-GALNT1-1+oe-EIF4G2组细胞凋亡率低于sh-GALNT1-1+oe-NC组,差异有统计学意义(P<0.001), 见图6C。Western blot检测结果显示, sh-GALNT1-1组Bcl-2蛋白表达低于sh-NC组, Bax、cleaved-caspase3、cleaved-caspase9蛋白表达高于sh-NC组,而sh-GALNT1-1+oe-EIF4G2组Bcl-2蛋白表达高于sh-GALNT1-1+oe-NC组,Bax、cleaved-caspase3、cleaved-caspase9蛋白表达低于sh-GALNT1-1+oe-NC组,差异有统计学意义(P<0.001), 见图6D。

A: CCK-8法检测AGS细胞活力; B: 克隆形成实验检测AGS细胞克隆形成能力; C: TUNEL实验检测AGS细胞凋亡率; D: Western blot检测凋亡相关蛋白表达。与sh-NC组比较, ***P<0.001; 与sh-GALNT1-1+oe-NC组比较, #P<0.05, ###P<0.001。图6 过表达EIF4G2能够部分逆转GALNT1敲低对胃癌细胞增殖和凋亡的作用
2.7 过表达EIF4G2能够部分逆转GALNT1敲低对胃癌细胞迁移和侵袭的作用
与sh-NC组比较, sh-GALNT1-1组细胞迁移和侵袭能力降低,差异有统计学意义(P<0.001); 与sh-GALNT1-1+oe-NC组比较, sh-GALNT1-1+oe-EIF4G2组细胞迁移和侵袭能力提升,差异有统计学意义(P<0.01或P<0.001), 见图7A、图7B。sh-GALNT1-1组MMP2、MMP9蛋白表达低于sh-NC组,差异有统计学意义(P<0.001); sh-GALNT1-1+oe-EIF4G2组MMP2、MMP9蛋白表达高于sh-GALNT1-1+oe-NC组,差异有统计学意义(P<0.05或P<0.001), 见图7C。

A: 划痕实验检测AGS细胞迁移能力; B: Transwell实验检测AGS细胞侵袭能力; C: Western blot检测转移相关蛋白表达。与sh-NC组比较, ***P<0.001; 与sh-GALNT1-1+oe-NC组比较, #P<0.05, ##P<0.01, ###P<0.001。图7 过表达EIF4G2能够部分逆转GALNT1敲低对胃癌细胞侵袭和迁移的作用
3 讨 论
胃癌是临床常见的胃肠道恶性肿瘤[1], 而肿瘤侵袭、转移和复发是胃癌患者生存率低、预后不良的重要因素[10]。由于胃癌缺乏明显的症状和敏感的生物标志物,大多患者确诊时病程已进入晚期,肿瘤细胞易侵入血液或淋巴管并向远处转移[11]。研究[10]报道,胃癌的发生发展通常与癌基因的激活或肿瘤抑制因子的失活有关。因此,揭示胃癌侵袭和转移的机制并探寻新的生物标志物对于提高早期胃癌诊断率及治疗效果具有重要意义。
蛋白糖基化修饰是常见的蛋白质翻译后修饰,在多数生物过程中起着广泛且高度特异的作用[7]。GALNT水平的改变与乳腺癌[12]、胶质瘤[13]、胃癌[14]等癌症有关,而GALNT家族成员GALNT6在胃癌组织中表达升高且与TNM分期、远处转移有关[14]。GALNT1作为常见的癌基因,可通过调控癌细胞增殖、迁移、侵袭等恶性表型而促进肿瘤进展[15]。本研究通过CCLE、GEPIA数据库分析和RT-qPCR、Western blot检测发现, GALNT1在胃癌组织和胃癌细胞中表达显著上调,且GALNT1高表达与胃癌患者低总体生存率密切相关。此外, GALNT1不足可导致胃癌细胞增殖能力降低和凋亡能力提升,并下调抗凋亡蛋白Bcl-2表达,上调促凋亡蛋白Bax、cleaved-caspase3、cleaved-caspase9表达。进一步实验结果显示,GALNT1敲低后,胃癌细胞迁移和侵袭能力显著减弱,且转移相关蛋白MMP2、MMP9活性降低。由此表明, GALNT1缺失可能通过抑制细胞增殖、迁移、侵袭并促进细胞凋亡而延缓胃癌进展。
EIF4F作为翻译起始复合体,可影响mRNA翻译,参与蛋白质合成[16]。EIF4G2是EIF4F复合物的关键组成成分,可参与PCBP2mRNA翻译[17]。本研究基于CCLE、GEPIA数据库分析和RT-qPCR、Western blot检测发现, EIF4G2在胃癌组织和胃癌细胞中表达显著上调,EIF4G2高表达与胃癌患者不良预后密切相关,且胃癌组织中EIF4G2表达与GALNT1表达呈正相关。机制实验结果证实, EIF4G2蛋白可与GALNT1mRNA结合并增强GALNT1mRNA稳定性。相关研究[18]表明, EIF4G2在胶质瘤细胞中表达上调并促进肿瘤细胞增殖。另有研究[12]发现,瘦素可能通过诱导EIF4G2在大鼠胃黏膜中表达而促进胃癌细胞增殖。本研究同样发现,过表达EIF4G2可部分恢复GALNT1缺失所致胃癌细胞增殖、迁移、侵袭能力降低和细胞凋亡能力提升。
综上所述, EIF4G2可促进胃癌细胞增殖、迁移、侵袭并抑制细胞凋亡,其机制可能与增强GALNT1mRNA稳定性有关。本研究验证了EIF4G2、GALNT1在胃癌中的致癌作用,可为以EIF4G2和GALNT1为靶点的胃癌治疗策略的发展提供参考。此外,本研究揭示了RNA结合蛋白EIF4G2与GALNT1mRNA间的调控新机制,对构建RNA结合蛋白调控网络具有重要意义。

